細菌的耐藥性與適應性
李 穎, 楊 帆
一些臨床重要病原菌耐藥性的迅速增長對抗感染治療構成了嚴重威脅,尋找細菌耐藥性增長的原因及相應防治措施成為當務之急。目前認為細菌耐藥性的傳播主要取決于病原菌(細菌本身的適應性)、抗菌藥物(抗生素選擇壓力)和宿主(免疫力)3個因素的相互作用[1-2]。眾多研究表明細菌耐藥性的增長與抗生素的選擇壓力有關,但過去很長一段時間的研究更多關注抗菌藥物的抗菌活性和細菌耐藥機制。近年來對于細菌耐藥性與其適應性關系的研究為分析細菌耐藥性增長的原因和探討如何降低耐藥性提供了新的視角,成為感染性疾病領域的一個研究熱點。
適應性(fitness)一般是指細菌為了生存與繁殖而調節自身代謝以適應環境條件(如溫度、pH、滲透壓、抗生素等)的能力[3]。病原體的適應性包含以下要素:在感染宿主和環境中不同菌株的繁殖(競爭)的相對效率;在宿主間的傳播能力;被感染宿主清除的情況[4]。本文主要就細菌的耐藥性與適應性的關系作一綜述。
一、耐藥菌株的適應性代價
有臨床和實驗室證據證實,肺炎鏈球菌在對青霉素的敏感性和其菌株毒力之間存在相關性。Ho等[5]測定了181株肺炎鏈球菌菌株的藥敏結果,血流感染分離株中青霉素耐藥肺炎鏈球菌(PRSP)占33.3%,明顯低于其他部位分離株PRSP比率(73.8%,P<0.01)。而在免疫功能健全的小鼠體內,接種野生型(wild)耐藥肺炎鏈球菌并不能誘發實驗性肺炎[6]。Azoulay-Dupuis等[7]研究了122株肺炎鏈球菌臨床株,其中32株毒力株均對青霉素敏感,而41株對青霉素耐藥或敏感性下降的菌株均無毒力,結果顯示侵襲性肺炎鏈球菌大部分是青霉素敏感的,非侵襲性菌株大多數是青霉素不敏感的。
在觀察到耐藥細菌的生長繁殖能力和致病力可能較敏感細菌低的現象后,國外很多學者進行了許多有關細菌耐藥性與適應性關系的研究(表1)。例如T rzcinski等[18]把肺炎鏈球菌青霉素敏感株,和插入青霉素結合蛋白基因的同源青霉素耐藥株同時接種于幼鼠鼻腔,并在其后4~5 d通過鼻腔沖洗液觀察兩者數量比值的變化,結果青霉素耐藥株在幼鼠上呼吸道定植力與敏感株相比隨著青霉素結合蛋白耐藥基因的表達數目增加而降低。比較腹腔內接種青霉素敏感株和插入pbp2b、pbpx基因的同源青霉素耐藥株的小鼠的半數致死量(LD50),結果顯示耐藥株的LD50[分別為(6.0±0.6)和(6.7±0.3)log10CFU/小鼠]比敏感株[(2.0±0.6)log10CFU/小鼠]高,耐藥株毒力較敏感株顯著下降[19]。Gillespie等[30]推測PRSP株適應性降低的主要原因可能是耐藥性菌株改變了細胞壁組成,由此導致的細胞壁合成所需的酶的底物、活性和親和力的變化增加了青霉素耐藥株的代謝負擔。適應性降低的現象同樣見于喹諾酮類和大環內酯類抗菌藥耐藥肺炎鏈球菌菌株[20-23]。
對其他細菌的研究也都顯示細菌獲得耐藥性突變后可引起其在體外或動物體內適應性降低的結果。如體外誘導的rpoB基因突變耐利福平結核分枝桿菌H37Rv(ATCC9360),與其同源敏感株共同孵育后計算兩者繁殖世代的比值,觀察到突變耐藥株的世代數較同源敏感株少,也就是說前者的體外競爭繁殖能力較后者低[15]。另外有2項關于鼠傷寒沙門菌的研究顯示,由于rpsL、rpoB和gyrA基因突變而分別對鏈霉素、利福平和萘啶酸耐藥的鼠傷寒沙門菌的大部分(6株/7株)對小鼠無毒力[8],而由于rpsL基因的核苷酸置換AAA42→AAC而對鏈霉素產生耐藥性的鼠傷寒沙門菌,核糖體蛋白S12的氨基酸置換(K42N)引起核糖體校對率(rate of ribosomal proofreading)的上升,從而使得蛋白合成和細菌生長速度減慢,細菌毒力降低[9]。Kanai等[24]在研究中發現,分離自同一患者分別對大環內酯類抗生素耐藥(23SrRNA基因突變)和敏感的幽門螺桿菌株,在單獨培養時未表現出生長速率的顯著性差異,當混合培養時,隨著連續傳代,耐藥株與敏感株菌落數量的比值逐漸減小,提示耐藥株在適應性上有所降低。一項通過測定耐喹諾酮類藥物銅綠假單胞菌的體外生長速率的研究結果顯示,前者的生長速率較其同源敏感株低[25]。Sánchez等[26]的研究表明,2株體外選擇的多重耐藥銅綠假單胞菌突變株(nalB和nfxB)在水中的生存力,產蛋白酶和菌株毒力均較敏感株有所下降,推測可能是由于外排泵耐藥機制不僅泵出了抗生素,也泵出了大量代謝產物,增加了菌株的代謝負擔。

表1 染色體靶位突變引起的耐藥性相關適應性代價Table 1.The biological cost of resistance conferred by target-altering chromosomal mutations
除了染色體靶位的突變,腸桿菌科細菌和其他革蘭陰性菌的耐藥性常由質粒介導。也有許多研究報道了與攜帶耐藥質粒相關的適應性代價,Lee等[31]的研究證實攜帶含有TcR(四環素耐藥)基因質粒pBR322的大腸埃希菌體外生長速率降低。Lenski等[32]分析與染色體靶位突變引起的細菌適應性下降機制不同的是,攜帶耐藥性相關質粒而導致適應性降低的原因可能是質粒在缺乏抗生素選擇壓力下不穩定狀態所造成的,這也就使得不攜帶耐藥質粒的敏感菌株生長繁殖更多。
這些研究提示細菌產生耐藥性常常改變了細菌的核糖體、DNA旋轉酶、RNA聚合酶、細胞壁等重要結構,導致細菌生長速度、定植能力和毒力等的下降,即細菌耐藥性的獲得需要付出適應性代價(fitness cost)[33]。因此,耐藥菌株的競爭優勢可能僅表現在存在抗生素選擇壓力時,當無抗菌藥選擇壓力時其適應性反而可能較敏感株低。
二、適應性代價的補償
多數研究已經證實耐藥會引起適應性代價,但也有研究觀察到一些無顯著適應性代價的耐藥突變(表1)。如Bj?rkman等[10]的研究發現,rpsL基因突變導致對鏈霉素耐藥的鼠傷寒沙門菌中部分菌株毒力并無明顯降低,在這些菌株同時發現了rpsD或rpsE基因突變。一項通過測定體外生長速率評價競爭莫匹羅星耐藥和糖肽類中介的金葡菌(GISA-2)、莫匹羅星敏感和糖肽類中介的金葡菌(GISA-1)和Mu50菌株生長情況的研究結果表明異亮氨酰-tRNA合成酶點突變的GISA-2菌株的體外生長繁殖能力無明顯降低[27]。Ramadhan等[29]在研究中比較了對萬古霉素耐藥和敏感的腸球菌的體外最大生長速率,未顯示前者適應性降低。
諸多實驗表明,這是由于耐藥菌株進一步發生補償突變,使得其適應性恢復(表2)。如Bj?rkman等[8]發現由于rpsL、rpoB和gyrA基因突變所致鏈霉素、利福平和萘啶酸耐藥的鼠傷寒沙門菌的多數(6株/7株)對小鼠無毒力,但這些耐藥菌株很快通過不同形式的補償突變在維持耐藥性的同時恢復菌株毒力。經過序列分析rpsL基因突變耐藥株存在rpsL基因內補償突變(42位點和93位點的ACA序列、AAC序列和CGT序列發生突變),一些突變株的毒力甚至超過了敏感株,提示可能存在rpsL基因外補償突變機制。而gyrA和rpoB基因突變耐藥株則通過基因內補償突變恢復了菌株毒力。而由于katG基因突變而對異煙肼產生耐藥性的結核分枝桿菌也通過基因外補償突變(高表達ahpC基因),在維持耐藥性的同時也恢復了毒力[34]。
同樣的對于攜帶耐藥質粒而使得細菌適應性降低的情況,也有報道補償突變的發生。McDermott等[35]的研究證實攜帶含有 TcR(四環素耐藥)基因的質粒pBR322的大腸埃希菌一開始最大生長速率降低,經連續傳代后獲得了適應性補償,由于耐藥質粒的結構和數目并無明顯變化,提示可能細菌染色體發生了補償突變。Bouma等[36]的研究也顯示雖然一開始攜帶pACYC184非結合質粒的大腸埃希菌的體外適應性是降低的,但當在抗菌藥選擇壓力下傳代500代時,細菌染色體出現了適應補償(而非質粒本身改變),從而使得細菌的適應性升高。其他研究結果也提示抗生素選擇壓力存在的情況下,大多數細菌通過染色體適應性突變從而克服攜帶質粒的適應性缺陷[30]。
細菌適應性代價及其補償在不同條件下有其不同的意義。如已知肺炎鏈球菌野生株可以通過PgdA和Adr 2種酶修飾肽聚糖的不同部位,PgdA對N-乙酰葡萄糖胺脫乙酰基,Adr乙酰化N-乙酰胞壁酸酯,從而造成對溶菌酶的耐受。Davis等[37]在研究中在溶菌酶 M(LysM+/+)和溶菌酶缺陷(LysM-/-)的小鼠模型中,同時接種肺炎鏈球菌野生株和pgdAadr突變株,結果發現在LysM+/+小鼠中野生株占絕對優勢,在LysM-/-小鼠中突變株占優勢,提示肽聚糖的修飾本身在降低了菌株在無溶菌酶存在條件下的競爭能力,但對溶菌酶的耐受性補償了其在寄殖中適應性代價(通常存在溶菌酶)。
目前大多數觀點認為適應性代價意味著耐藥株在無抗生素環境中適應性降低,生長速率較敏感株減慢,然而自然環境中的競爭使得耐藥菌株需要通過基因進化來克服這個缺陷重新獲得競爭力[30]。很多實驗已經證明盡管有時在缺乏抗生素的條件下適應性降低的無毒力耐藥株會回復突變成敏感株,但絕大多數都出現了通過補償突變(compensatory mutation)即第2位點突變(second site mutation),在不喪失耐藥性的同時來修復適應性和毒力。這也表明減少抗生素的使用可能并不能使已經出現在人體和環境中的耐藥細菌消失,第2靶位的補償突變能增加耐藥株的適應性,使這些菌株在沒有抗生素的環境中和敏感株成功競爭[30]。事實上臨床耐藥株可能已經適應了環境,補償了耐藥相關的適應性代價,從而引起耐藥細菌的廣泛傳播。
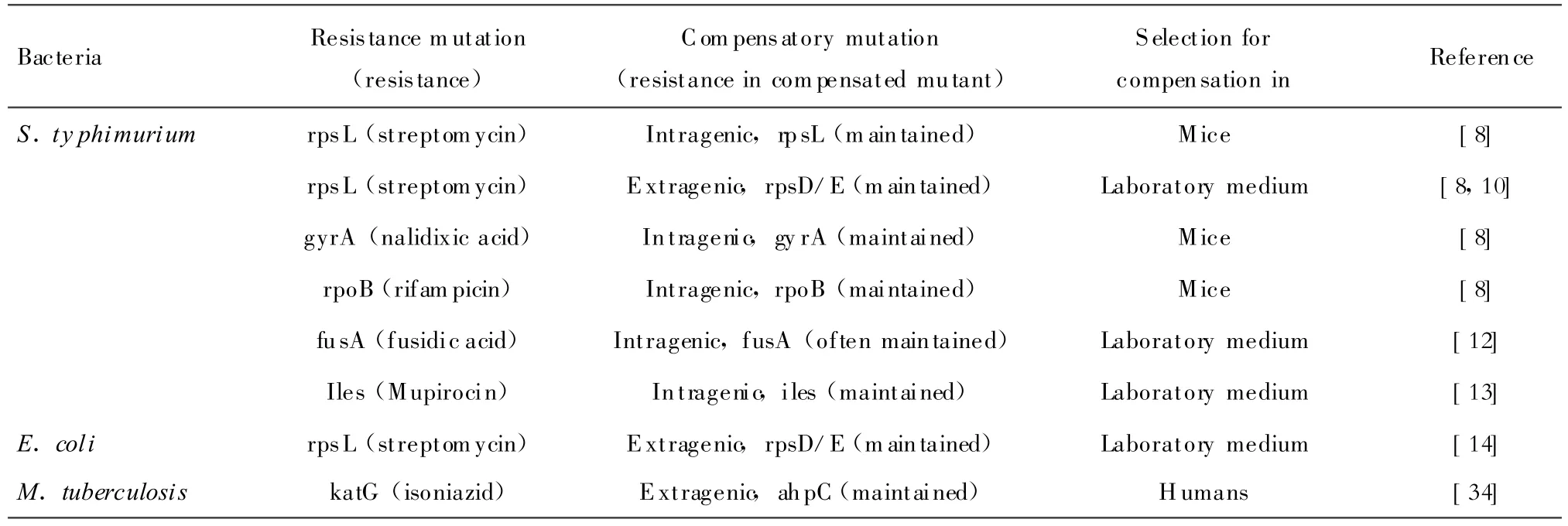
表2 染色體突變引起的適應性降低的改善和補償Table 2.Compensatory evolution and amelioration of fitness losses caused by chromosomal mutations
對細菌耐藥性與適應性關系的研究,是對細菌耐藥監測和耐藥機制研究的重要補充,有助于我們更好理解細菌耐藥性的發生機制和傳播規律,進而探索阻遏細菌耐藥性上升趨勢的更合理策略。
[1] Nuermberger EL,Bishai WR.Antibiotic resistance in Streptococcus pneumoniae:what does the future hold?[J].Clin Infect Dis,2004,38(Suppl 4):S363-371.
[2] Bj? rkman J,Nagaev I,Berg OG,et al.Effects of environment on compensatory mutations to ameliorate costs of antibiotic resistance[J].Science,2000,287(5457):1479-1482.
[3] Laurent F,Lelièvre H,Cornu M,et al.Fitness and competitive growth advantage of new gentamicin-susceptible MRSA clones spreading in French hospitals[J].J Antimicrob Chemother,2001,47(3):277-283.
[4] Andersson DI,Levin BR.The biological cost of antibiotic resistance[J].Curr Opin Microbiol,1999,2(5):489-493.
[5] Ho PL,Que T L,T sang DN,et al.Emergence of fluoroquinolone resistance among multiply resistant strains of Streptococcus pneumoniae in Hong Kong[J].Antimicrob Agents Chemother,1999,43(5):1310-1313.
[6] Azoulay-Dupuis E,Vallee E,Veber B,et al.In vivo efficacy of a new fluoroquinolone,sparfloxacin,against penicillin-susceptible and-resistant and multiresistant strains of Streptococcus pneumoniae in a mouse model of pneumonia[J].Antimicrob Agents Chemother,1992,36(12):2698-2703.
[7] Azoulay-Dupuis E,Rieux V,Muffat-Joly M,et al.Relationship between capsular type,penicillin susceptibility,and virulence of human Streptococcus pneumoniae isolates in mice[J].Antimicrob Agents Chemother,2000,44(6):1575-1577.
[8] Bj? rkman J,Hughes D,Andersson DI.Virulence of antibiotic-resistant Salmonellaty phimurium[J].Proc Natl Acad Sci U S A,1998,95(7):3949-3953.
[9] Maisnier-Patin S,Berq OG,Liljas L,et al.Compensatory adaptation to the deleterious effect of antibiotic resistance in Salmonella ty phimurium[J].Mol Microbiol,2002,46(2):355-366.
[10] Bj? rkman J,Samuelsson P,Andersson DI,et al.Novel ribosomal mutations affecting translational accuracy,antibiotic resistance and virulence of Salmonella ty phimurium[J].Mol Microbiol,1999,31(1):53-58.
[11] Giraud E,Cloeckaert A,Baucheron S,et al.Fitness cost of fluoroquinolone resistance in Salmonella enterica serovar Typhimurium[J].J Med Microbiol,2003,52(pt 8):697-703.
[12] Johansson U,Evarsson A,Liljas A,et al.The dynamic structure of EF-G studied by fusidic acid resistance and internal revertants[J].J Mol Biol,1996,258(3):420-432.
[13] Paulander W,Maisnier-Patin S,Andersson DI.Multiple mechanisms to ameliorate the fitness burden of mupirocin resistance in Salmonella typhimurium[J].Mol Microbiol,2007,64(4):1038-1048.
[14] Schrag SJ,Perrot V.Reducing antibiotic resistance[J].Nature,1996,381(6578):120-121.
[15] Billington OJ,McHugh TD,Gillespie SH.Physiological cost of rifampin resistance induced in vitro in Mycobacterium tuberculosis[J].Antimicrob Agents Chemother,1999,43(8):1866-1869.
[16] Wilson T M,de Lisle GW,Collins DM.Effect of inhA and katG on isoniazid resistance and virulence of Mycobacterium bovis[J].M ol Microbiol,1995,15(6):1009-1015.
[17] Li Z,Kelley C,Collins F,et al.Expression of katG in Mycobacterium tuberculosis is associated with it growth and persistence in mice and guinea pigs[J].J Infect Dis,1998,177(4):1030-1035.
[18] Trzcinski K,Thompson CM,Gilbev AM,et al.Incremental increase in fitness cost with increased beta-lactam resistance in pneumococci evaluated by competition in an infant rat nasal colonization model[J].J Infect Dis,2006,193(9):1296-1303.
[19] Rieux V,Carbon C,Azoulay-Dupuis E.Complex relationship between acquisition of beta-lactam resistance and loss of virulence in Streptococcus pneumoniae[J].J Infect Dis,2001,184(1):66-72.
[20] Johnson CN,Briles DE,Benjamin WH Jr,et al.Relative fitness of fluoroquinolone-resistant Streptococcus pneumoniae[J].Emerg Infect Dis,2005,11(6):814-820.
[21] Balsalobre L,de la Campa AG.Fitness of Streptococcus pneumoniae fluoroquinolone-resistantstrains with topoisomerase IV recombinant genes[J].Antimicrob Agents Chemother,2008,52(3):822-830.
[22] Rozen DE,M cGee L,Levin BR,et al.Fitness costs of fluoroquinolone resistance in Streptococcus pneumoniae[J].Antimicrob Agents Chemother,2007,51(2):412-416.
[23] Wolter N,Smith AM,Farrell DJ,et al.Heterogeneous macrolide resistance and gene conversion in the pneumococcus[J].Antimicrob Agents Chemother,2006,50(1):359-361.
[24] Kanai K,Shibayama K,Suzuki S,et al.Growth competition of macrolide-resistant and-susceptible Helicobacter pylori strains strains[J].Microbiol Immunol,2004,48(12):977-980.
[25] Kugelberg E,Lofmark S,Wretlind B,et al.Reduction of the fitness burden of quinolone resistance in Pseudomonas aeruginosa[J].J Antimicrob Chemother,2005,55(1):22-30.
[26] Sánchez P,Linares JF,Ruiz-D í ez B,et al.Fitness of invitro selected Pseudomonas aeruginosa nalB and nfxB multidrug resistant mutants[J].J Antimicrob Chemother,2002,50(5):657-664.
[27] Hurdle JG,O'Neill AJ,Chopra I.The isoleucyl-tRNA sy nthetase mutation V588F conferring mupirocin resistance in gly copeptide-intermediate Staphylococcus aureus is not associated with a significant fitness burden[J].J Antimicrob Che-mother,2004,53(1):102-104.
[28] Bj? rkman J,Andersson DI.The cost of antibiotic resistance from a bacterial perspective[J].Drug Resist Updat,2000,3(4):237-245.
[29] Ramadhan AA,Hegedus E.Survivability of vancomycin resistant enterococci and fitness cost of vancomycin resistance acquisition[J].J Clin Pathol,2005,58(7):744-746.
[30] Gillespie SH,M cHugh TD.T he biological cost of antimicrobial resistance[J].T rends Microbiol,1997,5(9):337-339.
[31] Lee SW,Edlin G.Expression of tetracycline resistance in pBR322 derivatives reduces the reproductive fitness of plasmidcontaining Escherichia coli[J].Gene,1985,39(2-3):173-180.
[32] Lenski RE,Simpson SC,Nguyen T T.Genetic analysis of a plasmid-encoded,host genotype-specific enhancement of bacterial fitness[J].J Bacteriol,1994,176(11):3140-3147.
[33] Enne VI,Delsol AA,Roe JM,et al.Rifampicin resistance and its fitness cost in Enterococcus faecium[J].J Antimicrob Chemother,2004,53(2):203-207.
[34] Sherman DR,Mdluli K,Hickey MJ,et al.Compensatory ahpC gene expression in isoniazid-resistant Mycobacterium tuberculosis[J].Science,1996,272(5268):1641-1643.
[35] McDermott PJ,Gowland P,Gowland PC.Adaptation of Escherichia coli growth rates to the presence of pBR322[J].Lett Appl Microbiol,1993,17(3):139-143.
[36] Bouma JE,Lenski RE.Evolution of a bacteria/plasmid association[J].Nature,1988,335(6188):351-352.
[37] Davis KM,Akinbi HT,Standish AJ,et al.Resistance to mucosal lysozyme compensates for the fitness deficit of peptidoglycan modifications by Streptococcus pneumoniae[J].PloS Pathog,2008,4(12):e1000241.

