H1N1流感病毒D151G突變株對奧司他韋的抗藥性研究及藥物篩選
石會娜,栗海波,劉 偉
鄭州大學生物工程系鄭州450001
(2011-01-14收稿 責任編輯 徐春燕)
2009年3月以來,一種新的H1N1流感病毒的暴發引起了全球越來越多的關注。世界衛生組織推薦奧司他韋為最為有效的抗病毒藥物。然而,截止到2010年2月,世界衛生組織宣布已有200多例H1N1病例對奧司他韋產生抗藥性[1]。神經氨酸酶(NA)在流感病毒復制、釋放和致病過程中起著非常重要的作用。奧司他韋是NA的抑制劑,能夠有效阻止流感病毒NA發揮以上功能,從而達到較好的治療效果。甲型流感病毒NA活性位點的結構在所有類中都是保守的,但是活性位點氨基酸殘基的改變有可能使病毒對奧司他韋產生抗藥性[2]。作者在GenBank中發現一株新H1N1流感病毒的NA活性位點151位由天冬氨酸突變成了甘氨酸(D151G),并通過分子動力學方法觀察了該流感病毒株對奧司他韋的抗藥性,再利用分子對接方法篩選出對這一病毒株的NA具有較強抑制作用的候選化合物。
1 材料與方法
1.1 分子動力學模擬
D151G突變株NA氨基酸序列由GenBank獲得(序列號:GQ369413.1),其結構是以野生型禽流感H5N1病毒NA和奧司他韋復合物(WT-OTV)的晶體結構(PDB Code:2HU4)為模板通過同源建模得到,兩者氨基酸序列相似性為90.4%,然后再將WT-OTV和D151G突變株NA的結構疊加,刪除掉WT-OTV的NA后就得到了D151G突變株NA與奧司他韋復合物(MT-OTV)的結構。
分別用WT-OTV和MT-OTV的結構作為初始結構進行分子動力學模擬。蛋白質的力場采用ff99力場[3],小分子的力場采用 gaff力場[4]。利用Gaussian 03軟件[5]的 HF/6-31G*方法優化小分子的結構,再應用antechamber程序產生gaff力場中缺失的小分子參數。復合物模型在進行動力學模擬前還需要利用Xleap程序對復合物進行處理,包括增加氫原子、增加抗衡離子(Na+)以使體系中性化和增加8.5?厚的基于TIP3P模型的八面體水環境。
分子動力學過程優化分為兩階段:首先約束溶質,對溶劑分子進行優化;再無約束優化整個系統。每個階段均采用最陡下降法優化2 500步,再采用共軛梯度法優化2 500步。優化之后用60 ps時間使系統升溫到298 K,在298 K下進行500 ps的無約束的動力學平衡,平衡之后再進行500 ps的分子動力學模擬,用MM-GBSA方法計算結合能。
1.2 藥物篩選 從ZINC數據庫(http://zinc.docking.org/)里篩選出和奧司他韋相似度為70%的小分子化合物,然后把這些小分子分別和D151G突變株的NA組成復合物,再采用Autodock 4.0程序[6]對這些復合物進行分子對接。對接選用的格點盒子大小為40點×40點×40點,格點間距0.375?。運用Lamarckian遺傳算法[6],對小分子進行100次的獨立對接實驗,能量極大值設為2 500 000,最大變異代數設為27 000,最后依據復合物的最低結合能來挑選出比MT-OTV結合能更小的小分子。
2 結果與討論
利用MM-GBSA方法計算出的各項能量值如表1所示,WT-OTV的結合能為 -14.0 kcal/mol(1 kcal=4.184 kJ),而實驗值為 -13.0 kcal/mol[7],由此可以看出此種方法得到的結合能跟實驗值非常吻合。
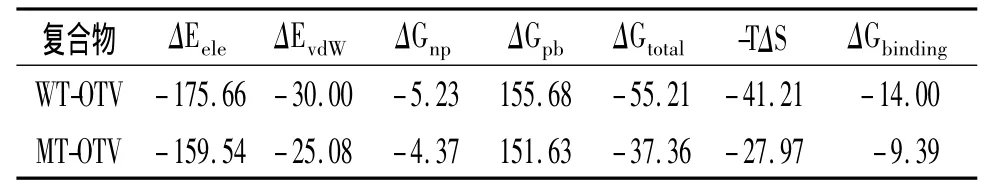
表1 MM-GBSA計算所得到的能量值 kcal/mol
MT-OTV的結合能比 WT-OTV降低了-4.61 kcal/mol,實驗[8]已證實 H274Y 突變流感病毒株對奧司他韋的敏感性比野生型流感病毒降低了1 466倍,而H274Y突變株的NA與奧司他韋預測的結合能為 -9.8 kcal/mol[9],由此可以看出 D151G 突變流感病毒株對奧司他韋也將表現出較強的抗藥性。
從ZINC數據庫里篩選出了21個和奧司他韋相似度為70%的小分子化合物,經Autodock 4.0軟件計算,有5個化合物與D151G突變的NA的結合能比與奧司他韋的結合能小。這5個化合物和奧司他韋的結構以及計算結合能如圖1所示,其中,4-[1-(1-金剛烷基氧化羧基)乙氨基]-3-氨基-4-氧代-丁酸與 D151G突變株 NA的結合能力最強,對D151G突變流感病毒株具有較強的抑制作用。

圖1 5種化合物和奧司他韋的結構式及計算結合能A:奧司他韋;B:3-氨基-4-氧代-4(1-茚酮-2-基)氨基丁酸;C:3-氨基-4-(4-甲基-2-氧代苯并吡喃-7-基)氨基-4-氧代丁酸;D:C的異構體;E:2-[2-氨基-3-(4-羥苯基)-丙醇]氨基-3-甲基-戊酸;F:4-[1-(1-金剛烷基氧化羧基)乙氨基]-3-氨基-4-氧代-丁酸。
總之,D151G突變流感病毒株對奧司他韋具有抗藥性;利用分子對接軟件從ZINC小分子數據庫中篩選出的5個小分子化合物中4-[1-(1-金剛烷基氧化羧基)乙氨基]-3-氨基-4-氧代-丁酸的結合能力最強,是較理想的藥物候選化合物。
[1] Update on oseltamivir resistant pandemic A(H1N1)2009 infl uenza virus:January 2010[J].Wkly Epidemiol Rec,2009,85(6):37
[2] Colman P,Varghese J,Laver W.Structure of the catalytic and antigenic sites in influenza virus neuraminidase[J].Nature,1983,303(5912):41
[3] Duan Y,Wu C,Chowdhury S,et al.A point-charge force field for molecular mechanics simulations of proteins based on condensed-phase quantum mechanical calculations[J].J Comput Chem,2003,24(16):1999
[4] Wang J,Wolf R,Caldwell J,et al.Development and tes ting of a general amber force field[J].JComput Chem,2004,25(9):1157
[5] Frisch M,Trucks G,Schlegel H,et al.Gaussian 03,revision B.03[M].Gaussian:Pittsburgh,PA,2003.
[6] Morris G,Goodsell D,Halliday R,et al.Automated docking using a Lamarckian genetic algorithm and an empirical binding free energy function[J].JComput Chem,1998,19(14):1639
[7] Malaisree M,Rungrotmongkol T,Nunthaboot N,et al.Source of oseltamivir resistance in avian influenza H5N1 virus with the H274Y mutation[J].Amino Acids,2009,37(4):725
[8] Hurt A,Ernest J,Deng Y,et al.Emergence and spread of oseltamivir-resistant A(H1N1)influenza viruses in Oceania,South East Asia and South Africa[J].Antiviral Res,2009,83(1):90
[9] Rungrotmongkol T,Malaisree M,Nunthaboot N,et al.Molecular prediction of oseltamivir efficiency against probable influenza A(H1N1-2009)mutants:molecular modeling approach[J].Amino Acids,2009,39(2):393