細胞培養穩定同位素標記技術的發展及其在腦病研究中的應用
朱 麗,劉 安,楊洪軍,陳 暢,李德鳳,吳傳鴻,高 健,李韶菁
(1.江西中醫學院,江西南昌 330006;2.中國中醫科學院中藥研究所,北京 100700;3.河北大學藥學院,河北保定 071002)
腦病是指腦組織結構和生理功能異常的疾病,腦萎縮和老年癡呆、腦癱、帕金森病、癲癇、腦血栓后遺癥和中風偏癱,被國際醫學界并稱為5大腦病。上述腦病,究其病因雖是腦神經和腦血管兩大因素,但其復雜性使之成為21世紀研究的熱點,醫學界的難題。對于腦病的研究,過去一般是采用非動態、局部、靶向性的方法。穩定同位素標記(stable isotope labeling with amino acids in cell culture,SILAC)技術,作為新興的定量蛋白質組學研究技術,可以明顯提高蛋白質鑒定的特異性,敏感性和準確性,是從系統角度研究復雜性問題,符合網絡藥理學的理念,可以全面揭示腦病的發生機制,為動態、系統地定性和定量分析腦病提供了有效工具。隨著SILAC技術的不斷發展,其應用范圍也從最初的細胞系擴展到整體動物水平,具體的應用策略也在不斷完善發展,本文主要就其進展及其在腦病研究中的應用做一概述。
1 SILAC技術的原理與優勢
1.1SILAC技術的基本原理SILAC技術是在細胞培養過程中利用穩定同位素標記的氨基酸結合質譜技術對蛋白表達進行定量分析的一種新技術。SILAC的基本原理是分別用天然同位素(輕型)或穩定同位素(重型)標記的必需氨基酸取代細胞培養基中相應氨基酸,細胞進行若干倍增周期后,穩定同位素標記的氨基酸完全參入到細胞新合成的蛋白質中,替代了原有氨基酸,不同標記的細胞按細胞數或蛋白量等比例混合,經分離、純化后進行質譜鑒定[1-2]。SILAC技術作為一種體內代謝標記技術,目前標記的氨基酸種類已擴展到亮氨酸、精氨酸、賴氨酸、甲硫氨酸和酪氨酸[3-4]等。
1.2SILAC技術的優勢SILAC技術較其他標記技術具有不可替代的優勢,它是一種體內代謝標記技術,標記效率高,不需要樣品預處理,具有高通量性等突出優勢;樣本需求量少、操作簡單、定量精確;適合于大分子量、復雜生物樣品分析,可以鑒定出更多的低豐度蛋白(包括細胞表面蛋白、細胞器特異性蛋白以及磷酸化蛋白或糖蛋白這些經過翻譯后修飾的蛋白);體內代謝標記與聚丙烯酰胺凝膠電泳(SDSPAGE)或色譜分離技術相結合,兼容疏水性蛋白和偏堿性蛋白,不受蛋白性質限制,可以研究某些疏水性蛋白質如膜蛋白、等電點極酸或極堿的蛋白質以及分子量極大或極小的蛋白質;可以進行細胞信號轉導的動態變化研究。SILAC技術除在全面、精確定量差異蛋白質組研究方面發揮作用外[5-6],其在研究生化代謝、分子信號通路、蛋白質翻譯后修飾[7](如磷酸化、糖基化、甲基化、乙酰化等)、時空動態變化的蛋白質[8]和蛋白質相互作用[9]等方面亦具有明顯優勢。目前SILAC結合質譜技術應用范圍已從細胞系擴展到亞細胞器、組織與動物整體水平[10-12],甚至已成功用于原代細胞培養[13-16]。
2 SILAC技術的發展
SILAC結合質譜技術作為定量蛋白質組學的一個重要方法,其應用處于不斷發展和創新中。其應用范圍從最初的細胞系擴展到整體動物水平。在細胞方面,從只用于傳代細胞的經典SILAC技術,發展到可以進行原代細胞培養的多重SILAC技術,進而發展到可以用于組織樣本的超級SILAC的方法。
2.1SILAC技術在細胞方面的研究進展
2.1.1經典SILAC技術經典的SILAC技術是一種完全標記的絕對蛋白質定量方法,穩定同位素標記的氨基酸與天然氨基酸化學性質基本相同,對細胞無毒性,因而它所標記的細胞和未標記細胞在生物學行為上幾乎沒有差異,標記效率可高達100%。經典的SILAC技術是分別用含輕型穩定同位素(天然同位素)和重型穩定同位素標記的必需氨基酸的培養基,對處于兩種不同狀態的細胞(如一種為正常細胞,另一種為病理狀態的細胞)分別進行培養標記,細胞經至少6個細胞倍增周期被完全標記后,將經過輕、重型穩定同位素標記的兩種細胞等數量混合,將分別提取的蛋白等比例混合后經SDS-PAGE分離,酶解提取肽,進行質譜檢測,通過質譜圖上一對輕重穩定同位素峰的比例可以反映對應蛋白在不同狀態下的表達水平(Fig 1)。經典的SILAC標記方法主要用于定量分析樣品間的蛋白質表達差異,可以應用于檢測蛋白質在某些生物過程中的動態變化。
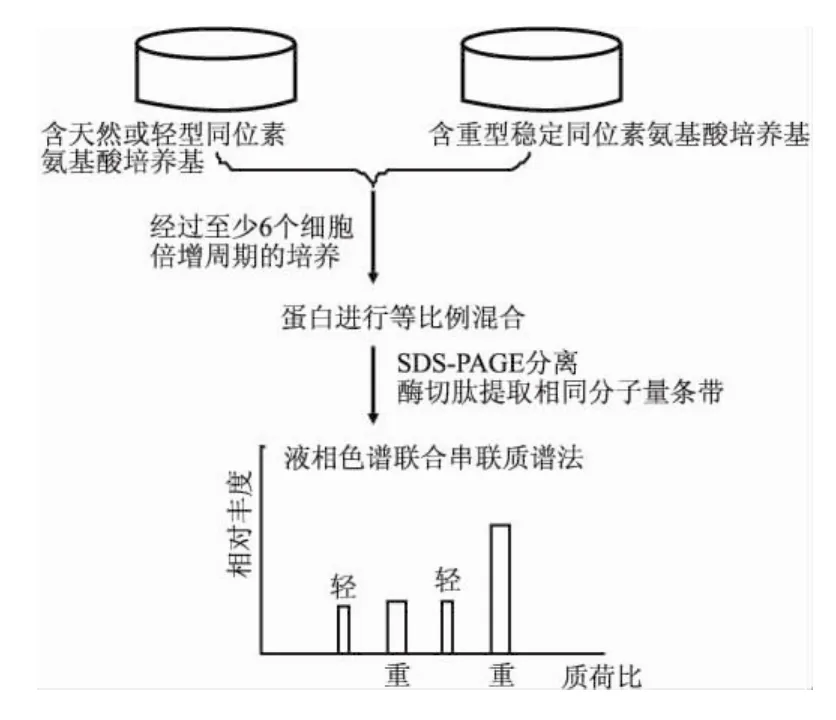
Fig 1 Experiment process of classic SILAC technology[1 -2]
2.1.2多重SILAC技術經典的SILAC技術不適用于不可傳代或者不穩定的細胞,多重SILAC技術是使用多組重質氨基酸進行標記,是一種相對定量的不完全標記技術[17](Fig 2),可用于原代細胞的培養。多重SILAC對同一種細胞標記兩種以上不同的重質氨基酸,細胞中所含重質氨基酸比重的動態變化反映了蛋白質表達量的不同,是一項相對精度較高的質譜定量技術。但由于SILAC技術需采用透析后血清,培養條件相對貧乏,為保持不可傳代細胞的生物學特性,需對細胞培養條件進行摸索,使之既滿足SILAC標記要求,同時也可以維持細胞的正常功能。
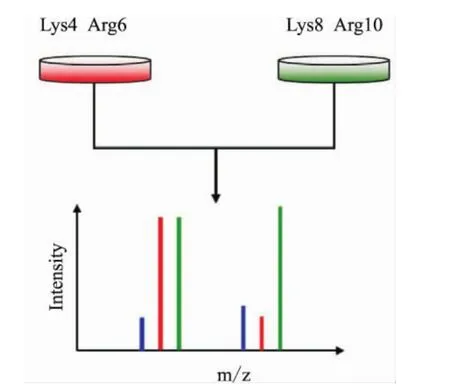
Fig 2 Multiplex SILAC[17] technology
2.1.3超級SILAC技術超級 SILAC(Super-SILAC)技術的出現擴大了SILAC技術的應用范圍,使之既能夠測出不可傳代細胞,而且可以對多種不同類型的細胞同時測定。Super-SILAC方法是將重質標記的細胞樣品作為參照體系,然后將這一參照樣品與組織樣品等量混合比較,不同的組織樣品之間以參照樣品的相對量為標準進行比較,這一方法使SILAC在組織樣本上的應用也得到推廣。這種Super-SILAC技術可以應用于蛋白質生物標識的發現。Geiger等[18]用重氨基酸標識了含有不同癌癥細胞系的混合物,用5種標記方法混合,精確測定出人類原發性腫瘤組織中的蛋白質(Fig 3)。
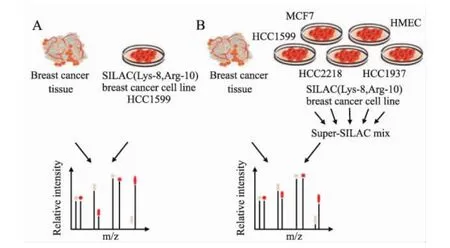
Fig 3 Heavy amino acid identity Super-SILAC technology[18]
2.2SILAC技術在整體動物方面的研究進展SILAC技術目前主要應用在細胞水平,但在整體動物方面的研究也開始進行。體內穩定同位素標記法有一個明顯優點是在樣品制備的初期,蛋白質就被標記,從而減少了樣品間由于實驗操作造成的差異,有利于提高定量的準確度。采用純系小鼠分別飼以含重型穩定同位素和含天然或輕型穩定同位素氨基酸的飼料,至少傳代6代以上(保證重型穩定同位素標記高的結合率)。然后對動物模型進行藥物處理,取樣后提取蛋白質,與重穩定性同位素標記的小鼠樣品進行等量混合,利用高分辨質譜尋找表達差異的標志蛋白,并對鑒定的蛋白質進行定量,可分析和研究藥物作用機制。Kruger等[12]將組織標記技術應用到基因敲除小鼠上(Fig 4),揭示了Kindlin-3蛋白在紅細胞中具有重要作用,是紅細胞發揮正常功能的必要因子。該方法是定量比較基因敲除小鼠蛋白質組的通用工具,可利用其確定蛋白質在復雜體內條件下的功能。
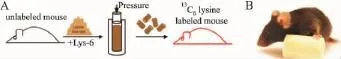
Fig 4 Mice are fed with heavy isotope[12]
3 SILAC技術在腦病研究中的應用
腦病是一類復雜的、難治愈的疾病,目前針對腦病的研究缺乏有效的方法。SILAC技術作為一種多靶點、多途徑的研究方法,能夠系統、動態地分析腦病發生和發展過程中蛋白質表達譜的變化,符合網絡藥理學的理念,為尋找腦中特異的蛋白標志物,進行疾病機制深入研究和藥物治療提供了新的思路。但目前SILAC技術在腦病研究中的應用尚處于起步階段,相關報道較少[19],主要有以下幾個方面:
以腦中風神經血管單元研究為例闡述SILAC的應用,神經血管單元作為一個獨立的功能單位,主要由原代神經元、星形膠質細胞、微血管內皮細胞及其基質組成,通過不同策略的SILAC技術進行對神經血管單元中不同細胞進行標記:針對原代神經元細胞和星形膠質細胞采用多重SILAC標記方法進行標記(multiplex SILAC labeling),微血管內皮細胞系為可傳代細胞,采用經典SILAC技術。然后對3種細胞加入損傷因素和藥物干預,對處理的不同標記的細胞等量混合,進行LC-MS分析,鑒定其中的蛋白質并進行定量。還可采用Super-SILAC技術直接提取腦組織,用重氨基酸標識神經血管單元里不同細胞的混合物,在組織水平上鑒定蛋白質。使用神經血管單元作為體外研究腦缺血的模型,有利于有效地將SILAC定量技術與腦卒中微環境的研究結合起來,而且利用這個模型發現的新靶標可以進一步在整體動物模型中進行驗證,形成了完整的腦卒中相關細胞的定量蛋白質組學SILAC研究策略。
Yang等[20]使用SILAC技術分析糖氧剝奪后sumo3表達的改變,找到了一些可能與sumo相關的信號通路和恢復缺血性應力的通路。
阿爾采末癥(Alzheimer’s disease,AD)是由于腦內神經結構發生病變而引起的老年性癡呆癥,有文獻證明小膠質細胞可能在其發病機制中發揮重要作用。Liu等[21]利用SILAC技術探討AD發病機制,通過內毒素預處理1h的小膠質細胞與空白組進行比較,確定了77種分泌蛋白,其中28種與細胞溶酶體相關,13種溶酶體蛋白表達差異有顯著性,證明了小膠質細胞釋放的一些溶酶體酶可能參與神經元損傷過程。應用SILAC技術能夠系統地解析疾病涉及信號網絡,預測某些關鍵信號蛋白,為深入的功能研究指出方向。
帕金森病(Parkinson’s disease,PD)是一種嚴重影響患者活動能力的中樞神經系統慢性疾病,目前公認其病因是神經細胞的退行性變,大腦黑質神經元數量減少,多巴胺合成不足,但其深入的病機尚不清楚。Jin等[22]利用SILAC技術比較魚藤酮對線粒體蛋白質(多巴胺)的影響,確定了1 722個蛋白質,在950個線粒體蛋白質中,在魚藤酮處理之后有110個有相對豐度的明顯差異變化。進一步表明這些蛋白質可能闡明PD發病機制。
4 展望
中藥具有多靶點、多向調節、防治結合的特點,雖早已被廣泛應用于腦病臨床治療,但由于其成分復雜,缺乏有效研究手段,作用機制無法闡釋清楚。將SILAC這種系統、高通量的定量蛋白質組技術應用于復雜腦病的研究,并將其作為治療腦病中藥研究的手段,探索適合中醫藥特點的新研究模式,是一個相對新的思路,但如何聯合不同策略的SILAC技術,有效并更適合應用于神經系統疾病如腦中風、AD、PD的研究,更深入地闡明中藥復方的藥效物質基礎、多成分交互作用機制及其組方的科學內涵,仍需進一步探討。
[1]Ong S E,Blagoev B,Kratchmarova I,et al.Stable isotope labeling by amino acids in cell culture,SILAC,as a simple and accurate approach to expression proteomics[J].Mol Cell Proteomics,2002,1(5):376-86.
[2]Chen X,Smith L M,Bradbury E M.Site-specific mass tagging with stable isotopes in proteins for accurate and efficient protein identification[J].Anal Chem,2000,72(6):1134 -43.
[3]Ong S E,Kratchmarova I,Mann M.Properties of13C-substituted arginine in stable isotope labeling by amino acids in cell culture(SILAC)[J].Proteome Res,2003,2(2):173 -81.
[4]Everiey PA,Krijgsveld J,Zetter B R,et al.Quantitative cancer Proteomics:stable isotope labeling with amino acids in cell culture(SILAC)as a tool for prostate cancer research[J].Mol Cell Proteomics,2004,3(7):729 -35.
[5]Rangiah K,Tippornwong M,Sangar V,et al.Differential secreted proteome approach in murine model for candidate biomarker discovery in colon cancer[J].Proteome Res,2009,8(11):5153 -64.
[6]Zhang G,Fenyo D,Neubert T A.Evaluation of the variation in sample preparation for comparative proteomics using stable isotope labeling by amino acids in cell culture[J].Proteome Res,2009,8(3):1285-92.
[7]Cunningham D L,Sweet S M,Cooper H J,et al.Differential phosphoproteomics of fibroblasr growth factor signaling:identification of Src family kinase-mediated phosphorylation events[J].Proteome Res,2010,9(5):2317 -28.
[8]Molina H,Parmigiani G,Pandey A.Assessing reproducibility of a protein dynamics study usingin vivolabeling and liquid chromatography tandem mass spectrometry[J].Anal Chem,2005,77(9):2739-44.
[9]Mann M.Functional and quantitative proteomics using SILAC[J].Nat Rev Mol Cell Biol,2006,7(12):952 -8.
[10]Ostasiewicz P,Zielinska D F,Mann M,et al.Proteome,phosphoproteome,and N-glycoproteome are quantitatively preserved in formalin-fixed paraffin-embedded tissue and analyzable by high-resolution mass spectrometry[J].Proteome Res,2010,9(7):3688 -700.
[11]Liao L,Park S K,Xu T,et al.Quantitative proteomic analysis of primary neurons reveals diverse changes in synaptic protein content in fmr 1 knockout mice[J].Proc Natl Acad Sci USA,2008,105(40):15281-6.
[12]Kruger M,Moser M,Ussar S,et al.SILAC mouse for quantitative proteomics uncovers kindling-3 as an essential factor for red blood cell function[J].Cell,2008,134(2):353 - 64.
[13]Qiu H,Wang Y.Quantitative analysis of surface plasma membrane proteins of primary and metastatic melanoma cells[J].Proteome Res,2008,7(5):1904 -15.
[14]Greco T M,Seeholzer S H,Mak A,et al.Quantitative mass spectrometry-based proteomics reveals the dynamic range of primary mouse astrocyte protein secretion[J].Proteome Res,2010,9(5):2764-74.
[15]Olstad E,Qu H,Sonnewald U.Long-term kainic acid exposure reveals compartmentation of glutamate and glutamine metabolism in cultured cerebellar neurons[J].Neurochem Int,2007,50(7-8):1004-13.
[16]Spellman D S,Deinhardt K,Darie C C,et al.Stable isotopic labeling by amino acids in cultured primary neurons:application to brain-derived neurotrophic factor-dependent phosphotyrosine-associated signaling[J].Mol Cell Proteomics,2008,7(6):1067 -76.
[17]Zhang G,Deinhardt K,Chao M V,et al.Study of neurotrophin 3 signaling in primary cultured neurons using multiplex stable isotope labeling with amino acids in cell culture[J].Proteome Res,2011,10(5),2546 -54.
[18]Geiger T,Cox J,Ostasiewicz P,et al.Super-SILAC mix for quantitative proteomics of human tumor tissue[J].Nat Methods,2010,7(5):383-5.
[19]趙 欣,蒲小平.蛋白質組學在藥物研究中的應用[J].中國藥理學通報,2009,25(8):988 -91.
[19]Zhao X,Pu X P.The application of proteomics technology in drug study[J].Chin Pharmacol Bull,2009,25(8):988 - 91.
[20]Yang W,Thompson J W,Wang Z,et al.Analysis of oxygen/glucose-deprivation-induced changes in SUMO3 conjugation using SILAC-based quantitative proteomics[J].Proteome Res,2011,11(2):1108-17.
[21]Liu J,Hong Z,Ding J,et al.Predominant release of lysosomal enzymes by newborn rat microglia after LPS treatment revealed by proteomic studies[J].Proteome Res,2008,7(5):2033 -49.
[22]Jin J,Davis J,Zhu D,et al.Identification of novel proteins affected by rotenone in mitochondria of dopaminergic cells[J].BMC Neurosci,2007,8:67.

