冷藏帶魚貯藏期間主要微生物動態變化的PCR-DGGE分析
藍蔚青,謝 晶
(上海海洋大學食品學院,上海水產品加工及貯藏工程技術研究中心,上海 201306)
冷藏帶魚貯藏期間主要微生物動態變化的PCR-DGGE分析
藍蔚青,謝 晶*
(上海海洋大學食品學院,上海水產品加工及貯藏工程技術研究中心,上海 201306)
本文研究了冷藏帶魚貯藏期間的主要微生物種群演替規律,為延長水產品貨架期提供參考。采用SDS化學裂解法,從冷藏帶魚中提取總細菌基因組DNA,并進行16S rRNA的V3可變區PCR擴增,再通過DGGE得到動態指紋圖譜。結果表明,貯藏初期冷藏帶魚樣品中的微生物群落豐富,嗜冷桿菌(Psychrobacter sp.)為前期的主要優勢菌。隨著貯藏時間的延長,僅有少數種類細菌存活并最終成為主導菌群。熒光假單胞菌(Pseudomonas fluorescens)在貯藏過程中比例逐漸增大,希瓦氏菌(Shewanella sp.)與弧菌(Vibrio sp.)比例也有明顯增加,在貯藏后期發展成為優勢腐敗菌。
帶魚,變性梯度凝膠電泳,微生物,動態變化
Key words:Trichiurus haumela;denaturing gradientgelelectrophoresis(DGGE);microorganisms;dynamic changes
帶魚(Trichiurus haumela)又名裙帶魚、牙帶魚、刀魚、白帶魚等,為鱸形目帶魚科帶魚屬,屬暖溫性近底層魚類,是我國四大經濟魚類之一,廣泛分布于印度洋和西太平洋的溫暖海區,因其身體側扁,呈帶狀,尾細長如鞭而得名[1]。由于帶魚捕獲后即刻死亡,且產品的加工特性和人們消費習慣的不同,其在運輸、加工與貯藏過程中易受細菌與酶的作用而腐敗變質,也易發生脂肪氧化酸敗[2]。在常規的冷藏條件下,帶魚貨架期通常為2~3d,極大程度地限制了水產品的流通運輸,使其貯運銷售遠不能滿足人們對產品貨架期與品質的要求[3]。近幾十年間,國外專家學者先后對新鮮、冷藏過程和腐敗魚類的細菌開展過部分研究工作[4-5],國內也有少部分相關報道。然而長期以來,人們研究微生物多樣性只局限于一部分能夠分離培養的微生物種類上[6-7],而對其中大量的未培養微生物不能深入研究,研究結果極具片面性,也不能全面反映微生物群落的真實狀態。變性梯度凝膠電泳 (DenaturingGradientGel Electrophoresis,DGGE)是目前在國內外應用最廣泛的分析微生物群落多樣性和監視種群動態的分子指紋技術,它不依賴于培養過程,大大縮短樣品的分析時間,而且還能顯示環境樣品中不可培養微生物的遺傳信息。1993年Muzyers等首次將變性梯度凝膠電泳技術應用于分子微生態學研究領域,并證實了這種技術在揭示自然界微生物區系的遺傳多樣性和種群差異方面具有獨特的優越性,可很好反映微生物的多樣性[8-10]。該法避免了經典微生物技術在微生物多樣性變化研究中的局限性,能更可靠、直接全面地認識微生物組成與有效分析復雜微生物的群落結構,可快速獲得環境樣品中各種微生物的菌落指紋和特征性核苷酸序列,以確定微生物的多樣性及其分類地位,揭示和豐富生態環境的微生物資源[11-12]。Maria B H 等[13-14]采用 PCR- DGGE 技術分別對氣調包裝的大比目魚與大西洋真鱈魚貯藏期間的主要微生物動態變化進行研究,發現假單胞菌(Pseudomonas spp.)為大比目魚的優勢腐敗菌,腐敗希瓦氏菌(Shewanella putrefaciens)與假單胞菌(Pseudomonas spp.)為大西洋真鱈魚中的主要優勢菌。蔡秋杏等[15]研究了液熏羅非魚片加工過程中的微生物群落,得出假單胞菌屬(Pseudomonas)與弧菌屬(Vibrio)細菌都具有使產品腐敗的潛能。本文應用PCR-DGGE指紋技術,研究了冷藏帶魚貯藏期間主要優勢菌的演替變化規律,為延長水產品貨架期提供參考。
1 材料與方法
1.1 材料與儀器
帶魚(Trichiurus haumela) 上海市浦東新區蘆潮港碼頭水產市場,條重0.25~0.50kg,冰藏條件下當日運送至實驗室進行實驗。EDTA 生工生物工程(上海)有限公司;KCl 天津永大化學試劑開發中心;Na2HPO4上海凌峰化學試劑有限公司;KH2PO4上海高信化玻儀器有限公司;SDS、TAE電泳緩沖液(50×TAE)、Acry-lamide丙烯酰胺、TEMED、引物:V3-1,V3-2,V3-3 生工生物工程(上海)有限公司;NaCl 上海市疾病預防控制中心;TaqDNA酶 日本 TaKaRa BIO株式會社;dNTP Mixture、10×PCR Buffer 日本TaKaRa BIO株式會社;Tris Formamide去離子甲酰胺 北京鼎國生物技術有限公司;Bis-acrylamide(BIO BASIC.INC)、過硫酸銨國藥集團化學試劑有限公司;6×Loading Buffer、100bp DNA Ladder、瓊脂糖、溴化已錠染色液(EB)、Solarbio SYB Green I核酸染料等。
Gilson微量移液器、凝膠圖像成像系統 德國Eppendorf公司;Mini高速離心機 德國Eppendorf公司;D-code system變性梯度凝膠電泳儀 美國BIO-RAD公司;CR21G himac高速冷凍立式離心機
日本 HITACHI;SS-325高壓蒸汽滅菌鍋 日本SANYO;MDF-U53Y超低溫冰箱 日本SANYO;隔水式電熱恒溫培養箱 上海躍進醫療器械廠;DHG-9053型電熱鼓風干燥箱 上海一恒科學儀器有限公司;壓力蒸汽滅菌器 上海華線醫用核子儀器有限公司;H-1微型混合器 上海康禾光電儀器有限公司;超凈工作臺 上海博迅實業有限公司;JY2001電子天平 上海精密科學儀器有限公司;pH計 上海精科科學儀器有限公司;DYY-6C型電泳儀 北京市六一儀器廠;TC—24/H(b)型基因擴增儀 杭州博日科技有限公司;QL-81旋渦振蕩器 海門市其林貝爾儀器制造有限公司;BCD-205T TCL冰箱TCL集團股份有限公司等。
1.2 實驗方法
1.2.1 原料處理 將帶魚洗凈,經“三去”處理(去頭、去尾、去內臟),無菌操作切成6~7cm長的魚段,隨機分組,放入PE保鮮袋,在(4±1)℃的冰箱中貯藏。
1.2.2 菌落計數 采用有氧平板菌落計數法,分別將第1、3、5、8d 冷藏帶魚的樣品按 GBT4789.2-2008法[16]進行處理。每組樣品3個重復,選3個合適的稀釋度,每個稀釋度5組平行,取各稀釋菌液0.1mL涂布于TSA培養基上,37℃培養48h后計數。
1.2.3 樣品細菌總DNA的提取 無菌條件下取樣品10.0g(3次重復),剪碎后放于90.0mL滅菌生理鹽水中,轉移到均質袋中均質2min,搖床振搖1h,將液體轉移至滅菌后的離心管中,4℃,4000r/min離心10min,取上清液于離心管中,4℃,10000r/min離心20min,將沉淀轉移至1.5mL離心管。采用SDS化學裂解法提取總基因組DNA[17],并對樣品處理階段作適當改良。
1.2.4 細菌16S rRNA的V3可變區PCR擴增 以提取的細菌總DNA為模板,細菌16S rRNA的V3可變區PCR引物如下:

PCR采用25.0μL體系,反應條件為2.5μL 10×PCR buffer;2.0μL dNTP(含 Mg2+);0.3μL TaqDNA酶;1.0μL V3-2;1.0μL V3-3;2.0μL DNA 模板;16.2μL ddH2O。PCR反應步驟為:a.95℃預變性3min;b.95℃變性1min;c.55℃退火1min;d.72℃延伸30s;e.72℃延伸10min;f.10℃保存;重復步驟 b、c、d、e25次。使用1.5%瓊脂糖凝膠電泳檢驗PCR擴增產物,產物約為200bp,獲得V3可變區的PCR產物用于DGGE電泳分析,PCR產物置于-20℃下保存備用。
1.2.5 變性梯度凝膠電泳(DGGE) 對細菌16S rRNA的V3區段的擴增產物進行DGGE分析。參照Muyzer[18-21]的方法變性梯度范圍為 30% ~60% ,其配制方法見表1,DNA擴增產物上樣量為10μL。采用Dcode DGGE系統(Bio Rad)進行電泳,電泳緩沖液為1×TAE緩沖液。
1.2.6 染色 電泳完畢后,用SYB green I(1×TAE,1∶10000)染色30min,棄去浸泡液,再在 ddH2O 中浸泡沖洗5min,通過UVP成像系統進行紫外照相。
1.2.7 DNA回收、純化和測序[22]使用圖像分析軟件Image Lab 3.0.1進行條帶一致性分析,將DGGE膠片放置于紫外燈下,切下不同位置的條帶,分裝入1.5mL的離心管中,在50μL的Elution buffer中靜置過夜。次日,將離心管在4℃,12000r/min下離心1min,所得的上清液作為模板進行PCR擴增。細菌16S rRNA的V3可變區PCR引物為:


表1 不同濃度梯度變性膠的配制Table 1 Deformation of the different gradient preparation
PCR采用25.0μL體系,反應條件為2.5μL 10×PCR buffer;2.0μL dNTP(含 Mg2+);0.3μL TaqDNA酶;1.0μL V3-2;1.0μL V3-3;2.0μL DNA 模板;16.2μL ddH2O。PCR反應步驟為:a.95℃預變性3min;b.95℃變性1min;c.55℃退火1min;d.72℃延伸30s;e.72℃延伸10min;f.10℃保存;重復步驟 b、c、d、e25次。使用1.5%瓊脂糖凝膠電泳檢驗PCR擴增產物,產物片段大小約為200bp。將其送至上海生工公司進行測序,登錄 NCBI(http://www.ncbi.nlm.nih,gov/blast/),將所得序列與數據庫中的已知序列進行比對。
2 結果與分析
2.1 冷藏帶魚貯藏期間細菌總數的變化
參照鮮帶魚國標[23]規定,細菌總數≤4lgCFU/g為一級鮮度,4lgCFU/g<細菌總數≤6lgCFU/g為二級鮮度。由表2可見,在冷藏的第3、5d,冷藏帶魚的細菌總數先后超過了一級與二級鮮度,細菌總數分別達到4.23lgCFU/g與6.58lgCFU/g,在冷藏的第8d,其細菌總數高達8.75lgCFU/g,樣品已完全腐敗。從細菌總數的數值變化程度和趨勢來看,符合帶魚在冷藏條件下的腐敗規律,所選的時間點具有代表性。

表2 冷藏帶魚貯藏期間的細菌總數變化Table 2 Changes of total viable counts in Trichiurus haumela during cold storage
2.2 細菌16S rRNA的V3可變區PCR擴增結果
以上述提取的微生物總基因組DNA為模板,用16S rRNA的V3可變區引物(帶GC夾子)進行PCR擴增,經1.5%瓊脂糖凝膠電泳檢測,4個樣品均獲得200bp左右大小的特異性擴增片斷,從圖1中來看,樣品均有較亮的擴增條帶,說明本實驗選用的PCR擴增條件合適,可用于后期的DGGE電泳分析。
2.3 冷藏帶魚貯藏期間細菌的DGGE圖譜分析與割膠測序
采用Dcode DGGE系統(Bio Rad)進行電泳,電泳緩沖液為1×TAE緩沖液,120V電泳4h左右。DGGE圖譜如圖2所示,3次重復每個取樣點的DGGE圖譜均無明顯差異。
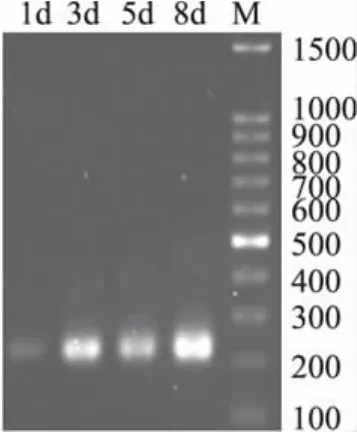
圖1 樣品中細菌16S rDNA的V3可變區PCR擴增產物電泳圖Fig.1 Representative electrophoresis photo of PCR amplification products of V3 regions of sample DNA

圖2 冷藏帶魚貯藏期間的細菌DGGE圖譜Fig.2 DGGE fingerprinting of PCR productsof Trichiurus haumela stored at 4℃
由圖2分析得出:樣品在貯藏的第1、3d,微生物條帶多,但強度相對較低。其中,條帶1在貯藏前期強度明顯,條帶8隨著貯藏時間的延長,強度逐漸增加。第5d時,條帶1強度明顯減弱,條帶8始終保持顯著亮度。條帶9與條帶10在貨架期結束時保持較清晰的亮度。4℃條件下,對保藏在離心管中的主要割膠條帶進行可變區V3擴增,經PCR擴增后,經1.5%瓊脂糖凝膠電泳檢測。由于條帶12、13與14在割膠回收后沒有得到純的DNA,經PCR擴增后不足以測序用,故后期未獲得這些條帶的相應序列。
2.4 冷藏帶魚貯藏過程中優勢菌的分析
通過對其余割膠回收條帶的PCR擴增產物送至上海生工測序,登錄NCBI,用BLAST軟件將所得序列在Genbank中與參考序列進行相似度比較,分析結果如表3。
DGGE圖譜上不同的條帶代表不同的微生物種類。由表 3可知,條帶 2、5、11分別為假單胞菌(Pseudomonassp.)、嗜水氣單胞菌(Aeromonas hydrophila)與殺魚假交替單胞菌(Pseudoalteromonas piscicida),其相似性在92%~94%,其余細菌相似性均在97%以上。條帶6、7經過核苷酸序列比對可知,其為南極細菌屬(Moritella)微生物,條帶3為假條帶號 GenBank登錄號 親緣關系最相似菌株 (%)交替單胞菌屬(Pseudoalteromona)微生物。結合圖2與表3比較,結果表明:帶魚樣品在冷藏過程中,其微生物具有顯著的差異性和明顯的動態演替規律。

表3 DGGE條帶分離的主要細菌16S rRNA部分序列相似性比較Table 3 Comparison of partial 16S rRNA sequencing fragments of dominating microbe similarity from DGGE bands
樣品在貯藏第 1d時,主要有嗜冷桿菌(Psychrobactersp.)、嗜水氣單胞菌(Aeromonas hydrophila)、熒光假單胞菌(Pseudomonas fluorescens)與弧菌(Vibrio sp.)等不同屬種的細菌。第3d時,主要有嗜冷桿菌(Psychrobacter sp.)、南極細菌(Moritella sp.)與熒光假單胞菌(Pseudomonas fluorescens),其中嗜水氣單胞菌(Aeromonas hydrophila)受到抑制,條帶強度明顯減弱。到第5d時,主要有熒光假單胞菌(Pseudomonas fluorescens)與弧菌(Vibrio sp.),嗜冷桿菌(Psychrobacter sp.)已基本抑制。到貯藏的第8d,帶魚樣品完全腐敗時,條帶8、9與10濃度最高,表明3種細菌所占比例相對最大。可見,熒光假單胞菌(Pseudomonas fluorescens)在貯藏期間比例逐漸增大,而希瓦氏菌(Shewanella sp.)與弧菌(Vibrio sp.)雖在初期不占主導,但隨著貯藏時間的延長,也逐漸發展成為主要腐敗菌,這與Gram[24]等關于特定腐敗菌(SSO)的理論相符。另外,在貯藏末期,還檢測到條帶11的微弱存在,主要是殺魚假交替單胞菌(Pseudoalteromonas piscicida)。
假交替單胞菌(Pseudoalteromonas sp.)與南極細菌(Moritella sp.)用傳統微生物方法培養難度大或不能培養,這就進一步證明了DGGE技術在研究微生物群落和種群多樣性方面的優越性。假交替單胞菌(Pseudoalteromonas sp.)主要分布于海洋環境中,其適應機制與存活策略具有多樣性和有效性[25]。Torben等采用實時定量PCR技術檢測了海洋樣品中假交替單胞菌的分布與豐度,結果表明假交替單胞菌在海洋環境中分布的廣泛性。南極細菌(Moritella sp.)屬菌株嗜鹽,生存在寒冷的海洋環境中,兼性厭氧為耐壓菌和兼性嗜壓深海菌[26]。由于PCR擴增產物片段大小僅為200bp,因此多數細菌僅能比對鑒定到屬,Shewan等[27]認為以食品保藏為目的的菌相分析將細菌種類分類到屬已經足夠,通過DGGE指紋技術則能清楚了解冷藏帶魚貯藏過程中主要優勢菌的種類與分布狀況。
3 結論
受到產品加工特性的限制,帶魚冷藏期間的腐敗問題一直困擾著廣大水產品生產加工企業。本研究采用PCR-DGGE指紋技術,對冷藏帶魚貯藏期間主要優勢菌的分布狀況進行了動態監測,并對其微生物群落結構進行分析。從中發現,嗜冷桿菌(Psychrobacter sp.)為帶魚貯藏初期的優勢菌,熒光假單胞菌(Pseudomonas fluorescens)在貯藏期間比例逐漸增大。希瓦氏菌(Shewanella sp.)與弧菌(Vibrio sp.)的比例明顯增加,在貯藏后期也發展成為優勢腐敗菌。在有氧冷藏的水產品中,腐敗菌主要為嗜冷性非發酵革蘭氏陰性桿菌。大多數產品中,假單胞菌、腐敗希瓦氏菌占絕對優勢[28]。其中,假單胞菌是一類嚴格好氧且適應能力極強的致腐菌,在有氧條件下,假單胞菌能迅速分解蛋白質,生長速率也顯著優于其他類細菌,是水產品的特定腐敗微生物[29-30]。腐敗希瓦氏菌大量存在于水和土壤中,水產品中攜帶較多,此前也將希瓦氏菌屬歸類到假單胞菌屬,被稱為腐敗交替假單胞菌。弧菌屬細菌多數存在于水生環境,尤其是海洋環境,為水產品中常見的一類菌。PCR-DGGE技術在食品微生物領域中具有較好的應用前景,它能快速監測食品成熟階段微生物的情況,評價產品中的微生物群落,并可根據環境變化分析食品中菌群的動力學規律,能大大提高微生物多樣性分析的準確性和真實性[31]。雖然其本身存在著一些不足,但由于該技術重復性高,且快速簡單等優點,能完全彌補傳統分離鑒定方法的不足,現已發展成為微生物學領域的重要研究手段。
[1]沈月新.水產食品學[M].北京:中國農業出版社,2001:23-24.
[2]丁仲田 .帶魚的氧化與黃變[J].肉品衛生,1996,12:14-17.
[3]Surendran P K,Joseph J,Shenoy A V,et al.Studies on spoilage of commercially important tropical fishes under iced storage[J].Fisheries Research,1989(7):1-9.
[4]Gram L,Huss H H.Microbiological spoilage of fish and fish products[J].International Journal of Food Microbiology,1996,33:121-137.
[5]Gillespie N C,Macrae I C.The bacterial flora of some Queensland fish and its ability to cause spoilage[J].Journal of Applied Bacteriology,1975,39:91-100.
[6]AmannR I,LudwigW,SchleferK H.Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J].Microbial Reviews,1995,59:143-169.
[7]Pace N R.A molecular view of microbial diversity and the biosphere[J].Science,1997,276:734-740.
[8]Muyzer G,De Waal E C,Uitterlinden A G.Profiling of complex microbialpopulations by denaturing gradientgel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J].Applied of Environment Microbiology,1993,59:695-700.
[9]Gurtler V,Garrie H D,Mayall B C.Denaturing gradient gel electrophoretic multilocussequence typing ofStaphylococcus aureus isolates[J].Electrophoresis,2002,23:3310-3320.
[10]Theelen B,Silvestri M,Gueho E,et al.Identification and typing of Malassezia yeasts using amplified fragment length polymorphisms(AFLP),random amplified polymorphic DNA(RAPD)and denaturing gradient gel electrophoresis(DGGE)[J].FEMS Yeast Research,2001(1):79-86.
[11]馬俊孝,季明杰,孔健.PCR-DGGE技術在微生物物種多樣性研究中的局限性及其解決措施[J].食品科學,2008,29(5):493-497.
[12]高菲,孫慧玲,許強,等.刺參消化道內含物細菌群落組成的PCR-DGGE 分析[J].中國水產科學,2010,17(4):671-680.
[13]Maria B H,Morten S,Bjorn T L,et al.Characteriasation of the dominantbacterialpopulation in modified atmosphere packaged farmed halibut(Hippoglossus hippoglossus)based on 16S rDNA-DGGE[J].Food Microbiology,2007,24:362-371.
[14]Maria B H,Bjorn T L,Morten S,et al.Characterisation of the bacterial flora of modified atmosphere packaged farmed Atlantic cod(Gadus morhua)by PCR-DGGE of conserved 16S rRNA gene regions[J].International Journal of Food Microbiology,2007,117:68-75.
[15]蔡秋杏,李來好,陳勝軍,等.液熏羅非魚片加工過程中微生物群落的 PCR-DGGE 分析[J].食品科學,2009,30(23):35-40.
[16]GBT 4789.2-2008,食品衛生微生物學檢驗——菌落總數測定[S].
[17]薩姆布魯克,拉塞爾.分子克隆實驗指南[M].第三版,北京:科學出版社,2002.
[18]Muyzer G.DGGE/TGGE a method for identifying genes from natural ecosystems[J].Current of Opinion Microbiology,1999,2:317-322.
[19]Muyzer G,Smalla K.Application of denaturing gradient gel electrophoresis(DGGE) and temperature gradient gel electrophoresis(TGGE)in microbial ecology[J].Antonie Van Leeuwenhoek,1998,73:127-141.
[20]Blixt Y,Borch E.Comparison of shelf life of vacuum-packed pork and beef[J].Meat Science,2002,60:371-378.
[21] NübelU,Engelen B,Felske A,etal.Sequence heterogeneities of genes encoding 16S rRNAs in Paenibacillus polymyxa detected by temperature gradient gel elelectrophoresis[J].Journal of Bacteriology,1996,178:5636-5643.
[22]何麗明,李志勇,吳杰,等.基于PCR-DGGE指紋的南海海綿共附生細菌優勢種群的揭示與系統發育分析[J].微生物學報,2006,3(46):487-491.
[23]中華人民共和國農牧漁業部.SC128-84.中華人民共和國農牧漁業部部標準——鮮帶魚[S].1984.11.
[24]Gram L,Dalgaard P.Fish spoilage bacteria-problems and solutions[J].Current Opinion in Biotechnology,2002,13:262-266.
[25]席宇,朱大恒,劉紅濤,等.假交替單胞菌及其胞外生物活性物質研究進展[J].微生物學通報,2005,32(3):108-112.
[26]楊秀霞.南極細菌Moritella sp.低溫脂肪酶基因的克隆與表達[D].青島:中國海洋大學,2004.06.
[27]Shewan J M.The microbiology of fish and fishery products-a progress report[J].Applied Microbiology,1971,34:299-315.
[28]許鐘,肖琳琳,楊憲時.羅非魚特定腐敗菌生長動力學模型和貨架期預測[J].水產學報,2005,29(4):540-546.
[29]劉壽春,周康,鐘賽意,等.淡水養殖羅非魚中病原菌與腐敗菌的分離與鑒定初探[J].食品科學,2008,29(5):327-331.
[30]許鐘,楊憲時,郭全友,等.波動溫度下羅非魚特定腐敗菌生長動力學模型與貨架期預測[J].微生物學報,2005,45(5):798-801.
[31]高蕙文,呂欣,董明盛.PCR-DGGE指紋技術在食品微生物研究中的應用[J].食品科學,2005,26(8):465-468.
PCR-DGGE analysis on the dynamic changes of microorganisms in cutlassfish(Trichiurus haumela)under the cold storage
LAN Wei-qing,XIE Jing*
(College of Food Science and Technology,Shanghai Ocean University,Shanghai Engineering Research Center of Aquatic Product Processing & Preservation,Shanghai 201306,China)
In this paper,the main microbe dynamic changes of cutlassfish(Trichiurus haumela)were studied under the cold storage(4±1)℃ so as to provide the experimental references for extending its shelf-life.The total bacterium DNA of Trichiurus haumela were extracted by SDS(Sodium Dodecyl Sulfate)pyrolysis method and the PCR amplification of V3 variable region of 16S rRNA were taken in order to get the dynamic changing fingerprint spectra by DGGE technology.It was claimed that there were abundant microbial population on the products in the initial stage and Psychrobacter sp.was the main bacterium firstly.The proportion of Pseudomonas fluorescens was increased from the initial periods.With the extension of storage time,the proportion of Shewanella sp.and Vibrio sp.also increased gradually and they took the place of Psychrobacter sp.to be the dominant bacteria finally.
TS254.1
A
1002-0306(2012)15-0118-05
2012-02-13 *通訊聯系人
藍蔚青(1977-),男,博士,助理研究員,研究方向:食品冷凍冷藏學。
“十二五”國家支撐計劃項目(2012BAD38B09);上海市科委工程中心建設(11DZ2280300);上海海洋大學博士啟動基金(A-2400-11-0202);上海市教育委員會重點學科建設項目(J50704)。

