穩定同位素技術研究濕地甲烷產生的微生物過程進展
李 清,林光輝
(1.清華大學 地球系統科學中心 地球系統數值模擬教育部重點實驗室,北京 100084;2.清華大學 深圳研究生院,海洋材料與技術學部,深圳 518055)
在經歷了約十年的平穩期后,2007年開始全球大氣甲烷濃度又出現增長,而且增長主要來自北半球[1]。Kai[2]通過大氣甲烷穩定同位素監測、大氣反演、地球系統模型預測等多種研究手段綜合分析認為,全球甲烷過去十年排放量增速出現下降的主要原因是北半球微生物資源減少,其中,亞洲特別是中國水稻田甲烷排放量的下降可能是主導因素[2]。Bousquet[3]研究發現,濕地甲烷排放量的變化是過去20年全球甲烷排放出現年際間波動最重要的原因。1999年以來,全球濕地甲烷排放量的下降抵消了人類活動引起的大氣甲烷濃度上升,如果濕地甲烷的排放量恢復到1990年的高排放量水平,全球大氣甲烷的濃度將在經歷這短暫的穩定之后重新開始增長[3]。古氣候學家的研究[4]也發現,最后一次冰期的消失更可能是由于北半球濕地的甲烷排放引起而非甲烷冰的融化。這些研究均說明了全球濕地特別是北半球濕地甲烷排放對于調節大氣甲烷濃度變化具有十分重要的作用。
濕地僅占陸地面積6%~7%,但其碳含量卻占陸地土壤碳儲量的68%[5],凈初級生產力占全球的10%。濕地由于較低的有機質分解率和較高的生產力而成為重要的碳匯,但同時也是重要的甲烷排放源。根據政府間氣候變化專門委員會(Intergovernmental Panel on Climate Change,IPCC)2007年的評估結果,濕地甲烷排放量占全球甲烷排放量的20%~39%。對于全球濕地甲烷的排放,目前不同的估算結果相差較大[6]。例如,Cao[7]利用過程模型研究發現,全球濕地的甲烷排放量為145 Tg·a-1,其中,天然濕地的排放量為92 Tg·a-1,水稻田的排放為53 Tg·a-1[7]。然而,Walter[8]對天然濕地的甲烷排放計算的結果則達到260 Tg·a-1,其中約60%釋放到大氣中,其他部分在排放到大氣之前就已經在土壤中被氧化了[8]。而Bastviken[9]研究發現,盡管未被計算在全球溫室氣體通量中,陸地湖泊、水庫和河流的總甲烷排放量事實上達到了103 Tg·a-1,大約抵消了目前陸地碳匯總量的25%[9]。
這種差異產生的重要原因是不同類型和不同區域濕地甲烷排放的空間格局以及隨時間的動態變化信息比較缺乏。而要得到這些結論,除了需要建立更多的甲烷通量數據監測網絡外,還需要對濕地甲烷產生的微生物過程進行深入的研究,從而為甲烷過程建模和通量數據分析提供支撐。
盡管國際上關于產甲烷相關微生物的研究非常多,以產甲烷菌(Methanogens)或甲烷氧化菌(Methanotrophs) 作為主題詞搜索web of knowledge,查找到的文獻超過10 000篇,但大多集中在微觀生物學領域。與全球變化以及溫室氣體產生機制等主題相關的研究主要包括甲烷相關微生物的種群多樣性和群落結構隨環境因子變化響應情況[10],包括對水位變化的響應[11],對溫度變化的響應[12],對營養元素濃度變化的響應[13],以及甲烷相關微生物在土壤中的垂直分布情況[14],產甲烷細菌的數量隨著季節變化以及土壤深度而變化[15]等。對于甲烷通量的空間格局與甲烷相關微生物的群落結構之間的直接關系研究還比較缺乏。而事實上,如果將產甲烷菌的數量、種群變化規律與甲烷排放量的變化直接相關聯將更具有科學與現實意義[16]。
本文在簡要介紹濕地甲烷產生的基本微生物過程后,重點闡明如何利用穩定同位素技術研究產甲烷的不同途徑和不同微生物種類在甲烷產生過程的作用,并分析該領域的最新研究進展和未來研究重點,以期為我國開展這方面的研究提供一些信息。
1 甲烷產生的基本微生物過程及其對甲烷排放計算的意義
目前的研究表明,濕地排放的甲烷主要是由產甲烷微生物在厭氧條件(Eh值小于-200 mV)下把乙酸、CO2或甲醇等作為電子受體,乙酸或H2作為電子供體,發生氧化還原反應而產生的,有機物分解產生甲烷的過程示于圖1。
其中,乙酸和H2是產甲烷微生物所利用的主要基質,其他的基質對于甲烷產生的貢獻一般不超過5%[18]。根據使用基質的不同,產甲烷微生物可以分為CO2型、甲基型和乙酸型三大類,產甲烷微生物的三種類型列于表1。
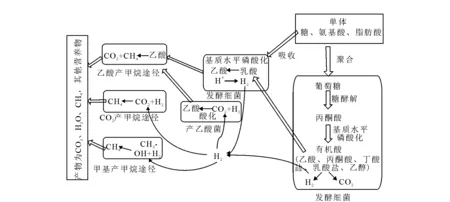
圖1 有機物分解產生甲烷的過程[17] Fig.1 Methane production pathways during organic matter decomposition[17]

類型所利用的基質代表性微生物CO2型CO2、CO和甲酸鹽甲酸甲烷桿菌(Methanobacterium)產甲烷球菌(Methanococcus)甲基型甲醇、甲胺、二甲胺、三甲胺甲硫醇和二硫醇甲烷八疊球菌(Methanosarcins)甲烷八疊球菌(Methanosarcina)乙酸型醋酸鹽甲烷絲菌(Methanothrix)鬃毛甲烷菌(Methanosaeta)
由于產甲烷微生物不能直接利用復雜化合物,因而其生存必須依賴其他至少三類微生物的存在,包括水解、發酵以及產氫產乙酸細菌。
以上分析可見,甲烷的產生主要由以下三個反應決定的:
CH3COOH→CO2+CH4
(1)
CO2+4H2→2H2O+ CH4
(2)
4 CH3OH→H2O+ CO2+3CH4
(3)
因而甲烷產生總量m可以表示為:
m=ma+mc+mi
(4)
或者
m=(fma+fmc+fmi)m,同時fma+fmc+fmi=1
(5)
其中,m代表甲烷產生的總量或者速率,ma代表來自于醋酸的甲烷,mc代表來自于CO2的甲烷,mi代表來自于其他化合物比如甲醇的甲烷;fma則表示來自于醋酸的甲烷的占總甲烷的比例,fmc表示來自于CO2的甲烷占總甲烷的比例,fmi則表示來自于甲基化合物等其他途徑的甲烷占總甲烷的比例。
由于反應(3)的發生主要是在高鹽的環境中,且甲烷的產生量很低[19],因而在大多數情況下甲烷的產生來自反應(1)和反應(2) ,因而上述方程可以進一步簡化為:
ma=(1-fmc)m
(6)
已有研究發現,fmc在不同的環境條件下不一樣,而且受到溫度、植被種類和富營養化程度等多個因素的影響[20],因而fmc是計算甲烷產生的各種過程模型中十分重要的參數。
2 穩定同位素技術在產甲烷途徑貢獻分析中的應用
目前,對于fmc的計算主要有3種方法[19]:(1) 放射性同位素示蹤,例如用14C標記區分碳酸氫根和醋酸中的碳然后測定產生的甲烷中的14C豐度進而計算碳酸氫根和醋酸各自的轉化速率;(2) 利用特異性的抑制劑比如CH3F抑制利用醋酸產生甲烷的反應,此時產生的甲烷則認為是來自于CO2;(3)利用穩定同位素的分餾效應,通過測定產物和底物的同位素比值來計算不同產甲烷途徑對于總甲烷的貢獻率。其中,方法(3)由于操作簡單,受實驗條件以及環境的影響小且更加精確而成為目前計算fmc最重要的方法之一[19]。
不同的產甲烷途徑中的同位素分餾過程如圖2所示,因而在不考慮反應(3)的發生時,可以得到以下質量平衡方程:
δCH4=fmcδmc+ (1-fmc)δma
(7)
解上述方程可得到:
fmc= (δCH4-δma) / (δmc-δma)
(8)
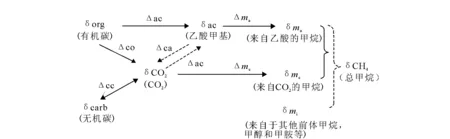
圖2 甲烷產生過程中的同位素分餾[19] Fig.2 Isotopic fractionation in methane producing processes[19]
如果甲烷產生反應的同位素效應是確定的,則:
δma=δac+Δma
(9)
δmc=δCO2+ Δmc
(10)
而對于反應A→B,通常需要定義一個同位素分餾因子αAB:
αAB= (δA + 103) / (δB + 103)
(11)
所以αma和αmc可以分別表示為:αma= (δac+ 103) / (δma+ 103)
(12)
αmc= (δCO2+ 103) / (δmc+ 103)
(13)
即δma和δmc可以重新表示為:
δma=(1/αma)(δac+ 103-αma103)
(14)
δmc=(1/αmc)(δCO2+ 103-αmc103)
(15)
由以上分析可見,不考慮反應(3)的情況下,根據方程(8)、 (14)和(15),如果已知αma和αmc的值,則fmc可以很容易通過測定CH4、CO2和乙酸甲基中的δ13C來確定。利用穩定同位素信號進行產甲烷途徑貢獻分析的研究總結列于表2,由表2可見,目前關于δac的信息十分缺乏,僅在Cape Lookout Bight和日本的水稻田土壤中有所研究,還有一些研究測定了總乙酸的δ13C,由于乙酸羧基和乙酸甲基的δ13C相差超過14‰[19],總乙酸的δ13C對于fmc的準確計算意義不大。

表2 利用穩定同位素信號進行產甲烷途徑貢獻分析[19]
另一方面,αma和αmc在研究中更難被確定。由于很多中間值不容易得到,通常用簡單的參數αc來對不同產甲烷途徑的貢獻進行區分。定義αc= (δCO2+ 103) / ( δCH4+ 103),通過對αc進行計算得出:當αc>1.065時,CO2途徑占主導;而當αc<1.055時,乙酸途徑占主導[21]。然而,這種方法只能進行粗略的定性區分,無法得到fmc的精確值。
為了精確計算fmc,可以利用特定培養基分別培養利用不同途徑產甲烷的微生物,希望對αma和αmc進行精確計算[19]。然而,αma和αmc受微生物種類、溫度以及可利用的H2濃度等因素影響變化很大,其中αmc在1.031~1.077,αma在1.007~1.027[19]。盡管范圍變化很大,但是能夠很清楚的看出αmc要顯著的大于αma。這些結果部分驗證了前文中對αc的計算得出的結論,同時也指出αma和αmc在各種特定類型生態系統中的確定還需要更多深入研究。
3 同位素標記技術在產甲烷微生物功能研究中的應用
由于αma和αmc受微生物種類的影響很大,因而對不同類型濕地中的微生物群落結構進行分析,特別是對產甲烷微生物種類、種群大小以及其功能情況進行研究將對αma和αmc的計算提供重要信息。利用分子生物學技術能夠對土壤微生物的種群數量、種群大小甚至功能基因的表達豐度等進行定性或半定量分析,比如Godin[22]利用末端限制性片段長度多樣性(Terminal Restriction Fragment Length Polymorphism ,T-RFLP)分析方法發現,泥炭地甲烷產生速率與產甲烷菌群落豐富度之間在春季沒有相關性,但是生長季節存在明顯相關關系,但這種分析更多的是起到指示和推測微生物在環境中的功能[22],難以將這些參數與微生物具體代謝反應過程間建立直接聯系。同位素標記技術與分子生物學技術的結合為建立微生物的群落結構、功能結構與代謝功能之間的關系研究提供了可能[22]。
John[24]利用放射性同位素14C標記法和T-RFLP法分別研究了鹽沼不同土壤深度的產甲烷微生物對不同類型底物的利用速率和產甲烷微生物的種群變化,表層土壤甲胺的利用速率最快,與此同時,表層土壤微生物的分布則以利用甲胺的甲烷八疊球菌(Methanosarcinales)的種群數量占據主導。深層土壤則是乙酸的利用速率最快,而同時主要利用乙酸的鬃毛甲烷菌(Methanosaeta)的種群數量占據主導地位[23]。
目前同位素標記中應用最為廣泛的是穩定同位素探測技術(Stable Isotope Probing,SIP),是指一類利用穩定同位素(比如13C,15N)標記微生物所利用的基質,然后對微生物經過代謝反應后被標記DNA或RNA等進行分析的技術,根據檢測的分子不同進而區分為DNA-SIP和RNA-SIP[24]。Lu等[26]在水稻田中加入13C標記的CO2后研究發現,水稻根系土壤中一類被稱為Rice ClusterⅠ的產甲烷微生物RNA中13C的比例顯著升高,說明其利用了水稻根呼吸產生的CO2,因而在水稻田甲烷產生中可能起著重要作用,盡管與其他幾種產甲烷微生物相比,其在水稻土壤中的相對數量并不是最高的[25]。這個結果顯示了同位素標記技術相對分子生物學技術的優勢,如果利用T-RFLP法對水稻根系周圍的產甲烷菌的種群數量進行研究,Rice ClusterⅠ會因為種群數量偏低而被認為其對水稻田甲烷產生的貢獻也偏低,如果將不同產甲烷途徑的貢獻率和同位素標記的結果結合起來,水稻田產甲烷微生物的種群數量和實際代謝功能與甲烷通量之間的關系將更加明確。Conrad 等[27]通過測定CO2和CH4的穩定同位素比值來粗略估算CO2途徑和乙酸發酵途徑的相對產甲烷貢獻率并結合T-RFLP技術發現,溫度高于40 ℃時,土壤產甲烷以完全CO2還原途徑為主,而溫度低于30 ℃時,CO2還原途徑和乙酸還原途徑同時存在[26]。Conrad等的研究很好的結合穩定同位素分餾效應和分子生物學技術,但沒有測定土壤乙酸中甲基的碳同位素比值,因而只能對不同產甲烷途徑的貢獻率進行粗略計算。
4 展 望
全球甲烷排放在2007年前的十年中增長速率下降,這引發了科學家們激烈的爭論,但參與討論的主要是大氣科學家,盡管濕地和微生物的作用可能是決定性因素[2],而濕地生態學家和微生物學家參與程度卻并不高。微生物種群的復雜性、微生物種群大小與其實際代謝功能之間的非線性關系、研究手段的缺乏可能是主要原因。由于研究手段的限制,利用穩定同位素分餾效應對不同類型的濕地中各種產甲烷途徑的比重進行精確分析還需要付出巨大的努力,尤其是αma和αmc受到的影響因素十分復雜,因而對于不同類型濕地中αma和αmc的確定將是甲烷產生相關微生物過程的研究重點。與此同時,盡管基因組學研究的發展和應用使微生物種群多樣性有了更為深入的研究,但許多微生物的代謝功能仍然很難確定[22],即便是已經進行了全基因組測序基因組的模式生物大腸桿菌(E. coli)基因組中仍然有大約50%的基因其功能是未知的[27]。而同位素標記技術特別是最近發展的SIP技術能夠通過標記微生物利用的底物來對微生物在環境中的功能進行研究。因此,如果將分子生物學技術、SIP和穩定同位素分餾效應等結合起來,可以更為深入研究濕地甲烷產生的微生物過程以及其與環境因子之間的關系,為減小濕地甲烷排放量估算的不確定性提供機理上的信息。
參考文獻:
[1] Rigby M, Prinn R, Frase P,et al. Renewed gro-wth of atmospheric methane[J]. Geophysical Research Letters, 2008, 35:L22805.
[2] Kai FM, Tyler SC, Randerson JT, et al. Reduced methane growth rate explained by decreased Northern Hemisphere microbial sources[J]. Nature, 2011, 476:194-197.
[3] Bousquet P, Ciais P, Miller JB, et al. Contribution of anthropogenic and natural sources to atmospheric methane variability[J]. Nature, 2006, 443:439-443.
[4] Fischer H, Behrens M, Bock M, et al. Changing boreal methane sources and constant biomass burning during the last termination[J]. Nature, 2008, 452:864-867.
[5] Schlesinger WH. Biogeochemistry: an analysis of global chang[M]. Academic press, 1997.
[6] Solomon S. Climate change 2007: the physical science basis: contribution of Working Group I to the Fourth Assessment Report of the Intergovernmental Panel on Climate Change[M]. Cambridge Univ Pr, 2007.
[7] Cao M, Gregson K, Marshall S. Global methane emission from wetlands and its sensitivity to climate change[J]. Atmospheric Environment, 1998, 32:3 293-3 299.
[8] Walter B, Heimann M, Matthews E. Modeling modern methane emissions from natural wetlands. I- Model description and results[J]. Journal of Geophysical Research D Atmos, 2001, 106:34.
[9] Bastviken D, Tranvik LJ, Downing JA, et al. Freshwater methane emissions offset the continental carbon sink[J]. Science, 2011, 331:50.
[10] Yavitt JB, Yashiro E, Cadillo-Quiroz H, et al. Methanogen diversity and community composition in peatlands of the central to northern Appalachian Mountain region[J].North America Biogeochemistry, 2012, 109:117-131.
[11] Yrj?l? K, Tuomivirta T, Juottonen H, et al. CH4production and oxidation processes in a boreal fen ecosystem after long‐term water table drawdown[J]. Global change biology, 2011, 17:1 311-1 320.
[12] Kim SY, Freeman C, Fenner N, et al. Functional and structural responses of bacterial and methanogen communities to 3-year warming incubation in different depths of peat mire[J]. Applied Soil Ecology, 2012, 57:23-30.
[13] Eriksson T, ?quist MG, Nilsson MB. Production and oxidation of methane in a boreal mire after a decade of increased temperature and nitrogen and sulfur deposition[J]. Global change biology, 2010, 16:2 130-2 144.
[14] Cadillo‐Quiroz H, Br?uer S, Yashiro E, et al. Vertical profiles of methanogenesis and methanogens in two contrasting acidic peatlands in central New York State, USA[J]. Environmental Microbiology, 2006, 8:1 428-1 440.
[15] 湯博, 唐杰, 吳俐莎, 等. 若爾蓋高原產甲烷菌數量的時空差異性[J]. 微生物學通報, 2010, 37:1 706-1 711.
Tang Bo, Tang Jie, Wu Lisha, et al. Time-space change of methanogen amount from zoige plateau[J]. Microbiology China, 2010, 37: 1 706-1 711(in Chinese).
[16] 金城. 高原濕地產甲烷菌[J]. 微生物學通報, 2010, 37:1 705.
Jin Cheng. Methanogensin zoige wetland[J]. Microbiology China, 2010, 37:1 705(in Chinese).
[17] Reddy KR, DeLaune RD. Biogeochemistry of wetlands: science and applications[M]. CRC, 2008.
[18] Segers R. Methane production and methane consumption: a review of processes underlying wetland methane fluxes[J]. Biogeochemistry, 1998, 41:23-51.
[19] Conrad R. Quantification of methanogenic pathways using stable carbon isotopic signatures: a review and a proposal[J]. Organic Geochemistry, 2005, 36:739-752.
[20] Conrad R. Contribution of hydrogen to methane production and control of hydrogen concentrations in methanogenic soils and sediments[J]. Fems Microbiology Ecology, 1999, 28:193-202.
[21] Hornibrook ERC, Longstaffe FJ, Fyfe WS. Evolution of stable carbon isotope compositions for methane and carbon dioxide in freshwater wetlands and other anaerobic environments[J]. Geochimica Et Cosmochimica Acta, 2000, 64:1 013-1 027.
[22] Godin A, McLaughlin JW, Webster K, et al. Methane and methanogen community dynamics across a boreal peatland nutrient gradient[J]. Soil Biology and Biochemistry, 2012, 48: 96-105.
[23] Chen Y, Murrell JC. When metagenomics meets stable-isotope probing: progress and perspectives[J]. Trends in microbiology, 2010, 18:157-163.
[24] John PR, Brock F, Banning N, et al. Changes in methanogenic substrate utilization and communities with depth in a salt-marsh, creek sediment in southern England. Estuarine[J]. Coastal and Shelf Science, 2012, 96: 170-178.
[25] Dumont MG, Murrell JC. Stable isotope probing - linking microbial identity to function[J]. Nature Reviews Microbiology, 2005, 3:499-504.
[26] Lu Y, Conrad R. In situ stable isotope probing of methanogenic archaea in the rice rhizosphere[J]. Science, 2005, 309:1 088-1 090.
[27] Conrad R, Klose M, Noll M. Functional and structural response of the methanogenic microbial community in rice field soil to temperature change[J]. Environmental Microbiology, 2009, 11:1 844-1 853.
[28] Serres MH, Gopal S, Nahum LA,et al. A functional update of the Escherichia coli K-12 genome[J]. Genome Biol, 2001, 2:1-35.

