長吻養殖群體與野生群體遺傳多樣性分析
肖明松 崔 峰 康 健 馬玉涵
(安徽科技學院生命科學學院, 鳳陽 233100)
1 材料與方法
1.1 材料
主要試劑 蛋白酶K、瓊脂糖、飽和酚購自上海生工生物工程技術有限公司, 引物DL1 (5′-AC CCCTGGCTCCCAAAGC-3′) 和 DH2 (5′-ATCTTAG CATCTTCAGTG-3′)[18]由上海生工生物工程技術有限公司合成,Taq酶、dNTPs 購自大連TaKaRa公司;其他試劑均為國產分析純。
主要儀器 PCR 儀、核酸分析儀和離心機為Eppendorf (德國), Alphalmager Is-2200凝膠圖像分析系統(美國), 電泳儀等常規儀器均為國產。
1.2 方法
模板 DNA的制備 每個樣品隨機取肌肉100—200 mg, 剪碎, 勻漿后置于1.5 mL 的微量離心管中, 加入含蛋白酶K 的裂解液(10 mmol/ L Tris-HCl, pH 8.0, 1 mmol/ L EDTA, pH 8.0, 1%SDS) ,55℃消化過夜。次日分別用Tris-飽和酚、氯仿/異戊醇(24∶1) 提取、純化DNA, 之后用?20℃冷凍的無水乙醇沉淀DNA, 自然干燥后, 溶于滅菌雙蒸水中,所得用基因組 DNA 樣品用紫外分光光度計測量DNA 樣品的濃度和純度, 同時輔以瓊脂糖凝膠電泳檢測基因組 DNA 的完整性并估測分子量, 置于?20℃冰箱保存備用。
PCR 反應 PCR 反應總體積為 30 μL, 其中含 10×buffer反應緩沖液 3.0 μL, 3 μL dNTP(2.5 mmol/L), 0.4 μmol/L 引物, 0.25 μLTaq酶, 25 ng DNA, 用滅菌雙蒸餾水補足體積。PCR反應條件為: 94℃下預變性5min, 94℃下變性30s, 58℃下退火1min, 72℃下延伸1min, 共35個循環, 最后72℃下延伸10min, 4℃保存。
瓊脂糖凝膠電泳 PCR 擴增產物在1.2 %的瓊脂糖凝膠(含0.5 g/mL EB) 上電泳, 120 V 穩壓電泳1h。電泳結果在Alphalmager Is-2200'凝膠圖像分析系統中拍照、保存。
DNA序列測定和分析 經2 %瓊脂糖凝膠電泳檢測后, 將 PCR 產物送往上海生工生物工程技術有限公司進行序列測定, 測得的序列用Clustal X 1.81 程序[19]進行比對, 并輔以人工校對, 進行同源性比較。所有序列均遞交 GenBank (序列號為JN131500-JN131517和 AB054127-AY297099)。利用MEGA 3.0 軟件包[20]分析序列特征、統計堿基組成和轉換與顛換值、計算遺傳差異和遺傳距離。采用鄰接法(Neighbor-joining tree, NJ)構建分子系統發育樹, 系統樹各分支的置信度由 1000次自舉法(Bootstrap)檢驗。利用DnaSP 4.0軟件[21]計算各群體的單倍型多樣度(H)、平均核苷酸差異數(K)、核苷酸多態性(Pi); Arlequin 3.1軟件[22]進行群體間的歐氏距離平方(Squared euclidean distance) 矩陣分子變異分析(AMOVA), 統計長吻養殖群體與野生群體間的遺傳分化指數(Fst), 并根據Nm≈(1?Fst)/(2Fst)得到群體間的基因流值(Nm)。為進行譜系生物地理學(Phylogeography)分析, 利用 TCS 1.21[23]軟件, 依據統計簡約原理[24], 構建單倍型的網狀圖。相對于傳統的系統發育分析方法, 網狀圖更能夠有效地揭示種群水平上的譜系關系(Genealogical relationship),更清楚地反映與種內基因進化有關的特性, 如是否保持祖先的單倍型、是否存在多重后代的單倍型以及種群間的序列差異較低的特性[25]。
2 結果
2.1 長吻線粒體控制區序列變異、單倍型和單倍型分布
8個, 缺失位點1個。含有4個轉換位點(Ts), 1個顛換位點(Tv), 平均轉顛換比(Ts/Tv)為8.27。在長吻野生群體中, 69個個體共檢測到 35個變異位點(占全部堿基數的4.43%)。其中簡約信息位點24個, 單突變位點10個, 缺失位點1個。含有5個轉換位點(Ts), 1個顛換位點(Tv), 平均轉顛換比(Ts/Tv)為9.80。在長吻養殖群體和野生群體中, 135個個體共檢測到42個變異位點, 其中簡約信息位點28個,單突變位點12個, 缺失位點2個(表1)。長吻養殖群體和野生群體的堿基變化不大(表2), 其中堿基G的含量顯著低于其他堿基的含量, A+T含量(60.6%)明顯高于C+G含量(39.4%)。
表1 長吻線粒體控制區單倍型分布及單倍型的變異位點Tab.1Alignment of mtDNAcontrol region haplotypes of variable positions and distribution of mtDNAcontrol region haplotypes of Leiocassis longirostris
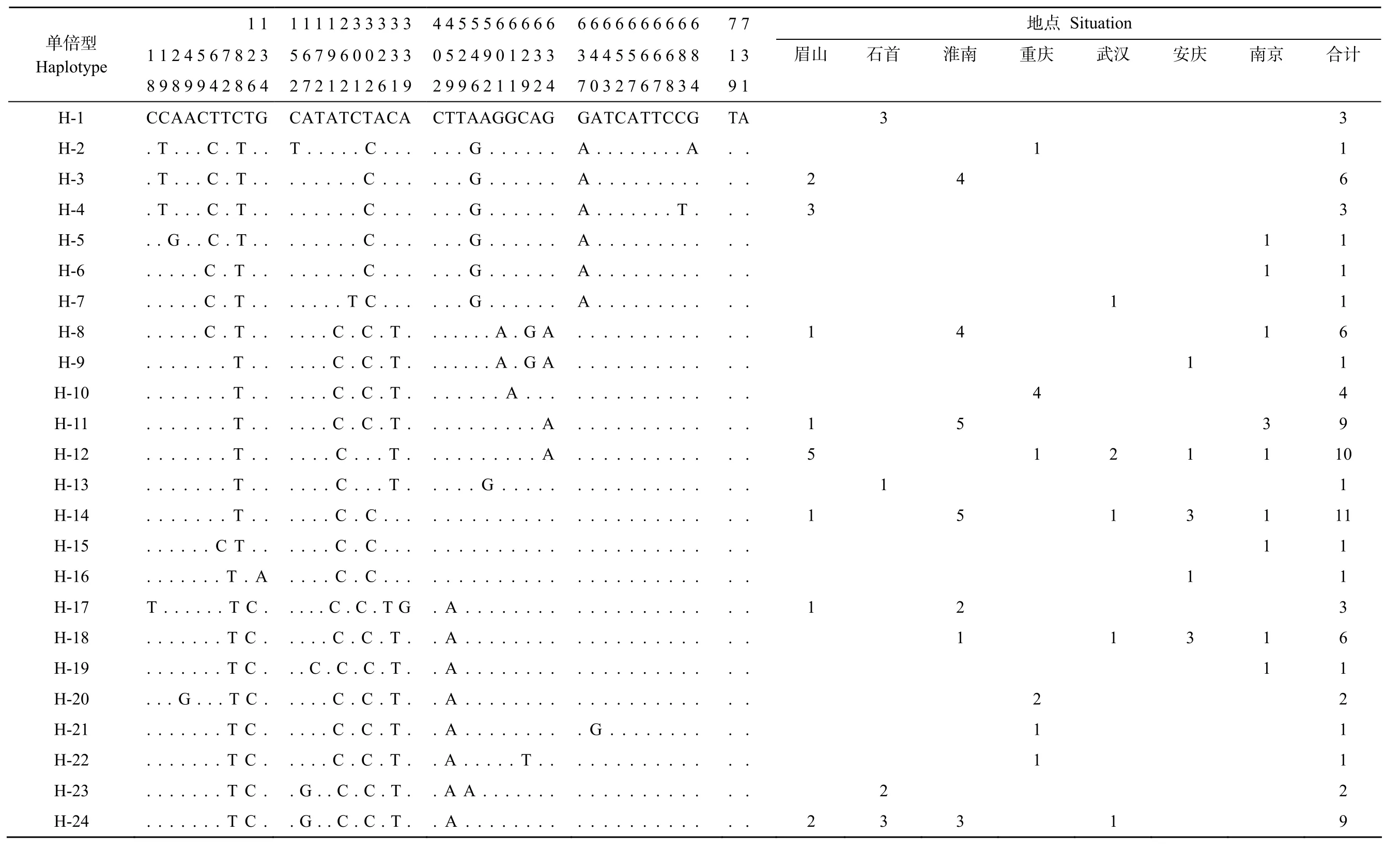
表1 長吻線粒體控制區單倍型分布及單倍型的變異位點Tab.1Alignment of mtDNAcontrol region haplotypes of variable positions and distribution of mtDNAcontrol region haplotypes of Leiocassis longirostris
計合316311161491 011 11136121129京111311111南慶安11313 ation漢武1 2111 Situ點地慶1 41 211重南淮4 455213首3 1 23石山2311511 2眉7 7 1 3 9 1 TA................................................................A........CG.....T................................TC..............................................AT..............................................TC......................................G.......6 6 6 6 6 6 6 6 6 6 3 4 4 5 5 6 6 6 8 8 7 0 3 2 7 6 7 8 3 4 GA.......... A.. A.. A.. A.. A.. A.. ... ... ... ... ... ... ... ... ... .AG G A G A.A.A.. ... ... .GC............A.A.A........................T......AG..4 4 5 5 5 6 6 6 6 6 0 5 2 4 9 0 1 2 3 3 2 9 9 6 2 1 1 9 2 4..............................TA.G.G.G.G.G.G..................G...................A...CT...................................A.A.A.A.A.A.A.A CA.1 1 1 2 3 3 3 3 3 6 7 9 6 0 0 2 3 3 7 2 1 2 1 2 6 1 9................G.......TA...........T.T.T.T.T.T....C..T.T.T.T.T.T.T.T TC.C.C.C.C.C T C..C.C.C.C.....C.C C..C.C.C.C.C.C.C.C.............C.C.C.C.C.C.C.C.C.C.C.C.C.C.C.C TA...................................C......G.G.CA....1 1 1 TG.. T 1 1 2 4 5 6 7 8 2 3 5 8 9 8 9 9 4 2 8 6 4 2.. ... ... ... ... ... ... ... ... ... ... ... ..A C. .C. .C. .C. .C. .C. .C. .C. .CTTC.T.T.T.T.T.C.T.C.T.C.C.T...T...T...C.T...T...T...C C T..T.T...T.T...T...T.T.T.T..AA...C..........CC.........T.T.T G...............................................T............G................e型倍單Haplotyp H-1 H-2 H-3 H-4 H-5 H-6 H-7 H-8 H-9 H-10 H-11 H-12 H-13 H-14 H-15 H-16 H-17 H-18 H-19 H-20 H-21 H-22 H-23 H-24
2.2 單倍型分布及單倍型聚類分析
利用DnaSP 4.0 軟件計算各群體的單倍型(表1)。由表1可知, 在長吻不同群體中, 135個體共檢測出46 個單倍型, 即H-1 - H-46。其中28個獨享單倍型, 占 20.74%,18個共享單倍型, 單倍型 H-8、H-11、H-12、H-14、H-18、H-24、H-37和 H-43為野生群體和養殖群體共享。另外, 在長吻養殖群體中, 66個體共檢測出 18 個單倍型, 在野生群體中 69個體共檢測出36 個單倍型(表略)。TCS 1.21軟件生成的統計簡約網狀圖顯示出星狀的分布態勢,沒有將 46個單倍型區分為對應不同地理區域或者地理種群的單系群。單倍型H- 14位于星狀圖的中心,其他單倍型則與其依短支相連(圖1)。依據溯祖理論 (Coalescence theory), 線粒體控制區單倍型的星狀拓撲結構表明所研究的種群經歷了明顯的種群擴張[26], 單倍型 H-14由于其分布最廣和享有該單倍型的個體在所有的種群中所占的比重也明顯的高于其他單倍型, 而且在網狀圖中處于基部位置, 說明單倍型 H-14可能是原始的單倍型。
表2 長吻序列堿基組成Tab.2 Nucleotide compositions of mtDNA control region in Leiocassis longirostris

表2 長吻序列堿基組成Tab.2 Nucleotide compositions of mtDNA control region in Leiocassis longirostris
群體 Populations T C A G眉山 Meishan 31.6 25.2 29.1 14.1石首 Shishou 31.5 25.2 29.1 14.2淮南 Huainan 31.5 25.3 29.0 14.2平均Average 31.6 25.2 29.0 14.2重慶 Chongqing 31.5 25.3 29.1 14.1武漢 Wuhan 31.5 25.3 29.1 14.1安慶 Anqing 31.5 25.3 29.0 14.2南京 Nanjing 31.5 25.3 29.1 14.1平均 Average 31.5 25.3 29.1 14.1

圖1 基于線粒體控制區單倍型的統計簡約網狀圖Fig.1 The statistical parsimony network inferred from mtDNA control region haplotypes
2.3 群體遺傳多態性參數統計
利用 DnaSP 4.0 軟件計算各群體的單倍型多樣性(Hd)、平均核苷酸差異數(K)、核苷酸多態性(Pi)。由表3可知, 長吻4個野生群體的單倍型多樣性Hd值均較高, 而從衡量群體遺傳水平的K值和Pi值來看, 重慶野生群體明顯高于其他群體。長吻3個養殖群體的平均單倍型多樣性和平均核苷酸多樣性相對較低 (Hd=0.8867±0.0013,Pi=0.0056±0.0013)。
2.4 群體遺傳分化
群體間的序列差異反映的是生物間親緣關系的遠近, 使用MAGA 3.0 軟件中Tamura-Nei 模型計算群體內及群體間的遺傳距離(表4)。結果表明, 四川眉山和湖北石首人工養殖群體間的親緣關系較遠,而安徽淮南養殖群體與湖北石首和四川眉山人工養殖群體間的親緣關系較近, 長吻養殖群體與野生群體存在一定的遺傳距離。分子變異分析(AMOVA)的結果表明, 長吻群體內存在較高的遺傳變異(96.42%), 而群體間的遺傳變異較小(3.21%) (表 5),分子遺傳變異主要來自地理區內群體內個體間。7個群體兩兩比較的遺傳分化指數(Fst)顯示, 安慶群體和石首群體之間的遺傳分化最大(Fst=0.1125), 而武漢群體和安慶群體之間的遺傳分化最小(Fst=0.0014), 其中長吻養殖群體間的遺傳分化高于野生群體間的遺傳分化。 此外, 群體間的遺傳距離分析與遺傳分化指數的分析結果是一致(表 4)。Arlequin 3.1 軟件統計養殖群體與野生群體間的遺傳分化指數和基因流分別為Fst=0.0358、Nm=13.47。
表3 長吻7個群體遺傳多樣性統計參數Tab.3 Demographic parameters estimated from seven Leiocassis longirostris populations

表3 長吻7個群體遺傳多樣性統計參數Tab.3 Demographic parameters estimated from seven Leiocassis longirostris populations
群體 Populations 單倍型多樣性 Hd 平均核苷酸差異數 K 核苷酸多樣性 Pi眉山 Meishan 0.8972±0.0361 4.8854 0.0064±0.0013石首 Shishou 0.8580±0.0570 4.3500 0.0049±0.0021淮南 Huainan 0.9048±0.0260 4.9444 0.0056±0.0011平均 Average 0.8867±0.0013 4.7266 0.0056±0.0013重慶 Chongqing 0.9470±0.0320 6.5580 0.0083±0.0008武漢 Wuhan 0.9810±0.0310 6.1520 0.0078±0.0007安慶 Anqing 0.9170±0.0420 3.9580 0.0050±0.0006南京 Nanjing 0.9430±0.0540 4.2860 0.0054±0.0007平均 Average 0.9736±0.0070 5.3030 0.0087±0.0015
表4 長吻群體間的遺傳分化指數(左下角)和遺傳距離(右上角)Tab.4 Fixation Index (F) (below) and genetic distance (above) in three populations of Leiocassis longirostris
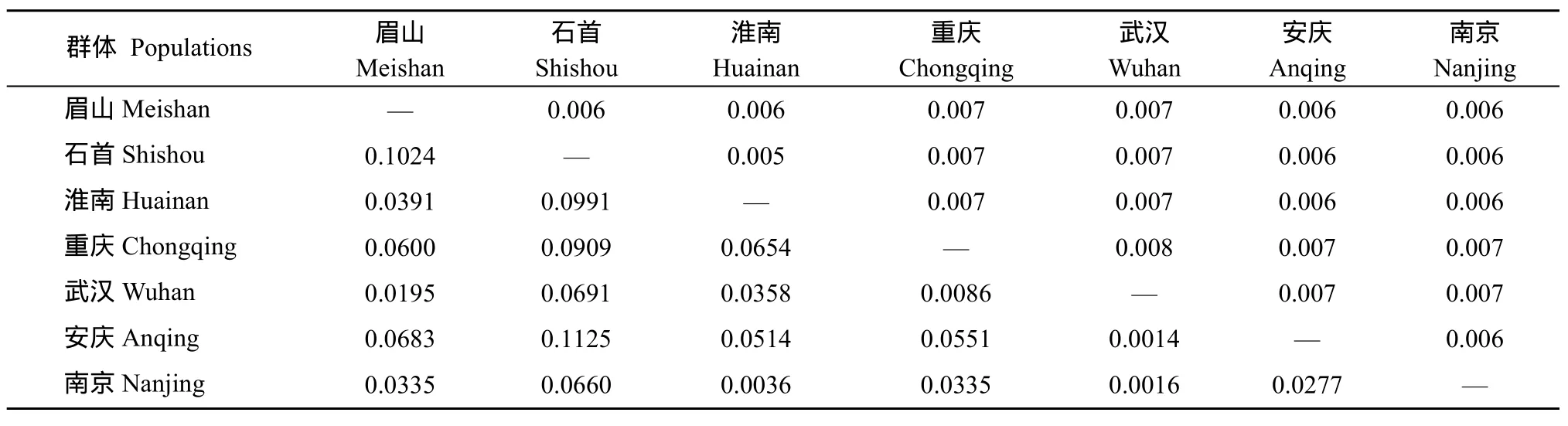
表4 長吻群體間的遺傳分化指數(左下角)和遺傳距離(右上角)Tab.4 Fixation Index (F) (below) and genetic distance (above) in three populations of Leiocassis longirostris
群體 Populations 眉山Meishan石首Shishou淮南Huainan重慶Chongqing武漢Wuhan安慶Anqing南京Nanjing眉山Meishan — 0.006 0.006 0.007 0.007 0.006 0.006石首Shishou 0.1024 — 0.005 0.007 0.007 0.006 0.006淮南Huainan 0.0391 0.0991 — 0.007 0.007 0.006 0.006重慶Chongqing 0.0600 0.0909 0.0654 — 0.008 0.007 0.007武漢Wuhan 0.0195 0.0691 0.0358 0.0086 — 0.007 0.007安慶Anqing 0.0683 0.1125 0.0514 0.0551 0.0014 — 0.006南京Nanjing 0.0335 0.0660 0.0036 0.0335 0.0016 0.0277 —

表5 長吻?群體分子變異分析結果Tab.5 Analysis of molecular variance (AMOVA) for the Leiocassis longirostris populations
2.5 群體間的親緣關系及聚類分析

圖2 長吻各單倍型基于線粒體控制區序列構建的NJ樹Fig.2 NJ tree based on the control region of 7 populations of Leiocassis longirostris
3 討論
3.1 長吻群體的遺傳多樣性
遺傳多樣性的研究是生物多樣性研究的重要內容, 只有通過遺傳多樣性的研究才能從本質上揭示物種多樣性的起源、變異和進化。核苷酸多態性(Pi)作為一個衡量群體間遺傳多態性的重要指標, 表示各種 mtDNA 單倍型在群體中所占的比例。由實驗結果可知, 在長吻養殖群體中66個個體線粒體控制區790 bp的序列中, 總核苷酸突變位點為27個,共檢測出18個單倍型。長吻野生群體中69個個體共檢測到35個變異位點, 36個單倍型。與長吻野生群體相比, 養殖群體的單倍型多樣性和核苷酸多樣性水平均較低(Hd=0.8867±0.0013,Pi=0.0057±0.0013)。另外, 從 3個養殖群體內部的核苷酸位點突變情況來看, 安徽淮南群體內有22個核苷酸位點的變異, 湖北石首群體共產生12個變異位點, 四川眉山群體有17個變異位點, 分別只占總群體變異的81.48%、44.44%和62.96%。這表明遺傳變異主要存在于群體內, 群體間的基因變異較小。安徽淮南群體的基因多樣性水平最高, 石首和眉山河群體較低。Wang,et al.[27]對取自長江上游和下游的野生長吻進行 PCR–RFLP分析, 發現野生長吻遺傳多樣性低, 種群數量下降, 這與已有野生種群的研究相一致[28,29]。而Yang,et al.[30]利用線粒體控制序列和核SSR標記研究長江上游、中游和下游野生長吻遺傳變異和種群結構, 表明長江野生長吻單倍型多樣性較高, 核苷酸多樣性水平較低(Hd=0.9770±0.0041,Pi=0.0081±0.0043)。上述研究結果表明, 長吻長期的人工養殖已使得該群體的遺傳多樣性水平明顯降低, 反映了長吻作為我國鯰形目分布最廣且產量較高的特產淡水名貴經濟魚類特殊的生命進化歷程和適應復雜環境的能力。造成這一現象的主要原因可能是長吻的基礎群體數量較小、養殖過程的近交及遺傳漂變等。而相對較高Hd值、低Pi值表明長吻這個群體可能是由一個較小的有效群體迅速增長, 盡管變異導致單倍型的多態性的積累, 但核苷酸序列的多樣化還未能積累[31]。高的Hd為通過分子選育等技術有效防止品種退化提供了前提條件, 并為保持遺傳多樣性水平, 維持雜交選育優勢提供了重要基礎。這也在一定程度上說明定期補充不同地區不同群體的個體作為親魚進行繁育有利于保持長吻人工繁殖群體的遺傳多樣性, 避免近親繁殖和瓶頸效應。
3.2 長吻群體的遺傳結構分析
[1] He X F, Su L D, Zhou G R,et al.Study on domestication and reproduction test ofLeiocassis longirostris[J].Freshwater Fisheries, 1985, (1): 14—17 [何學福, 蘇良棟, 周貴榮, 等.長吻的蓄養繁殖試驗研究.淡水漁業, 1985, (1): 14—17]
[2] Fang J Q.Collection and domestication ofLeiocassis longirostrisfrom the Yangtze River [J].Scientific Fish Farming,1999, (7): 19 [方建清.長江長吻的收集與蓄養.科學養魚, 1999, (7): 19]
[3] Liu J K, He B W.The Chinese Freshwater Fisheries Science(third edition) [M].Beijing: Science and Technology Press.1992, 304 [劉建康, 何碧梧.中國淡水魚類養殖學(第三版).北京: 科學技術出版社.1992, 304]
[4] Gong J H.Test on cage culture wildLeiocassis longirostrisin reservoir [J].Reservoir Fisheries, 2006, 26(1): 54 [龔建輝.水庫網箱養殖野生長吻試驗.水利漁業, 2006, 26(1):54]
[5] Wu Q J.Population ecology ofLeiocassis longirostris(Gunther) (Pisces, Bagridae) with reference to the problem of maximum sustained yield [J].Acta Hydrobiologica Sinica,1975, 5(3): 387—408 [吳清江.長吻種群生態學及其最大持續漁獲量的研究.水生生物學集刊, 1975, 5(3): 387—408]
[6] Xiao M S, Wan Q.The biology and culture prospect ofLeiocassis longirostrisin the Yangtze River [J].Journal of Anhui Technical Teachers College, 2001, 15(3): 49—51 [肖明松, 萬全.長江長吻的生物學習性及其發展前景.安徽技術師范學院學報, 2001, 15(3): 49—51]
[7] Zhang Y Y.Researches and development ofLeiocassis longirostrisGunther [J].Southwest China Journal of Agricultural Sciences, 1998, S1(11): 134—139 [張義云.長吻(Leiocassis longirostrisGunther) 的研究與開發.西南農業學報, 1998, S1(11): 134—139]
[8] Mo Y X, Wang X Q, Mo Y L.Morphological and histological observations of digestive system inLeiocassis longirostris[J].Journal of Hunan Agricultural University(Natu-ral Sciences), 2004, 30(3): 267—271 [莫艷秀, 王曉清, 莫永亮.長吻消化系統的形態學與組織學觀察.湖南農業大學學報(自然科學版), 2004, 30(3): 267—271]
[9] Luo M, Jiang L K, Liu Y,et al.Comparative study on isoenzymes inLeiocassis longirostris[J].Chinese Journal of Applied & Environmental Biology, 2000, 6(5): 447—451[羅曼, 蔣立科, 劉穎, 等.野生與養殖長吻血液及不同器官同工酶的比較.應用與環境生物學報, 2000, 6(5):447—451]
[10] Wan Q, Liu E S, Shen D L,et al.Analysis on karyotype ofLeiocassis longirostrisGunther [J].Journal of Anhui Agricultural University, 2002, 29(2): 182—184 [萬全, 劉恩生,申德林, 等.長吻染色體組型分析.安徽農業大學學報,2002, 29(2): 182—184]
[11] Xiao M S, Yang G.Isolation and characterization of 17 microsatellite loci for the Chinese longsnout catfishLeiocassis longirostris[J].Molecular Ecology Resources, 2009, 9(3):1039—1041
[12] Yu Y Y, Xiao M S, Chen L,et al.Isolation and characterization of microsatellite loci in the longsnout catfish (Leiocassis longirostris) [J].Aquaculture Research, 2009, 40(2):246—248
[13] Bermingham E, Avise J C.Molecular zoogeography of freshwater fishes in the southeastern United States [J].Genetics, 1986, 113(4): 939—965
[14] Grunwald C, Stabile J, Waldman J R,et al.Population genetics of shortnose sturgeonAcipenser brevirostrumbased on mitochondrial DNA control region sequences [J].Molecular Ecology, 2002, 11(10): 1885—1898
[15] Mabuchi K, Senou H, Suzuki T,et al.Discovery of an ancient lineage ofCyprinus carpiofrom Lake Biwa, central Japan, based on mtDNA sequence data, with reference to possible multiple origins of koi [J].Journal of Fish Biology,2005, 66(6): 1516—1528
[16] Kyle C J, Wilson C C.Mitochondrial DNA identification of game and harvested freshwater fish species [J].Forensic Science International, 2007, 166(1): 68—76
[17] Teletchea F.Molecular identification methods of fish species:reassessment and possible applications [J].Reviews in Fish Biology and Fisheries, 2009, 19: 265—293
[18] Zhang Y, Zhang E, He S P.Studies on the structure of the control region of the bagridae in China and its phylogentic significance [J].Acta Hydrobiologica Sinica, 2003, 27(5):463—467 [張燕, 張鶚, 何舜平.鲿中國科魚類線粒體DNA控制區結構及其系統發育分析.水生生物學報, 2003,27(5): 463—467]
[19] Thompson J D, Gibson T J, Plewniak F,et al.The Clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools [J].Nucleic Acids Research, 1997, 24: 4876—4882
[20] Kumar S, Tamura K, Nei M.Mega 3.0: Integrated software for molecular evolutionary genetics analysis and sequence莫永亮.長吻遺傳多樣性的RAPD分析.江西水產科技,2010, 122(2): 13—16]
[29] Wang H Y, Huang W Q.Preliminary analysis on the genetic diversity in four populations ofLeiocassis longirostrisby using microsatellite markers [J].Journal of Henan Agricultural Sciences, 2011, 40(2): 146—148 [王紅瑩, 黃文清.應用微衛星標記分析長江流域長吻4 個群體的遺傳多樣性.河南農業科學, 2011, 40(2): 146—148]
[30] Yang G, Xiao M S, Yu Y Y,et al.Genetic variation at mtDNA and microsatellite loci in Chinese longsnout catfish(Leiocassis longirostris) [J].Molecular Biology Reports,2012, 39(4): 4605—4617
[31] Avise J C.Phylogeography the History and Formation of Species [M].Cambridge, Massachusetts London, England:Harvard University Press.2000, 213—285
[32] Meng W, Guo Y, Hai S,et al.Genetic structure and diversity ofSchizothorax biddulphipopulations [J].Acta Hydrobiologica Sinica, 2012, 36(5): 851—857 [孟瑋, 郭焱, 海薩,等.塔里木裂腹魚群體遺傳結構及遺傳多樣性分析.水生生物學報, 2012, 36(5): 851—857]
[33] Masatoshi N.Molecular Population Genetics and Evolution[M].Beijing: Agriculture Press.1975, 121—133 [根井正alignment [J].Briefings in Bioinformatics, 2004, 5: 150—163
[21] Rozas J, Sánchez-del Barrio J C, Messeguer X,et al.DNA polymorphism analyses by the coalescent and other methods[J].Bioinformatics, 2003, 19: 2496—2497
[22] Excoffier L, Laval G, Schneider S.Arlequin ver 3.0.An integrated software package for population genetics data analysis[J].Evolutionary Bioinformatics Online, 2005, 1: 47—50
[23] Clement M, Posada D, Crandall K A.TCS: a computer program to estimate gene genealogies [J].Molecular Ecology,2009, 9: 1657—1660
[24] Templeton A K.Crandall K A, Sing C F.A cladistic analysis of phenotypic associations with haplotypes inferred from restriction endonuclease mapping and DNA sequence data.III.Cladogram estimation [J].Genetics, 1992, 132: 619—633
[25] Posada D, Crandall K A.Evaluation of methods for detecting recombination from DNA sequences: Computer simulations[J].Proceedings of the National Academyof Sciences of the United States of America, 2001, 98: 13757—13762
[26] Slatkin M, Hudsun R R.Pairwise comparisons of mitochondrial DNA sequences in stable and exponentially growing populations [J].Genetics, 1991, 129: 555—562
[27] Wang Z W, Zhou J F, Ye Y,et al.Genetic structure and lowgenetic diversity suggesting the necessity for conservation of the Chinese longsnout catfishLeiocassis longirostrisGünther(Pisces: Bagriidae) [J].Environmental Biology of Fishes,2006, 75: 455—463
[28] Mo Y X, Wang X Q, Mo Y L.RAPD analysis of genetic diversity ofLeiocassis longirostris[J].Journal of Jiangxi Fisheries Science, 2010, 122(2): 13—16 [莫艷秀, 王曉清,利.分子群體遺傳學與進化論.北京: 農業出版社.1975,121—133]
[34] Wright S.The gentical structure of population [J].Annals of Eugenics, 1951, 15: 323—334
[35] Wright S.Evolution in Mendelian populations [J].Genetics,1931, 16: 97—159
[36] Han X L, Xu J R, Li X R,et al.Analysis of genetic diversity ofElopichthys bambusaby AFLP Markers [J].Journal of Nan jing Normal University(Natural Science Edition), 2009,32(1): 110—114 [韓曉磊, 徐建榮, 李小蕊, 等.鳡魚群體遺傳多樣性的 AFLP 分析.南京師大學報(自然科學版),2009, 32(1): 110—114]
[37] Yang X, Yang J F, Tang M L,et al.Intraspecific genetic polymorphisms ofSiniperca ScherzeriSteindacher and molecular identification withSiniperca chuatis[J].Acta Hydrobiologica Sinica, 2007, 31(6): 891—895 [楊星, 楊軍峰, 湯明亮, 等.斑鱖種內遺傳多態性以及與翹嘴鱖的分子鑒別.水生生物學報, 2007, 31(6): 891—895]

