DNA序列堿基組合的頻率矩陣及其應用
李玉雙,劉倩,張昱
(1.燕山大學 理學院,河北 秦皇島066004;
2.石家莊郵電職業技術學院 計算機系,河北 石家莊050021)
隨著生物科學技術的迅猛發展,生物信息學越來越受到人們的重視,各種研究方法相繼產生[1].近年來,數學模型被引入到該領域,對生物信息學本身而言,這是一次從量變到質變的飛躍.眾所周知,數學模型在生物序列和結構的比較中起到了很好的研究效果,在理論方面給出了很好的解釋,如幾何表示模型[2]、字統計模型[3]和馬爾科夫模型[4]等 .隱馬爾科夫模型在生物信息學的一系列問題都得到成功應用,如多序列比對[5]、基因識別[6]和蛋白質二級結構預測[7]等.伴隨生物研究中數學模型和算法的不斷完善,產生了許多強有力的生物信息分析工具,如進化分析、聚類分析等,部分有效的分析工具極大地依賴于生物序列和結構的比較.序列和結構的比較是最重要和最常用的原始操作,是許多其他復雜操作的基礎.序列的相似性分析是生物序列和結構比較中的一個重要問題.從序列分析角度,判定兩條序列同源與否的一個主要依據是探尋它們之間的相似性.文獻[8]提出了轉移矩陣,將DNA序列看成是離散的馬爾科夫鏈,分別以堿基A,T,C和G在序列中出現的次數作為基準來構造轉移矩陣,進而刻畫11個物種的β-globin基因第一個外顯子編碼序列的差別.本文以序列的長度作為基準,基于堿基組合在DNA序列中出現的頻率,構造了DNA序列的頻率矩陣.
1 堿基組合的頻率矩陣
1.1 頻率矩陣的定義
給定長為n的生物序列l=l1l2l3…ln,li∈S,S={A,T,C,G}為堿基集合.記 AA在序列中出現的次數為nAA,則定義PAA=nAA/n.同理,可分別定義PAT,PAC,PAG,PTA,PTT,PTC,PTG,PCA,PCT,PCC,PCG,PGA,PGT,PGC,PGG.這里稱 AA,AT,AC,AG,TA,TT,TC,TG,CA,CT,CC,CG,GA,GT,GC,GG為堿基組合.定義該序列對應的頻率矩陣P為

由此可知,對應于文獻[8]刻畫的11個物種的β-globin基因第一個外顯子編碼序列,可以分別定義相應的頻率矩陣,其堿基如表1所示 .表1中:1~11個物種分別是人類(human),家山羊(goat),負鼠目(opossum),原雞(gallus),狐猴(lemur),小鼠(mouse),兔子(rabbit),老鼠(rat),大猩猩(gorilla),牛科動物(bovine),黑猩猩(chimpanzee).
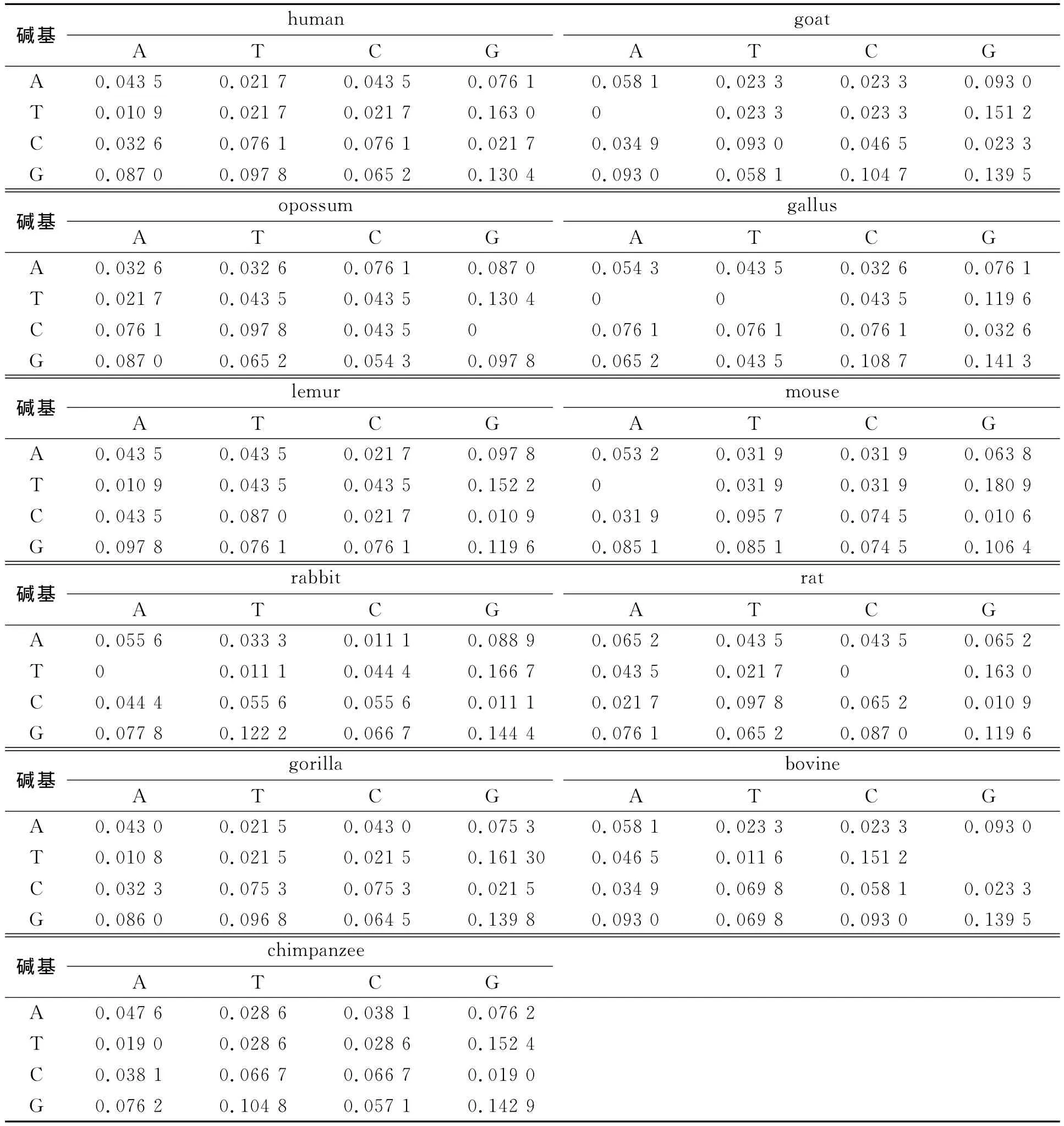
表1 11個物種的頻率矩陣Tab.1 Frequency matrix of eleven species
從表1可以看到:11個物種中TG出現的頻率都是最高,其次是GG,而TA和CG頻率較低.這說明在β-globin基因的編碼序列中TG和GG相對來說出現頻繁,而TA和CG相對出現次數較少,有些物種甚至沒有出現 .從單個物種來說,opossum和gallus又有些特殊的地方,例如TG中頻率較其他物種偏低,CA中頻率較高.這說明了在11個物種的β-globin基因的編碼序列中opossum和gallus有著特殊性.上述結果與代琦等[8]的結論基本一致.
1.2 頻率矩陣的性質

根據頻率矩陣的性質1),可以計算出11個物種堿基含量的向量,即

對于序列的最后一個堿基,雖然它的含量不能通過上述向量中的對應值精確體現(由于計算的是堿基組合),但由于其他3個堿基的含量恰好就是向量中的對應值,所以能夠很容易得到最后一個堿基的含量.如在human中,堿基A的含量是0.184 8,堿基T的含量是0.217 3,堿基C的含量是0.206 5,則堿基G的含量是0.391 4.圖1為11個物種的堿基含量分布柱狀圖,可以更直觀的展現堿基A,T,C,G在11個物種中的分布情況.

圖1 堿基在11個物種中的分布圖Fig.1 Distribution of nucleotide of eleven species
觀察11個堿基含量向量及圖1可以看出:11個物種序列中堿基G的含量都較高,堿基A的含量分布較為均勻;相比其他物種,gallus堿基G的含量明顯偏低,lemur堿基C的含量偏低,opossum堿基G的含量偏低;human和gorilla的堿基含量幾乎相等.眾所周知,研究DNA序列的特殊區域能為基因組的組織結構和生物作用提供更加豐富的信息.這里借助堿基含量向量及圖1可以很容易的得出特殊堿基組合的含量,如GC含量.GC含量為基因組提供了數量以及性質上的重要信息,GC含量高的DNA序列要比GC含量低的DNA序列更加穩定[9].
根據頻率矩陣的性質2),可以計算出11個物種的堿基轉移向量,即

通過比較堿基含量向量和堿基轉移向量不難發現,每個物種的兩個向量總有兩個分量是相等的.因為前者忽略了序列的最后一個堿基,后者忽略了序列的第一個堿基.如果一個序列首尾堿基相同,則這個序列對應的兩個向量一定相等.從這個意義上來說,堿基轉移向量也能夠反映出各個堿基在序列中的含量分布.此外,除首尾堿基相同的序列(注:這11個物種首尾堿基都不同),不用計算通過比較兩個向量就能確定每個物種中各個堿基的含量,如human的堿基轉移向量的最后一個分量即為堿基G的含量0.391 2,這與前面計算的結果一致(微小誤差是由于計算時舍位引起的).
2 序列相似性分析
由于生物序列有其進化上的生物學意義,因此比較兩條生物的相似性時,不能完全使用計算機科學中的模式匹配,常會借助“距離”來反映,如向量的歐氏距離、協方差距離、夾角距離等.文中引入矩陣的2-范數對11個物種進行相似性比較.
設P1和P2為兩個物種的頻率矩陣,令Q=|P1-P2|,則Q的2-范數計算公式為
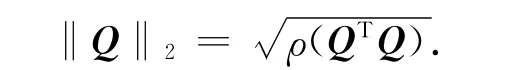
利用2-范數的計算公式來求兩個物種的相似性大小,即求得的范數越小,代表兩個物種所刻畫的DNA序列越相似,兩個物種越接近;反之,它們刻畫的DNA序列差別越大.利用2-范數的計算公式和常用的歐式距離公式計算得到的11個物種的相似性矩陣,如表2,3所示.
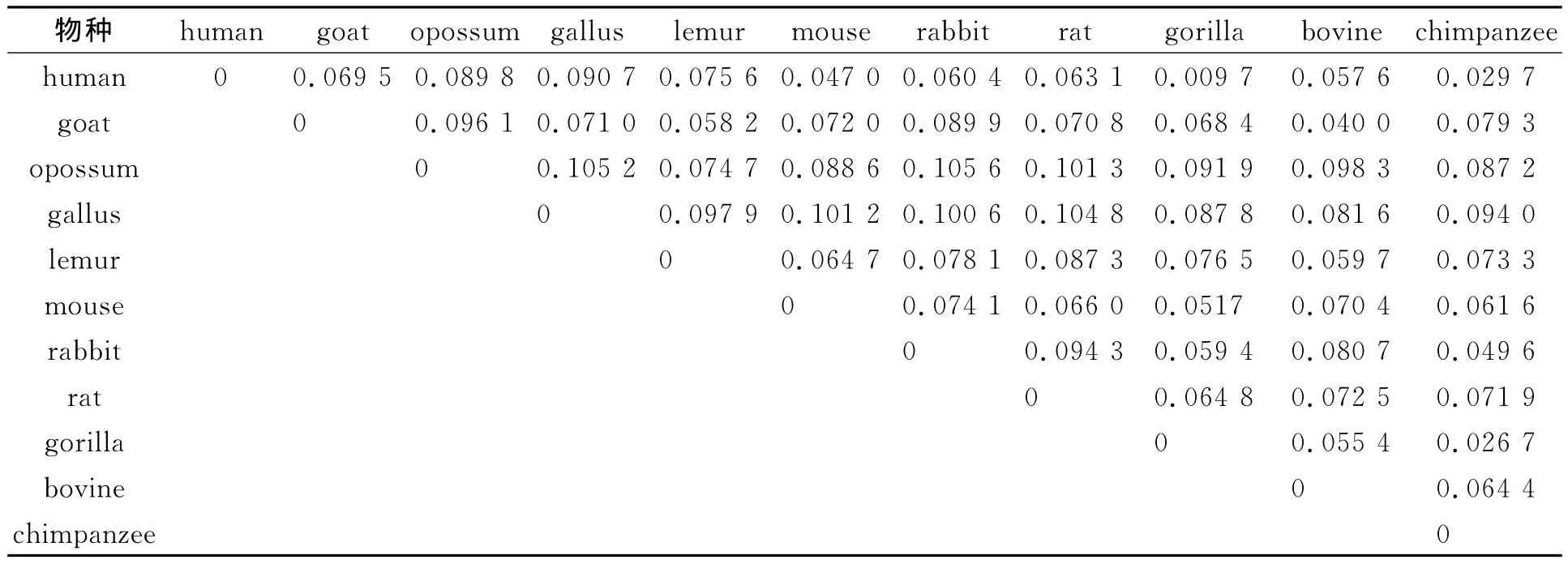
表2 由2-范數算得的11個物種的相似性矩陣Tab.2 Similarity matrix of eleven species based on the 2-norm

表3 由歐氏距離算得的11個物種的相似性矩陣Tab.3 Similarity matrix of eleven species based on the Euclidian distance
比較表2,3可知:2-范數法要比常用的歐氏距離法好,但從整體上看兩個方法求得的結果基本一致.即human和gorilla相似性非常高,human和chimpanzee,gorilla和chimpanzee相似性也很高,goat和bovine相似性較高;相比之下,opossum和其他物種相似性較低,這與opossum是與其他哺乳動物親緣較遠的哺乳動物相符合;Gallus和其他物種相似性也較低,這與Gallus是唯一的非哺乳動物相符合.這些結論都與相關的文獻結果一致[2,8].
3 結論
介紹一種利用DNA序列堿基組合的頻率矩陣來刻畫物種相似性的方法 .該矩陣的每一個分量都能夠反映出對應堿基組合在序列中的含量分布情況,其行和能反映每個堿基在序列中的含量分布情況,列和能反映堿基突變的情況,而所有元素值之和為定值.相較文獻[8]中的轉移矩陣,頻率矩陣能夠更好地從整體上反映出DNA序列中堿基以及堿基組合的含量分布,顯示出序列堿基突變的情況.
文中引入矩陣的2-范數對11個物種進行相似性比較,結果顯示該方法要優于上述常用的距離分析方法.頻率矩陣的應用在物種的相似性比較方面得到了很好的體現,借助矩陣2-范數和柱狀圖所得到的結果對物種的進化分析有一定的參考價值.
[1] 王勇獻,王正華.生物信息學導論:面向高性能計算的算法與應用[M].北京:清華大學出版社,2011:28-72.
[2] XIE Guo-sen,MO Zhong-xi.Three 3Dgraphical representations of DNA primary sequences based on the classifications of DNA bases and their applications[J].J Theor Biol,2011,269(1):123-130.
[3] VINGA S,GOUVEIA-OLIVEIRA R,ALMEIDA J S.Comparative evaluation of word composition distances for the recognition of SCOP relationships[J].Bioinformatics,2004,20(2):206-215.
[4] PHAM T D,ZUEGG J.A probabilistic measure for alignment-free sequence comparison[J].Bioinformatics,2004,20(18):3455-3461.
[5] 羅澤舉,宋麗紅.隱馬爾可夫模型的多序列比對的研究[J].計算機工程與應用,2010,46(7):171-174.
[6] 豐月姣,賀興時.二階隱馬爾科夫模型在基因識別中的應用[J].佳木斯大學學報,2009,27(6):940-942.
[7] 石峰,莫忠息,張楚瑜.隱馬爾可夫模型-改進的預測蛋白質二級結構方法[J].生物數學學報,2004,19(2):233-237.
[8] 代琦.生物序列、結構比較中若干數學模型研究及應用[D].大連:大連理工大學,2009:17-71.
[9] GAO F,ZHANG C T.GC-Profile:A web-based tool for visualizing and analyzing the variation of GC content in genomic sequences[J].Nucleic Acids Res,2006,34:686-691.

