貴人香冰酒大生產過程中酵母菌群結構及動態變化
何 娟,曹培鑫,黃英子,劉延琳*
(1.西北農林科技大學 葡萄酒學院,陜西 楊凌 712100;2.陜西省葡萄與葡萄酒工程技術研究中心,陜西 楊凌 712100)
冰酒(icewine)即冰葡萄酒,指環境溫度達到-8℃以下,葡萄在葡萄樹上自然冰凍,此時葡萄中的一部分水分蒸發,一部分結冰,葡萄汁的糖、酸得到濃縮,風味、香氣變濃,將這種葡萄采摘、壓榨,流出少量濃縮的葡萄汁經低溫保糖發酵而成的葡萄酒[1]。冰酒因其獨特、濃郁、甜香醇厚的風味,漫長的釀造過程而身價倍增,被譽為“液體黃金”。
冰酒的發酵過程是一個復雜的微生物動態變化及生化過程,包含了不同酵母屬種此消彼長的變化,研究發酵不同階段酵母菌的消長規律,有利于冰酒發酵過程中的微生物控制,也為研究酵母菌對葡萄酒風味的影響提供依據[2]。冰酒原料的高糖、高酸環境及其發酵過程要求的低溫等特殊環境對發酵過程酵母動態變化有重要影響[3]。
我國作為世界少有的冰酒釀造國之一,有關我國冰酒酵母資源的研究意義重大。本研究通過采用甘肅祁連酒莊冰酒主栽品種貴人香(Italian Riesling)為對象,監測冰酒大生產發酵過程中酵母菌菌群的多樣性和動態變化。利用WLN營養瓊脂、26S rDNA D1/D2區域的多態性和Interdelta序列指紋圖譜分析對發酵過程中不同階段分離的酵母進行分類鑒定,跟蹤商業酵母在冰酒發酵過程中的變化及其與野生酵母的競爭狀況,有利于明確商業酵母是否在嚴苛的冰酒發酵過程中取得優勢地位,為冰酒發酵過程中的微生物控制和冰酒風味改良與提升提供依據。
1 材料與方法
1.1 實驗材料
于2011年11月從甘肅祁連葡萄酒有限公司葡萄基地采集釀造冰白主栽品種貴人香(糖度35°Bx,總酸8.1g/L),于60kL的圓柱形不銹鋼發酵罐發酵釀造冰白葡萄酒,在大生產中分離酵母180株進行分類鑒定。
商業酵母LVCB:荷蘭DSM公司。
1.2 培養基
菌株的分離培養采用酵母浸出粉胨葡萄糖瓊脂培養基(yeast extract peptone dextrose medium,YEPD)[2];菌株的初步形態分類采用WLN(Wallerstein laboratories nutrient)營養培養基[4]。
1.3 方法
1.3.1 菌株分離
貴人香葡萄采收后,除梗、破碎、取汁,均勻加入60mg/L的SO2,葡萄汁澄清后,按2×106CFU/mL接入商業酵母LVCB,控溫12~16℃。在發酵過程中,分別于接種前、接種后、起酵時、糖消耗1/4、糖消耗2/3、終止發酵六個時期取樣1mL,用無菌水稀釋至合適梯度,取100μL涂布于YEPD平板上,加入100mg/L氯霉素抑制細菌生長,28℃倒置培養3d,每個時期共挑取單菌落30個,于20%甘油中-20℃保藏。
1.3.2 WLN培養基聚類分析
將分離到的180株菌,于YEPD培養基活化12h后,接種于WLN培養基上,28℃倒置培養5d,觀察并記錄菌落顏色和形態,參照PALLMANN C L等[4]中方法進行聚類分析,進行26S rDNA D1/D2區序列的分子鑒定補充,對得到的釀酒酵母進行Interdelta指紋圖譜分析。
1.3.3 DNA提取
采用石英砂破壁提取法[5]。
1.3.4 26S rDNA D1/D2區域PCR擴增
使用引物NL1(5′-GCATATCAATAAGCGGAGGAAAAG-3′)和NL4(5′-GGTCCGTGTTTCAAGACGG-3′)進行26S rRNA D1/D2區的擴增[6]。PCR反應體系(50μL體系):10×PCR buffer 5μL;25mmol/L MgCl23μL;10mmol/L dNTP 1μL;10μmol/L引物各1μL;Taq酶1.5U,l0ng~lμg/μL模版DNA 1μL;最后加雙蒸水定容至50μL。PCR循環為:95℃預變性5min,94℃變性1min,52℃退火1min,72℃延伸1min 20s;循環36次;72℃保溫8min。擴增產物經1%瓊脂糖凝膠檢測后,送生工生物工程(上海)股份有限公司進行純化和測序分析。
1.3.5 Interdelta序列擴增
采用正向引物delta12(5′-TCAACAATGGAATCCCAAC-3′)和反向引物delta21(5′-CATCTTAACACCGTATATGA-3′)進行擴增反應[7]。擴增體系為50μL:1×PCR buffer;2.5mmol/L MgCl2;0.2mmol/L dNTP;引物δ12和δ21各為0.2mmol/L;Taq酶0.08U;模版DNA(l0ng/L~lμg/μL);加滅菌雙蒸水定容至50μL。擴增反應條件:95℃4min(預變性);94℃30s(變性),46℃30s(退火),72℃1min 30s(延伸),循環35次;72℃10min(補充延伸)。
擴增產物經2%瓊脂糖凝膠檢測(電泳時間為2h),之后置于凝膠成像儀內拍照。
1.3.6 數據處理
將測序得到26S rDNA序列結果經BLAST軟件分析及序列相似性計算。對Interdelta指紋圖譜的帶型進行人工讀帶并統計,將特定位置上有DNA條帶的計為1,無DNA條帶的計為0,對釀酒酵母進行基因型。
2 結果與分析
2.1 發酵過程中酵母菌的種類及其數量變化
冰酒大生產發酵過程中共分離菌株180株,其中6株由于保存不當被污染,剩余174菌株通過WLN營養培養基的培養后,根據菌落顏色和形態的不同,可初步聚為12類(見圖1),參照PALLMANN C L等[4]、楊瑩等[8-9]描述的WLN分類鑒定結果和26S rDNA D1/D2鑒定表明,所獲酵母菌分屬于9屬10種(見表1):釀酒酵母(Saccharomyces cerevisiae)、戴爾有孢圓酵母(Torulaspora delbrueckii)、核果梅奇酵母(Metschnikowia aff.Fructicola)、檸檬形克勒克酵母(Kloeckera apiculata)、嗜高壓有孢漢生酵母(Hanseniaspora osmophila)、發酵畢赤酵母(Pichia fermentans)、粟酒裂殖酵母(Schizosaccharomyces pombe)、耐熱克魯維酵母(Lachancea thermotolerans)、淺白隱球酵母(Cryptococcus albidus)、葡萄汁有孢漢遜酵母(Hanseniaspora uvarum),分別占到了分離總菌株的86.21%、0.57%、2.87%、4.60%、1.15%、1.15%、1.15%、0.57%、0.57%、1.15%。其中,非釀酒酵母24株,釀酒酵母150株。菌落類型D(K.apiculata)有D1和D2兩個不同形態;菌落類型J(H.uvarum)有J1和J2兩個不同形態。各個時期酵母菌種群分布及其動態變化見表2。
由表2可知,接種前S.cerevisiae的比例高達63%,這可能包括酒廠設備上的常用商業酵母的及部分野生酵母,同時也包含分屬5屬5種的具有高糖耐受性的非釀酒酵母;接種后,S.cerevisiae的比例已達到81.5%;隨著發酵進行,發酵液中酒精含量不斷增加,S.cerevisiae比例逐漸升高達到90%以上,此時大量非釀酒酵母由于酒精耐受性不夠而大量死亡[10],而發酵末期還存在少量高耐酒精孢圓酵母(T.delbrueckii),發酵畢赤酵母(P.fermentans)。
2.2 發酵過程中釀酒酵母的菌株變化
從150株釀酒酵母中選取85株Interdelta序列擴增后,得到2種基因型分別為已接種的商業酵母LVCB(Ⅰ)和1種野生酵母(Ⅱ)(見圖2)。發酵過程中各個時期釀酒酵母各基因型的動態變化(見表3)。在冰酒大生產發酵過程中,接種前有66.7%的商業酵母LVCB存在可能是因為酒廠常年使用LVCB釀酒,致使部分商業酵母LVC殘留于車間地面、釀酒設備、空氣等。而野生酵母多樣性不豐富,這可能跟冰酒原料高糖、高酸環境對酵母生長產生的嚴苛條件有關。商業酵母基本主導了整個冰酒發酵,在發酵中后期更是成為絕對的發酵優勢菌,但是野生釀酒酵母在發酵初中期都表現出了競爭的潛力。

圖1 12株酵母在WLN培養基上的菌落形態Fig.1 Colony morphology of twelve yeast species on WLN medium

表1 大生產發酵中分離菌株的WLN形態描述及鑒定Table 1 Morphological descriptions and identification of yeast colonies on WLN medium isolated from industrial fermentation
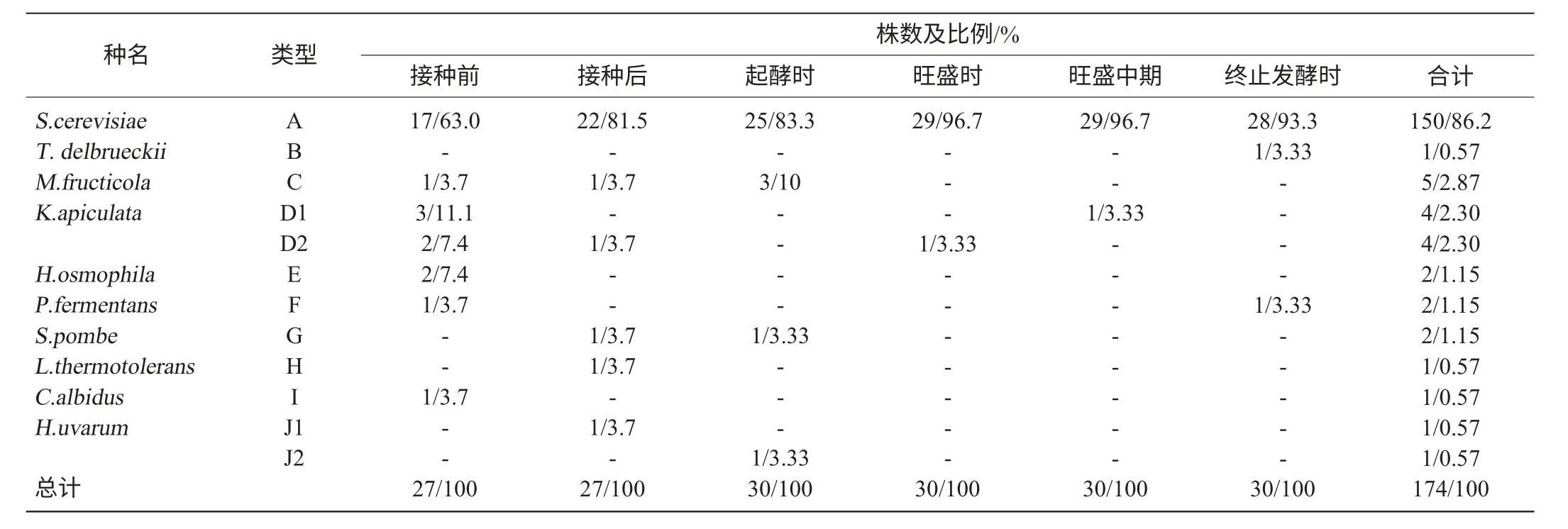
表2 發酵各階段酵母菌種類、數量和比例Table 2 Species,quantity and proportion of yeast during different fermentation stages
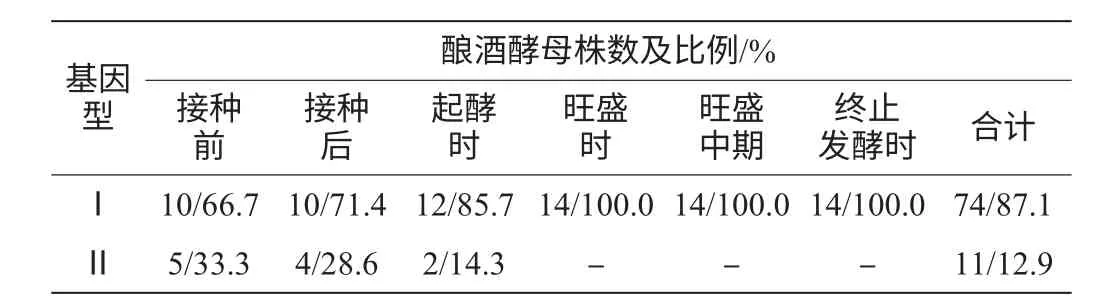
表3 釀酒酵母各基因型在發酵過程中的分布和比例Table 3 Distribution and proportion of S.cerevisiaein genotypes during different fermentation stages

圖2 釀酒酵母的Interdelta圖譜Fig.2 Interdelta sequence of S.cerevisiaein
3 結論
我國冰酒釀造所用酵母大多為國外引進商業酵母,而有關商業酵母能否在嚴苛的冰酒發酵過程中取得優勢地位且冰酒發酵過程中酵母菌種群及動態變化研究甚少。本研究通過對甘肅祁連貴人香冰葡萄酒大生產發酵過程中酵母菌群的動態變化研究,分離到的酵母菌分屬9屬10種,除了常見的葡萄酒相關酵母外[11-13],還發現了Metschnikowia aff.Fructicola、Hanseniaspora osmophila、Cryptococcus albidus等不太常見的非釀酒酵母。這些非釀酒酵母的存在是否與冰酒特殊環境及此地區地理環境有關還需進一步的研究。此外,在菌株水平上對S.cerevisiae進行區分,得到接種的商業酵母及一種野生酵母,并證實商業酵母基本主導了整個冰酒發酵,但是野生釀酒酵母在發酵初中期都表現出一定的競爭潛力。與其他地區大量的野生酵母資源相比[14-16],本實驗分離得到的釀酒酵母少,這可能跟冰酒發酵的高糖、高酸、低溫環境及商業酵母對本土酵母的排擠作用有關。
[1]DONALD J.Anatomy of winery[M].Toronto:Key Porter Books Ltd.1998.
[2]宋育陽,裴穎芳,王國平,等.黑比諾葡萄接種發酵過程酵母菌的變化監控[J].中國食品學報,2010,10(2):125-130.
[3]裴廣仁,李記明,趙玉平,等.冰葡萄釀酒酵母的篩選[J].食品與發酵工業,2010(6):98-102.
[4]PALLMANN C L,BROWN J A,OLINEKA T L,et al.Use of WL medium to profile native flora fermentations[J].Am J Enol Vitic,2001,52(3):198-203.
[5]周小玲,沈 微,饒志明,等.一種快速提取真菌染色體DNA 的方法[J].微生物學通報,2004,31(4):89-92.
[6]KURTZMAN C P,ROBNETT C J.Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit(26S)ribosomal DNA partial sequences[J].Anton Leeuw,1998,73(4):331-371.
[7]LEGRAS J L,KARST F.Optimization of interrelate analysis forSaccharomyces cerevisiaestraincharacterization[J].FEMS Microbiol Lett,2003,221(2):249-255.
[8]楊 瑩,徐艷文,薛軍俠,等.WL 營養瓊脂對葡萄酒相關酵母的鑒定效果驗證[J].微生物學雜志,2007,27(5):75-78.
[9]WANG C,LIU Y.Dynamic study of yeast species andSaccharomyces cerevisiaestrains during the spontaneous fermentations of Muscat blanc in Jingyang,China[J].Food Microbiol,2013,33(2):172-177.
[10]BEZERRA B,BAFFI M,GOMES E,et al.Yeast diversity isolated from grape musts during spontaneous fermentation from a Brazilian Winery[J].Curr Microbiol,2013,67(3):356-361.
[11]SUN H,MA H,HAO M,et al.Identification of yeast population dynamics of spontaneous fermentation in Beijing wine region,China[J].Ann Microbiol,2009,59(1):69-76.
[12]LI E,LIU C,LIU Y.Evaluation of yeast diversity during wine fermentations with direct inoculation and pied de cuve method at an industrial scale[J].J Microbiol Biotech,2012,22(7):960-966.
[13]LI E,LIU A,XUE B,et al.Yeast species associated with spontaneous wine fermentation of Cabernet Sauvignon from Ningxia,China[J].World J Microbiol Biotechnol,2011,27(10):2475-2482.
[14]CAPECE A,ROMANIELLO R,SIESTO G,et al.Diversity ofSaccharomyces cerevisiaeyeasts associated to spontaneously fermenting grapes from an Italian heroic vine-growing area[J].Food Microbiol,2012,31(2):159-166.
[15]SCHULLER D,CARDOSO F,SOUSA S,et al.Genetic diversity and population structure ofSaccharomyces cerevisiaestrains isolated from different grape varieties and winemaking regions[J].Plos One,2012,7(2):e32507.
[16]MUNOZ-BERNAL E,RODRIGUEZ M,BENITEZ P,et al.Molecular analysis of red wine yeast diversity in the Ribera del Duero DO(Spain)area[J].Arch Microbiol,2013,195(5):297-302.

