一株分離微擬球藻LAMB0002的鑒定*
梁 鑫,朱葆華,楊官品,潘克厚**
(中國海洋大學1.海水養殖教育部重點實驗室,2.海洋生命學院,山東 青島 266003)
微擬球藻屬包括Nannochloropsisoculata(Doop)D.J.Hibberd(Hibberd 1981),NannochloropsissalinaD.J.Hibberd (Hibberd 1981),Nannochloropsis gaditanaLubián (Lubián 1982),Nannochloropsis granulataKarlson & Potter[1],Nannochloropsislimnetica[2]和Nannochloropsisoceanica[3]6個 種,都是單細胞藻,屬于真眼點藻綱[4]。這些藻含有多種色素,如葉綠素a、玉米黃素、角黃素等[5]和豐富的長鏈多不飽和脂肪酸,尤其是二十碳五烯酸(C20∶5,eicosapentenoic acid,EPA),一種人體需要的重要多不飽和脂肪酸[6]。另外,微擬球藻還是水產養殖廣泛使用的餌料[7]和生物柴油候選生產藻[8]。微擬球藻是小于5μm的球形細胞[9],形態非常簡單,難以用形態學方法進行描述和準確鑒定。因此,微擬球藻的種間鑒定主要依據指示基因序列[1-3,10],但至今尚未有廣泛認同的基因序列作為種鑒定的分子條碼,選擇適用基因或者基因組合進行鑒定目前進行了較多的探索[11-12]。本研究基于形態觀察、色素和脂肪酸組成分析、18SrRNA基因和rbcL基因分子系統學分析對一株分離的微擬球藻進行了鑒定,希望建立一種適用單細胞微擬球藻的分類體系,使微擬球藻的分類鑒定更快速、準確。
1 材料及方法
實驗材料是一株分離的微擬球藻藻株(Nannochloropsissp.)LAMB0002。
N.sp.LAMB0002經擴大培養后,利用平板涂布法2次分離單細胞,在錐形瓶中擴大培養。全過程在無菌條件下操作。培養條件為:溫度(20±1)℃;光照60~80μmol·m-2·s-1,光周期為12h∶12h。光源為白熾燈,每天定時搖瓶3次。
1.1 形態學觀察
在光學顯微鏡 (Nikon 50i)下觀察N.sp.LAMB0002。將培養至指數中期的N.sp.LAMB0002離心收集,加入用磷酸緩沖液配制的5%戊二醛,4℃固定過夜。然后參考鐘秀容等的方法[13],制作切片。用日立透射電鏡(Hitachi H-7000Elctron Microscope)觀察。
1.2 生理生化分析
色素分析采用Zapata等的方法[14]。將15mL培養至指數中期的N.sp.LAMB0002在弱真空(<0.03 MPa)微光的條件下過濾到47mm Whatman GF/F玻璃纖維濾膜上,再用甲醇提取色素。使用的色譜設備為Waters Alliance HPLC系統,并配有Waters2996二極管陣列檢測器(1.2nm光學分辨率)和 Waters2475多波長熒光檢測器。使用的液相色譜固定相為C8色譜柱,流動相:A=甲醇∶乙睛∶吡啶水溶液(50∶25∶25 v/v/v),B=甲醇∶乙睛∶丙酮(20∶60∶20v/v/v)。
按照Lepage和Roy的方法提取脂肪酸[15]。取進入指數生長末期N.sp.LAMB0002,離心收集,冷凍干燥。將十九烷酸(Nonadecanoic acid,SIGMA)作為內標進行相對定量分析。脂肪酸組成分析采用美國Agilent 6890series GC system氣相色譜儀、氫火焰離子化檢測器和DB-225毛細管柱(30m×320μm×0.15 μm,50%-cyanopropylphenyl-dimethylpolysiloxane)。標準品為Accustandard FAMQ-005。根據內標的量計算各種脂肪酸所占藻粉干重的比值。再用SPSS軟件做各藻株之間脂肪酸的差異性分析。
1.3 分子系統學分析
離心收集對數生長期的藻細胞,用去離子水沖洗3次,去掉細胞表面的鹽類等物質。用CTAB法提取微擬球藻DNA[16],-20℃貯存備用。通過設計(見表1)的18SrRNA基因和rbcL基因引物對這2個目的片段進行PCR擴增。回收擴增產物,送至上海博尚公司測序。將測序得到的序列在GenBank進行Blast N比對,并下載相關的序列。用 MEGA4.1采用鄰接法(Neighbor-Joining Method,NJ)和最大簡約法(Maximum porosity,MP)經Consensus程序計算多數一致樹。構建系統樹,用自舉檢驗(Bootstrap test)估計系統樹分支節點的置信度,重復次數1 000。
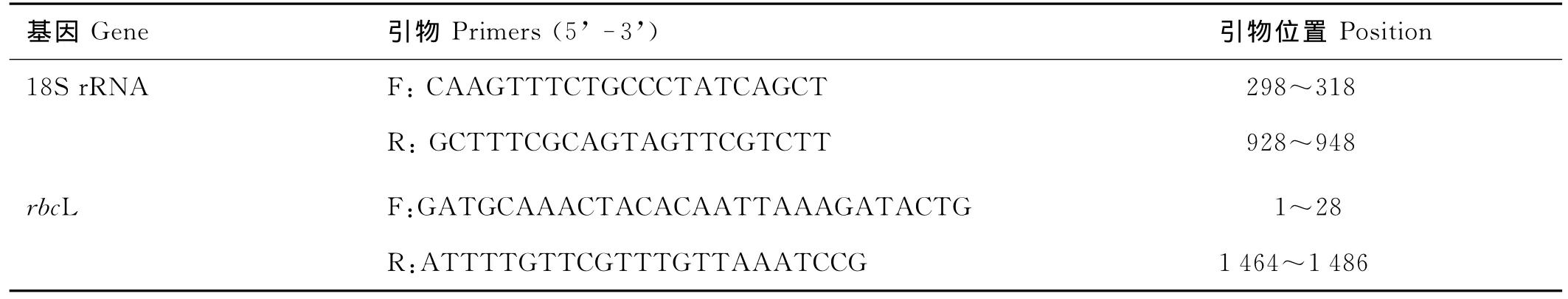
表1 本研究所用引物Table 1 Primers used in this study
2 結果
2.1 形態學觀察
在顯微鏡下觀察,發現N.sp.LAMB0002細胞(見圖1)單個呈球形、梨形或卵圓形,2~5μm,細胞壁光滑,胞內有一個杯狀或側生塊狀的黃綠色素體。細胞內常有不動的、顆粒狀的內含物。眼點紅色,沒有蛋白核。這些特征與微擬球藻特征相符[7],將其與本實驗室的4株微擬球藻進行比較。結果見圖1。
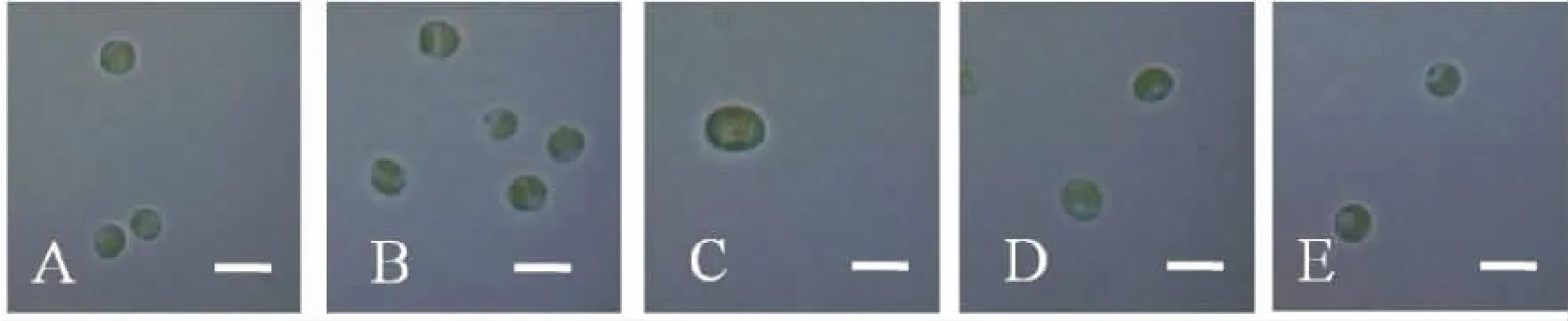
圖1 不同微擬球藻藻株的形態Fig.1 Morphology of different Nannochloropsis strains
幾株微擬球藻都是不游動的單細胞藻,外形沒有明顯區別,內含物顆粒狀,且都有一個黃綠色色素體。N.oceanic細胞呈球形或卵圓形,色素體杯狀或塊狀,常可見眼點(見圖1B);N.salina細胞呈長柱形,相比其他藻株較大,常可見眼點(見圖1C);N.oculata細胞呈球形或卵圓形,不常觀察到眼點(見圖1D、E);N.oculataCS-189(見圖1D)和N.oculataCS-192(見圖1E)在光學顯微鏡下差別不明顯。光鏡下,N.sp.LAMB0002與其他幾株微擬球藻十分相似,難以將其鑒定到種。
用透射電鏡觀察N.sp.LAMB0002,N.salinaCS-190、N.oculataCS-189和N.oculataCS-192。結果如圖2所示。
幾株微擬球藻都呈現細胞核、葉綠體、線粒體和液泡等結構,葉綠體片層結構十分清晰。N.sp.LAMB0002細胞卵圓形,胞內有2個較大的線粒體和1個明顯的液泡(見圖2A)。細胞核外膜和葉綠體外膜形成一個連續體(Nucleus-plastid continuum,NPC)[17],核內膜與葉綠體內膜直接接觸,將核與葉綠體完全分隔開來形成封閉的分隔[18]。NPC結構出現在整個細胞周期中,跟細胞有絲分裂有關。然而,在NPC中細胞核與葉綠體分裂是如何調適的卻是完全未知的[19]。N.oculataCS-189細胞中間有一個較大的脂肪滴(見圖2B)。N.oculataCS-192細胞壁上有一個乳突結構(見圖2C),這個結構曾出現在N.oculata、N.gaditana和N.salina中,被描述為栓狀結構[20],但該結構功能未知[3]。N.salinaCS-190,細胞形狀為長卵形(見圖2D),與其他幾株微擬球藻相比體積較大,這與光鏡觀察結果相符。幾株微擬球藻的超微結構沒有明顯區別,電鏡觀察難以將微擬球藻各個種相互區別。

圖2 各株微擬球藻超微結構Fig.2 Ultrastructure of different Nannochloropsis strains
2.2 生理生化分析
N.sp.LAMB0002的色素洗脫峰如圖3所示。N.sp.LAMB0002的主要色素為紫黃素、無隔藻黃素和葉綠素a,百合黃素、玉米黃素、角黃素和一些未知胡蘿卜素含量較少,除此之外還有痕量的β-胡蘿卜素。
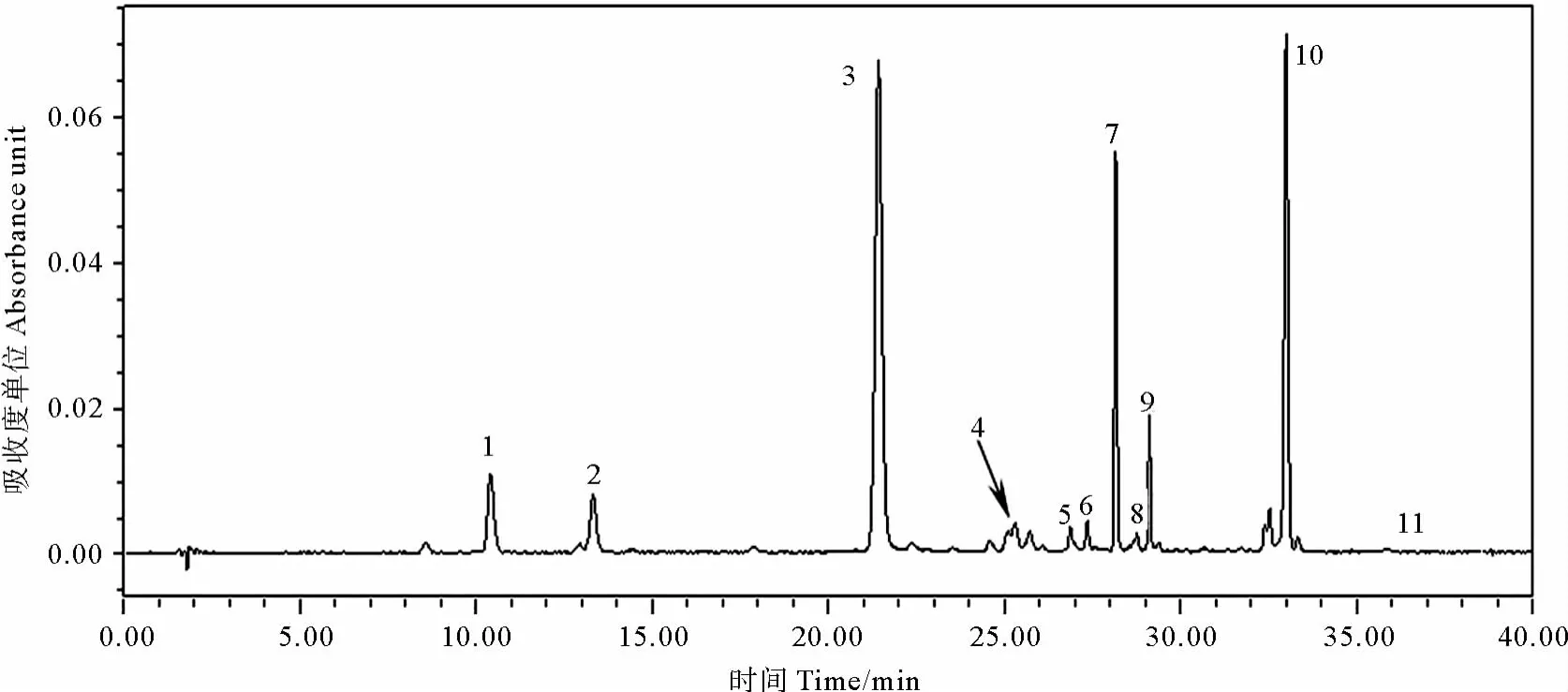
圖3 N.sp.LAMB0002高效液相色譜圖Fig.3 HPLC profile for N.sp.LAMB0002
微擬球藻屬只含葉綠素a,不含葉綠素b和葉綠素c,特征色素為紫黃素和無隔藻黃素[5]。因此,確定N.sp.LAMB0002為微擬球藻。
氣象色譜分析N.sp.LAMB0002、N.oceanicLAMB0001、N.salinaCS-190、N.oculataCS-189和N.oculataCS-192的幾種主要脂肪酸所占藻粉干重的比值見表2。
N.sp.LAMB0002的主要脂肪酸為C14∶0、C16∶0、C16∶1、C18∶0、C18∶1N9、C18∶2T、C20∶4和C20∶5(EPA),其中含量較高為C16∶0、C16∶1和C20∶5,分別能達到藻粉干重的1.44%、1.61%和1.09%。5株微擬球藻所含的主要脂肪酸種類相同,沒有特殊的脂肪酸將其區分。5株中C14∶0、C16∶1、C20∶4和C20∶5的含量并沒有顯著差異,其中N.oceanicLAMB0001含量最高,N.sp.LAMB0002次之。其余幾種主要脂肪酸含量在不同藻株中有顯著差異,但差異沒有明顯規律,不足以將不同種微擬球藻分開。因此,難以用脂肪酸的組成和含量區別微擬球藻各個種。
2.3 分子系統學分析
N.sp.LAMB0002的18SrRNA 基因與N.oceanicaLAMB0001(HQ201714.1)相 似 度 達 到98.7%。N.sp.LAMB0002的rbcL基因部分序列與N.oceanicaLAMB0001(HQ201714.1)相似度達到99.8%。將這2個序列與其他相關序列比對建立系統進化樹(見圖4和5)。
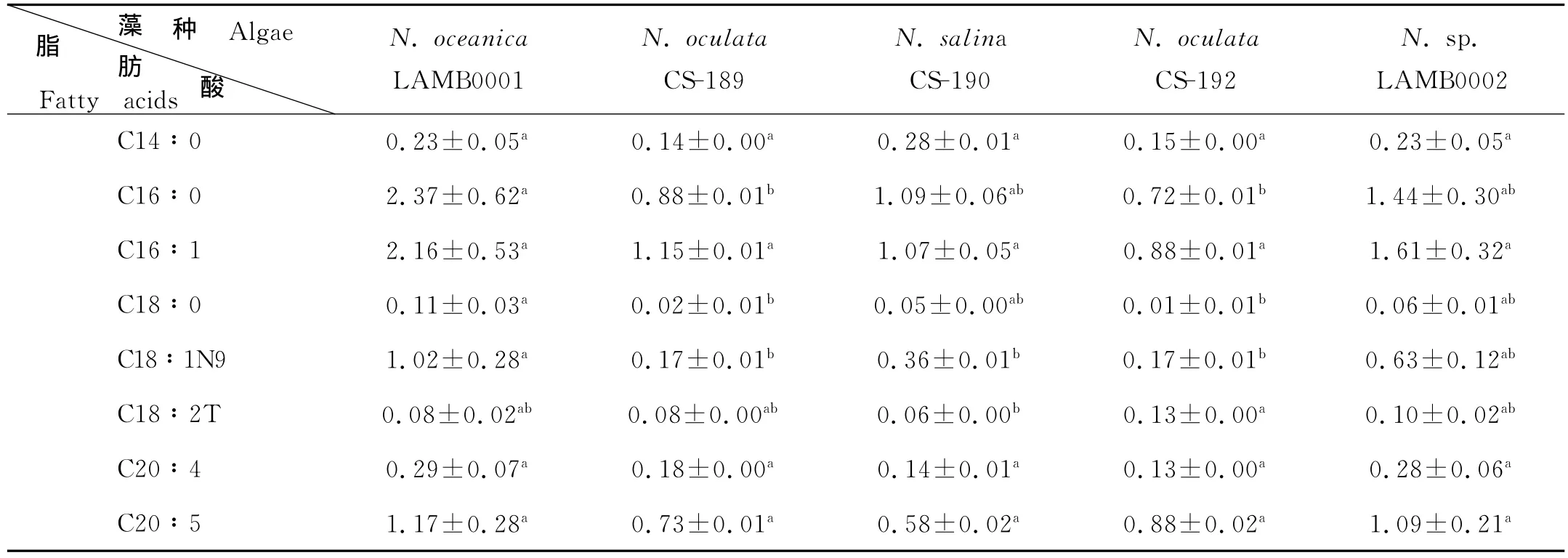
表2 5株微擬球藻主要脂肪酸占干重百分比Table 2 Percentile contents of main fatty acids in dry biomass of 5 Nannochloropsis strains
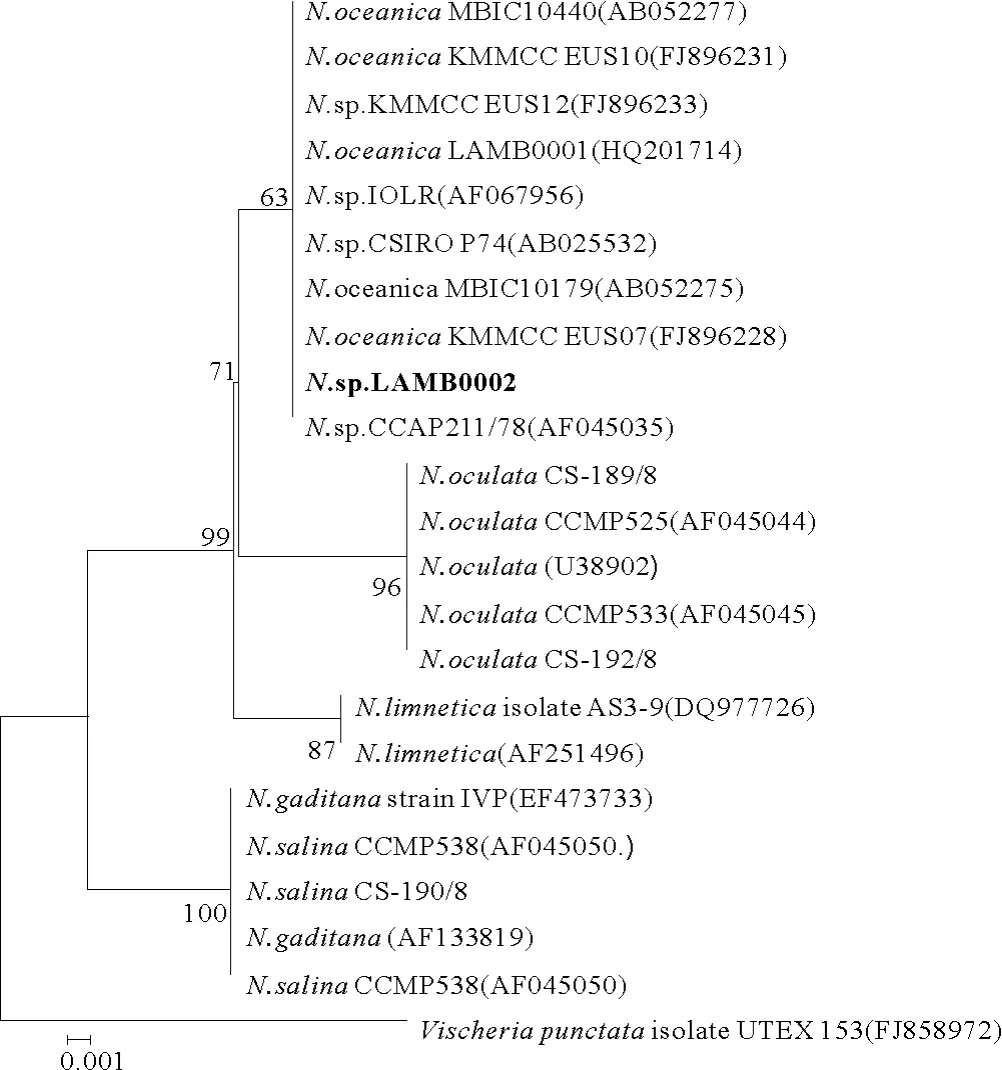
圖4 基于18SrRNA基因構建NJ樹(Tamura-Nei distances)Fig.4 Neighbor-Joining tree(Tamura-Nei distances)based on 18SrRNA gene sequences
以Vischeriapunctata為外類群基于18SrRNA基因分別構建了NJ樹和MP樹(未顯示)。2種進化樹都表明N.sp.LAMB0002與N.oceanica親緣關系較為接近。以Goniochlorissculpta為外類群基于rbcL基因分別構建了NJ樹和MP樹(未顯示)。2種進化樹都將N.sp.LAMB0002分到N.oceanica分枝中。結果表明,N.sp.LAMB0002是N.oceanica。從以上進化樹還可以看出rbcL基因與18SrRNA基因相比,能將微擬球藻各種完全分開,更能反映種間的差距。通過形態觀察、生理生化分析和分子系統學分析將N.sp.LAMB0002鑒定為N.oceanicLAMB0002。
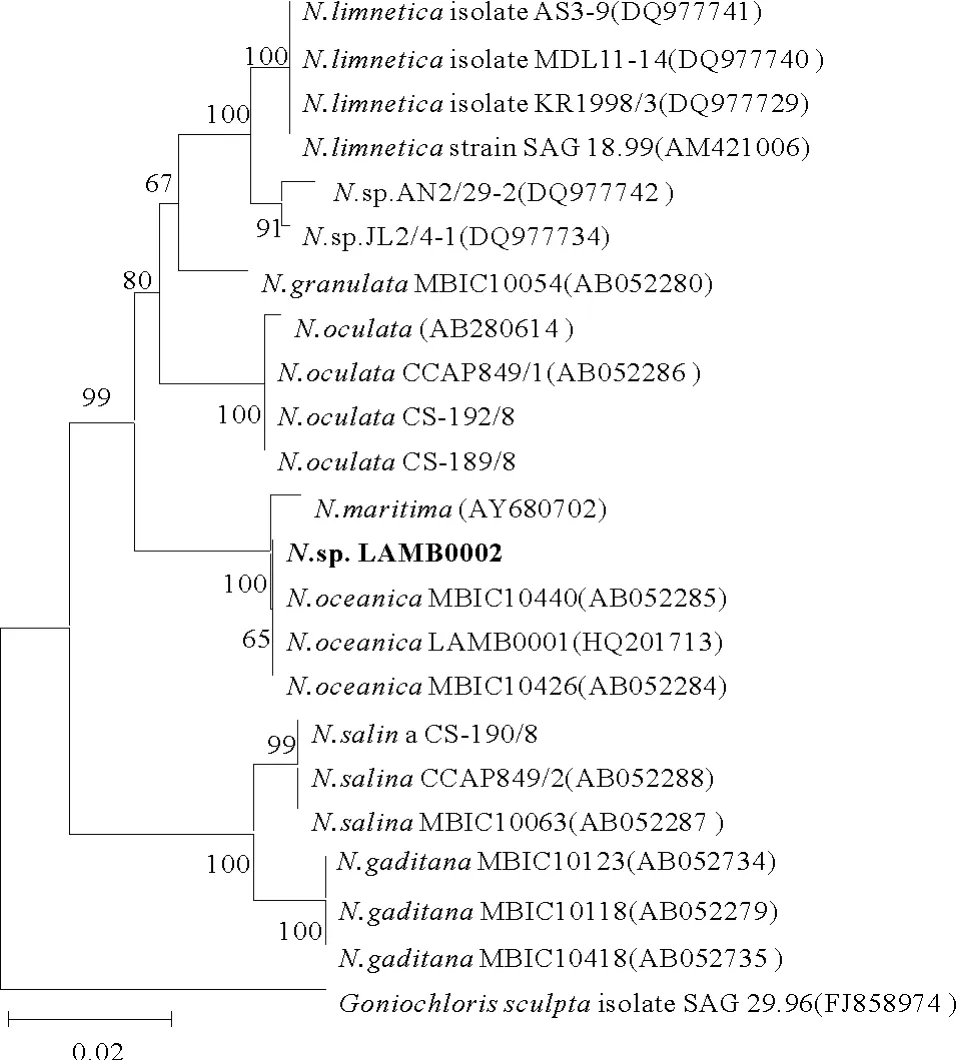
圖5 基于rbcL基因構建NJ樹(Tamura-Nei distances)Fig.5 Neighbor-Joining tree(Tamura-Nei distances)based on rbcL gene sequences
3 結論
形態學鑒定是鑒定微藻最直接的方法,對于很多種間形態相差較大的藻,形態學描述是最直接明確的手段。但是對于像微擬球藻這樣體積微小、結構簡單、種間差異微小的單細胞藻,形態學分類鑒定工作難以進行。不同生境條件下的同種微藻形態也有區別,這更加大了形態學鑒定的難度。這項工作對鑒定者的知識背景和經驗要求嚴格,要廣泛開展微藻鑒定的工作僅僅依靠形態學是遠遠不夠的。形態學鑒定受人為因素影響大,不同研究者的鑒定結果可能有所偏差,難以標準化。因此,鑒定工作需要更快、更準確的標準化的方法。
不同藻所含有生化成分不同,有些特殊的生化成分可以成為藻類分類鑒定的輔助工具。如微擬球藻含有較高的EPA[6],無隔藻黃素,角黃素,β-胡蘿卜素和僅有的葉綠素a(不含葉綠素b和葉綠素c),這些能有效地將微擬球藻與其他微藻區別開來[5]。雨生紅球藻含有含量較高的蝦青素,被認為是天然蝦青素的重要來源[21]。1948年 Millner等[22]提出以脂肪酸含量差異作為標準的化學分類方法;Graeve等[23]認為脂肪酸不僅可以作為分類的標準,還可以作為分析藻類進化關系的工具。至今,這種分類鑒定方法存在較大爭議[24-26]。如同其他的生化成分,脂肪酸的組成和含量也會隨著不同的培養條件有所變化[26],但是這能否將不同的種和株系分開,這些生化成分是否足夠保守,能否闡述不同藻株之間的進化關系,都是需要研究的問題。若利用不同生化成分快速鑒定微藻,必須有一個資料齊備的數據庫。這需要各地研究者的長期積累和共同努力。所以,目前用生化成分對微擬球藻鑒定只是一個輔助手段。
DNA條形碼技術(DNA barcoding)是現在浮游生物分類鑒定中應用最為廣泛的分子手段[27-29]。該技術旨在用一個或幾個基因實現快速鑒定。這項技術可以適用于不同生活階段的物種,生物體的組織部分與培養環境無關,也相對客觀。這種技術的應用不需要專業的訓練(除了對于PCR相關知識的了解)。目前DNA條形碼的數據庫主要有Barcode of Life Database(BOLD)、Canadian Centre for DNA Barcoding(CCDB),International Barcode of Life Project(iBOL)和Consortium for the Barcode of Life(CBOL)。除此之外,CBOL還有一個專注于植物陸地植物條形碼的組織 Royal Botanic Gardens,Kew(http://www.kew.org/barcoding/)。
在動物中,Cytochrome oxidaseⅠ(COⅠ)基因對于種間的鑒定非常有效,在大多數動物中有很好的分辨力[30]。植物中的線粒體基因組進化較慢,核酸替換率低。因此,排除了COⅠ作為植物條形碼的可能性[31]。CBOL推薦的2個質體基因組合rbcL+matK成為現在普遍承認的植物條形碼[32]。藻類中還沒有通用的條形碼,無論是在大型藻還是微藻中作為條形碼的候選基因很少。目前的研究都是針對特定種,選擇可能成為其條形碼的基因或基因組合,且這些研究主要集中在紅藻[33-34]、褐藻[35]、硅藻[36-37]和甲藻[28],在其他藻類中的研究很少。
在微擬球藻中,18SrRNA基因是最早提出用于分類鑒定的序列,在N.salina和N.gaditana中18S rRNA基因只有一個堿基的差異[10],微小的差異不足以將微擬球藻中各個種分開。rbcL基因的進化速度較18SrRNA基因快,不僅能分開微擬球藻屬的各個種,而且同種間的不同株系之間也有堿基的差異。對于微擬球藻的分類鑒定,rbcL基因比18SrRNA基因更有效[3]。rbcL基因具有通用、易擴增和易比對的特點,并且該基因數據庫相對豐富。但能否將rbcL基因作為微擬球藻的候選條形碼還需要大量的工作,涵蓋更多的微擬球藻種和株系。而該基因是否能作為微藻的候選條形碼則需要更多更全面的工作和研究。
該方法中的形態學指標和生理生化指標鑒定未能實現微擬球藻的種間鑒定,且電鏡(無論掃描電鏡或是透射電鏡)的樣品制備過程十分繁復,制作切片也需要進行專業的訓練,否則會影響鑒定。對于硅藻和甲藻而言,掃描電鏡是非常好的鑒定工具。利用掃描電鏡能將在光學顯微鏡下難以觀察到的硅殼的花紋細節和甲板的細節放大,這些硅殼和甲板上的細節就是鑒定的依據。但近幾年來,硅藻和甲藻的分子系統學分析層出不窮,且該方法不僅能快速準確地實現鑒定,還能省去長時間培養、制作切片等費時費力的步驟。對于微擬球藻而言,光鏡或是電鏡觀察都不能實現快速、準確的鑒定。
依據生理生化指標的鑒定方法所需的藻液量大,所需的培養時間長,而且只能作為輔助的鑒定手段,難以達到大規模、快速鑒定的目的。在本研究中脂肪酸組成和色素組成的分析都無法實現微擬球藻的種間鑒定。因此,依據分子標簽序列進行鑒定是實現微擬球藻快速、準確的鑒定的最好方法。
對于野外大規模樣品的快速鑒定,可以先用光學顯微鏡觀察的方法初步推斷未知藻株的分類地位,再用分子標簽序列進行鑒定。該微擬球藻的鑒定方法是否適用于微型海洋微藻的鑒定還需要進行進一步驗證。
微藻種類繁多,形態各異,生化組成各不相同,進化速度也有很大差異,所以微藻的鑒定工作仍未形成標準。本研究根據形態學、生化成分和分子手段共同鑒定,準確的將一株未知的微擬球藻鑒定到種,初步形成了準確、快速的微藻分類體系,為今后形成系統的微藻分類體系打下了基礎。
[1]Karlson B,Potter D,Kaglenstierna M,et al.Ultrastructure,pigment composition,and 18SrRNA gene sequence forNannochloropsisgranulatasp.nov.(Mondopsidaceae,Eustigmatophyceae),a marine ultraplankter isolated from the Skagerrak,northeast At-lantic Ocean[J].Phycology,1996,35:253-260.
[2]Krienitz L,Hopporle D,Stich H B,et al.Nannochloropsislimnetica(Eustigmatophyceae),a new species of picoplankter from freshwater[J].Phycologia,2000,39:219-227.
[3]Suda S,Atsumi M,Miyashita M.Taxonomic characterization of marineNannochloropsisspecies,N.oceanicsp.nov.(Eustigmatophyceae)[J].Phycologia,2002,41(3):273-279.
[4]Hibberd D J.Notes on taxonomy and nomenclature of algal Eustigmatophyceae and Tribophyceae (synonym Xanthophyceae)[J].Botanical Journal of the Linnean Society,1981,82(2):93-119.
[5]Lubián L M,Montero O,Moreno-Garrido I,et al.Nannochloropsis(Eustigmatophyceae)as a source of commercially valuable pigments[J].J Appl Phycol,2000,12:249-255.
[6]Zou N,Richmond A.Effect of light-path length in outdoor flat plate reactors on output rate of cell mass and EPA inNannochloropsissp.[J].J Biotenchnol,1999,70:351-356.
[7]Apt K E,Behrens P W.Commercial developments in microalgal biotechnology[J].Journal of Phycology,1999,35:215-226.
[8]Gouveia L,Oliverara C.Microaglae as a raw material for biofuels production[J].J Ind Microbiol Biotechnol,2009,36:269-274.
[9]Fawley K P,Fawley M W.Observations on the diversity and ecology of freshwaterNannochloropsis(Eustigmatophyceae),with descriptions of new taxa[J].Protist,2007,158:325-336.
[10]Anderson R A,Brett R W,Potter D.Phylogyeny of the Eustigmatophyceae based upon 18SrRNA gene,with emphasis onNannochloropsis[J].Protist,1998,149:61-74.
[11]Pennisi E.Wanted:A barcode for plants[J].Science,2007,318:190-191.
[12]Kress W J,Erickson D L.DNA barcodes:Genes,genomics,and bioinformatics[J].Proceedings of the National Academy of Science USA,2008,105:2761-2762.
[13]鐘秀容,陳連云,陳文列.單細胞海藻的透射電鏡樣品制備 [J].電子顯微學報,2005,24(4):434-434.
[14]Zapata M,Rodríguez F,Garrido J L.Separation of chlorophylls and carotenoids from marine phytoplankton:a new HPLC method using a reversed phase C8:column and pyridine-containing mobile phases[J].Marnie Eeology Progress Series,2000,195:29-45.
[15]Lepage G,Roy C.Improved recovery of fatty acids through direct transesterification without prior extraction or purification[J].J Lipid Res,1984,25:1391-1396.
[16]陳穎,劉根齊,李文彬,等.三種小球藻DNA提取方法的比較[J].植物生理學通報,2001,37(3):242-244.
[17]Hashimoto H.The ultrastructural features and division of secondary plastids[J].J Plant Res,2005,118:163-172.
[18]Van den Hoek C,Mann D G,Jahus H M C.Algae-An Introduction to Phycology[M].New York:Cambridge University Press,1995.
[19]Murakami R,Hashimoto H.Unusual nuclear division inNannochloropsisoculata(Eustigmatophyceae,Heterokonta)which may ensure faithful transmission of secondary plastids[J].Protist,2009,160:41-49.
[20]Santos L M A,Lee Dale G F.Some notes on the ultrastructure of small azoosporic members of the algal class Eustigmatophyceae[J].Nova Hedwigia,1995,60:219-225.
[21]Johnson E A,An G H.Astaxanthin from microbial sources[J].Critical Reviews in Biotechnology,1991,11:297-326.
[22]Millner H W.The fatty acids of Chlorella[J].Biochem,1948,176:813-817.
[23]Graeve M,Kattner G,Wiencke C,et al.Fatty acid composition of arctic and Antarctic marcroalgae:indicator of phylogentic and trophic relationship[J].Marine Ecol Prog Series,2002,231:67-74.
[24]Khotimchenko S U.Fatty acid composition of 12species of Chlorophyceae from the Senegalese coast[J].Phytochemistry,1992,31:2739-2741.
[25]Khotimchenko S V.Fatty acids of brown algal the Russian far east[J].Phytochemistry,1998,49(8):2363-2369.
[26]Carpenter E J,Harvey H R,Fry B,et al.Biogeochemistry tracers of the marineTrichodesmiumcganobacterium[J].Deep-Sea Research PartⅠ,1997,44:27-38.
[27]Bucklin A,Wiebe P H,Smolenack S B,et al.DNA barcodes for species identification of euphausiids(Euphausiacea,Crustacea)[J].J Plantkton Res,2007,29:483-493.
[28]Lin S,Zhang H,Hou Y,et al.High-level diversity of dinoflagellates in the Natural Environment,revealed by assessment of mitochondrial cox1and cob genes for dinoflagellate DNA barcoding[J].Appl Environ Microbiol,2009,75:1279-1290.
[29]Webb K E,Baenes D K A,Clark M S,et al.DNA barcoding:a molecular tool to identify Antarctic marine larvae[J].Deep-Sea Research PartⅡ,2006,53:1053-1060.
[30]Jinbo U,Kato T,Ito M.Current progress in DNA barcoding and future implications for entomology[J].Entomological Science,2011,14:107-124.
[31]Fazekas A J,Burgess K S,Kasanakarti P R,et al.Multiple multilocus DNA barsodes from the plastids genome discriminate plant species equally well[J].PLoS One,2008,3:2802.
[32]CBOL Plant Working Group.A DNA barcode for land plants[J].Proceedings of the National Academy of Science,2009,106:12794-12797.
[33]Clarkston B E,Saunders G W.A comparision of two DNA barcode markers for species discrimination in the red algal family Kallymeniaceae(Gigartinales,Florideophyceae),with a description ofEuthoratrimburtoniisp.nov.[J].Botany,2010,88:119-131.
[34]Saunders G W.Applying DNA barcoding to red macroalgal:a preliminary appraisal holds promise for future applications[J].Phil Trans R Soc B,2005,360:1879-1888.
[35]Lee S R,Oak J H,Keum Y S,et al.Utility ofrbcS gene as a novel target DNA region for brown algal molecular systematic[J].Phycological Resarch,2011,59:34-41.
[36]Hamsher S E,Evans K M,Mann D G,et al.Barcoding Diatoms:Exploring alternatives to COⅠ-5P[J].Protist,2011,162:405-422.
[37]Moniz M B J,Kaczmarska I.Barcoding of Diatoms:Nuclear encoded ITS revisited[J].Protist,2010,161:7-34.

