利用PCR擴增及基因合成構建Adnectin半隨機核糖體展示文庫
郭永紅,問嬌,王俊紅(西安交通大學第二附屬醫院,西安 710004)
?
利用PCR擴增及基因合成構建Adnectin半隨機核糖體展示文庫
郭永紅,問嬌,王俊紅(西安交通大學第二附屬醫院,西安 710004)
摘要:目的構建Adnectin半隨機的核糖體(mRNA)展示文庫。方法分析Adnectin核糖體展示文庫結構序列的編碼氨基酸序列,利用無意突變建立酶切位點,使用PCR擴增和基因合成兩種方法相結合,構建Adnectin半隨機核糖體展示文庫,通過限制性內切酶及DNA測序證實序列正確性,對文庫轉化菌的滴定和插入失活藍白菌斑篩查和計數測定計算文庫的庫容。結果經測序證實文庫結構的正確性,文庫的庫容為1.54×1013/mL。結論成功構建Adnectin半隨機的核糖體展示文庫,方法簡單,庫容量大,該文庫的建成為親和各種相關蛋白的Adnectin結合序列奠定基礎。
關鍵詞:Adnectin;PCR擴增;基因合成;核糖體展示文庫
Adnectins是基于Ⅲ型纖維連接蛋白第10結構域(10Fn3)所設計衍生的半人工蛋白分子,可以模擬抗體結合靶蛋白分子,具有高親和力、高特異性、反復使用不產生抗體等優點[1],被認為是有極好應用前景的蛋白藥物[2]。核糖體展示技術是利用PCR技術擴增目的基因文庫,同時引入啟動子、核糖體結合位、間隔序列、5′端和3′端莖環結構,然后在體外無細胞系中進行轉錄和翻譯[3],利用體外表達體系以mRNA—核糖體—抗體三元復合物的形式將基因型和表型聯系起來用于抗體開發的技術。采用體外構建、篩選的方法改造抗體,避免了噬菌體展示技術轉化步驟的限制,更易構建出高容量、高質量的文庫篩選出特異性蛋白,為制備小分子蛋白質或多肽開辟了一條新途徑[4]。2012年6月~2013年3月,本研究利用基因合成和PCR擴增兩者相結合方法,成功地構建了Adnectin半隨機的核糖體(mRNA)展示文庫,該文庫的建成為親和各種相關蛋白的Adnectin結合序列奠定了基礎。
1材料與方法
1.1材料pGEM-Teasy載體及菌種Ecoli DH5α和TOP10菌種均由本實驗室保存,質粒pES+G18602由華廣生物有限公司提供。限制性內切酶、DNA Marker、DNA連接試劑盒均購自TaKaRa寶生物工程(大連)有限公司。PCR產物純化試劑盒和凝膠回收試劑盒為Qiagen quick Gel Extraction Kit(Cat28704);質粒小量抽提試劑盒及基因合成為上海生物工程技術服務有限公司提供,其余試劑均為分析純。
1.2方法
1.2.1基因合成結合PCR擴增構建文庫設計方法Adnectin核糖體(mRNA)展示文庫結構和序列:T7 promoter-5′-stem loop-ribosome binding site-Adnectin library-spacer-3′-stem loop,使用基因合成方法合成文庫的兩側翼序列:T7 promoter-5′-stem loop-ribosome binding site-SpeI-spacer-Hind Ⅲ-3′-stem loop序列,使用PCR方法合成半隨機文庫-SpeI-spacer-Hind Ⅲ序列,使用限制性內切酶Spe Ⅰ和Hind Ⅲ雙酶切后將半隨機文庫插入到合成兩翼序列載體中,使半隨機文庫5′端加載核糖體展示需要的T7啟動子、莖環結構、核糖體結合位點和起始密碼子,3′端加載莖環結構和間隔序列,建立插入Adnectin核糖體(mRNA)展示文庫的質粒載體。半隨機文庫的酶切點Spe Ⅰ的選擇是依據對Adnectin核糖體(mRNA)展示文庫結構序列的編碼氨基酸序列分析后,使用無意突變后確定,第152及155位核苷酸為突變處,原序列ACCAGC突變后ACTAGT,編碼相同的氨基酸-TS-,ACTAGT序列成為了可以使用的Spe Ⅰ酶識別點。把需要構建的如下序列分成了兩個區,使用PCR方法擴增半隨機文庫-Spe Ⅰ-spacer-Hind Ⅲ序列,由GATCAG開始到AAGCTTG,余序列由基因合成后連接入pES+G18602質粒。需要構建的全序:ATACGAAATTAATACGACTCACTATAGG-GAGACCACAACGGTTTCCCTCTAGAAATAATTTTG-TTTAACTTTAAGAAGGAGATATATCCATGGACTAC-AAAGACGTTTCTGATGTTCCGAGGGACCTGGAAGT-TGTTGCTGCGACCCCCACTAGTCTACTGATCAGCT-GGNNBNNBNNBNNBNNBNNBNNBTATTACAGGAT-CACTTACGGAGAAACAGGAGGAAATAGCCCTGTC-CAGGAGTTCACTGTGCCTNNBNNBNNBNNBNNBG-CTACCATCAGCGGCCTTAAACCTGGAGTTGATTAT-ACCATCACTGTGTATGCTGTCACTNNBNNBNNBNN-BNNBNNBNNBNNBNNBNNBCCAATTTCCATTAATT-ACCGAACAGAAATTGACAAACCATCCCAGGGCTTT-AATGAAAAGCTTGATCCATTCGTTTGTGAATATCA-AGGCCAATCGTCTGACCTGCCTCAACCTCCTGTCA-ATGCTGGCGGCGGCTCTGGTGGTGGTTCTGGTGGC-GGCTCTGAGGGTGGTGGCTCTGAGGGTGGCGGTTC-TGAGGGTGGCGGCTCTGAGGGAGGCGGTTCCGGTG-GTGGCTCTGGTTCCGGTGATTTTGATTATGAAAAG-ATGGCAAACGCTAATAAGGGGGCTATGACCGAAA-ATGCCGATGAAAACGCGCTACAGACCGCACACCT-TACTGGTGTGCGG。其中N表示A、C、G、T 4種堿基隨機;B表示C、G兩種隨機堿基;TAATA為T7促進子;ATGGAC開始為文庫序列;GATCCA開始為間隔序列;ACCGC開始為莖環結構。
1.2.2PCR擴增半隨機文庫方法首先,使用含有第三helix bundle的F1引物和R1引物做PCR擴增,獲得產物作為二次PCR的模板;再使用F2和R2引物進行PCR擴增,產物作為反應模板,使用含有第二helix bundle的F3引物和R3引物擴增后產物用于下一次PCR反應模板,在以后的PCR反應中F向引物依次增加,R向引物一直使用R3引物,直至F6和R3引物擴增后得到產物。半隨機文庫PCR引物:F1:GTGTAT GCTGTCACTN NBNNBNNBNN BNNBNNBNNB NNBNNBNNBC CAATTTCCAT T;R1:GTTTGTCA ATTTCTGTTC GGTAATTAAT GGAAATTGG;F2:T CAG CGGCCTTAAA CCTGGAGTTG ATTATACCAT CACTGTGTAT GCTGTCAC-T;R2:GCTTTTCATT AAAGCCCTGG GATGGTTTGT CAATTTCTG;F3:CCTGTCCAGG AGTTCACTGT GCCTNNBNNB NNBNNBNNBG CTACCATCAG CGGCC;R3:CAA GCTTTTCATT AAAGCCC;F4:TATTACAGG ATCACTTACG GAGAAACAGG AGGAAATAGC CCTGTCCAGG AGTTC;F5:GTCTACT GATCAGCTGG NNBNNBNNBN NBNNBNNBNN BTATTACAGG ATCACTTAC;F6:CA CTAGTCTACT GATCAGCTGG。其中N表示A、C、G、T 4種堿基隨機,B表示C、G兩種隨機堿基。
1.2.3半隨機文庫的構建方法PCR反應條件:95 ℃ 5 min;95 ℃ 40 s;56 ℃ 1 min;72 ℃ 1 min;18個循環;最后72 ℃延伸10 min,以25 μL PCR反應體系進行。PCR產物進行2%瓊脂糖凝膠電泳鑒定,擴增成功的PCR產物以膠回收試劑盒進行純化回收,為增加文庫容量,每次PCR使用5 μL產物做模板,在5個平行管內擴增,產物混合后作為模板使用。將PCR純化產物與pGEM-Teasy質粒載體16 ℃連接過夜,最后的PCR產物全部插入到pGEM-Teasy質粒載體轉導Top10受體菌,取感受態菌100 μL加重組體10 μL,常規方法操作,共轉導100管受體菌,50管菌體做LB(Luria-Bertani)—氨基芐青霉素(Amp)固體培養基培養,挑取單菌落進行Spe Ⅰ及Hind Ⅲ雙酶切鑒定,切膠回收酶切產物。
1.2.4重組pES+G18602載體的構建方法DNA連接試劑盒連接,16 ℃過夜。將連接產物轉化感受態DH5α,37 ℃培養16~18 h,將PCR產物連接入含有合成基因的pES+G18602載體,挑取單菌落進行酶切鑒定,篩選正確的菌落20個克隆送上海生工生物工程技術服務有限公司測序,采用Pubmed Blast進行序列檢索比對。
1.2.5文庫容量計算方法50管菌體經倍比稀釋轉化大腸桿菌Top10,取轉化菌10 μL涂布于表面涂有X-gal(Galactoside)和IPTG(Isopropyl β-D-1-thiogalactopyranoside)的含Amp瓊脂糖平板上,37 ℃過夜培養。藍白班篩選計數細菌轉化克隆和計算文庫容量。
2結果
2.1PCR產物電泳結果見圖1。
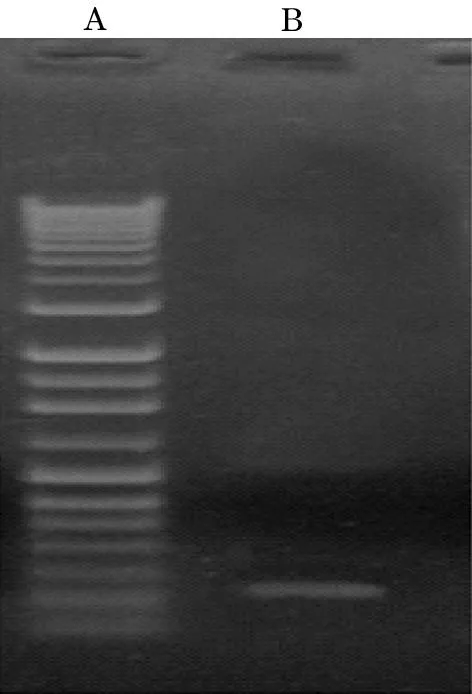
注:A為DNA Marker,分子量從下到上分別是100、200、300、400、500、600、750、1 000、1 314、1 614、2 011、3 000、4 000、5 000、6 000和12 000 bp;B為PCR擴增產物,分子量為263 bp。
圖1PCR產物的瓊脂糖凝膠電泳鑒定圖
2.2重組pGEM-Teasy-Adnectin質粒酶切電泳鑒定結果見圖2。

注:A為DNA Marker,分子量從下到上分別為100、200、300、400、500、600、750、1 000、1 314、1 614、2 011、3 000、4 000、5 000、6 000和12 000 bp;B為限制性內切酶SpⅠ及HindⅢ酶切產物,分子量為263 bp。
圖2重組pGEM-Teasy-Adnectin質粒酶切電泳圖
2.3文庫轉化菌克隆的測序結果20個送檢測序的克隆,僅有13個克隆測序顯示具有完整的three-helix bundle結構,經Blast比對序列完全符合,見插頁Ⅰ圖1。其余7個克隆僅有two-helix bundle結構,產生的原因可能和該插入序列多發夾結構使測序信號中斷有關,可能也同PCR擴增產生錯誤有關。
2.4文庫容量計算結果對文庫轉化菌的滴定和插入失活藍白菌斑篩查和計數,最終獲得純化的PCR產物15.4 μg,文庫的庫容為1.54×1013/mL。
3討論
10Fn3獨特的結構和良好的生物物理特性決定其最適合作為蛋白支架。10Fn3是最早被研究、也是最具臨床應用前景的支架蛋白[5~7],其結構與抗體可變區相似,適合裝配多功能分子;10Fn3不包含二硫鍵或自由巰基,這種結構特點保證了其在高溫和還原環境下的穩定性,并且能在細菌內高效表達活性產物[8];10Fn3作為蛋白支架,其本身無毒性和免疫原性;10Fn3的另一個優勢是它可以天然適合多聚化產生多功能連接分子。10Fn3中的3個環狀結構(BC、DE和FG)在結構上與抗體分子的互補決定區(CDR)中的H1、H2及H3同源,改變該處的氨基酸序列則可以使其理論上能與任何配體分子特異結合[9]。
Adnectins是基于10Fn3所設計的一類半人工蛋白分子,對于目標靶分子高親和力和高特異性的特性使得Adnectins被認為是未來最具應用前景的蛋白藥物[10~12]。
目前,報道篩選出的Adnectins有20個左右,靶分子包括泛素、TNF-α、溶菌酶、SARS病毒N蛋白、EGFR、VEGF-R2等[13~16],篩選方法包括噬菌體展示系統、酵母展示技術、酵母雙雜交技術及核糖體mRNA展示技術等,篩選出的Adnectin與靶分子的親和力不乏與抗原抗體之間親和力相媲美者。其中,針對VEGF-R2的Adnectin和CT-322已經批準進入二期臨床試驗,用于多形膠質母細胞瘤、非小細胞肺癌及轉移性結腸癌的治療[17~19]。
核糖體展示技術是針對大容量基因庫高效篩選功能基因的有效方法之一,其相比于目前的體內展示技術(噬菌體展示技術、酵母表面展示技術)有兩個關鍵的特性:首先,篩選不受轉化效率的限制,可針對大容量基因庫進行富集篩選;其次,在PCR過程中可引入點突變改變功能蛋白的專一性和親和性,同時通過核糖體—蛋白質—mRNA三聯體的配體結合反應選擇功能蛋白。通過核糖體展示文庫篩選Adnectins,可以構建更大庫容的庫。經過參閱相關文獻,設計選擇合成長片段和PCR的方法構建用于轉錄和篩選的DNA鏈。半隨機文庫的酶切點Spe Ⅰ的選擇是依據對原Adnectin核糖體(mRNA)展示文庫結構序列的編碼氨基酸序列分析后,使用無意突變后確定,第152及155位核苷酸為突變處,原序列ACCAGC突變后ACTAGT,編碼相同的氨基酸-TS-,ACTAGT序列成為了可以使用的Spe Ⅰ酶識別點。使用PCR方法擴增半隨機文庫-Spe Ⅰ-spacer-Hind Ⅲ序列,使用基因合成方法合成文庫的兩側翼序列,即T7 promoter-5′-stem loop-ribosome binding site—Spe Ⅰ-spacer -Hind Ⅲ-3′-stem loop序列。這樣可以使用內切酶Spe Ⅰ和Hind Ⅲ雙酶切后將半隨機文庫文庫插入到合成兩翼序列載體中,使半隨機文庫文庫5′端加載核糖體展示需要的T7啟動子、莖環結構、核糖體結合位點和起始密碼子,3′端加載莖環結構和間隔序列,建立插入Adnectin核糖體(mRNA)展示文庫的質粒載體,且整個核糖體展示模板不存在終止密碼子,保證mRNA-核糖體—蛋白質以復合物的形式存在。目前,DNA合成技術已非常成熟,能保證合成的長DNA鏈的準確性;PCR過程中以多投入引物、減少循環次數的方法減少偏倚;每次反應后進行長度檢測,將終產物克隆入質粒載體,進行多個克隆的序列分析,推算文庫的多樣性;通過這些措施,保證文庫的質量,而引物的設計是決定文庫多樣性的關鍵。經限制性內切酶及DNA測序證實了文庫結構的正確性和文庫序列的多樣性,對文庫轉化菌的滴定和插入失活藍白菌斑篩查和計數測定計算,文庫的庫容為1.54×1013/mL。
綜上所述,使用基因合成和PCR擴增兩者相結合的方法,成功的構建了Adnectin半隨機的核糖體(mRNA)展示文庫。該文庫構建方法較之前傳統方法簡單,成本低,庫容量大,為構建親和各種相關蛋白的Adnectin結合序列奠定了基礎。
參考文獻:
[1] Lipovsek D. Adnectins: engineered target-binding protein therapeutics[J]. Protein Eng Des Sel, 2011,24(1-2):3-9.
[2] Ackermann M, Carvajal IM, Morse BA, et al. Adnectin CT-322 inhibits tumor growth and affects microvascular architecture and function in Colo205 tumor xenografts[J]. Int J Oncol, 2011,38(1):71-80.
[3] Mattheakis LC, Bhatt RR, Dower WJ, et al. An in vitro ploysome display system for identifying ligands from very large peptide libraries[J]. Proc Natl Acad Sci USA, 1994,91(19):9022-9026.
[4] Yan X, Xu Z. Ribosome-display technology: applications for directed evolution of functional proteins[J]. Drug Discovery Today, 2006,11(19-20):911-916.
[5] Gebauer M, Skerra A. Engineered protein scaffolds as next-generation antibody therapeutics[J]. Curr Opin Chem Biol, 2009,13(3):245-255.
[6] Beck A, Wurch T, Bailly C, et al. Strategies and challenges for the next generation of therapeutic antibodies[J]. Nat Rev Immunol, 2010,10(5):345-352.
[7] Koide A, Bailey CW, Huang X, et al. The fibronectin type Ⅲ domain as a scaffold for novel binding proteins[J]. J Mol Biol, 1998,284(4):1141-1151.
[8] Batori V, Koide A, Koide S. Exploring the potential of the monobody scaffold: effects of loop elongation on the stability of a fibronectin type Ⅲ domain[J]. Protein Eng, 2002,15(12):1015-1020.
[9] Bloom L and Calabro V. FN3: a new protein scaffold reaches the clinic[J]. Drug Discov Today, 2009,14(19-20):949-955.
[10]Lipovsek D. Adnectins: engineered target-binding protein therapeutics[J]. Protein Eng Des Sel, 2011,24(1-2):3-9.
[11] Xu L, Aha P, Gu K, et al. Directed evolution of high-affinity antibody mimics using mRNA display[J]. Chem Biol, 2002,9(8):933-942.
[12] Hackel BJ, Wittrup KD. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold[J]. Protein Eng Des Sel, 2010,23(4):211-219.
[13] Lipovsek D, Lippow SM, Hackel BJ, et al. Evolution of an interloop disulfide bond in high-affinity antibody mimics based on fibronectin type Ⅲ domain and selected by yeast surface display: molecular convergence with single-domain camelid and shark antibodies[J]. J Mol Biol, 2007,368(4):1024-1041.
[14] Liao HI, Olson CA, Hwang S, et al. mRNA display design of fibronectin-based intrabodies that detect and inhibit severe acute respiratory syndrome coronavirus nucleocapsid protein[J]. J Biol Chem, 2009,284(26):17512-17520.
[15] Hackel BJ, Ackerman ME, Howland SW, et al. Stability and CDR composition biases enrich binder functionality landscapes[J]. J Mol Biol, 2010,401(1):84-96.
[16] Getmanova EV, Chen Y, Bloom L, et al. Antagonists to human and mouse vascular endothelial growth factor receptor 2 generated by directed protein evolution in vitro[J]. Chem Biol, 2006,13(5):549-556.
[17] Dineen SP, Sullivan LA, Beck AW, et al. The Adnectin CT-322 is a novel VEGF receptor 2 inhibitor that decreases tumor burden in an orthotopic mouse model of pancreatic cancer[J]. BMC Cancer, 2008,8:352.
[18] Molckovsky A, Siu LL. First-in-class, first-in-human phase I results of targeted agents: highlights of the 2008 American society of clinical oncology meeting[J]. J Hematol Oncol, 2008,1:20.
[19] Ackermann M, Carvajal IM, Morse BA, et al. Adnectin CT-322 inhibits tumor growth and affects microvascular architecture and function in Colo205 tumor xenografts[J]. Int J Oncol, 2011,38(1):71-80.
Construction semi-random Adnectin ribosome display library by PCR amplification
and gene synthesis
GUOYong-hong,WENJiao,WANGJun-hong
(TheSecondAffiliatedHospitalofXi′anJiaotongUniversity,Xi′an710004,China)
Abstract:ObjectiveTo construct semi-random Adnectin ribosome display library. MethodsAdnectin ribosome display library was constructed by PCR amplification combining with gene synthesis method. PCR products contained the restrictive sites of SpeⅠbased on nonsense mutation. The recombinant plasmids were identified by restriction endonuclease enzyme analysis and DNA sequence. The positive recombinant clones were screened and library capacity was measured by blue-white test. ResultThe library was confirmed by sequencing and library capacity was 1.54×1013/mL. ConclusionsTo corstruct successful Adnectin semi-random ribosomes display library (mRNA) used simple and easy method. The built library laid the foundation for Adnectin affinity with various related protein.
Key words:Adnectin; PCR amplification; gene synthesis; ribosomes display library
(收稿日期:2014-09-24)
中圖分類號:Q784
文獻標志碼:A
文章編號:1002-266X(2015)04-0004-04
doi:10.3969/j.issn.1002-266X.2015.04.002
通信作者簡介:王俊紅(1970-),男,碩士,副主任醫師,研究方向為基因治療機制。E-mail: jhwangdoctor@sohu.com
作者簡介:第一郭永紅(1971-),女,博士,副主任醫師,研究方向為肝硬化及肝癌的分子機制。E-mail: xiaoqing9759@sina.com
基金項目:國家自然科學基金青年科學基金項目(81100300)。

