SARIMA模型在戊型肝炎發病趨勢預警預測中的應用
祝寒松,黃文龍,謝忠杭,陳光敏,曹 洋,章燦明,陳 武,歐劍鳴,洪榮濤
SARIMA模型在戊型肝炎發病趨勢預警預測中的應用
祝寒松1,黃文龍1,謝忠杭1,陳光敏1,曹 洋2,章燦明1,陳 武1,歐劍鳴1,洪榮濤1
目的 采用SARIMA模型對福建省戊肝發病趨勢進行預測,為預警和風險評估提供定量數據。方法 以Eviews5.0對福建省2004年1月-2015年12月戊肝的月發病例數進行SARIMA分析。結果 2004年1月-2014年8月福建省戊肝月發病序列呈先升后降的趨勢和周期性波動,取自然對數和1次1階非季節差分后序列得到平穩化,模型SARIMA (0,1,1)(1,0,1)12和SARIMA(2,1,0)(1,0,1)12參數有統計學意義,殘差為白噪聲,后者為最優模型,表達式為:(1+0.61L+0.23L2)(1-0.89L12)(1-L)log(yt)=(1-0.89L12)εt。靜態回代預測值和實際值吻合較好,相對誤差取絕對值后均數為13.06%。2014年1-8月預測值的相對誤差較小(5月份除外),預測標準誤(S.E)較小。結論 運用SARIMA模型可對戊肝發病趨勢進行較準確的短期預測,可為及時、科學地研判風險提供可靠的數據基礎。
季節時間序列模型;戊肝;預警;預測;風險評估
戊型肝炎(戊肝,Hepatitiv E)是由戊型肝炎病毒引起的一種急性人獸共患病,目前在全球各地廣泛傳播,嚴重危害人類健康,據估計20億的世界人口有可能被感染Hepatitiv E病毒的風險,死亡率也占0.5%,并且有可能在許多發展中國家導致大暴發,近年來發病率在我國呈逐年上升趨勢[1-3]。為了對戊肝發病趨勢進行預測和預警,考慮到其發病具有一定的周期性,而季節時間序列模型(Seasonal Autoregressive Integrated Moving Average Model, SARIMA)可用于對存在周期性變化的時間序列進行預測,因此,本文擬采用SARIMA模型對福建省戊肝發病趨勢進行定量預測,以期為風險評估提供基礎資料,現報告如下。
1 材料與方法
1.1 材料 來自福建省各級衛生機構通過中國疾病預防控制信息系統的報告(按“現住址”、“終審”選項下載)。
1.2 方法 采用SAS9.2軟件對數據進行匯總、清洗,運用Eviews 5.0軟件對數據進行SARIMA建模和分析,α=0.05(雙側)。
1.2.1 原理 ARIMA模型通常借助時間序列的隨機特性來描述事物的發展變化規律,從而解釋并預測時間序列的變化發展規律,而SARIMA模型是ARIMA模型的擴展和改進,包含了季節性或周期性因素[4-5],對于存在季節性的非平穩時間序列不能直接建立ARIMA模型,可考慮進行非季節差分和季節性差分使其平穩化,基本模型:SARIMA(p,d,q)(P,D,Q)S,表達式為:φp(L)ΦP(LS)(1-L)d(1-LS)Dyt=θq(L)ΘQ(LS)εt。其中,p、q分別是非季節自回歸過程AR和移動平均過程MA的階數;d、D分別是序列yt的非季節差分和季節差分階數;P、Q分別是季節自回歸過程SAR和移動平均過程SMA的階數。φp(L)、ΦP(L)分別是非季節自回歸過程AR和季節自回歸過程SAR的滯后算子多項式,θq(L)、ΘQ(LS)分別是非季節移動平均過程MA和季節移動平均過程SMA的滯后算子多項式,(1-L)d、(1-LS)D分別是對序列yt的非季節差分和季節差分滯后算子,S是季節差分的步長,εt是殘差序列(白噪聲序列)。
1.2.2方法 ①繪制原序列的曲線圖以識別其基本形式,加入截距項和時間趨勢項做ADF單位根檢驗和Q統計量以判斷平穩性。若為非平穩,可考慮對其取自然對數后做非季節差分和(或)季節性差分使之滿足平穩性條件。②識別新序列自相關函數(autocorrelation function, ACF)和偏相關函數(partial autocorrelation function, PACF) 的SARIMA模型形式。③估計模型參數,對結果進行t檢驗(參數檢驗),通過Q檢驗進行殘差分析(白噪聲檢驗)。如果殘差序列不是白噪聲序列,說明殘差序列中還存在有用的信息未被提取出來,需要對原模型進一步調整,以便得到更合適的模型。④利用調整R2、Akaike info criterion (AIC)準則和Schwarz criterion (SC)準則評價其相對優勢,調整R2越大、AIC和SC越小,一般認為越好。⑤利用所估計的SARIMA模型,進行回代評價和預測。
2 結 果
2.1 原序列分析 2004年1月-2014年8月福建省戊肝月發病序列呈先升后降的趨勢和周期性波動(圖1),ADF單位根檢驗,P=0.00<0.05,但自相關函數呈現指數衰減,且衰減速度緩慢,所以認為該序列不平穩(圖2)。
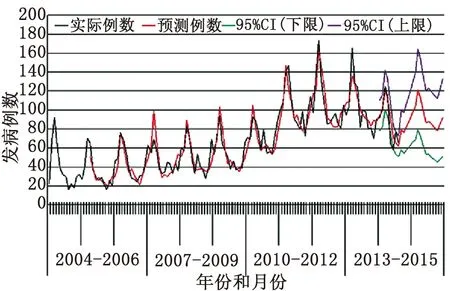
圖1 福建省2004-2015年戊肝月發病例數的實際值和預測值
Fig.1 Observed and predicted values of hepatitis E in Fujian Province, per month from 2004 to 2015
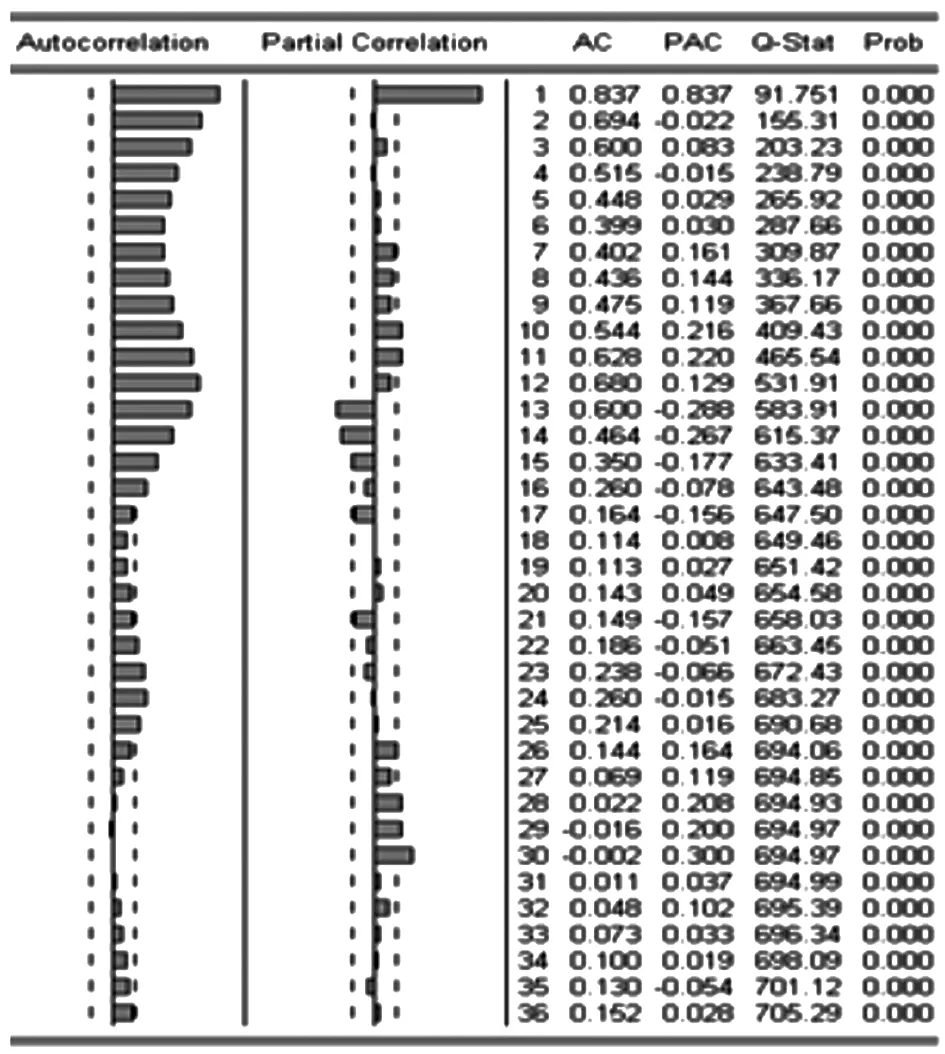
圖2 福建省2004年1月-2014年8月戊肝月發病數序列的自相關和偏相關圖
Fig.2 ACF and PACF diagram of hepatitis E incidence in Fujian Province, per month from January 2004 to August 2014
2.2 平穩化 采用2種方法對原序列進行處理,第1種是取自然對數和1次1階非季節差分,再做1次12階季節差分;第2種是只取自然對數和1次1階非季節差分。通過比較,后者效果更好,現只對其進行分析:差分后的序列已接近平穩(圖3),ADF單位根檢驗P=0.00<0.05;ACF和PACF顯示,序列平穩性已得到明顯改進,滯后1階和7階顯著地不為0,其它近似為1個平穩過程(圖4)。因此,可認為序列已得到平穩化。
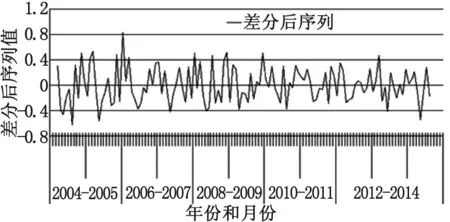
圖3 經自然對數和1次1階非季節差分后的序列
Fig.3 Time series diagram after conducting natural logarithm and once non-seasonal of lag 1 difference
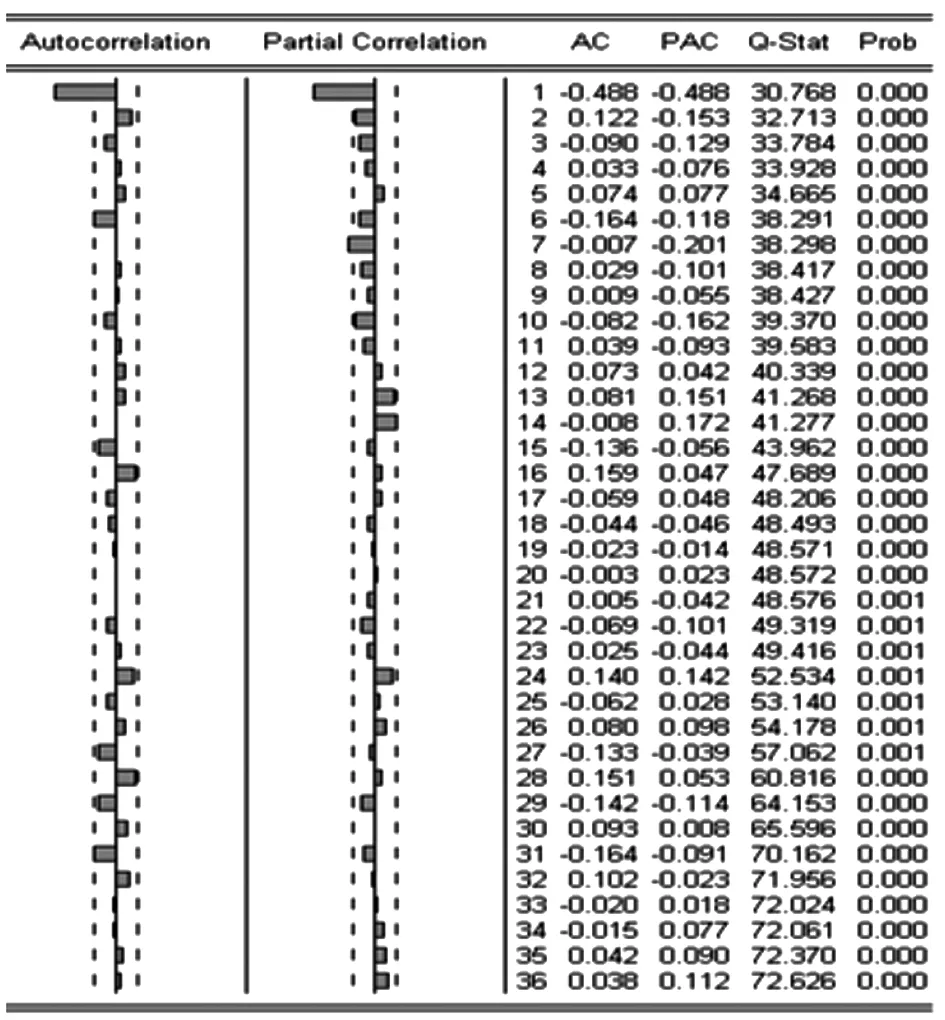
圖4 序列經差分后的自相關和偏相關圖
Fig.4 ACF and PACF diagram after conducting natural logarithm and a non-seasonal difference
2.3 建模 由2.2差分可知d=1、D=0,ACF滯后1階呈截尾,PACF滯后2階呈截尾或者拖尾至2階。通過嘗試,2組模型通過t檢驗(p<0.05)和Q檢驗(全部p>0.05,已呈明顯的白噪聲序列):SARIMA(0,1,1)(1,0,1)12、SARIMA(2,1,0) (1,0,1)12(表1)。通過調整R2、AIC和SC比較,SARIMA(2,1,0) (1,0,1)12擬合優度較好(表2),Q檢驗見圖5,表達式為:(1+0.61L+0.23L2)(1-0.89L12)(1-L)log(yt)=(1-0.89L12)εt。
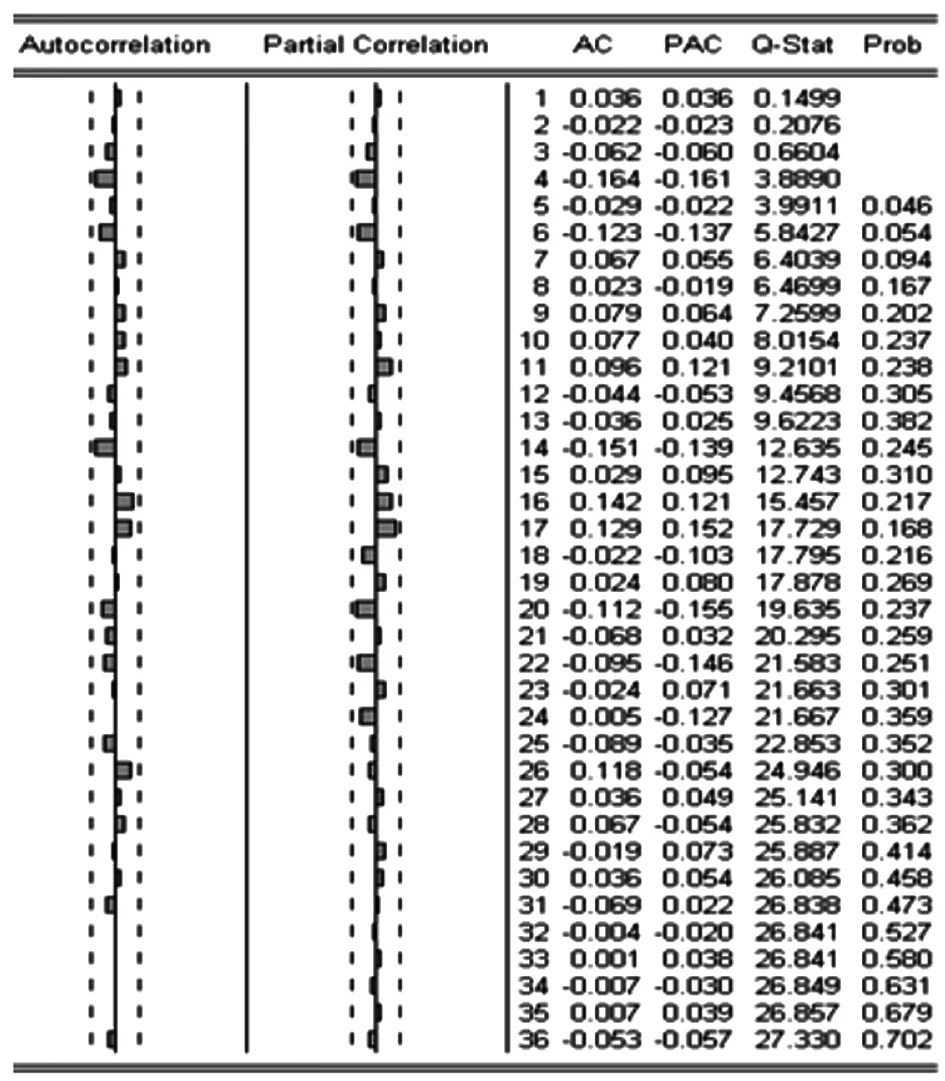
圖5 SARIMA(2,1,0)(1,0,1)12模型的Q檢驗
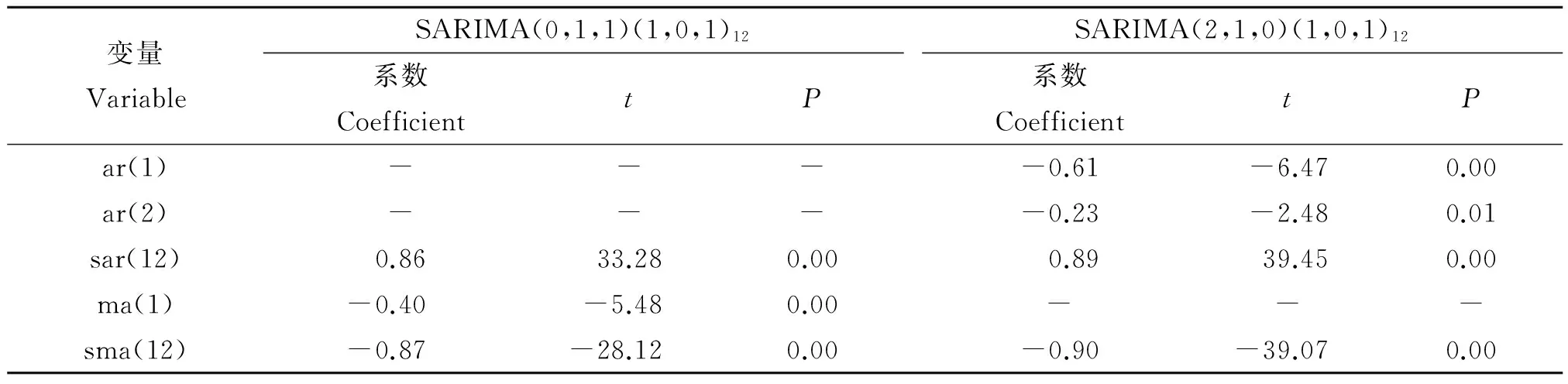
變量VariableSARIMA(0,1,1)(1,0,1)12系數CoefficienttPSARIMA(2,1,0)(1,0,1)12系數CoefficienttPar(1)----0.61-6.470.00ar(2)----0.23-2.480.01sar(12)0.8633.280.000.8939.450.00ma(1)-0.40-5.480.00---sma(12)-0.87-28.120.00-0.90-39.070.00

表2 2組SARIMA模型的擬合優度比較
2.4 預測 采用SARIMA(2,1,0)(1,0,1)12模型回代靜態預測評估,擬合結果顯示,預測值和實際值吻合較好,2004年1月-2005年3月的實際值經差分已去除(圖1),2005年4月-2014年8月相對誤差均數為0.34%,取絕對值后相對誤差均數為13.06%。2014年1-8月預測值的相對誤差較小(5月份除外),預測標準誤(S.E)較小(表3)。2014年9月-2015年12月進行動態預測,S.E逐漸較大(圖1),2014年9-12月的S.E和95%CI上下限見表3。
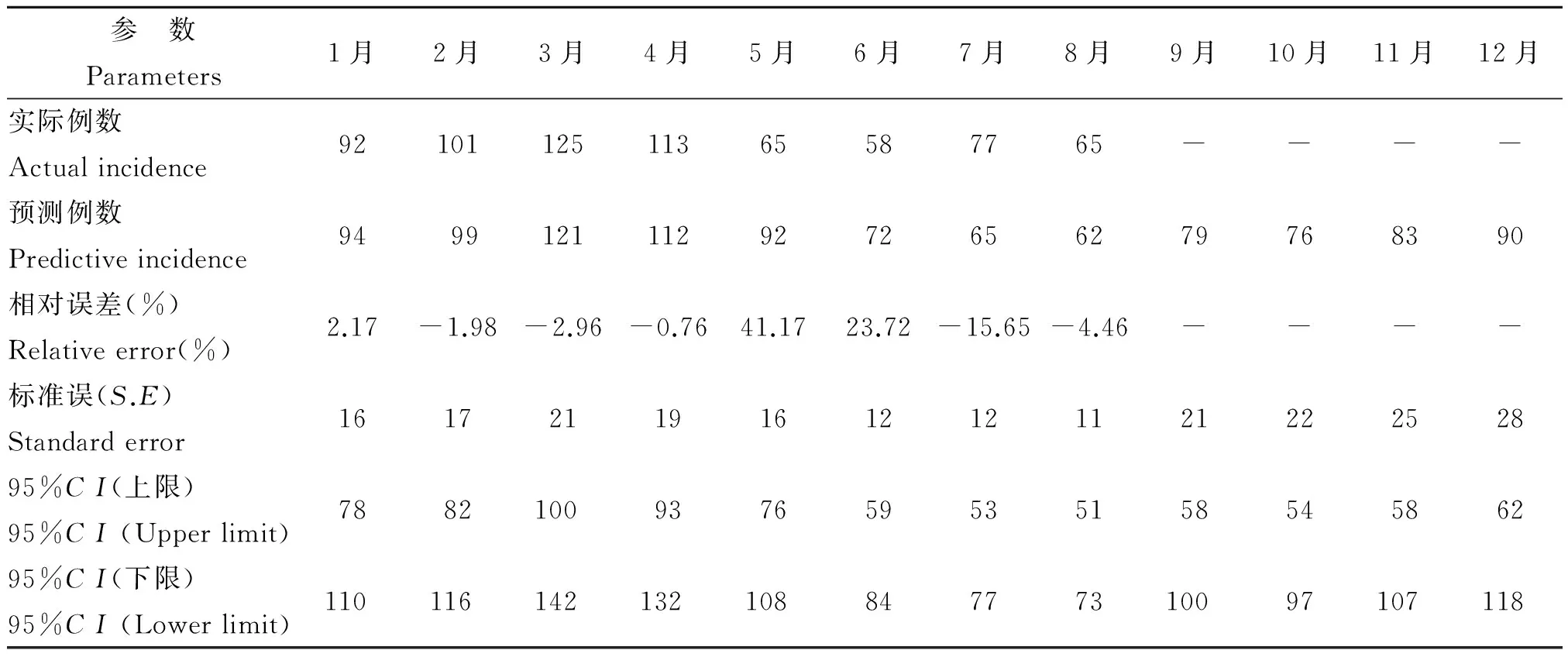
表3 福建省2014年戊肝預測情況
3 討 論
戊肝傳播途徑多樣,其中以糞-口途徑傳播為主,而且有證據顯示通過攝食受感染的肉類可以引起人獸共患傳播,易引起食物或水源性流行暴發,另外,普通人群戊肝病毒的感染率較高,提示亞臨床感染的存在,也是無癥狀感染、重癥肝炎、不明原因肝炎以及暴發性急性肝炎的重要原因之一[6-7],然而,目前該病尚缺乏特效的治療方法,也沒有特異性免疫制劑可供預防,而福建省省戊肝發病總體呈上升態勢。因此,加強戊肝監測資料分析并予以預測預警以嚴防流行和暴發顯得尤為重要。
但傳染病的暴發流行受到多種不確定因素的影響,這使得在傳染病早期的預測預警上存在著諸多困難,導致了傳染病早期預防控制工作一直較為滯后,因此如何及時有效地預測預警傳染病的暴發流行一直是傳染病預測控制工作的重點[8-10]。
應用模型對傳染病的發病及流行強度進行預測預警有多種方法:灰色動態模型GM(1,1)將原始序列累加、求均值而生成新的數列,使得GM(1,1)模型預測精度降低;利用人工神經網絡(ANN)模型預測發病率,其難度在于確定網絡結構,隱含層節點數太少,預測精度無法保證,節點數太多,又易陷入局部極小值,因此如何選擇一個最佳的網絡結構,成為一個關鍵問題;傳統的時間序列模型要求序列具有平穩的線性趨勢,但許多傳染病的發病情況有著明顯的季節性和周期性,如果不考慮這些因素的影響,做出的預測往往不精確[11-12]。而SARIMA模型適用于多種復雜的時間序列模式,可將擬合誤差作為重要因素納入模型中,因此預測精度較高,作為疾病風險評估、預防和控制的監測數據,這對于規劃公共衛生干預措施是一個非常有價值的工具[13-14]。
福建省戊肝月發病序列總體呈現明顯的周期性波動(圖1),而且自相關函數呈指數緩慢衰減,為非平穩性,因此該序列比較適合建立SARIMA模型定量預測月(周)發病數(率),可以為傳染病風險評估提供基礎數據。
分析顯示,SARIMA(2,1,0)(1,0,1)12模型擬合效果較好,回代預測值和實際值相當吻合(平均誤差為13.06%),對2014年的預測標準誤較低,表明該模型預測較為準確、精度較高,可以認為它對于原序列是個理想的模型。但是也要注意到,圖1中紫色和綠色間的2014-2015年預測置信區間逐漸變寬,從而表明預測期越往后,模型的預測精度越差,因此比較適用于短期預測。在實際應用中,應該不斷加入新的實際值,然后修正模型和重新擬合預測值[15],所以對于2015年,在2014年的實際值納入序列矯正之后再做以預測,結果才能更可靠。
值得一提的是,除5月份外,2014年1-8月的相對誤差較小,是較為成功的模型預測。至于5月份,其實際值偏低,且低于預測值的95%CI下限,同時大幅低于2011-2013年同期實際值(113、104和124例),需要從網絡直報、自然環境因素和防控力度的加強等多方面因素考慮。另外,本研究顯示,2014年2-4月和7、8月實際值均不高于95%CI上限預警值,實際情況也無流行和暴發,與預測基本一致。
但也應注意到,由于SARIMA模型也有不足之處,一是建立合理的模型需要大量的觀測數據(至少50個樣本);二是該模型是基于正態假設,對于罕見病,即便是對數或平方根轉換后也不是十分合適。所以對于小樣本,可以結合泊松回歸模型預測,但對于大樣本,前者的預測精確度高于后者[16]。
[1]Lin J, Norder H, Uhlhorn H, et al. Novel hepatitis E like virus found in Swedish moose[J]. J Gen Virol, 2014, 95(Pt 3): 557-570. DOI: 10.1099/vir.0.059238-0
[2]Kuniholm MH, Purcell RH, Mc Quillan GM, et al. Epidemiology of hepatitis E virus in the United States: results from the Third National Health and Nutrition Examination Survey, 1988-1994[J]. J Infect Dis, 2009, 200(1): 48-56. DOI: 10.1086/599319
[3]Wei S, Lu YH, Gao MY, et al. Time series analysis of hepatitis E incidence in China[J]. Chin J Health Statistics, 2012, 29(6): 808-811. (in Chinese) 魏珊, 陸一涵, 高眉揚, 等. 我國戊型肝炎發病例數的時間序列分析[J]. 中國衛生統計, 2012, 29(6): 808-811.
[4]Moosazadeh M, Nasehi M, Bahrampour A, et al. Forecasting tuberculosis incidence in Iran using Box-Jenkins models[J]. Iran Red Crescent Med J, 2014, 16(5): 1-6. DOI: 10.5812/ircmj.11779
[5]Kam HJ, Sung JO, Park RW. Prediction of daily patient numbers for a regional emergency medical center using time series analysis[J]. Healthc Inform Res, 2010, 16(3): 158-165. DOI: 10.4258/hir.2010.16.3.158
[6]Wang HR, Yan YS, Xiao JX, et al. Seroepidemiological investigation and analysis of hepatitis virus infection among various groups of population in Fujian province[J]. Chin J Zoonoses, 2007, 23(4): 370-372. (in Chinese) 王惠榕, 嚴延生, 蕭劍雄, 等. 福建省不同人群中戊型肝炎病毒感染的血清流行病學調查分析[J]. 中國人獸共患病學報, 2007, 23(4): 370-372
[7]Arends JE, Ghisetti V, Irving W, et al. Hepatitis E: An emerging infection in high income countries[J].J Clin Virol, 2014, 59(2): 81-88. DOI: 10.1016/j.jcv.2013.11.013
[8]Yang Z, Ye ZH, You AG, et al. Application of multiple seasonal ARIMA model in prediction of tuberculosis incidence[J]. Chin J Public Health, 2013, 29(4): 469-473. (in Chinese) 楊召, 葉中輝, 尤愛國, 等. 乘積季節ARIMA模型在結核病發病率預測中的應用[J]. 中國公共衛生, 2013, 29(4): 469-473.
[9]Song Q. On the weight convergence of Elman networks[J]. IEEE Trans Neural Netw, 2010, 21(3): 463-480. DOI: 10.1109/TNN.2009.2039226
[10]Lai SJ, Li ZJ, Jin LM, et al. Evaluation content and their indicators of early warning system for infectious disease outbreak[J]. Chin J Epidemiol, 2009, 30(6): 637-641. (in Chinese) 賴圣杰, 李中杰, 金連梅, 等. 傳染病暴發早期預警系統評價內容及其指標[J]. 中華流行病學雜志, 2009, 30(6): 637-641.
[11]Bras AL, Gomes D, Filipe PA, et al. Trends, seasonality and forecasts of pulmonary tuberculosis in Portugal[J]. Int J Tuberc Lung Dis, 2014, 18(10): 1202-1210. DOI: 10.5588/ijtld.14.0158
[12]Shen TQ, Liu WD, Hu JL, et al. The application of x-11-ARIMA process in dysentery prediction[J]. Chin J Health Statistics, 2014, 31(3): 395-398. (in Chinese) 申銅倩, 劉文東, 胡建立, 等. X-11-ARIMA過程在痢疾疫情預測中的應用研究[J]. 中國衛生統計,2014, 31(3): 395-398.
[13]An SY, Zhao Z, Guo JQ, et al. Forecasting measles epidemic situation by applying the time series model in Liaoning Province[J]. Chin J Health Statistics, 2014, 31(5): 781-783. (in Chinese) 安淑一, 趙卓, 郭軍巧, 等. 應用時間序列模型預測遼寧省麻疹疫情[J]. 中國衛生統計, 2014, 31(5): 781-783.
[14]Feng HF, Duan GC, Zhang RG, et al. Time series analysis of hand-foot-mouth disease hospitalization in Zhengzhou: establishment of forecasting models using climate variables as predictors[J]. PLoS One, 2014, 9(1): 1-10. DOI: 10.1371/journal.pone.0087916
[15]Mehdi K, Mehdi B, Heiazi SR. Combining seasonal ARIMA models with computational intelligence techniques for time series forecasting[J]. Soft Comput, 2012, 16(6): 1091-1105. DOI: 10.1007/s00500 -012-0805-9
[16]Hu WB, Tong SL, Mengersen K, et al. Weather variability and the incidence of cryptosporidiosis: comparison of time series poisson regression and SARIMA models[J]. Elsevier Inc, 2007, 17(9): 679-688. DOI: 10.1016/j.annepidem.2007.03.020
SARIMA model for incidence trend and prediction of hepatitis E
ZHU Han-song1,HUANG Wen-long1,XIE Zhong-hang,CHEN Guang-min,CAO Yang2,ZHANG Can-ming1,CHEN Wu1,OU Jian-ming1,HONG Rong-tao1
(1.FujianProvincialCenterforDiseaseControl&Prevention,Fuzhou350001,China;2.ChineseCenterforDiseaseControlandPrevention,Bejing102206,China)
We forecasted the incidence trends of hepatitis E in Fujian Province by SARIMA model, in order to provide quantitative data for early warning and risk assessment. The monthly cases of hepatitis E in Fujian Province from January 2004 to December 2015 were analyzed for SARIMA using Eviews 5.0 software. Results showed that monthly incidence sequences of hepatitis E in Fujian Province, from January 2004 to August 2014, showed a trend of increased and then decreased, as well as cyclical fluctuations. And it was into smoothed after done natural logarithm and once non-seasonal of lag 1 difference. SARIMA (0,1,1)(1,0,1)12and SARIMA(2,1,0) (1,0,1)12were statistically significant,and the residual was white noise, in which the latter was the optimal model, expressed as: (1+0.61L+0.23L2)(1-0.89L12)(1-L)log(yt)=(1- 0.89L12)εt. Static back generation forecast and actual values were in good agreement, in which the mean of relative error was 13.06% after taking the absolute value. Relative error of predictive value in 2014 January to August (except in May) was small, and the prediction standard error (S.E) small, too. Incidence trends of hepatitis E could short-term predicted through SARIMA model, thus it could provide reliable data base in order to judge the risk of infectious disease more timely and scientifically.
SARIMA; hepatitis E; warning; forecast; risk assessment
Horg Rong-tao, Email:hrt@fjcdc.com.cn
洪榮濤,Email:hrt@fjcdc.com.cn
1.福建省疾病預防控制中心,福建省人獸共患病研究重點實驗室,福州 350001; 2.中國疾病預防控制中心,應急中心監測預警與風險評估辦公室,北京 102206
10.3969/cjz.j.issn.1002-2694.2015.02.014
R373
A
1002-2694(2015)02-0158-05
2014-09-12;
2014-11-23


