基于核糖體RNA ITS1-5.8S-ITS2基因序列的十二指腸鉤蟲分子系統發育分析
汪家旭,蘇成豪,黃建煒 ,葉 曦,李國偉
基于核糖體RNA ITS1-5.8S-ITS2基因序列的十二指腸鉤蟲分子系統發育分析
汪家旭1,蘇成豪1,黃建煒1,葉 曦2,李國偉2
目的 通過克隆十二指腸鉤蟲的ITS1-5.8S-ITS2,初步分析鉤口屬的系統發育,構建基于ITS1、ITS2的圓線目線蟲的系統進化樹,為進一步研究其遺傳進化關系奠定基礎。方法 從廈門海滄東孚鎮收集標本,分離,形態鑒定為十二指腸鉤蟲,分別克隆了供試鉤蟲的ITS1-5.8S-ITS2序列,經NCBI網站的BLAST比對以及基于ITS1和 ITS2序列的鉤口屬的系統發育分析。結果 供試鉤蟲的ITS1、5.8S和ITS2序列,它們的長度分別為366 bp、153 bp和221 bp,登陸http://www.ncbi.nlm.nih.gov進行BLAST,在數據庫中沒有與之相匹配的序列,將獲得的序列作為新的序列上傳至GenBank中進行注冊,各序列的GenBank登錄號分別為:5.8S rRNA基因:EU344796、ITS1-5.8S-ITS2: EU344797。結論 從系統進化樹中,本實驗的供試十二指腸鉤蟲均與GenBank中已公布的十二指腸鉤口線蟲自然聚類在一起。
十二指腸鉤蟲;ITS1-5.8S-ITS2基因; 系統發育分析
Supported by the Key Construction Project Fund of the Xiamen Medical Center (No. [2012]285)
真核生物細胞核的核糖體RNA基因簇在不同的真核生物中存在于不同的染色體上[1],真核生物細胞核的核糖體RNA基因簇中的串聯重復序列,在進化上表現出相當的一致性,并且在種內的獨特性序列的同質性相對較高,但其它非獨特性序列仍有一定的差異性[2]。本文試驗標本源自廈門市海滄區東孚鎮純十二指腸鉤蟲感染者,首先克隆分離株5. 8 SrDNA基因、ITS1和ITS2的完整序列;然后,利用ITS1和ITS2的序列進行蟲種的分子鑒定以及分析鉤口屬常見鉤蟲的系統發育關系,結果報告如下。
1 材料與方法
1.1 供試蟲種 結合人體腸道寄生蟲的年度常規糞檢普查,收集海滄區東孚鎮洪塘村鉤蟲感染患者糞便,按常規“T”試管濾紙培養法,于27 ℃恒溫箱,獲得試驗所需鉤蚴。經幼蟲形態學鑒定為純十二指腸感染患者。收集的十二指腸鉤蚴用PBS或生理鹽水(pH 7.3)洗滌3遍, 無水乙醇洗滌3遍,-20 ℃或-70 ℃保存備用。
1.2 細菌菌株及質粒 大腸桿菌(Escherichiacoli)DH5α菌株由本室保存,TA克隆載體pMD18-T及其試劑盒購自TAKALA公司。
1.3 引物 參照文獻[3]設計擴增鉤蟲18S rRNA基因的引物:NC18SF1 5’-AAAGATT A GCCATGCA-3’ (forward)和NC5BR 5’-GCAGGTTCACCTACAGAT-3’ (reverse)。參照文獻[4]設計擴增鉤蟲ITS1-5.8S-ITS2基因的引物:NC16 5’-AGTTCAAT CGCAATGGCTT-3’ (forward)和NC2 5’-TTAGTTTCTTTTCCTCCGCT-3’ (reverse) 。用于菌落PCR篩選的引物為TA克隆載體pMD18-T的通用引物M13-47:5’-CGCCAGGGTTTTCCCAGTCACGAC-3’(forward)和RV-M:5’-GAGCGGA TAACAATT TCACACAGG-3’(reverse)。測序引物為M13-47和RV-M。
1.4 鉤蟲基因組DNA粗提物的制備 鉤蟲基因組DNA粗提物的制備參照Subbotin等[5]的方法并稍作修改。
1.5 鉤蟲ITS1-5.8S-ITS2基因的克隆 PCR擴增鉤蟲ITS1-5.8S-ITS2基因的引物為NC16和NC2,模板為上述鉤蟲基因組DNA粗提物。PCR的反應條件均為:95 ℃預變性5 min;95 ℃變性30 s,55 ℃退火45 s,72 ℃延伸1 min,循環數30;72 ℃延伸10 min。PCR產物進行1%瓊脂糖凝膠電泳鑒定。
1.6 PCR擴增產物 PCR擴增產物按Gel extraction kit的操作指南進行純化、連接、轉化、菌落PCR篩選等。菌落PCR反應為陽性的克隆,加適量含30%甘油和Amp(100 μg/mL)的LB培養液,于-70 ℃冷凍保藏。選取陽性克隆2個,送上海英俊公司測序。
1.7 基于ITS區的鉤蟲種類的分子鑒定 測序后得到的供試鉤蟲樣品的ITS1-5.8S-ITS2序列,通過NCBI網站的nucleotide blast程序進行比對分析,根據最高同源性原則,分別確定供試鉤蟲樣品的ITS1、5.8S和ITS2序列;然后,用生物軟件Clustalx 1.8對供試鉤蟲的ITS1和ITS2序列與從GenBank中搜尋的其他鉤口屬線蟲的ITS1和ITS2序列進行比對分析;最后,通過生物軟件MEGA 3.1[6]分別構建基于ITS1和ITS2序列的鉤口屬線蟲的系統進化樹,并根據與供試鉤蟲親緣關系最近GenBank中已公布的鉤蟲種類來判斷供試鉤蟲樣品的種類。
1.8 圓線目的系統發育分析 分別從GenBank中搜尋圓線目線蟲的ITS1、ITS2序列,分別編輯為適合生物軟件Clustalx 1.8和MEGA 3.1分析的文件形式。先用Clustalx 1.8軟件進行比對分析,然后,用MEGA 3.1軟件分別構建基于ITS1、ITS2的圓線目線蟲的系統進化樹。
2 結 果
2.1 鉤蟲 ITS1-5.8S-ITS2基因的PCR擴增 以NC16和NC2為引物,以基因組DNA粗提物為模板,PCR擴增鉤蟲的ITS1-5.8S-ITS2基因。如圖1所示,在約900 bp處均有一條特異的擴增條帶,與預期的片段大小相符合。
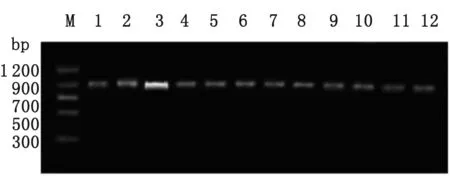
M: DNA marker.
圖1 鉤蟲ITS1-5.8S-ITS2基因的PCR擴增
Fig.1 PCR amplification of ITS1-5.8S-ITS2 gene of hookworm
2.2 菌落PCR篩選與測序分析 ITS1-5.8S-ITS2基因的PCR擴增產物與pMD18-T的連接產物轉化大腸桿菌DH5α,經藍白斑篩選后,先分別挑取9個白色菌落于LB培養液中進行培養;然后,分別制備用于菌落PCR的模板;最后,用pMD18-T載體的通用引物M13-47和RV-M,進行菌落PCR篩選。從圖2可以看出,在ITS1-5.8S-ITS2重組子的菌落PCR篩選中,除了5和7號克隆為陰性外,其余克隆均為陽性,將其中的1和3號送上海英俊公司測序,測序引物為M13-47。
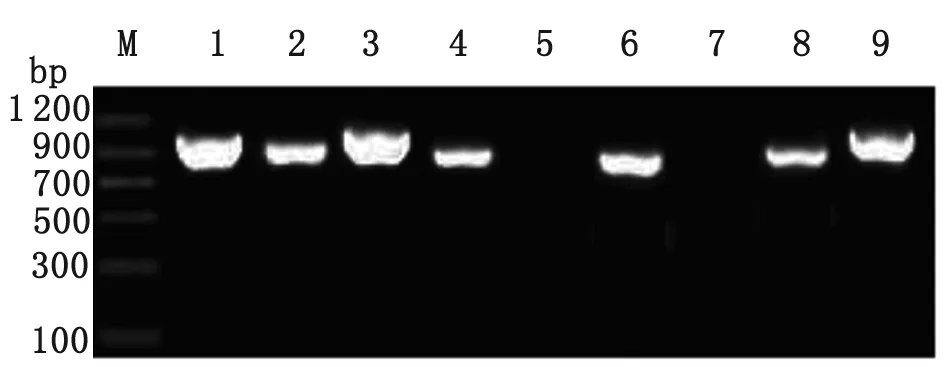
M: DNA marker;PCR products of bacterium colony 1-9, respectively.
圖2 ITS1-5.8S-ITS2重組子的菌落PCR篩選
Fig.2 Screening of ITS1-5.8S-ITS2 recombinants by bacteria colonies PCR
測序獲得的序列全長836 bp,通過NCBI網站的nucleotide blast程序進行比對分析,根據最高同源性原則,分別確定供試鉤蟲樣品的ITS1、5.8S和ITS2序列,結果見圖3。從圖3可以看出,克隆到的供試鉤蟲的ITS1和ITS2的序列與已公布的十二指腸鉤口線蟲的同源性分別為99%和100%。
根據比對結果,本研究克隆到的ITS1-5.8S-ITS2序列的結構組成如圖4所示,從圖中可以看出,克隆到的供試鉤蟲的5.8S rDNA、ITS1和ITS2的長度分別為153 bp、366 bp和221 bp。

圖3 供試鉤蟲ITS1和ITS2序列的BLAST比對結果

The sequences of ITS1, 5.8S rRNA and ITS2 were indicated, respectively.
2.3 用于鉤蟲種類分子鑒定的ITS1和ITS2序列 運用NCBI網站的CoreNucleotide程序,從GenBank+EMBL+DDBJ+PDB數據庫中分別搜尋到已公布的5種鉤口屬線蟲的ITS1和ITS2序列,同時以1種板口屬線蟲作為群外種(見表1)。

表1 ITS1和ITS2序列被用于分子鑒定的鉤蟲種類
2.4 鉤蟲種類的分子鑒定與鉤口屬系統發育分析 先用生物軟件Clustalx 1.8對供試鉤蟲的ITS1和ITS2序列與從數據庫中搜尋到的其他鉤蟲的ITS1和ITS2序列進行多序列比對分析。然后,通過生物軟件MEGA 3.1,采用鄰接法分別構建基于ITS1和ITS2序列的鉤口屬線蟲的系統進化樹。其中,采用的取代模型為Nucleotide:p-distance,間隙區采用完全刪除的方式,并以美洲板口線蟲線蟲作為群外種。最后通過自舉分析進行置信度檢測,自舉數據集為1 000次。
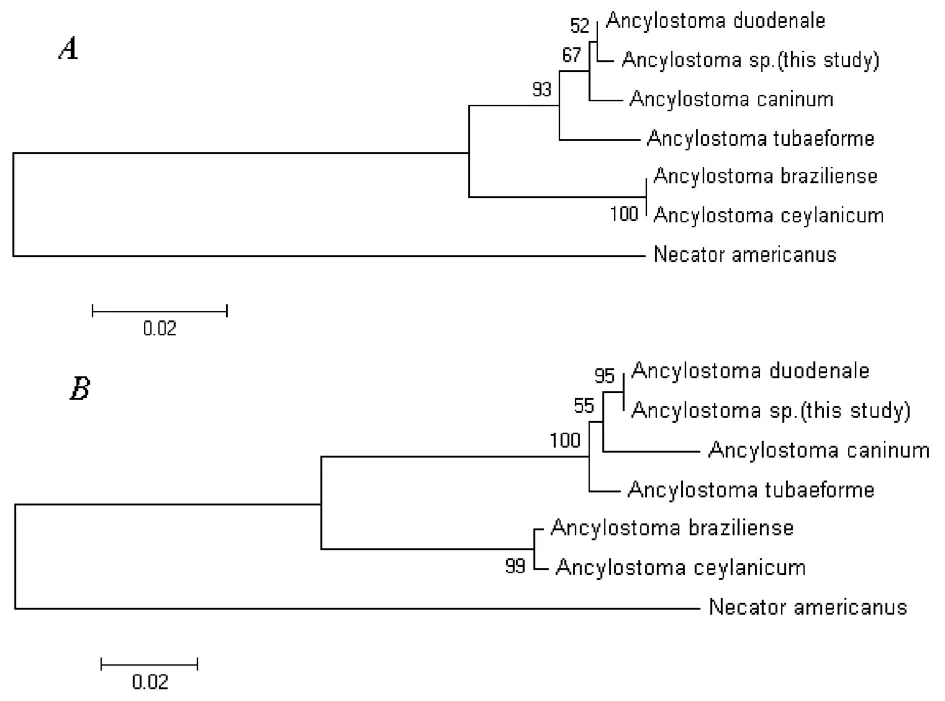
A , B: The phylogenetic relationships of the genusAncylostomainferred from ITS1 and ITS2 sequences, respectively.
圖5 鉤蟲種類的分子鑒定與鉤口屬系統發育分析
Fig.5 Molecular identification of species of hookworm and phylogenetic analysis of Genus:Ancylostoma
基于ITS1和ITS2序列的鉤口屬線蟲的系統進化樹,分別如圖5 A和B所示。在兩支系統進化樹中,本實驗的供試鉤蟲均與GenBank中已公布的十二指腸鉤口線蟲自然聚類在一起,因此,可以判定供試鉤蟲為十二指腸鉤口線蟲。
3 討 論
3.1 供試鉤蟲的ITS1-5.8S-ITS2克隆 本研究利用擴增線蟲rDNA的通用引物,克隆了供試鉤蟲的ITS1-5.8S-ITS2。將獲得的序列在GenBank中進行注冊,各序列的GenBank登錄號分別為:5.8S rRNA基因為EU344796、ITS1-5.8S-ITS2 為EU344797。
由于實驗過程中,鉤蟲蟲體無法大量獲得,因此不能按常規的酚-氯仿抽提法提取供試鉤蟲的基因組DNA。作者采用的做法是制備微量的鉤蟲基因組DNA粗提物,即將少量的鉤蟲幼蟲蟲體撕裂成數斷;轉入200 μL的Eppendorf 管中,加入線蟲裂解緩沖液,利用液氮罐,將蟲體反復凍融使蟲體破裂;最后通過蛋白酶 K 的消化作用,使鉤蟲基因組DNA釋放出來;經離心,上清即為鉤蟲基因組DNA粗提物。實驗結果證實,上述方法可以獲得符合要求的鉤蟲的基因組DNA。
克隆到的ITS1-5.8S-ITS2序列全長836bp,通過NCBI網站的nucleotide blast程序進行比對分析,結果表明,供試鉤蟲的ITS1和ITS2的序列與分別為99%和100%(圖3)。ITS1序列與已公布的十二指腸鉤口線蟲的同源性沒有達到100%,可能是不同地理株系之間在ITS1區具有遺傳多樣性。根據最高同源性原則,分別確定供試鉤蟲的ITS1、5.8S和ITS2序列,它們的長度分別為366 bp、153 bp和221 bp(圖4)。
3.2 基于ITS1和ITS2序列的鉤蟲種類的分子鑒定與鉤口屬系統發育分析 分別以ITS1和ITS2序列對5種鉤口屬線蟲進行屬內系統發育分析,同時以1種板口屬線蟲作為群外種。從兩支系統進化樹中(圖5A和B)可以看出,本實驗的供試鉤蟲均與GenBank中已公布的十二指腸鉤口線蟲自然聚類在一起,因此,可以判定供試鉤蟲為十二指腸鉤口線蟲。
在基于ITS1和ITS2序列所構建的2個系統進化樹中,鉤口屬的5種線蟲被聚類為2個姐妹群,其中,十二指腸鉤口線蟲、犬鉤口線蟲和貓鉤口線蟲組成1個類群,巴西鉤口線蟲和錫蘭鉤口線蟲組成另1個類群。這些結果說明,十二指腸鉤口線蟲與犬鉤口線蟲的親緣關系最近,其次是貓鉤口線蟲;而巴西鉤口線蟲和錫蘭鉤口線蟲的親緣關系最近。
[1]Indik ZK, Tartof KD. Long spacers among ribosomal genes ofDrosophilamelanogaster[J]. Nature, 1980, 284(5755): 477-479.
[2]Hills DM, Dixon MT. Ribosomal DNA: molecular evolution and phylogenetic inference[J]. Quarterly Rev Biol, 1991, 66(4): 411-453.
[3]Chilton NB, Huby-Chilton F, Gasser RB. The evolutionary origins of nematodes within the order Strongylida are related to predilection sites within hosts[J]. Mol Phylogenetics Evol, 2006, 40(1): 118-128.
[4]Chilton NB, Huby-Chilton F, Gasser RB. First complete large subunit ribosomal RNA sequence and secondary structure for a parasitic nematode: phylogenetic and diagnostic implications[J]. Mole Cell Probes, 2003, 17(1): 33-39.
[5]Subbotin SA, Vierstraete A, De Ley P. Phylogenetic relationships within the cyst- forming nematodes (Nematoda: Heteroderidae) based on analysis of sequences from the ITS Regions of ribosomal DNA[J]. Mol Phylogenetics Evol, 2001, 21(1): 1-16.
[6]Kumar S, Tamura K, Nei M. MEGA3: Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment[J]. Brief Bioinformatics, 2004, 5: 150-163.
[7]Hotez PJ, Hawdon J, Schad GA. Hookworm larval amphiparatenesis: theCaenorhabditiselegansDaf-c paradigm[J]. Parasitol Today, 1993, 9: 23-26.
[8]Hawdon JM, Jones BF, Homan DR, et al. Cloning and characterization ofAncylostoma-secreted protein. A novel protein associated with the transition to parasitism by infective hookworm larvae[J]. Biolog Chem, 1996, 22: 6672-6678.
[9]Hawdon JM, Datu B. The second messenger cyclic GMP mediates activation inAncylostomacaninuminfective larvae[J]. Int Parasitol, 2003, 33: 787-793.
[10]Zhan B, Liu Y, Badamchian M, et al. Molecular characterization of theAncylostoma-secreted protein (ASP) family from the adult stage ofAncylostomacaninum[J]. Int Parasitol, 2003, 33: 897-907.
Molecular phylogenetic analysis ofAncylostomaduodenalesbased on ITS1-5.8SrRNA-ITS2 gene sequence
WANG Jia-xu1,SU Cheng-hao1,HUANG Jian-wei1,YE Xi2,LI Guo-wei2
(1.XiamenCenterforDiseaseControlandPrevention,Xiamen361021,China;2.CenterforDiseaseControlandPreventionofHaicangCity,Haicang361026,China)
ITS1-5.8S-ITS2 genes ofAncylostomaduodenaleswas cloned and sequenced in this study. We collected specimens from Dongfu Town, Haicang District, Xiamen City after morphological identification forAncylostomaduodenale. Hookworm ITS1-5.8S-ITS2 sequence was tested, and the length of ITS1, 5.8S and ITS2 sequences were 366 bp, 153 bp and 221 bp, respectively. The genes of 5.8S rRNA and ITS1-5.8S-ITS2 as new sequences were registered in GenBank, and their number was EU344796 and EU344797, respectively. Based on ITS1 and ITS2 sequence and phylogenetic analysis ofAncylostoma, results have further confirmed that above materials wasAncylostomaduodenales, and the molecular level were accordance well with that of morphological identification.
Ancylostomaduodenale; ITS1-5.8S-ITS2 gene; phylogenetic analysis
1.廈門市疾病預防控制中心,廈門 361021; 2.廈門市海滄區疾病預防控制中心,廈門 361026
10.3969/cjz.j.issn.1002-2694.2015.07.010
R383.1
A
1002-2694(2015)07-0640-05
2014-06-10;
2015-03-30
廈門市醫學中心/重點專科建設項目基金(廈衛科教中醫[2012]285號)

