貝酵母耐硫相關基因FZF1的克隆與分析
賀 笑,張先昂,劉小珍,李升星,張漢堯
(西南林業大學云南省高校林木遺傳改良與繁育重點實驗室,云南昆明 650224)
貝酵母耐硫相關基因FZF1的克隆與分析
賀 笑,張先昂,劉小珍,李升星,張漢堯*
(西南林業大學云南省高校林木遺傳改良與繁育重點實驗室,云南昆明 650224)
FZF1基因在釀酒酵母中對亞硫酸鹽轉運蛋白編碼基因SSU1具有正向調節作用,但在貝酵母中尚無相關報道,本研究擬通過對該基因進行克隆和生物信息學分析為后續研究做出參考。首先對貝酵母FZF1基因進行克隆,并利用在線分析工具ProtParam、ProtScale、TMHMM、PredictProtein、Swiss-Model等軟件對其編碼蛋白質的基本理化性質進行分析,同時預測了該基因所編碼蛋白質的二級結構和三級結構。結果表明:該核苷酸序列含有一個長900 bp的開放閱讀框,可編碼299個氨基酸;編碼的蛋白質為在細胞核中行使調控功能的親水蛋白,含有18個絲氨酸(S)激酶潛在磷酸化位點、一個Coil區和4個鋅指結構域,與釀酒酵母FZF1基因所編碼的蛋白質結構和性質極為相似。可初步認為貝酵母FZF1基因與細胞的耐硫性相關;而貝酵母FZF1基因所編碼的蛋白質中僅有4個鋅指,則可能是貝酵母硫耐受能力比釀酒酵母弱的重要原因。
貝酵母,FZF1,序列分析,耐硫基因
葡萄酒生產過程中,需要多種抗氧化劑的作用,抑制細菌的生長,從而保證葡萄酒的品質與風味,亞硫酸作為最常見的一種抗氧化劑,廣泛用于釀酒工藝[1-2]。然而,過高濃度的亞硫酸會對細胞產生不同程度的毒害作用[3]。亞硫酸鹽會直接破壞細胞的組織結構,它能與膜受體結合,阻礙細胞膜正常活動[4],導致細胞內容物外流[5];與ATP酶結合,影響細胞中ATP的合成[6];與葡萄糖、丙酮酸等代謝產物結合,阻礙細胞的正常代謝活動[7-8]。所以在釀酒工藝中,亞硫酸鹽的耐受能力也成為篩選釀酒菌株的重要指標之一。
釀酒酵母中,SSU1編碼的蛋白SSU1p可以調節細胞的硫代謝[9-10],而在硫代謝過程中,酵母菌會產生亞硫酸鹽,這是一種具有潛在毒性的中間代謝產物[11-12],與耐硫相關的基因SSU1(Sulfite sensitivity protein,SSU1)在細胞調節中可作為“硫泵”,將對細胞具有毒害作用的亞硫酸鹽泵出細胞外,在保證細胞正常生命活動的同時,也形成了釀酒過程中酒品的特殊風味,從而提高了葡萄酒的品質。而與釀酒酵母耐硫相關的另一個基因FZF1(Five zine finger,FZF1)在“泵硫”過程中也發揮至關重要的作用。FZF1被發現在SSU1的轉錄過程中起著正向調節的作用。即在釀酒酵母硫代謝過程中,SSU1基因編碼的亞硫酸鹽轉運蛋白會調節酵母細胞的亞硫酸鹽代謝,過量的亞硫酸鹽可以通過亞硫酸鹽轉運蛋白運輸到細胞外,而正向調節基因FZF1可加強細胞內亞硫酸鹽的輸出量[13],從而提高酵母的硫耐受性,進而也可提高釀造葡萄酒中芳香物質硫醇的含量,最終達到提高葡萄酒品質的目的。

表1 進行蛋白質分析的軟件、網址及內容
貝酵母與釀酒酵母同屬于狹義釀酒酵母屬,二者在各性狀上都極為相似,但在“長相思”等酒種釀造工藝后期,貝酵母在無氧呼吸產生酒精方面具有更為重要的作用[14-16]。在貝酵母中,張太奎等[17]對耐硫相關基因SSU1進行了初步的分析。作為亞硫酸鹽轉運蛋白編碼基因SSU1轉錄過程中的正向調節基因FZF1,更具有分析研究的重要意義。
本實驗通過對貝酵母FZF1基因進行克隆,測序分析,再借助多種在線分析工具對該基因的結構和功能進行了詳細的分析及預測,從而探討貝酵母的FZF1基因在“泵硫”的過程中是否具有釀酒酵母FZF1基因對SSU1基因轉錄進行正向調節的相同作用。為后期進一步研究貝酵母硫代謝的詳細途徑奠定了基礎。
1 材料與方法
1.1 材料與儀器
菌株 香格里拉縣奔子欄鄉葡萄分離得到的貝酵母菌株BZL5;酵母膏、蛋白胨、瓊脂、葡萄糖、TIANamp Yeast DNA Kit、RNase A、Lyticase、山梨醇 TIANGEN公司;Loading Buffer、核酸染料 Bioteke Corporation;DNA Marker Ⅶ 莊盟生物有限公司;100 bp Ladder DNA Marker Biomed公司;Taq 酶(5000U)、dNTP(10 mM) NEB北京分公司;引物 生工生物工程(上海)股份有限公司。
DK98-1數顯恒溫水浴 天津泰斯特儀器有限公司;Centrifuge 5810R離心機 德國Eppendorf公司;PYX-DHG-9023-A干燥箱 上海新諾儀器設備有限公司;TP650 PCR儀 日本TaKaRa公司;Pico17 小型離心機 美國Thermo公司;DYY-10C電泳儀 北京百晶生物技術有限公司;GBOXHR 凝膠成像分析系統 美國Syngene公司;溫控搖床 德國Sartorius;YXQ-LS-75SII滅菌鍋 上海博訊實業有限公司;SW-CJ-2FD超凈工作臺 蘇州凈化工程公司;移液槍 德國Eppendorf公司。
1.2 實驗方法
1.2.1 貝酵母基因組DNA的獲得 利用酵母基因組DNA提取試劑盒提取高質量的樣品DNA。用瓊脂糖凝膠電泳檢測。
1.2.2 貝酵母FZF1基因的擴增 利用Primer Premier5,確定了上游引物Primer FZF1L1:5′ TACGGGTTGACCACTCCAAT 3′、下游引物Primer FZF1R1:5′ CTCAAGTTGGTACCACCCAA 3′。
進行PCR擴增:25 μL體系為:5 μL高質量DNA,1 μL Primer FZF1L1,1 μL Primer FZF1R1,2.5 μL 10×PCR Buffer,0.3 μL Taq酶,0.4 μL dNTP,14.8 μL ddH2O。
PCR反應程序:95 ℃預變性5 min,95 ℃變性30 s,60 ℃退火30 s,72 ℃延伸60 s,進行35個循環,72 ℃最后延伸7 min,4 ℃forever。PCR產物于-20 ℃保存。
1.2.3 貝酵母FZF1基因的測序與分析 PCR產物經過瓊脂糖凝膠電泳檢測后,委托生工生物工程(上海)股份有限公司進行測序。將測序結果提交到NCBI,利用Open Reading Fame Finder工具查找這條核苷酸序列中的開放閱讀框。預測該核苷酸序列所翻譯的蛋白質,然后利用在線分析工具對所預測蛋白質的基本理化性質、親疏水性、跨膜區、信號肽、Coil區、結構域、亞細胞定位等進行分析,最后預測該蛋白質的二級結構和三級結構。具體分析內容如表1。
2 結果與分析
2.1 貝酵母基因組DNA及PCR產物的檢測
將提取獲得的貝酵母基因組DNA通過瓊脂糖凝膠電泳檢測,結果見圖1。1、2、3、4、5、6號菌株均提出質量較好的基因組DNA。

圖1 貝酵母基因組DNA電泳圖譜Fig.1 Electrophoretogram of genomic DNA from S. bayanus
確定提取得到貝酵母基因組DNA后,擴增貝酵母FZF1基因,PCR檢測結果見圖2。3號、5號菌株擴增出了FZF1基因片段。

圖2 FZF1基因擴增結果Fig.2 Amplification results of FZF1 gene

表2 貝酵母FZF1基因編碼蛋白質的氨基酸組成及親疏水性
2.2 貝酵母FZF1基因編碼蛋白質的一級結構分析
將PCR結果測序后得到一條長為1603 bp的核苷酸序列。利用NCBI中的BLAST工具進行序列比對,只有兩條巴斯德酵母的序列與之同源性比例為5%和4%,說明該序列是一條新基因序列。然后再利用Open Reading Fame Finder工具查找這條核苷酸序列中的開放閱讀框。根據六種不同的編碼方式得到的結果顯示,該基因序列中含有一個長度為900 bp的開放閱讀框,位于基因的563~1462 bp之間,可能與遺傳信息的表達密切相關。該段CDS區可以翻譯成一條含有299個氨基酸的蛋白質,繼而得到其氨基酸序列,見圖3。

圖3 貝酵母FZF1基因編碼的蛋白質序列Fig.3 The protein sequence encoded by the FZF1 gene of S.bayanus
2.2.1 理化性質分析 將氨基酸序列提交到在線分析工具ExPASy ProtParam中,對該蛋白質序列的組分及基本理化性質進行分析。結果顯示:該蛋白質的分子式為C1520H2345N431O468S13,分子質量為34561.7,理論pI值為6.80,氨基酸組成和每個氨基酸的親疏水性分值見表2。氨基酸組成上Ser有33個,所占比例為11%,Leu有32個,所占比例為10.7%,這兩種氨基酸含量相對較高,其他氨基酸比例相差不大。不穩定系數為48.05,所以該蛋白質屬于不穩定蛋白。脂肪系數為69.46,總平均親水性為-0.812,為親水蛋白。
2.2.2 疏水性分析 利用ProtScale進行蛋白質親疏水性分析,見圖4。根據20種氨基酸疏水特性的參數,較高正值的氨基酸具有較強的疏水性,而較低負值的氨基酸具有較強的親水性[18]。由圖可知,根據正負分值比例,此預測蛋白質屬于親水蛋白。ProtScale圖形輸出顯示該蛋白質最高分值峰為1.344,最低分值峰為-3.100,不存在高分值(Scare>1.5)峰,所以該蛋白質可能沒有潛在的跨膜區。在36、99、120、140、193、243、248氨基酸位點附近的分值分別為-2.578、-2.678、-2.056、-1.456、-1.778、-2.233、-2.311,這些區域屬于高親水性,可能在這些位點存在折疊情況。

圖4 貝酵母FZF1基因編碼的蛋白質的親疏水性Fig.4 Hydrophobicty of protein encoded by the FZF1 gene of S.bayanus
2.2.3 蛋白質信號肽和亞細胞定位預測 組成生物體的蛋白質大多數是在細胞質中的核糖體上合成的,各種蛋白質合成之后要分別運送到細胞中的不同部位,以保證細胞生命活動的正常進行。有些蛋白質要通過內質網膜進入到內質網腔內,最終成為分泌蛋白;有的蛋白質則需要穿過各種細胞器的膜,進入細胞器內,構成細胞器蛋白。分泌蛋白的N端都有一段約15~35個氨基酸的疏水性肽端,其功能是引導蛋白質多肽鏈穿過內質網膜進入腔內,稱為信號肽[19]。由前文蛋白質的親疏水性分析顯示,該蛋白質為水溶性蛋白,應無信號肽,SignalP 3.0 server 分析結果與預測一致。
亞細胞定位與蛋白質的功能存在著非常密切的聯系。通過TargetP在線軟件分析該蛋白質的亞細胞定位,結果如圖5(a)。該蛋白質在線粒體存在的可能性為0.136,在分泌通路存在的可能為0.064,而在其他位置的可能性為0.908,即該蛋白質很有可能存在于除線粒體和分泌通路以外的位置。進一步用SPORT軟件進行亞細胞定位分析,使用k-NN近鄰算法,結果如圖5(b),該蛋白質主要分布在細胞核中,可能性為95.7%,也有可能在線粒體中出現,可能性僅為4.3%。由此可知,該蛋白質很有可能為一種在細胞核中進行代謝調控的轉錄因子。
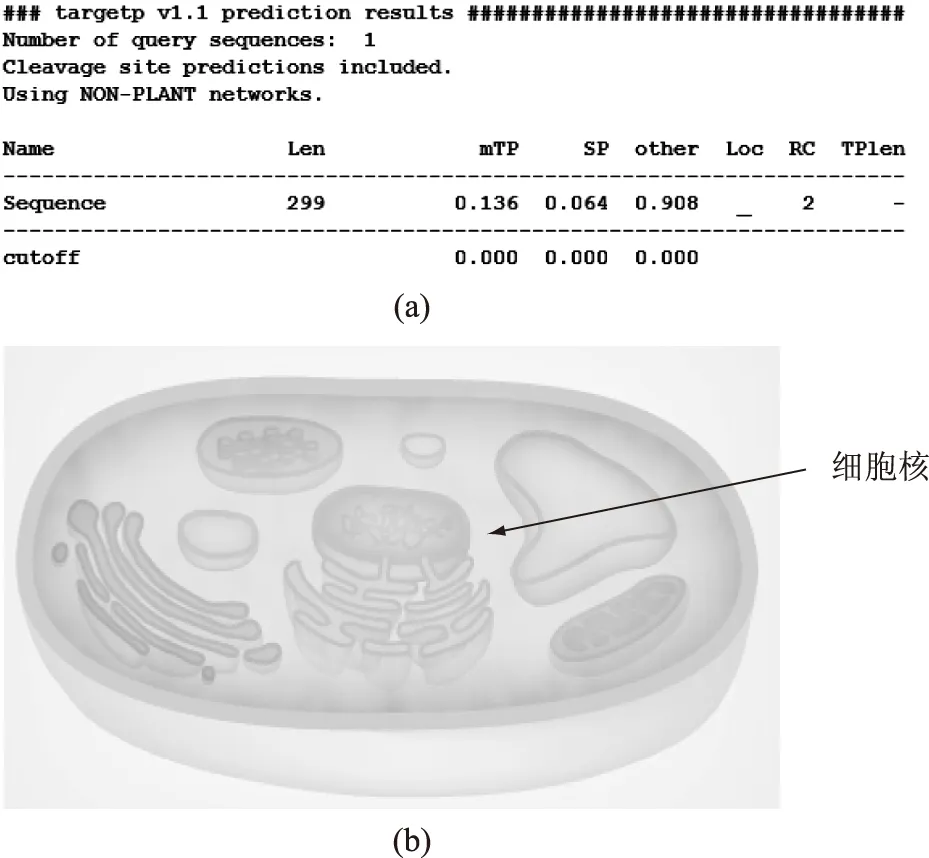
圖5 FZF1編碼蛋白質的亞細胞定位Fig.5 Subcellular localization of protein encoded by the FZF1 gene注:a圖為TargetP預測結果;b圖為PSORT預測結果。
2.2.4 激酶磷酸化修飾位點預測 氨基酸翻譯后通常會加入一些生物化學官能團,使其在化學性質或者結構上發生變化,被稱為蛋白質翻譯后修飾。其中磷酸化是將一個磷酸基團導入一個有機分子。此作用在生物化學中占有重要地位。蛋白質的磷酸化可發生在許多種類的氨基酸上,其中以絲氨酸為多,接著是蘇氨酸。而酪氨酸和組氨酸則相對較少的磷酸化發生。通過KinasePhos在線分析工具來預測此蛋白質的磷酸化位點和修飾激酶,為了減少假陽性率,當限定值為90%時,在線提交蛋白質序列,對四種蛋白質進行勾選,預測結果如表3,含有18個絲氨酸(S)激酶潛在磷酸化位點。這些激酶磷酸化位點對蛋白質修飾加工后的功能行使具有重要的意義。

表3 FZF1編碼蛋白質的激酶磷酸化位點預測
注:S指激酶磷酸化位點。
2.3 貝酵母FZF1基因編碼蛋白質的二級結構預測
2.3.1 Coil區分析 卷曲螺旋是蛋白質中由2~7條α螺旋鏈互相纏繞形成類似麻花狀結構的總稱。目前已發現的天然卷曲螺旋的主要存在形式為由2~5條α螺旋鏈相互纏繞而形成的平行或反平行同寡聚體或異寡聚體[20]。卷曲螺旋式控制蛋白質寡聚化的元件,含有卷曲結構的蛋白質主要是一些轉錄因子、骨架蛋白、動力蛋白等,在機體內執行這分子識別、代謝調控、細胞分化等生物學功能。利用COILS在線分析工具,基于Lupas算法,預測該蛋白質的卷曲螺旋,如圖6所示,該蛋白質殘基在3個不同窗口(Window 14、21、28)均顯示卷曲螺旋區域,文本數值(未列出)結果顯示,卷曲螺旋序列位于104~117區域框內。

圖6 FZF1基因編碼蛋白質的Coil區分析Fig.6 Coil area analysis of protein encoded by the FZF1 gene
2.3.2 跨膜區分析 通過TMPRED、TMHMM在線軟件分析該蛋白質的跨膜螺旋區,未發現可能的跨膜螺旋區,即該蛋白質不跨膜。通常蛋白質含有跨膜區,即可能作為膜受體、錨定蛋白或離子通道蛋白,均不溶于水,所以該預測蛋白質為水溶性蛋白。這與蛋白質的親疏水性性質一致。TMHMM預測結果如圖7,沒有跨膜區信號。
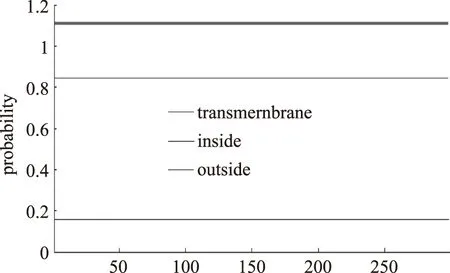
圖7 FZF1基因編碼蛋白質的跨膜區分析Fig.7 Transmembrane domains of protein encoded by the FZF1 gene
2.3.3 蛋白質二級結構預測 利用PredictProtein在線分析平臺預測該蛋白質的二級結構,結構表明,FZF1的二級結構主要以環(loop)為主,高達67.22%,其次是螺旋(helix),占有31.77%,僅含有1%的折疊(sheet)。
2.4 貝酵母FZF1基因編碼蛋白質的結構域
2.4.1 結構域分析 結構域是在蛋白質三級結構中介于二級和三級結構之間的可以明顯區分但又相對獨立的折疊單元,每個結構域自身形成緊實的三維結構,可以獨立存在或折疊。使用SMART在線服務器分析該蛋白質的結構功能域,結果如圖8。整個氨基酸序列中,在第12~36位、第42~66位、第72~94位、第246~273位氨基酸位點出分別存在一個C2H2型鋅指結構域。在第216~213位氨基酸位點處存在一個組成低復雜序列區。
鋅指結構域是一種相當小的蛋白模體,通過許多類似手指狀的突出物與靶分子相聯系,這些區域大多數情況下會結合鋅離子,但時常也會與其他的金屬離子相結合。許多情況下,一些家族成員會通過構成鹽橋使類似手指狀的折疊更加堅固。鋅指結構域能夠與DNA、RNA和蛋白質等相結合,綁定特征依氨基酸序列的不同而有所差異。在不斷的進化過程中,鋅指結構域已經形成了穩固的支架結構。不僅在形成蛋白質的折疊等方面發揮作用,而且對于基因的轉錄調控方面具有重要意義。C2H2型鋅指結構域作為這個家族中最經典的結構類型,由一個短的β折疊和一個α螺旋形成四面體結構,在真核生物中,它作為轉錄因子,與DNA或RNA特異性結合,從而對基因的表達起著抑制或增強的作用。在預測蛋白質的二級結構時,含有31.77%的螺旋結構,可能就是構成鋅指結構域的重要結構。

圖8 FZF1基因編碼蛋白質的結構域分析Fig.8 Structure domain analysis of protein by the FZF1 gene
2.4.2 motif搜索 模體是序列中局部的保守區域,或者是一組序列中共有的一小段序列模式。一般指構成任何一種特征序列的基本結構,但多數情況下是指可能具有分子功能、結構性質或家族成員相關的任何序列模式。模體作為結構中的亞單元,表現結構域的各種生物學功能。
利用PROSITE數據庫對該蛋白質進行motif搜索,結果如圖9。該蛋白質中有3處與指定的模板相匹配,具體為第12~36位、第42~66位、第72~94位的氨基酸序列分別存在一個鋅指結構域,這與SMART在線服務器分析結果相一致。FZF1基因編碼的蛋白質可能是通過這3個C2H2型鋅指結構域與SSU1基因特異性結合,從而增強了SSU1基因的表達。

圖9 FZF1基因編碼蛋白質的motif分析Fig.9 Motif analysis of protein encoded encoded by the FZF1 gene

圖10 FZF1基因編碼蛋白質的三級結構預測Fig.10 Three-dimensional structure prediction of protein encoded by the FZF1 gene
2.5 貝酵母FZF1基因編碼蛋白質的三級結構預測
利用同源建模的網絡綜合服務器Swiss-Model Workspace對貝酵母FZF1基因編碼的蛋白質進行三級結構的預測。預測的三級結構模型見圖10,由在線分析的結果可知:貝酵母FZF1基因編碼的蛋白質基于數據庫中搜索到的模型,比對建模,一致性達到42.62%。由模型評估結果可知,基于QMEANscore4記分值估算,可信度為0.54,表示模型質量中等偏高,較可信。下載PDB圖片,利用蛋白質結構可視化工具Swiss-PdbViewer對預測的三級結構模型圖進行修飾分析,見圖11,預測的三級結構中含有兩個鋅指結構域,與前文結構域的預測基本一致。該預測蛋白質主要由無規則卷曲和螺旋結構組成,與前文二級結構的預測相呼應。

圖11 Swiss-Pdb Viewer修飾下的三級結構Fig.11 The three-dimensional structure modified by Swiss-Pdb Viewer
2.6 貝酵母與釀酒酵母所編碼蛋白質序列的比對
將貝酵母預測編碼的蛋白質序列和釀酒酵母預測編碼的蛋白質利用MEGA 5.05進行序列比對,結果如圖12。分別比對了含有299個氨基酸的序列,其中保守氨基酸有192個,差異氨基酸107個,一致率為64.21%。

圖12 貝酵母與釀酒酵母所編碼蛋白質序列的比對結果Fig.12 Alignment results of protein sequence coded by S. cerevisiae and S.bayanus
3 討論
本研究首次對貝酵母FZF1基因進行了克隆分析,結果顯示該核酸序列可以編碼一條含有299個氨基酸的蛋白質,分子式為C1520H2345N431O468S13,結構不穩定,屬于親水性蛋白,沒有信號肽和跨膜結構域,亞細胞定位在細胞核中發揮作用,含有典型的C2H2鋅指結構域,二級結構中以環(loop)為主,其次是螺旋結構,預測的三級結構也基本與二級結構相符。
貝酵母FZF1基因所編碼的蛋白質序列雖然與釀酒酵母FZF1基因所編碼的蛋白質序列比對后一致率為64.21%,但是兩者蛋白質結構性質差異不甚明顯。釀酒酵母FZF1基因所編碼的蛋白質也是典型的親水性蛋白,沒有信號肽和跨膜結構域,主要在細胞核中發揮作用,氨基酸序列中有一處Coil區,唯一有差異的是,釀酒酵母FZF1基因所編碼的蛋白質中含有五個C2H2型鋅指結構域,而貝酵母FZF1基因所編碼的蛋白質中僅有4個。鋅指結構域的主要作用是與DNA結合而行使基因調控的作用,釀酒酵母FZF1基因與SSU1基因結合,從而可調節酵母亞硫酸鹽的耐受能力[3]。鋅指結構域的多少可能與酵母對亞硫酸鹽耐受能力的強弱相關。而貝酵母FZF1基因所編碼的蛋白質中僅有4個鋅指,則可能是貝酵母硫耐受能力比釀酒酵母弱的重要原因。蛋白質的特殊結構決定了蛋白質的特殊功能,在具有相似結構的前提下分析推測,貝酵母FZF1基因與釀酒酵母FZF1基因可能具有相似的功能,與酵母亞硫酸鹽的耐受能力相關。Tomoko[21]和Iijima[22]等先前就認為貝酵母SSU1基因編碼的蛋白質可能具有亞硫酸鹽“鹽泵”的功能,進而可以推測與之相關的FZF1基因也與亞硫酸鹽“鹽泵”相關,而有關貝酵母FZF1基因則還沒有相關的報道。當然,本文推測結論還需要后續進行功能確證等進一步驗證。
4 結論
本研究通過對貝酵母FAF1基因的克隆與分析,得出該基因包含一條長度為900 bp的開放閱讀框,編碼一條含有299個氨基酸親水蛋白,該蛋白主要以無規則卷曲為主,含有四個鋅指結構域,在細胞核中行使功能。與釀酒酵母FZF1基因所編碼的蛋白質序列比對結果一致性達64.21%,結構上主要區別在于,比釀酒酵母FZF1基因所編碼的蛋白質少一個鋅指結構域,這可能是貝酵母耐硫性稍遜色的重要原因。
亞硫酸在葡萄酒的釀造和品質形成過程中,起著至關重要的作用。但是由于亞硫酸鹽對細胞的毒害作用,所以對硫的耐受能力成為篩選釀酒菌株的重要指標之一。本研究通過生物信息學分析的知識初步預測了貝酵母FZF1基因所編碼的蛋白質,并對該蛋白質的基本理化性質和結構做出了詳細的預測,為貝酵母FZF1基因的進一步研究和貝酵母硫耐受性的更深層次探索做出了一定的參考。
[1]陳葉福,沈世超,王艷,等. SSU1多拷貝表達對釀酒酵母二氧化硫生成量的影響[J]. 微生物學報,2008,48(12):1609-1615.
[2]Taylor S L,Higley N A,Bush R. Sulfites in food:uses,analytical methods,residues,fate,exposure assessment,metabolism,toxicity,and hypersensitivity[J]. Adv. Food Res,1986,30:1-7.
[3]Avram D,Leid M,Bakalinsky A T. Fzflp ofSaccharomycescerevisiaeis a positive regulator of SSU1 transcription and its first zinc finger region is required for DNA binding[J]. Yeast,1999,15:473-480.
[4]Divol B,du Toit M,Duckitt E. Surviving in presence of sulphur dioxide:strategies developed by wine yeasts[J]. Appl Microbiol Biotechnol,2012,95(3):601-613.
[5]Hinze H,Holzer H. Analysis of the energy metabolism after incubation ofSaccharomycescerevisiaewith sulfite or nitrite[J]. Arch Microbiol,1986,145(1):27-31.
[6]Hinze H,Holzer H. Effect of sulfite or nitrite on the ATP content and the carbohydrate metabolism in yeast[J]. Z Lebensm Unters Forsch,1985,181(2):87-91.
[7]Schimz KL,Holzer H. Rapid decrease of ATP content in intact cells ofSaccharomycescerevisiaeafter incubation with low concentrations of sulfite[J]. Arch Microbiol,1979,121(3):225-229.
[8]Rankine B C,Pocock K F. Influence of yeast strain on binding of sulphur dioxide in wines,and on its formation during fermentation[J]. J Sci Food Agric,1962,20(2):104-109.
[9]Park H,Bakalinsky. SSU1 mediates sulphite efflus in Sacharomyces cerevisiae[J]. Yeast,2000(16):881-888.
[10]Tiziana N,Viviana C,Alessio G,et al. A sulphite-inducible
form of the sulphite efflux gene SSU1 in aSaccharomycescerevisiaewine yeast[J]. Microbiology,2010,156(6):1686-1696.
[11]Avram D,Bakalinsky A T. SSU1 encodes a plasma membrane protein with a central role in a network of proteins conferring sulfite tolerance in Saccharomyces cerevisiae[J]. Bacteriol,1997,179(18):5971-5974.
[12]Donalies U E B,Stahl U. Increasing sulphite formation inSaccharomycescerevisiaeby overexpression of MET14 and SSU1[J]. Yeast,2002,19:475-484.
[13]Breitwieser W,Price C,Schuster T. Identification of a gene encoding a novel zinc finger protein inSaccharomycescereviaiae[J]. Yeast,1993(9):551-556.
[14]王慶國,劉天明. 酵母菌分類學方法研究進展[J]. 微生物學雜志,2007,27(3):96-101.
[15]Ciani M E,Kerala(India),Sipiczki M. Taxonomic and physiological diversity of saccharomyces bayanus,in biodiversity and biotechnology of wine yeasts[M]. Research Signpost,2002,53-69.
[16]Naumov G I,Naumova E S,Martynenko N N and Masneuf-Pomaréde I. Taxonomy,ecology,and genetics of the yeast saccharomyces bayanus:a new object for science and practice[J].Mikrobiologiya,2011,80(6):723-730.
[17]張太奎,朱方明,劉小珍,等. 貝酵母SSU1基因的克隆與分析[J]. 中國食品學報,2014,14(9):195-199.
[18]閻隆飛,孫之榮. 蛋白質分子結構[M]. 北京:清華大學出版社,1999:17-18.
[19]劉雅婷,李正躍,朱有勇,等. 植物病原菌Pseudomonas syringae pv. tomato基因組中的信號肽分析[J]. 遺傳,2005,27(6):959-964.
[20]魏香,曾憲綱,周海夢. 蛋白質結構中卷曲螺旋的研究進展[J]. 中國生物化學與分子生物學報,2004,20(5):565-571.
[21]Tomoko F. Functional analysis of SSU1 genes in lager brewing yeast[J]. Bioscience & Industry,2003,61(12):809-810.
[22]Iijima K,Ogata T. Construction and evaluation of self-cloning bottom-fermenting yeast with high SSU1 expression[J]. Journal of Appl Microbiology,2010,109(6):1906-1913.
Cloning and sequence analysis of theFZF1 gene concerning sulfur tolerance fromSaccharomycesbayanus
HE Xiao,ZHANG Xian-ang,LIU Xiao-zhen,LI Sheng-xing,ZHANG Han-yao*
(Key Laboratory for Forest Genetic and Tree Improvement & Propagation in Universities of Yunnan Province,Southwest Forestry University,kunming 650224,China)
TheFZF1 gene ofSaccharomycescerevisiaewas found to be a positive regulator ofSSU1 transcription,but there was no report about theFZF1 gene ofSaccharomycesbayanus. In this study,the genes were cloned and the content of bioinformatics was analyzed by online tools for setting the foundation of the further research. Here theFZF1 gene ofS.bayanuswas cloned and the physical and chemical properties ofFZF1 protein were analyzed by online analytical tools,such as ProtParam,ProtScale,TMHMM,PredictProtein and Swiss-Model. At the same time,the protein secondary structure and tertiary structures were predicted. The results indicated that theFZF1 gene contained an opening reading frame(ORF)of 900 bp encoding a 299 predicted amino acids. The protein was a hydrophilic protein exercising their control functions in the nucleus. It contained 18 serine kinase potential phosphorylation sites,a coil domain and 4 zinc finger domains. The protein encoded byFZF1 gene ofS.cerevisiaewas very similar with the predicted protein. So theFZF1 gene ofS.bayanuswas inferred to be associated with the sulfur resistance. And the protein encoded by theFZF1 gene ofS.bayanusonly contains 4 zinc finger domains. It was one of the most important reasons that the sulfur tolerance of mostS.bayanuswas weaker than mostS.cerevisiae.
Saccharomycesbayanus;FZF1;sequencing analysis;sulfur tolerance gene
2015-02-12
賀笑(1991-),女,在讀碩士,研究方向:林業生物技術,E-mail:smile1165279688@sina.com。
*通訊作者:張漢堯(1975-),男,博士,教授,研究方向:植物和微生物分子遺傳,E-mail:hanyaoz@163.com。
國家自然科學基金項目(31360404);教育部歸國人員啟動基金項目(212209)。
TS201.3
A
1002-0306(2015)23-0166-07
10.13386/j.issn1002-0306.2015.23.026

