基于改良ERIC-PCR技術快速鑒別人參和西洋參
王 銳,楊 果,王昊楠,李明華,姚仕鑫(遼寧工程技術大學理學院,遼寧阜新123000)
基于改良ERIC-PCR技術快速鑒別人參和西洋參
王銳,楊果,王昊楠,李明華,姚仕鑫
(遼寧工程技術大學理學院,遼寧阜新123000)
為建立快速、準確的鑒定人參和西洋參并測定二者在混合物中含量的方法,將ERIC-PCR(Enterobacterial Repetitive Intergenic Consensus-Polymerase chain reaction)引物改良進行溫度梯度PCR擴增,分析人參(Panax ginseng C.A.Mey,PG)和西洋參(Panax quinquefolium L.,PQ)電泳條帶并尋找其特異性條帶,結果發現一對特異性引物ER1和EL2,當退火溫度為46.0℃時,只出現2.8 kb條帶為西洋參,而出現2.8 kb和3.4 kb條帶為人參。通過對混合樣品中3.4 kb條帶的定量分析,可以準確的定量西洋參的含量,系統誤差僅為5.2%,RSD為3.5%,說明該方法可以快速、準確的鑒定西洋參和人參,并且可以準確測定混合物中西洋參的含量。
ERIC-PCR,西洋參,人參,DNA條形碼
人參(Panax ginseng C.A.Mey,PG)和西洋參(Panax quinquefolium L.,PQ)均為五加科(Araliaceae)人參屬(Panax)植物,兩者形狀和化學成分相似,又都是著名的強壯滋補藥,易于混淆,人參性溫偏于助陽,西洋參性涼偏于滋陰,藥效差異很大[1-4]。隨著人們對美容、延緩衰老類滋補品需求的不斷增加,西洋參及其制品消費量大,價格昂貴,貨源偏緊,市場上偽品不斷出現,尤其以人參摻入或冒充西洋參,并加工成飲片、粉末或膠囊,傳統的性狀鑒別法、顯微鑒別法鑒別難度很大,依靠分析檢測人參中某些化學成分的理化性狀鑒別法快速、準確地鑒別人參和西洋參制品尚有一定的局限性[5]。因此需要建立一種適用于各種人參、西洋參形態的快速、客觀、穩定、易重復的鑒定方法。
ERIC-PCR被廣泛應用于細菌的分類和鑒定[6-9]。ERIC-PCR屬于半隨機擴增,即當基因組中含有ERIC時,擴增產物通常一端含有ERIC,另一端隨機匹配;當基因組中不存在ERIC時,擴增產物具有特殊的隨機擴增:其引物較長(22 bp),可產生具有較高可重復性和一定特異性的指紋圖譜[10]。Gillings發現ERIC可以在植物和動物中擴增出相應的特征產物[11]。基于此本文首次將ERIC-PCR引物末端堿基進行隨機化改良,通過對比每對引物溫度梯度PCR結果,尋找人參或西洋參的特異條帶,建立準確、快速、穩定的人參和西洋參鑒別和定量方法,有利于在功能性食品行業中快速準確地鑒定西洋參。
1 材料與方法
1.1材料與儀器
人參、西洋參藥材各10批樣品,每批樣品的狀態、產地見表1;植物基因組DNA提取試劑盒NEP003-1北京鼎國昌盛生物技術有限責任公司;DNA marker Ladder III 100 bp北京鼎國昌盛生物技術有限責任公司;100g-Biowest agarose瓊脂糖上海浩然生物技術有限公司;實驗所用其他試劑均為分析純或優級純;水滅菌超純水。
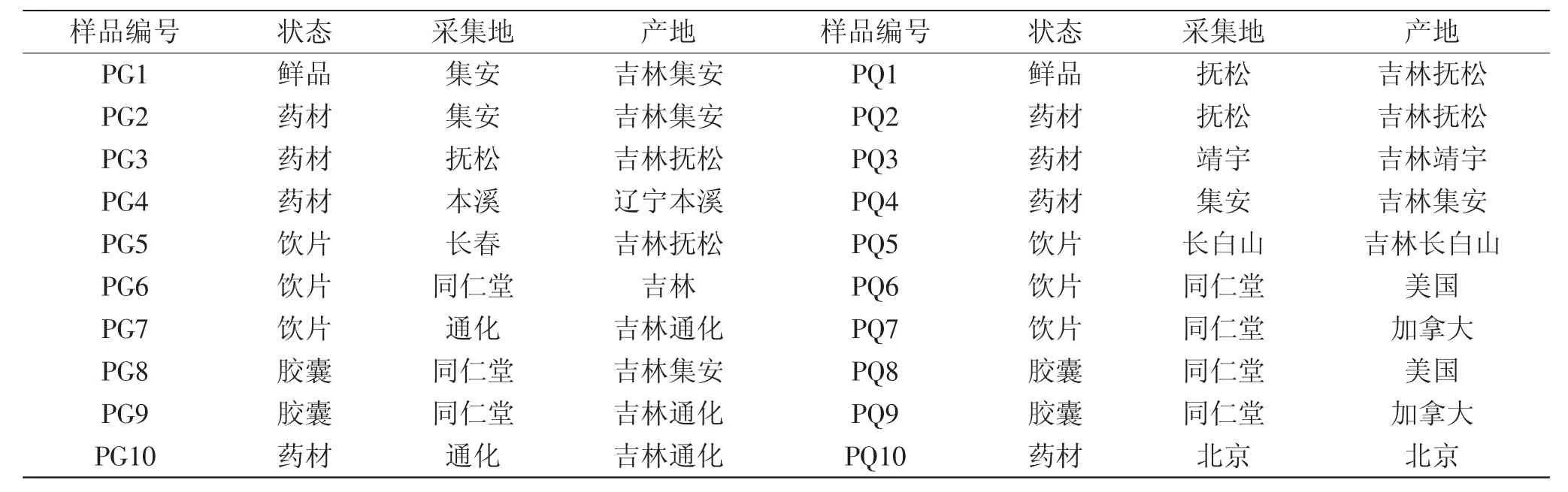
表1 供試人參(PG)和西洋參(PQ)樣品來源Table 1 Resources of Panax ginseng(PG)and Panax quinquefolium(PQ)
TC-512型PCR擴增儀北京眾益中和生物技術有限公司;LG2020D型電泳凝膠成像系統上海圣科儀器設備有限公司;HC-3018R高速冷凍離心機安徽科大中佳;LDZX-50FBS翻蓋型壓力蒸汽滅菌器上海申安醫療器械廠。
1.2實驗方法
1.2.1DNA的提取用70%乙醇漂洗人參和西洋參藥材或飲片,擦干,60℃干燥,粉碎,過篩(40目)制成粉末;膠囊直接將人參或西洋參粉末倒出用于實驗。按批次取人參和西洋參粉末各100 mg,放入液氮冷凍過的研缽內,加入適量液氮研磨成細粉。嚴格按照植物基因組DNA提取試劑盒說明書操作提取DNA,并以1%瓊脂糖凝膠電泳檢測DNA的完整性;取少量DNA樣品稀釋100倍,用紫外分光光度計測定波長在260 nm處的吸光度值A,通過公式濃度(μg·mL-1)= A260×50×稀釋倍數,計算DNA的濃度,并使DNA模板終濃度為200 ng。
1.2.2人參和西洋參的鑒定參照文獻[12]的方法,對人參和西洋參藥材、飲片和膠囊進行品種鑒定。
1.2.3ERIC-PCR擴增引物的改良將ERIC-PCR的引物:ER和EL的末位堿基C和G分別隨機化[13],并對引物重新編號,結果見表2。所有引物均由上海生工生物技術有限公司合成,并經聚丙烯酰胺凝膠電泳(PAGE)純化。

表2ERIC-PCR改良引物Table 2 The modified primers of ERIC-PCR
1.2.4人參和西洋參特異性條帶的確定以人參和西洋參標準品PG1和PQ1的DNA為模板,以改良的引物兩兩配對進行溫度梯度PCR擴增,25 μL反應體系中含有10×PCR Buffer(+Mg2+)2.5 μL,dNTP(2.5 mmol·L-1)2.0 μL,引物10 μmol·L-1各1 μL,TaqE(5 U·μL-1)0.3 μL,模板DNA 200 ng,加ddH2O至25 μL。
ERIC-PCR擴增程序:預變性95℃,7 min;變性溫度94℃,1 min;退火溫度40~62℃,50 s;延伸65℃,8 min;總延伸65℃,16 min;4℃保溫30 min結束反應。每對改良引物重復三次溫度梯度PCR,分別取10 μL反應產物在含EB的1.0%的瓊脂糖凝膠上電泳(100 V)1 h,用電泳凝膠成像系統觀察、拍照。對照片進行比對分析,確定人參和西洋參可能的特異性引物和退火溫度。
1.2.5適用范圍實驗以PG1和PQ1為標準品,按照1.2.4項確定的特異性引物和退火溫度、擴增程序,檢驗其余西洋參和人參樣品基因組DNA的擴增頻率和特異性。
1.2.6人參和西洋參混合樣品的測定將西洋參(PQ1)和人參(PG1)粉末按不同比例混合,總重量為100 mg,其中西洋參分別占0%、10%、20%、40%、60%、80%和100%。按1.2.1項下方法提取人參和西洋參混合樣品DNA,用紫外分光光度計測定DNA濃度,并調節濃度使標準品DNA濃度和5批待測的混合樣品DNA濃度一致,以1.2.5項下方法進行PCR擴增,精確量取PCR產物10 μL在含EB的1.0%的瓊脂糖凝膠上電泳(100 V)1 h,用電泳凝膠成像系統觀察、拍照。用Quantity One 4.6.2軟件分析照片,測定特異性條帶的峰密度(Peak Density),以峰密度值為縱坐標,以西洋參的百分含量為橫坐標做標準曲線。
準確稱量5個批次(PQ1、PQ4、PQ6、PQ9和PQ10)的西洋參粉末各50 mg,共5份;在每一份西洋參粉末中隨機加入任意批次的人參粉末50 mg,分別編號為PGQ1、PGQ4、PGQ6、PGQ9和PGQ10。按標準曲線方法進行PCR和電泳、測定特異條帶的峰密度值,計算人參的百分含量。
2 結果與分析
2.1人參和西洋參的鑒定
參照文獻[12],將采集或購買的人參和西洋參樣品的ITS2(Internal Transcribed Spacer 2,內部轉錄間隔區2)序列在中藥材DNA條形碼鑒定系統(http:// www.tcmbarcode.cn)上進行物種鑒定,鑒定結果顯示采集或購買的人參和西洋參樣品均為真品。
2.2人參和西洋參特異條帶的確定
通過對比西洋參和人參的溫度梯度照片,找出PQ1或PG1的特異性條帶,該特異性條帶對應的引物和相應的退火溫度就是快速鑒別PQ1和PG1的特異性引物和退火溫度,將該條件下的PCR產物放在同一塊瓊脂糖凝膠上進行電泳,結果見圖1。從圖1可以看出改變ERIC-PCR引物末尾堿基和退火溫度可以在PQ1和PG1中產生更多的、可重復、穩定的特異性指紋圖譜,這也在一定程度上反映PQ1和PG1基因組的單核苷酸多態性(SNP)。單核苷酸多態性(SNP)具有分布廣泛、數量眾多、易于批量檢測等優點。在物種鑒定、物種起源與親緣關系、遺傳育種等領域得到了廣泛的應用,特別是在區分近緣種的研究中非常有效[14]。

圖1 西洋參和人參可能特異性引物鑒別電泳圖Fig.1 The electrophoretograms of P.ginseng and P.quinquefolius possible specific primers.
2.3適用范圍實驗結果
選用不同產地、不同藥店出售藥材、飲片和膠囊作為實驗材料,具有廣泛的代表性。對收集到的代表性樣品各10批進行檢測,結果顯示:只有一對特異引物ER1和EL2在退火溫度為46.0℃條件下按照1.2.4項操作,才能將PQ和PG完全清晰地區分開(圖2)。從圖2中可看出2.8 kb條帶為PQ和PG共有帶,3.4 kb條帶為人參特異性帶。這與PQ和PG的IST2條形碼鑒定結果一致。實驗重復三次,結果均相同,說明特異性引物ER1和EL2在退火溫度為46.0℃條件下可準確、快速而穩定鑒別PQ和PG,而且不受物種產地和形態的影響,說明該方法重復性、穩定性很好,適用范圍廣。

圖2 西洋參和人參鑒別電泳圖Fig.2 The identification electrophoretogram of P.quinquefolius and P.ginseng
2.4人參和西洋參混合樣品的測定結果
圖3顯示了西洋參和人參混合樣品的測定結果。利用Quantity One 4.6.2軟件分析照片,讀取3.4 kb條帶的峰密度(Peak Density)值,以峰密度值為橫坐標,以西洋參的百分含量為縱坐標,用Origin 9.4繪制標準曲線,標準方程為y=-0.03x+0.98,r=0.9991,說明西洋參含量在0%~100%線性關系良好。

圖3 不同比例PQ和PG混合物電泳圖Fig.3 The electrophoretogram of different ratio mixtures of P.quinquefolius and P.ginseng
待測樣品(PGQ1、PGQ4、PGQ6、PGQ9和PGQ10)測定結果見表3。由表3可知,采用特異性引物(ER1和EL2)和退火溫度(46.0℃)進行ERIC-PCR擴增,用Quantity One 4.6.2軟件測定特異性條帶峰密度值的方法測定西洋參和人參混合物中的西洋參的含量準確度較高,平均加樣回收率為105.2%,相對標準偏差為3.5%,系統誤差率僅為5.2%。因此該方法不僅可以快速、準確、穩定的鑒別西洋參和人參,而且可以準確的測定西洋參的百分含量。

表3 西洋參和人參混合物中PQ含量的測定Table 3 The determination of PQ in the mixture of P.quinquefolius and P.ginseng
3 結論
通過ERIC-PCR技術找到鑒別西洋參和人參的一對特異性引物——ER1和EL2,當退火溫度為46.0℃時,人參的PCR產物有一條特異性條帶,其長度為3.4 kb。通過這對引物可以準確的對人參和西洋參混合物中的人參或西洋參進行定量,無需昂貴的儀器和測序。從DNA的提取、特異性PCR擴增到數據分析可在一日內完成。該方法在食品藥品領域還具有較大的潛在應用前景,特別是在功能性食品行業中通過快速準確檢測物種近緣種,為規范保健食品市場服務提供理論基礎。
[1]詹鑫婕,田程,張媛,等.基于ITS2條形碼SNPs的人參和西洋參PCR-SSCP分子鑒別研究[J].中國中藥雜志,2012,37(24):3748-3751.
[2]中華人民共和國衛生部藥典委員會.中華人民共和國藥典中藥薄層色譜集[M].北京:人民衛生出版社,2010:20-23.
[3]Li W,Gu C,Zhang H,et al.Use of high-performance liquid chromatography-tandem mass spectrometry to distinguish Panax ginseng CA Meyer(Asian ginseng)and Panax quinquefolius L.(North American ginseng)[J].Analytical chemistry,2000,72(21):5417-5422.
[4]曹智,張燕娣,許永華,等.人參有效成分及其藥理作用研究新進展[J].人參研究,2012,24(2):39-43.
[5]宋沁馨,馮芳,張心悅,等.SNP測定結合芯片電泳法快速鑒別人參和西洋參[J].藥物分析雜志,2009(1):1-5.
[6]Sharples G J,Lloyd R G.A novel repeated DNA sequence located in the intergenic regions of bacterial chromosomes[J]. Nucleic acids research,1990,18(22):6503-6508.
[7]廖永紅,任文雅,孫寶國,等.米醋沉淀中Bacillus subtilis DNA提取及ERIC-PCR體系條件優化[J].食品工業科技,2011(6):212-215.
[8]丁久法,潘迎捷,陳洪友,等.副溶血性弧菌ERIC-PCR分型及毒力基因檢測研究[J].食品工業科技,2010(8):137-141.
[9]郭丹鳳,張昭寰,肖莉莉,等.不同耐藥性致病性副溶血性弧菌的生長特性比較研究[J].食品工業科技,2014,35(19):137-141.
[10]陳迎春,曹又方,趙立平.大腸桿菌MG1655菌株ERIC-PCR圖譜主帶序列組成分析[J].微生物學通報,2002,29(6):28-32.
[11]Gillings M,Holley M.Repetitive element PCR fingerprinting(rep-PCR)using enterobacterial repetitive intergenic consensus(ERIC)primers is not necessarily directed at ERIC elements[J]. Letters in applied microbiology,1997,25(1):17-21.
[12]陳士林,姚輝,韓建萍.中藥材DNA條形碼分子鑒定指導原則[J].中國中藥雜志,2013,38(2):141-148.
[13]Versalovic J,Koeuth T,Lupski R.Distribution of repetitive DNA sequences in eubacteria and application to finerpriting of bacterial enomes[J].Nucleic acids research,1991,19(24):6823-6831.
[14]鄒喻蘋,葛頌.新一代分子標記——SNPs及其應用[J].生物多樣性,2004,11(5):370-382.
Study on rapid identification of Panax quinquefolius and Panax ginseng based on improved ERIC-PCR
WANG Rui,YANG Guo,WANG Hao-nan,LI Ming-hua,YAO Shi-xin
(College of Science,Liaoning Technology University,Fuxin 123000,China)
Studied on the rapid identification and quantification of PQ and PG using improved ERIC-PCR,and a pair of new primers(ER1 and EL2)was found.When the annealing temperature was 46.0℃,the genome DNA PCR results showed one specific 2.8 kb band for PQ while two bands of 2.8 kb and 3.4 kb repectively for PG. It also was possible to quantify PG in mixed PQ and PG sample by quantification of the 3.4 Kb band,and the systematic error was only 5.2%and RSD was 3.5%.The study showed that this method was rapid and accurate to identify PQ and PG and to quantify PG in mixed PQ and PG.
improved ERIC-PCR;Panax quinquefolius;Panax ginseng;DNA barcodes
TS252.1
A
1002-0306(2015)20-0080-04
10.13386/j.issn1002-0306.2015.20.008
2015-02-05
王銳(1978-),女,碩士,講師,研究方向:功能性食品開發,E-mail:527021510@qq.com。
國家大學生創新創業訓練計劃項目(201410147045);十二五農村領域國家科技計劃課題(2012BAD22B00)。

