流感病毒兩種反向遺傳學操作系統的初步構建
連孟洋,王 昕,彭 博,武偉華,劉 慧,董方圓,丁小滿,房師松,鄭 青
2.深圳市疾病預防控制中心,深圳 518055;
3.中山大學公共衛生學院,廣州 510080
當今世界處于后流感大流行期,甲型流感病毒在大范圍內季節性傳播,并感染包括人、禽類等多個物種,故世界各地都積極致力對此病毒的研究。甲型流感病毒是單股負鏈分節段的RNA病毒,病毒基因組約13.6kb,分為8個大小不等的獨立片段,攜帶至少13種病毒蛋白[1]。流感病毒基因組RNA含多個開放閱讀框,并與3′和5′端非編碼區相連[2]。近年來,負鏈RNA病毒的基因操作基于反向遺傳學技術獲得突破性進展,同時流感病毒反向遺傳學研究也在其生命周期等方面取得很大進展[3]。就甲型流感病毒而言,Luytjes和 Enami等[4-5]建立基于體外重組核糖核蛋白復合體(vRNPs)轉染輔助病毒感染性細胞的反向遺傳學系統,vRNPs是由體外轉錄產生的RNA及純化的病毒核蛋白(NP)和聚合酶蛋白(PB1,PB2和PA)孵育而成。輔助病毒則可作為病毒核蛋白和聚合酶蛋白的來源以幫助vRNPs在細胞內復制。隨后,Zobel等[6]和 Neumann等[7]提出依賴 RNA 聚合酶I的體內合成vRNA的反向遺傳學系統,成功獲得流感病毒cDNA 的精確復制[7]。1996年,Pleschka等[8]提出不依賴輔助病毒的反向遺傳學系統—利用人RNA聚合酶I啟動子及丁型肝炎病毒基因組核糖酶來完成反向遺傳操作。同年,Lutz等[9]得出甲型流感病毒表達報告基因可作為檢測和定量病毒復制的工具。隨后,Neumann等[10]同樣不依賴輔助病毒,利用12個質粒共轉染293T細胞,在人RNA聚合酶Ⅰ啟動子、小鼠RNA聚合酶Ⅰ終止子以及RNA聚合酶Ⅱ的調控下,成功包裝 A/WSN/33(H1N1)病 毒 粒 子。Hoffmann 等[11]在 Neumann[10],Fodor[12]等研究成果的基礎上于2000年建立了基于8質粒的雙向RNA聚合酶Ⅰ/聚合酶Ⅱ轉錄系統,此系統允許甲型流感病毒基于8個質粒拯救其子代病毒,使所需質粒數量由12/17降低到8,減少其所需時間及成本,促進雙向的反向遺傳學系統的發展。
本研究基于甲型流感病毒H1N1分離株A/Puerto Rico/8/34的基因組,分別構建依賴輔助病毒和宿主細胞內合成vRNPs的反向遺傳學操作系統,并嘗試利用上述兩系統表達EGFP,為本課題后期反向遺傳學系統的建立提供借鑒。
1 材料與方法
1.1 材料
1.1.1 病毒毒株 人源流感病毒 A/Puerto Rico/8/34(H1N1),由深圳市疾病預防與控制中心提供。
1.1.2 載體 克隆載體 pBluescript II sk(+)(Takara公司),pcDNA3.1(+)(Invitrogen公司)。
1.1.3 大腸桿菌菌種和細胞系 大腸桿菌JM109感受態細胞 (Takara公司);MDCK細胞,293T細胞,由深圳市疾病預防與控制中心提供。
1.1.4 引物
1.1.4.1 構建POL I系統(pBluescript II sk(+)-tI-EGFP-PIh),設計引物見表1。
1.1.4.2 構建表達載體 pcDNA3.1-PB2 、pcDNA3.1-PB1、pcDNA3.1-PA、pcDNA3.1-NP 設 計引物見表2。

表1 構建pBluescript II sk(+)-tI-EGFP-PIh載體的引物Tab.1 Primers for pBluescript II sk(+)-tI-EGFP-PIh
上述引物均合成于Takara公司。
1.1.5 主要試劑 PCR擴增試劑盒(Takara公司),質粒DNA抽提純化試劑盒(Takara公司),T4 DNA連接酶(Takara公司),PCR產物回收試劑盒(QIAGEN公司),膠回收試劑盒(QIAGEN公司),無內毒素的質粒DNA抽提純化試劑盒(QIAGEN公司),限制性內切酶 HindIII、XhoI、BamhI、NotI、XbaI(NEB 公 司 ),轉 染 試 劑 盒 Lipofectamine2000(Invitrogen 公 司 ),細 胞 培 養 基DMEM(Gibco公司)、Opti-MEM(Gibco公司),胎牛血清FBS(Gibco公司),雙抗青霉素、鏈霉素(Invitrogen公司),EDTA 胰酶(Gibco公司),TPCK處理胰酶(Gibco公司)。

表2 構建pcDNA3.1-PB2、pcDNA3.1-PB1、pcDNA3.1-PA、pcDNA3.1-NP載體的引物Tab.2 Primers for pcDNA3.1-PB2 、pcDNA3.1-PB1、pcDNA3.1-PA、pcDNA3.1-NP
1.2 方法
1.2.1 載體的構建
1.2.1.1 POL I系統(pBluescript II sk(+)-tI-EGFP-PIh)的構建 經文獻所述,因鼠源聚合酶I終止子序列末端6個腺嘌呤(A)堿基的差異[13],在此設計兩個POL I系統(POL I系統1和POL I系統2)。
為了構建POL I系統,首先利用聚合酶I元件引物擴增聚合酶I元件1和2序列,然后分別使用相應的引物將人POL I啟動子PIh、鼠的終止子tI及EGFP基因克隆出,隨后基于嵌合引物合成tI-EGFP-PIh基因片段,最后經限制性內切酶Hind III和Xho I酶切,T4DNA連接酶連接,將其克隆至pBluescript II sk(+)載體。
1.2.1.2 表達載體pcDNA3.1-PB2 、pcDNA3.1-PB1、pcDNA3.1-PA、pcDNA3.1-NP的構建 PB2、PB1、PA、NP基因的cDNA和載體pcDNA3.1經限制性內切酶酶切,連接獲得重組質粒pcDNA3.1-PA 、pcDNA3.1-PB1、pcDNA3.1-PB2、pcDNA3.1-NP。
上述所有重組質粒均經過菌液PCR、雙酶切鑒定和測序分析以保證載體的正確。
1.2.2 細胞轉染
1.2.2.1 MDCK細胞轉染 MDCK細胞在25m2細胞培養瓶中長至90%左右,用預溫的PBS液洗3次,EDTA胰酶消化細胞并按照1∶10的比例鋪6孔細胞培養板。待細胞密度為80%~90%時,用PBS液洗3次,將培養基換為病毒接種液,隨后用10μL流感病毒H1N1毒株(血凝價為1∶4)感染細胞。感染2h后,用PBS液洗3次,按照Lipofectamine 2000說明書進行操作,將純化質粒pBluescript II sk(+)-tI-EGFP-PIh-1和 pBluescript II sk(+)-tI-EGFP-PIh-2分別轉染至 MDCK細胞中。
1.2.2.2 293T細胞轉染 293T細胞在25m2細胞培養瓶中長至90%左右,用PBS液洗3次,按照1∶6的比例鋪6孔細胞培養板。待細胞密度為80-85%時,用 PBS液 洗3次,按 照 Lipofectamine2000說明書進行操作,將純化質粒pcDNA3.1-PB2 、pcDNA3.1-PB1、pcDNA3.1-PA、pcDNA3.1-NP 與 pBluescript II sk(+)-tI-EGFPPIh-1/2分別共轉染293T細胞。
2 結 果
2.1 POL I系統(pBluescript II sk(+)-tI-EGFPPIh)的構建 研究構建的 pBluescript II sk(+)-tI-EGFP-PIh載體的示意圖,見圖1。
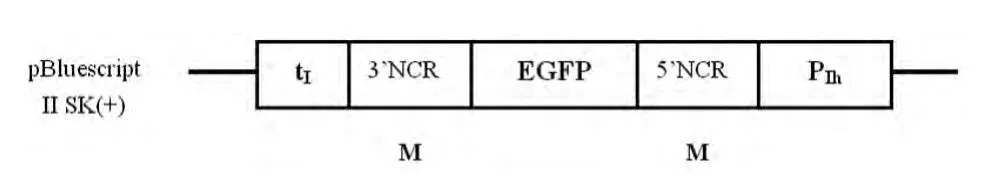
圖1 pBluescript II sk(+)-tI-EGFP-PIh載體的示意圖Fig.1 Schematic diagram of pBluescript II sk(+)-tI-EGFPPIh.
2.2 表達載體pcDNA3.1-PB2 、pcDNA3.1-PB1、pcDNA3.1-PA、pcDNA3.1-NP的構建,見圖2。
2.3 細胞轉染
2.3.1 依賴于輔助病毒的轉染,見圖3。
轉染時,需先用流感病毒 H1N1(血凝價為1∶4)感染MDCK細胞2h,隨后將含有POL I系統的 pBluescript II sk(+)-tI-EGFP-PIh-1/2 重組質粒轉染MDCK細胞,同時,分別將pEGFP-N1和pBluescript II sk(+)質粒轉染MDCK細胞作為陽性和陰性對照。轉染后12h,用熒光顯微鏡觀察到細胞開始出現熒光,在轉染48h時,熒光最強。同時,分別經 pBluescript II sk(+)-tI-EGFP-PIh-1和pBluescript II sk(+)-tI-EGFP-PIh-2轉染的兩組細胞,其熒光強度沒有明顯差別,見圖4。
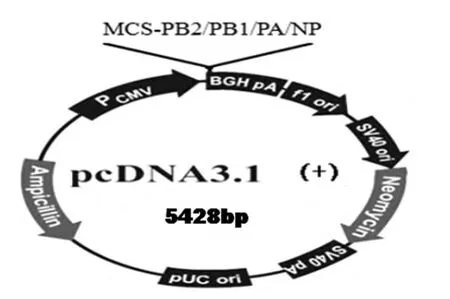
圖2 pcDNA3.1-PB2 、pcDNA3.1-PB1、pcDNA3.1-PA、pcDNA3.1-NP表達載體的示意圖Fig.2 Schematic diagram of expression vectors--- pcDNA3.1-PB2、pcDNA3.1-PB1、pcDNA3.1-PA、pcDNA3.1-NP.
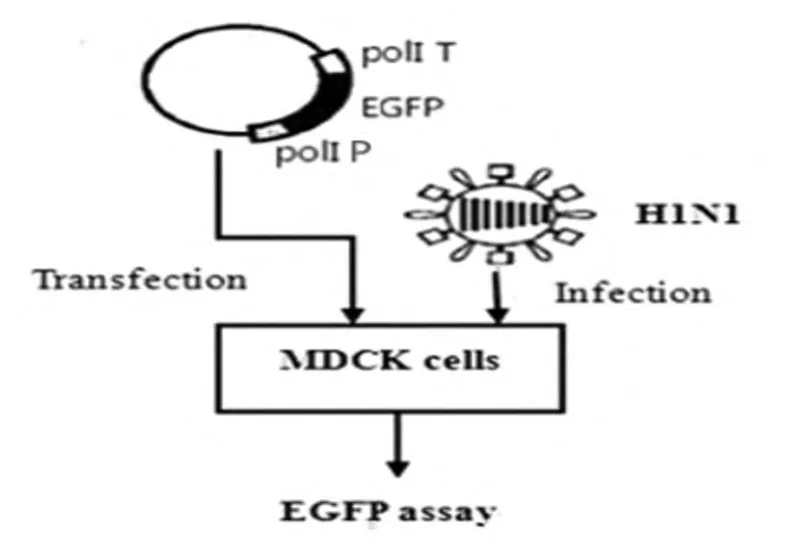
圖3 依賴于輔助病毒的POL I系統轉染示意圖Fig.3 Schematic diagram of transfection that POLI system relying on helper virus.
2.3.2 依賴宿主細胞內合成RNPs的轉染,見圖5。
將表達載體pcDNA3.1-PB2、pcDNA3.1-PB1、pcDNA3.1-PA、pcDNA3.1-NP與 POL I系統質粒pBluescript II sk(+ )-tI-EGFP-PIh-1/2 共 轉 染293T細胞,同時,分別設pEGFP-N1轉染陽性對照組和pBluescript II sk(+)轉染陰性對照組。轉染12h后,在熒光顯微鏡下可觀察到熒光現象,細胞培養至48h時,熒光強度逐漸增強。與上組依賴輔助病毒的轉染實驗相同,分別經POL I系統pBluescript II sk(+)-tI-EGFP-PIh-1和 pBluescript II sk(+)-tI-EGFP-PIh-2 轉 染 的 兩 組 細 胞,其 熒 光 強 度均較低,見圖6。
上述實驗結果表明:依賴輔助病毒和依賴宿主細胞內合成RNPs的轉染系統均可使外源基因EGFP在哺乳動物細胞中表達。
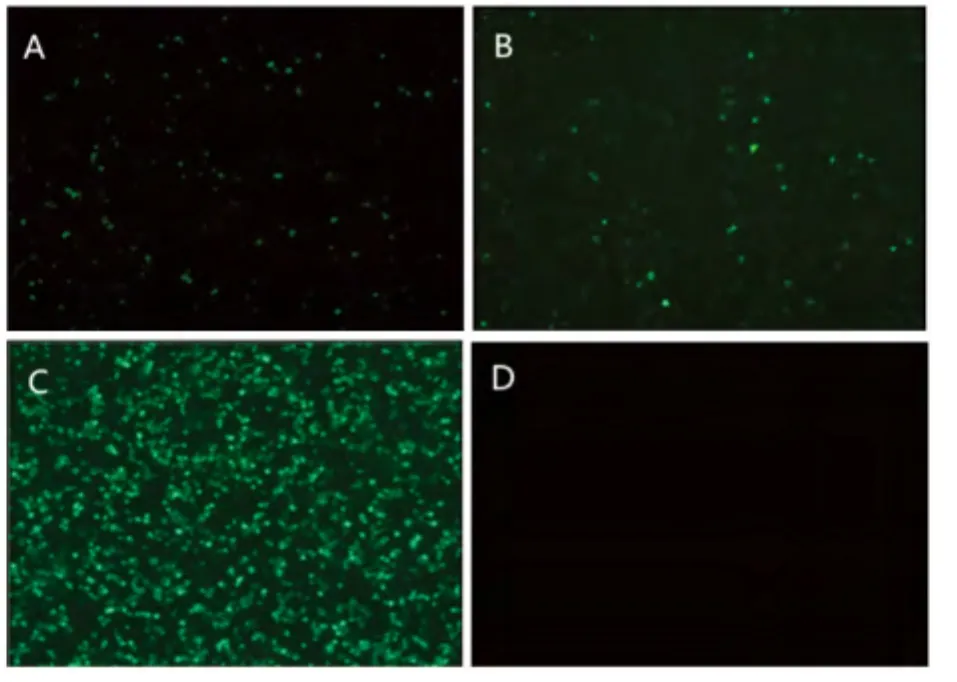
圖4 依賴輔助病毒的POL I系統轉染MDCK細胞48h的綠色熒光蛋白表達Fig.4 The expression of Green Fluorescent protein at 48h post-transfection that POLI system based upon helper virus-infection.
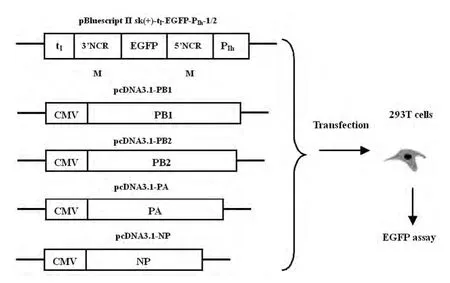
圖5 依賴宿主細胞內合成vRNPs的轉染Fig.5 The transfection of virus ribonucleoprotein complex(vRNPs)that synthetised in the host cell.
3 討 論
流感病毒等負鏈RNA病毒因其RNA為負極性,故其vRNA對細胞不具感染性。對流感病毒而光強度較弱,其原因可能與轉染效率有關,因五個質粒共轉染293T細胞需要五個質粒進入同一個細胞,此項工作較為困難;并且五質粒轉染時所加的轉染試劑對細胞有很大傷害;同時與宿主細胞內vRNPs表達效率有關。綜上所述,依賴輔助病毒或vRNPs的反向遺傳學操作系統均可驅動外源蛋白的表達,促進我們更進一步來研究流感病毒的反向遺傳學。言,最小病毒復制單位為vRNPs,它由聚合酶蛋白(PB2,PB1,PA)、核蛋白(NP)及vRNA 組成。此vRNPs既可以在細胞外[4],亦可在細胞內合成[7],但最終都進入細胞核,使得流感病毒RNA經復制、轉錄為 mRNA、cRNA 及負鏈vRNA。因此,vRNPs可作為病毒復制的前提,使得病毒蛋白在其基礎上表達。
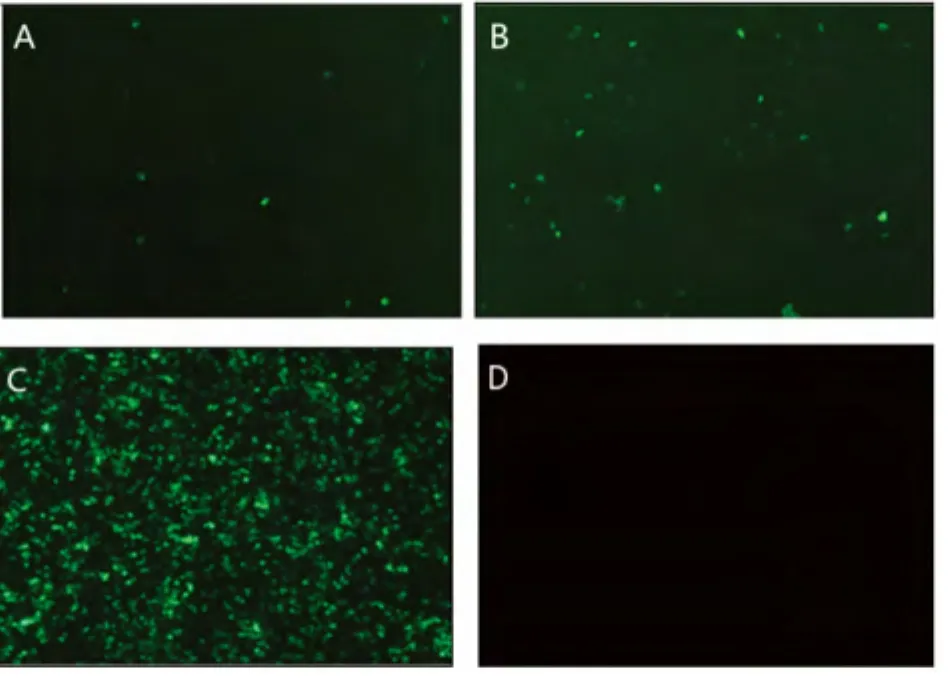
圖6 vRNPs系統轉染293T細胞48小時的綠色熒光蛋白表達Fig.6 The expression of Green Fluorescent protein at 48h post-transfection that vRNPs system.
本研究構建了依賴輔助病毒和宿主細胞內合成核糖核蛋白復合體兩個反向遺傳學操作系統,并在其基礎上表達外源綠色熒光蛋白。在實驗中,兩個反向遺傳學操作系統均可使綠色熒光蛋白表達,在熒光顯微鏡下均可觀察到熒光的發生,由此說明:本實驗所構建的聚合酶I系統在驅動外源蛋白在細胞內表達上是有效的。從圖片顯示,兩個轉染系統的熒光強度不高。由于POLI系統中的啟動子為人RNA聚合酶I啟動子,且RNA聚合酶I轉錄系統具有種屬特異性,但基于高等哺乳動物RNA POLI啟動子亦可推動低等哺乳動物RNA轉錄啟動的理論[14],本研究中兩個反向遺傳學系統均可驅動EGFP轉錄的啟動。同時,本實驗所構建的POLI系統中的啟動子序列前加有增強子,因此在宿主細胞中POLI系統的轉錄效率應較高,對熒光強度的影響較小。在依賴輔助病毒的轉染系統中,熒光強度不高的原因主要有:脂質體2000對MDCK細胞的轉染效率本就不高,轉染試劑亦對細胞有損傷;此外,因輔助病毒滴度較低,其對MDCK的感染效率也是一個因素。在細胞內合成vRNPs的轉染體系中,熒
[1]Hsu JT,Yeh JY,Lin TJ,et al.Identification of BPR3P0128as an inhibitor of cap-snatching activities of influenza virus[J].Antimicrob Agents Chemother,2012,56(2):647-657.
[2]Desselberger U,Racaniello V R,Zazra J J,and Palese P.The3′and 5′-terminal sequences of influenza A,B and C virus RNA segments are highly conserved and show partial inverted complementarity[J].Gene,8,1980,8(3):315-328.
[3]Wit E,Spronken MJ,Bestebroer TM,et al.Efficient generation and growth of influenza virus A/PR/8/34from eight cDNA fragments[J].Virus Res,2004,103(1-2):155-161.
[4]Luytjes W,Krystal M,Enami M,et al.Amplification,expression,and packaging of a foreign gene by influenza virus[J].Cell,1989,59(6):1107-1113.
[5]Enami M,Luytjes W,Krystal M,et al.Introduction of site-specific mutations into the genome of influenza virus[J].Proc Natl Acad Sci USA ,1990(10):3802-3805.
[6]Zobel A,Neumann G,Hobom G.RNA polymerase I catalysed transcription of insert viral cDNA[J].Nucleic Acids Res,1993,21(16):3607-3614.
[7]Neumann G,Zobel A,Hobom G.RNA polymerase immediated expression of influenza viral RNA molecules[J].Virol,1994,202(1):477-479.
[8]Pleschka S,Jaskunas SR,Engelhardt OG,et al.A plasmidbased reverse genetics system of influenza A virus[J].J Virol,1996(70):4188-4192.
[10]Lutz A,Dyall J,Olivo PD,Pekosz A.Virus-inducible reporter genes as a tool for detecting and quantifying influenza A virus replication[J].Journal of virological methods,2005,126(1-2):13-20.
[10]Neumann G,Watanabe T,Ito H,et al.Generation of influenza A viruses entirely from cloned cDNAs[J].Proc Natl A cad Sci,1999(96):9345-9350.
[11]Hoffmann E,Neumann G,Hobom G,et al."Ambisense"approach for the generation of influenza a virus:vRNA and mRNA synthesis from one template[J].Virol,2000,267(2):310-317.
[12]Fodor E,Devenish L,Engelhardt OG,et al.Rescue of influenza A virus from recombinant DNA[J].J Virol,1999(73):9679-9682.
[13]Zobel A,Neumann G,Hobom G.RNA polymerase I catalysed transcription of insert viral cDNA[J].Nucleic Acids Research,1993,21(16):3607-3614.
[14]Heix J,Grummt I.Species specificity of transcription by RNA polymerase I[J].Current Opinion in Genetics & Development,1995,5(5):652-656.

