應用微陣列芯片分析糖尿病大鼠下肢動脈硬化miRNAs差異表達譜*
孫 平, 肖 樂, 龔昆梅△, 王昆華
(1昆明理工大學醫學院, 2云南省第一人民醫院, 3昆明醫科大學第一附屬醫院,云南 昆明 650034)
?
應用微陣列芯片分析糖尿病大鼠下肢動脈硬化miRNAs差異表達譜*
孫平1,肖樂2,龔昆梅2△,王昆華3
(1昆明理工大學醫學院,2云南省第一人民醫院,3昆明醫科大學第一附屬醫院,云南 昆明 650034)
[摘要]目的: 通過建立糖尿病大鼠模型,檢測糖尿病大鼠動脈硬化下肢與糖尿病大鼠無動脈硬化下肢的動脈組織中微小RNAs(miRNAs)的差異表達情況,探究異常表達的miRNAs參與糖尿病大鼠下肢動脈硬化可能的分子機制。方法: 選取建模成功的患有糖尿病下肢動脈硬化和患有糖尿病但無下肢動脈硬化的大鼠,取出下肢動脈組織,分別提取總的miRNAs,用miRNAs微陣列芯片進行雜交檢測,經過芯片掃描和數據分析,再用RT-qPCR驗證芯片的掃描分析結果,最終獲得糖尿病大鼠下肢動脈硬化的miRNAs差異表達譜。結果: 篩選出10個與糖尿病大鼠下肢動脈硬化有關的miRNAs,即 rno-miR-206-3p、rno-miR-133a-5p、rno-miR-133b-3p、rno-miR-133a-3p、rno-miR-325-5p、rno-miR-675-3p、rno-miR-411-5p、rno-miR-329-3p、rno-miR-335和rno-miR-126a-3p,這10個異常表達的mi-RNAs都上調。RT-qPCR證實,其中9個miRNAs與芯片檢查結果一致,僅僅rno-miR-335的PCR檢測結果與基因芯片檢測結果相反,表達下調。結論: 有一群miRNAs在糖尿病大鼠下肢動脈硬化中起著重要作用,其表達過程很可能影響著動脈硬化的進程。
[關鍵詞]微小RNAs; 微陣列; 糖尿病; 下肢動脈硬化
微小RNAs(microRNAs,miRNAs)是一類21~25 個核苷酸組成的,不編碼蛋白質的,多物種高度保守性、時序性和組織特異性表達的,通過抑制靶mRNA翻譯或促其降解而參與控制基因表達的非編碼小RNA,作為生物體正常生長發育的重要基因調控分子備受關注。現在許多研究證實,糖尿病、多種腫瘤、心血管疾病、動脈粥樣硬化等都存在miRNAs的異常表達或缺失[1-7],miRNAs很可能參與了這些疾病發生的基因調控。臨床中發現許多糖尿病下肢動脈硬化病人,表現為間隙性跛行、休息痛和缺血性壞疽,與單純的僅患有下肢動脈硬化的病人比較,病情嚴重,更加不易治療,治療效果差,容易反復。為了準確了解其發病機制,做好對糖尿病下肢動脈硬化的有效預防和治療,我們搜索了相關文獻,可是迄今為止尚未見到有關miRNAs表達譜在糖尿病下肢動脈硬化中被研究及臨床應用的報道。因此為了補充這方面研究,我們首先建立動物模型,選取建模成功的患有糖尿病下肢動脈硬化(diabetic rats with lower limb arteriosclerosis,DAS)和患有糖尿病但無下肢動脈硬化(diabetic rats with normal lower limb,DN)的大鼠,利用微陣列技術平臺[8-10],在髂動脈組織中篩查miRNAs異常表達譜,為進一步研究miRNAs與人患有糖尿病下肢動脈硬化的發病關系提供動物實驗基礎。
材料和方法
1材料
SD大鼠購自常州卡文斯實驗動物有限公司。微陣列芯片選取建模成功后的DN大鼠3只和DAS大鼠3只,DN為對照組,DAS為實驗組。根據用于RNA抽提的組織樣品采集規范流程分別采取其下肢動脈組織,迅速置于液氮中(-196 ℃),用于以后分子實驗。miRNAs微陣列芯片和質控探針由Affymetrix提供,采用的芯片型號為Affymetrix GeneChip miRNA 4.0 Array,其miRNA探針序列信息來源于最新Sanger miRBase 20.0數據庫(http://microrna.sanger.ac.uk/sequences/),數據庫收錄了來自約40篇新文獻報道的3 355條新的發夾前體序列和5 393條新的成熟體miRNA,其中大鼠miRNA增至728條。
2方法
2.1樣本總miRNA的抽提根據Invitrogen TRIzol說明書分別提取DN大鼠和DAS大鼠的總RNA,然后用QIAGEN RNeasy Mini Kit進行純化,將總RNA通過微離心過濾柱得到miRNA,樣品量至少2 μg。
2.2樣本總miRNA質量檢測(1) NanoDrop 2000紫外分光光度計檢測樣品總miRNA在260 nm和280 nm下的吸光度(A)值,檢測miRNA濃度和純度,按以下公式計算總 RNA 的濃度,即總 RNA 濃度(mg/L)=A260×稀釋倍數×40 mg/L,以A260/A280值來檢測總RNA 的純度,A260/A280值在 1.9~2.1之間時,可認為 RNA 的純度較好。(2) 瓊脂糖凝膠電泳檢測miRNA的完整性,1.5%瓊脂糖凝膠電泳檢測,28S和18S核糖體RNA條帶非常亮而濃,上面一條帶(28S)的密度大約是下面一條帶(18S)的2倍。
2.3樣品miRNA熒光標記進行Poly (A) 加尾,FlashTag Biotin HSR 連接,生物素標記。
2.4微陣列芯片雜交、芯片掃描及數據分析把生物素標記好的樣品加入到配置好的雜交液中(2×hybridization, 27.5% formamide, DMSO, 20× eukaryotic hybridization controls, control oligonucleotide B2),離心PCR孵育后再離心,將雜交液注入芯片中,把芯片放在雜交爐架子上后放在雜交爐里雜交,每個樣品單獨雜交一張芯片。經過一番洗滌操作后,把芯片放在 Affymetrix 的掃描儀中,點擊Affymetrix Launcher 中的 AGCCScan Control,點擊工具欄中的 Start進行芯片掃描生成CEL原始數據文件數據,然后運用Array-Pro(MediaCyber-netics)軟件對雜交圖像進行數字化轉換,獲得實驗組與對照組檢測信號的比值(log2)和t檢驗的P值,篩選出有意義的差異表達miRNAs。其中為了得到芯片合理準確的質量評估,對每種類型的樣本進行不少于3次重復,同時為了保證芯片在探針集水平的評估結果,芯片內每個探針雜交布置5個重復,所得數據用SAM法進行分析[11]。
2.5RT-qPCR驗證查閱相關文獻[12]根據miRNA基因芯片檢測結果,對篩選出的差異表達的10個miRNA進行進一步驗證,內參照選擇snRNA U6(表1)。cDNA序列的翻譯、酶切位點分析、引物設計以及擴增產物、熔解曲線都是運用生物分析軟件DNAMAN(5.0版)所得。反轉錄程序: 25 ℃ 5 min; 50 ℃ 60 min; 70 ℃ 15 min。PCR反應程序(擴增和熔解曲線): 95 ℃ 10 s; 60 ℃ 30 s, 70 ℃ 45 s,重復40個循環。在PCR反應體系中加入過量SYBR染料,按Ct值來計算各基因的相對表達水平。
3統計學處理
用統計軟件包SPSS 10.0進行統計學分析。多組間比較采用單因素方差分析,兩兩比較采用q檢驗,以P<0.05為差異有統計學意義。
結果
1總RNA提取質量分析
瓊脂糖凝膠電泳后有清晰的28S和18S兩條條帶,上面一條帶(28S)的密度大約是下面一條帶(18S)的2倍,低分子量RNA(tRNA和5S核糖體RNA等)條帶微弱。由表2可知,所測RNA總量和濃度滿足儀器上樣(miRNA芯片要求:濃度不低于50 mg/L,總量不低于1 μg),A260/A280值介于1.9~2.1之間,質檢綜合評定合格,可以繼續進行miRNA芯片檢測。

表1 用于RT-qPCR驗證的引物序列
R: reverse; F: forward.
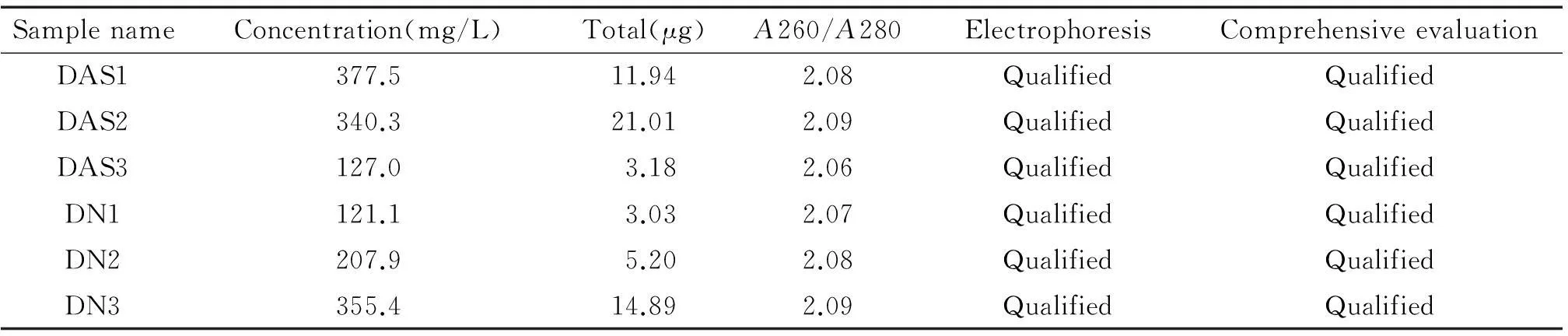
表2 總RNA樣本質檢報告
2miRNA微陣列芯片表達譜分析結果
微陣列芯片雜交結果分析顯示,與患有糖尿病但無下肢動脈硬化的大鼠相比,患有糖尿病下肢動脈硬化的大鼠下肢動脈組織中異常表達的miRNA有10個,分別是rno-miR-206-3p、rno-miR-133a-5p、rno-miR-133b-3p、rno-miR-133a-3p、rno-miR-325-5p、rno-miR-675-3p、rno-miR-411-5p、rno-miR-329-3p、rno-miR-335和rno-miR-126a-3p,均為表達上調,其中上調明顯的是rno-miR-206-3p、rno-miR-133a-5p和rno-miR-675-3p,見圖1、表3。
3RT-qPCR驗證miRNA基因芯片差異表達譜結果
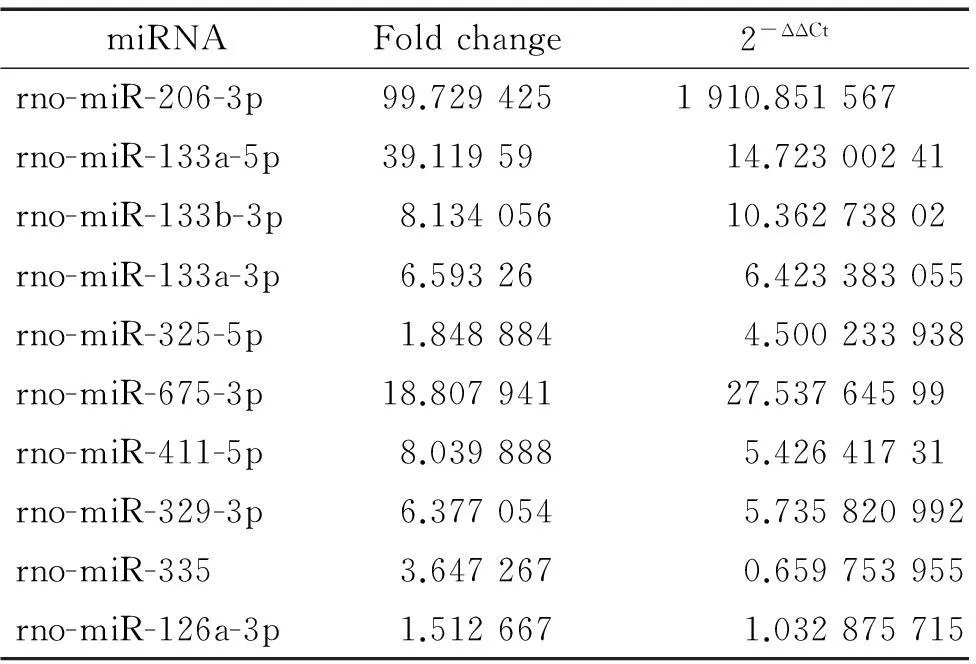
miRNA基因芯片中這10種miRNA在組織中表達差異較大,相比DN,都在DAS組織中表達上調。運用RT-qPCR技術對miRNA基因芯片篩選出差異表達的10個miRNAs進行驗證工作,設定miRNA在DN組織中表達量為對照,計算miRNA在DAS組織內相對表達水平(圖2)。對比DAS與DN miRNA的RT-qPCR結果與基因芯片結果,這10種miRNA的PCR檢測結果與基因芯片結果雖然在數值上有些差異,但是趨勢基本一致,僅僅rno-miR-335的PCR檢測結果與基因芯片檢測結果相反,為表達下調,見圖3、表4,所以miRNA芯片結果基本可靠。RT-qPCR驗證也顯示rno-miR-206-3p、rno-miR-133a-5p和rno-miR-675-3p在DAS下肢動脈組織高表達。
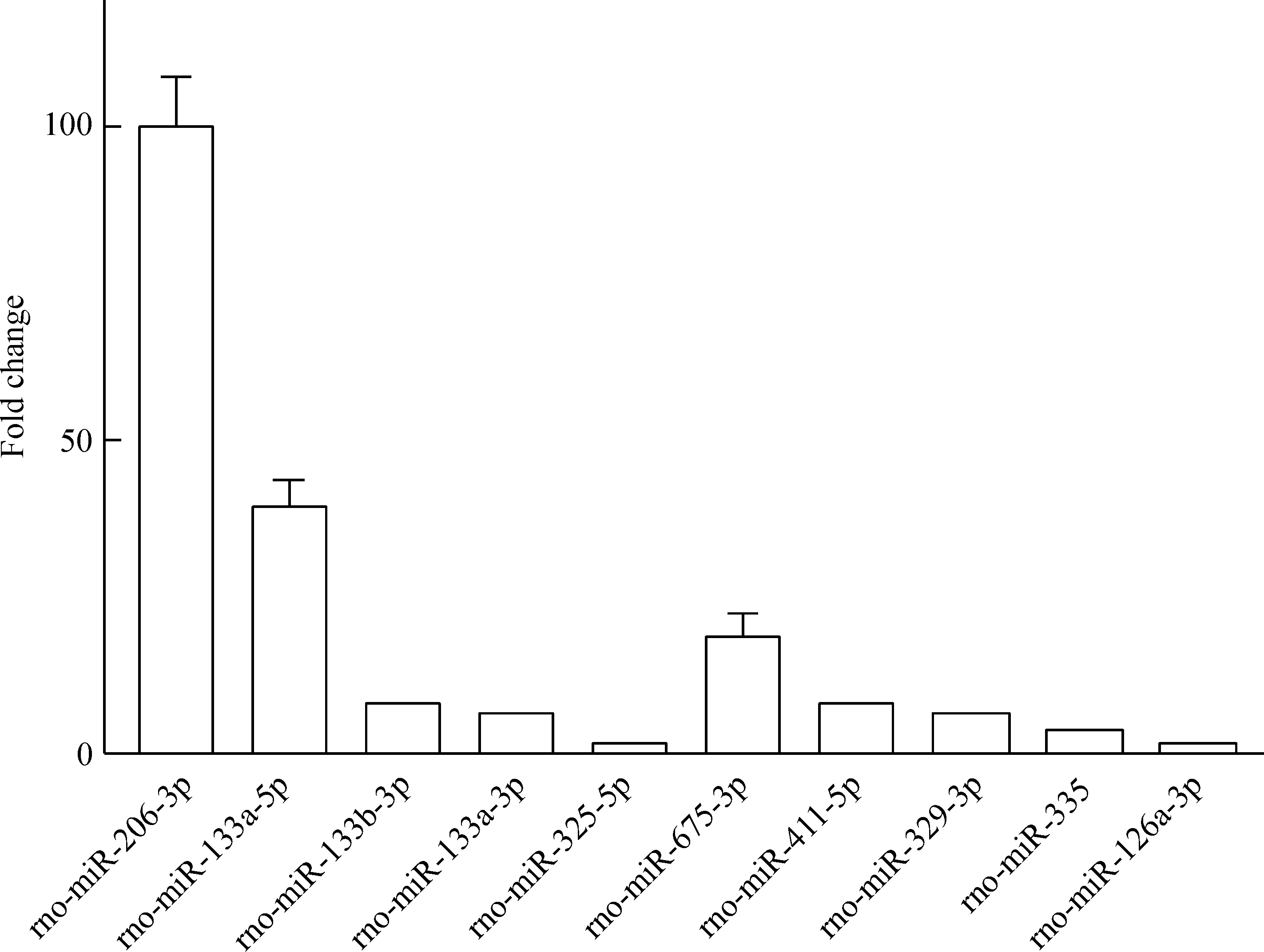
Figure 1. Differentially expressed miRNAs detected by microRNA chips. Mean±SD.n=3.
圖1芯片中miRNA的差異表達
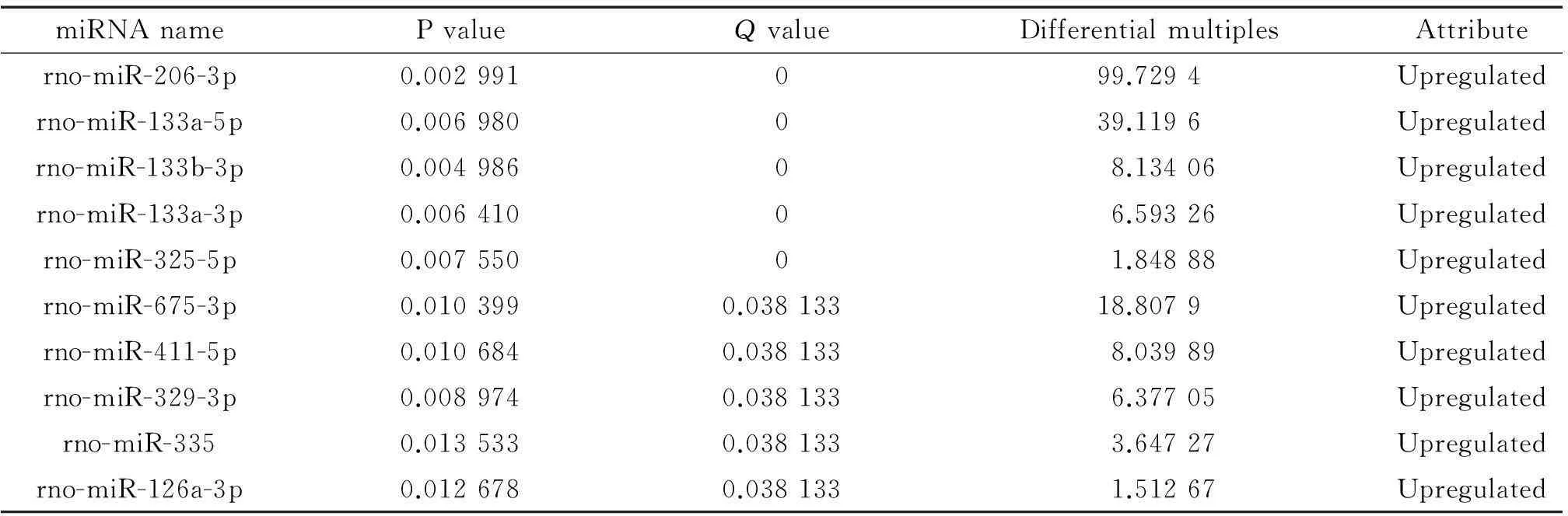
表3 DAS中異常miRNA表達譜
討論
查閱相關文獻,報道克隆技術和Northern blot分析技術也經常運用于miRNAs的確認[13],且費用相對來說比較低,可是對大量miRNA的差異表達譜篩選,二者存在著工作量大,靈敏度低的缺點,難以在單一一次實驗中分析出數百個miRNA的差異表達情況,非常不便于對差異基因的篩選。隨著能同時檢測成百上千差異基因的高通量基因芯片技術的普及與推廣,我們選擇Affymetrix提供的miRNA基因芯片技術,采用的芯片型號為Affymetrix GeneChip miRNA 4.0 Array,其miRNA探針序列信息來源于最新Sanger miRBase 20.0數據庫(http://microrna.sanger.ac.uk/sequences/),能夠檢測約28 700個注釋完好基因的約31 000個轉錄本。其中為了得到芯片合理準確的質量評估,每種類型的樣本不少于3次重復,同時為了保證芯片在探針集水平的評估結果,芯片內每個探針雜交布置5個重復,運用了μParaflo?技術,芯片內和不同芯片間陣列點的一致性都非常好,所以miRNA芯片是研究大量樣本和樣本中大量miRNA最常用的高通量方法[14-15]。
雖然高通量技術檢測出來的信息穩定性差,錯誤率高,且費用也比較昂貴,但是隨著近幾年基因芯片技術的發展,數據庫的增強,芯片數據準確率逐年攀升。因為芯片存在較大錯誤率,所以還需RT-qPCR和Northern blot實驗的驗證,對后續功能檢測確定,這個步驟也是非常關鍵和必要的。
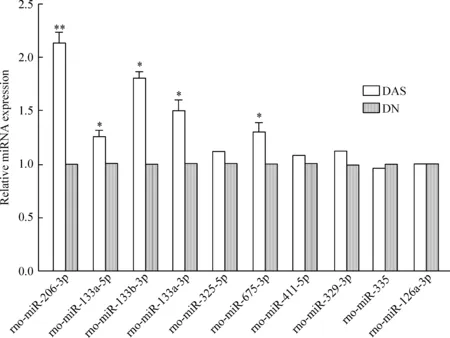
Figure 2. The expression levels of miRNAs determined by RT-qPCR detection. Mean±SD.n=3.*P<0.05,**P<0.01 vs DN.
圖2RT-qPCR檢測 miRNA相對表達水平
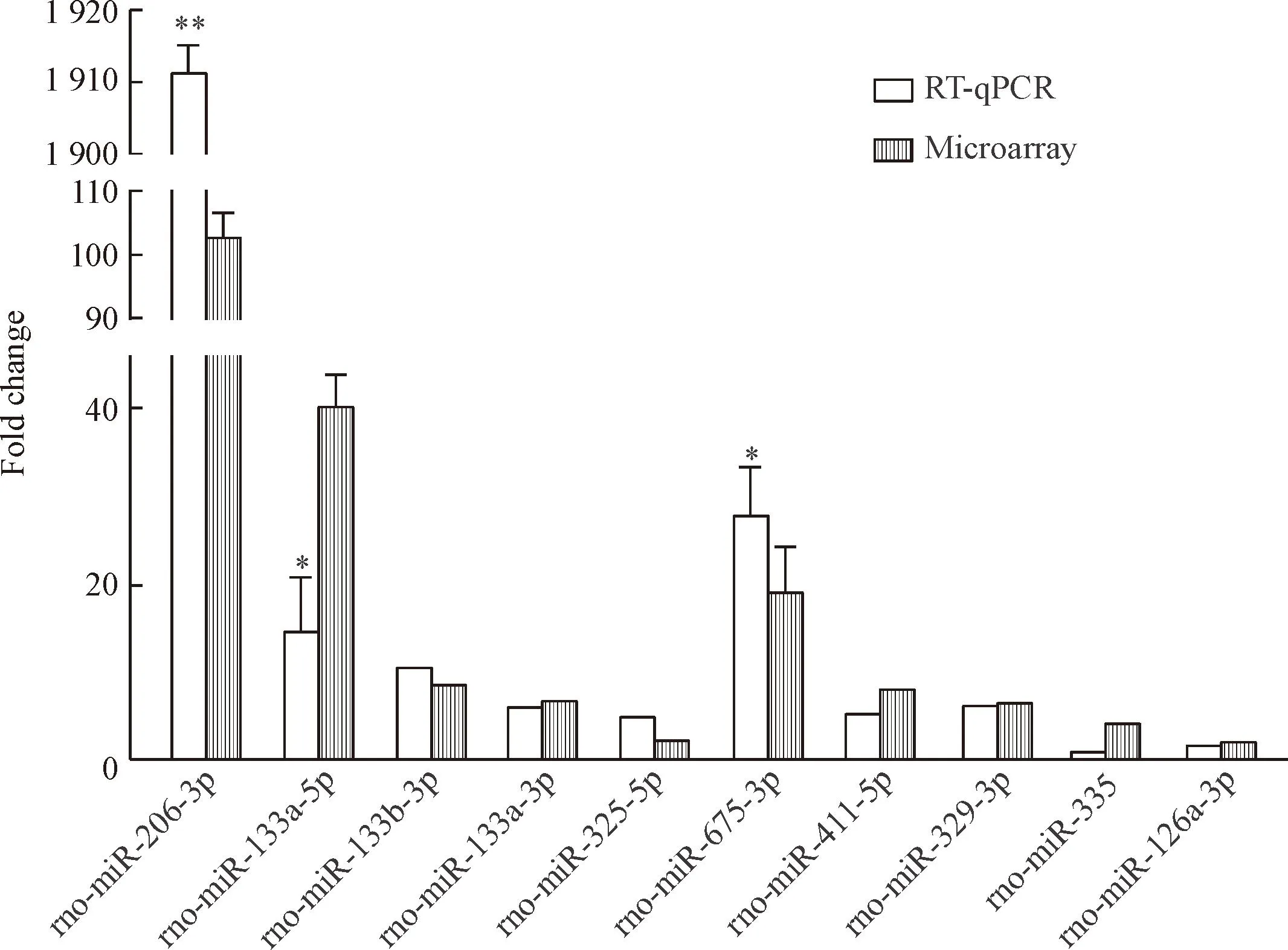
Figure 3. Comparison of the microarray and RT-qPCR results. Mean±SD.n=3.*P<0.05,**P<0.01 vs microarray.
圖3芯片和RT-qPCR結果的對比
由于動脈硬化具體詳細的發病機制還未闡明,同時鮮有關于糖尿病下肢動脈硬化的miRNA差異表達報道,本實驗率先將miRNA芯片技術應用到糖尿病下肢動脈硬化發病機制的研究中去,做出差異表達譜,篩選出差異表達的miRNAs,為研究糖尿病下肢動脈硬化發病機制提供了重要實驗補充。本次實驗數據來自動物模型不同的病例樣本,選自所有芯片表達趨勢一致的差異miRNA,差異具有統計學顯著性。基因芯片顯示有10個明顯差異表達的miRNA,RT-qPCR驗證與芯片趨勢一致,只是rno-miR-335在實時定量PCR中表達為下調,與芯片結果相反。進行RT-qPCR數據與基因芯片對比,DAS相對DN miRNA差異表達最為明顯的是rno-miR-206-3p、 rno-miR-133a-5p和rno-miR-675-3,為表達明顯上調。基于篩選出的關鍵miRNA,利用網上共享數據庫Gene Ontology(GO)分析(http://www.geneontology.org)進行GO分類,包括GO分析、Passway分析、基因網絡分析、GSEA分析和KEGG Pathway分析和了解相關的信號通路和基因功能,后期將針對關鍵miRNA構建miRNA擬似物和miRNA抑制物寡核苷酸,進行過表達和抑制實驗。根據現有實驗數據顯示,有一群miRNAs在糖尿病大鼠患有下肢動脈硬化中起著重要作用,其表達過程很可能影響著血管動脈硬化的進程。本次實驗數據為以后進行miRNA靶向調節基因表達研究,闡明糖尿病下肢動脈硬化發病基因調控機制提供了重要實驗數據和理論依據。
表4RT-qPCR檢測結果與miRNA芯片結果比較
Table 4.The results of RT-qPCR detection (2-ΔΔCt) for validating the results of microarray
miRNAFoldchange2-ΔΔCtrno-miR-206-3p99.7294251910.851567rno-miR-133a-5p39.1195914.72300241rno-miR-133b-3p8.13405610.36273802rno-miR-133a-3p6.593266.423383055rno-miR-325-5p1.8488844.500233938rno-miR-675-3p18.80794127.53764599rno-miR-411-5p8.0398885.42641731rno-miR-329-3p6.3770545.735820992rno-miR-3353.6472670.659753955rno-miR-126a-3p1.5126671.032875715
[參考文獻]
[1]Ye S, Yang L, Zhao X, et al. Bioinformatics method to predict two regulation mechanism: TF-miRNA-mRNA and lncRNA-miRNA-mRNA in pancreatic cancer[J]. Cell Biochem Biophys, 2014, 70(3):1849-1858.
[2]Bommer GT, Gerin I, Feng Y, et al. p53-mediated activation of miRNA34 candidate tumor-suppressor genes[J]. Curr Biol, 2007, 17(15):1298-1307.
[3]Baranwal S, Alahari SK. miRNA control of tumor cell invasion and metastasis[J]. Int J Cancer, 2010, 26(6):1283-1290.
[4]Cha YH, Kim NH, Park C, et al. MiRNA-34 intrinsically links p53 tumor suppressor and Wnt signaling[J]. Cell Cycle, 2012,11(7):1273-1281.
[5]王霞,杜杰, 李匯華. miRNA-155在心血管疾病中的作用[J].生理科學進展, 2013, 44(5):377-380.
[6]Figueira MF, Monnerat-Cahli G, Medei E, et al. MicroRNAs: potential therapeutic targets in diabetic complications of the cardiovascular and renal systems[J]. Acta Physiol (Oxf), 2014, 211(3):491-500.
[7]Li T, Yang GM, Zhu Y, et al. Diabetes and hyperlipidemia induce dysfunction of VSMCs: contribution of the metabolic inflammation/miRNA pathway[J]. Am J Phy-siol Endocrinol Metab, 2015, 308(4):E257-E269.
[8]Ramírez-Gordillo D, Powers TR, van Velkinburgh JC, et al. RNA-Seq and microarray analysis of theXenopusinner ear transcriptome discloses orthologous OMIM? genes for hereditary disorders of hearing and balance[J]. BMC Res Notes, 2015, 8:691.
[9]Ashaari NS, Ramarad S, Khairuddin D, et al. Development of repeatable arrays of proteins using immobilized DNA microplate (RAPID-M) technology[J]. BMC Res Notes, 2015, 8:669.
[10]Fraiture MA, Herman P, Taverniers I, et al. Current and new approaches in GMO detection: challenges and solutions[J]. Biomed Res Int, 2015,2015:392872.
[11]Tusher V, Tibshirani R, Chu G. Significance analysis of microarrays applied to the ionizing radiation response[J]. Proc Natl Acad Sci U S A, 2001, 98(9):5116-5121.
[12]Sempere LF, Freemantle S, Pitha-Rowe I, et al. Expression profiling of mammalian microRNAs uncovers a subset of brain-expressed microRNAs with possible roles in murine and human neuronal differentiation[J]. Genome Biol, 2004, 5(3):R13.
[13]Kim VN. MicroRNA biogenesis: coordinated cropping and dicing[J]. Nat Rev Mol Cell Biol, 2005, 6:376-385.
[14]Connon SA, Giovannoni SJ. High-throughput methods for culturing microorganisms in very-low-nutrient media yield diverse new marine isolates[J] Appl Environ Microbiol, 2002, 68(8):3878-3885.
[15]Persson KE, Lee CT, Marsh K, et al. Development and optimization of high-through put methods to measure plasmodium falciparum-specific growth inhibitory antibodies[J]. J Clin Microbiol, 2006, 44(5):1665-1673.
(責任編輯: 陳妙玲, 羅森)
Microarray-based miRNAs profiling in lower limb arteriosclerosis of diabetic rats
SUN Ping1, XIAO Le2, GONG Kun-mei2,
(1MedicalFacultyofKunmingUniversityofScienceandTechnology,2FirstPeople′sHospitalofYunnanProvince,3FirstAffiliatedHospitalofKunmingMedicalUniversity,Kunming650034,China.E-mail:kunhuagongkunmei@163.com)
[ABSTRACT]AIM: To establish the profiling of microRNAs (miRNAs) in the lower extremity arterial tissue between diabetic rats with lower limb arteriosclerosis (DAS) and diabetic rats with normal lower limb (DN), and to explore the possible molecular mechanisms involved in aberrant miRNA expression in DAS. METHODS: The rat models of DAS and DN were successfully established. The respective lower extremity arterial tissue was isolated. The total miRNAs were purified for a hybridization detection by miRNA microarray. The results of chip scanning and data were analyzed and verified by RT-qPCR. RESULTS: Ten miRNAs related to DAS, including rno-miR-206-3p, rno-miR-133a-5p, rno-miR-133b-3p, rno-miR-133a-3p, rno-miR-325-5p, rno-miR-675-3p, rno-miR-411-5p, rno-miR-329-3p, rno-miR-335 and rno-miR-126a-3p, were determined. All 10 abnormally expressed miRNAs were up-regulated. The validating results of RT-qPCR confirmed 9 of the miRNAs in line with chip expression. Just rno-miR-335 showed the opposite between PCR detection and microarray result. CONCLUSION: A group of miRNAs in diabetic rats suffering from lower limb arteriosclerosis plays an important role in the vascular atherosclerosis process. The abnormal expression of miRNAs is likely to affect the vascular atherosclerosis process.
[KEY WORDS]MicroRNAs; Microarray; Diabetes mellitus; Lower limb arteriosclerosis
[文章編號]1000- 4718(2016)05- 0917- 06
[收稿日期]2015- 11- 27[修回日期] 2016- 02- 16
*[基金項目]國家自然科學基金資助項目(No. 81260066; No. 81360069); 云南省科技廳-昆明醫科大學聯合專項基金資助項目(No. 2012FB089; No. 2013FB199)
通訊作者△Tel: 0871-63638564; E-mail: kunhuagongkunmei@163.com
[中圖分類號]R587.1; R363
[文獻標志碼]A
doi:10.3969/j.issn.1000- 4718.2016.05.025
雜志網址: http://www.cjpp.net
·短篇論著·

