甘肅高山細毛羊和湖羊HIF-2α基因遺傳特性與高原低氧適應性分析
黨鵬舉,李少斌,王繼卿,劉 秀,洪 魏,胡 江,羅玉柱
(1.甘肅省草食動物生物技術重點實驗室,甘肅省牛羊基因改良工程實驗室,甘肅農業大學 動物科學技術學院,甘肅蘭州 730070;2.浙江賽諾生態農業有限公司,浙江臨安 311311)
2.2 SSCP檢測結果
甘肅高山細毛羊和湖羊HIF-2α基因遺傳特性與高原低氧適應性分析
黨鵬舉1,李少斌1,王繼卿1,劉 秀1,洪 魏2,胡 江1,羅玉柱1
(1.甘肅省草食動物生物技術重點實驗室,甘肅省牛羊基因改良工程實驗室,甘肅農業大學 動物科學技術學院,甘肅蘭州 730070;2.浙江賽諾生態農業有限公司,浙江臨安 311311)
為探討HIF-2α基因核苷酸序列變異與綿羊高原適應的相關性,以540只(甘肅高山細毛羊200只,湖羊340只)綿羊為研究對象,采用PCR-SSCP技術分析HIF-2α基因第11外顯子和內含子部分區段在甘肅高山細毛羊和湖羊中單核苷酸多態性(SNPs)。結果顯示:HIF-2α基因擴增片段在2個綿羊品種中共檢測到4種等位基因(A、B、C和D)和10種基因型(AA、BB、CC、DD、AB、AC、BC、AD、BD和CD),存在6個SNPs位點,其中外顯子11檢測到5個SNPs位點(G/A、G/C、C/T、T/C、G/A、),內含子11檢測到1個SNPs位點(G/A),甘肅高山細毛羊未檢測到DD基因型。2個綿羊品種的優勢基因型均為AB,基因型頻率分別為34.50%和16.47%。HIF-2α基因在2個綿羊品種中具有豐富的多態性,且等位基因頻率在品種間存在極顯著差異(P<0.01)。推測在高海拔低氧適應過程中,HIF-2α基因的基因型可能受到了選擇,隨著長期的高海拔低氧環境的選擇,有利于低氧適應的基因型在高海拔綿羊品種中受到選擇并富集,攜帶基因型AA、BB和AB的綿羊可能更加適應高海拔低氧環境,而攜帶基因型CD、BD和DD的綿羊對高海拔低氧環境適應的能力可能較差。
HIF-2α基因;甘肅高山細毛羊;適應性;多態性
低氧誘導因子(Hypoxia-inducible factor,HIFs)是一種氧依賴的轉錄激活因子,在缺氧調節過程中發揮核心作用,由一個調節表達的亞基(HIF-α)和一個持續表達的亞基(HIF-β)組成[1],同時結合著一個缺氧反應元件(Hypoxia response element,HRE)。它是由Semenza等[2]于1992年發現的,與生物體的生長、發育及一些疾病的發病機理都存在著密切的關系。HIF的活性主要由α亞基決定,而α亞基的穩定性、DNA結合活性和轉錄又受到嚴格調控,其中氧誘導的羥化酶作用是最主要的調控途徑。低氧時,脯氨酸羥化酶功能喪失,不能使HIF-α亞基羥基化,從而導致HIF-α降解受阻而聚集增多;常氧條件下,脯氨酸羥化酶使HIF-α亞基羥基化,最終導致 HIF-α亞基降解[3]。目前,發現的HIF-α亞基主要有3種:HIF-1α、HIF-2α和HIF-3α[4]。HIF-2α是由Tian等[5]克隆出的HIF家族新成員,與HIF-1α具有48%的結構同源性。Yi等[6]發現HIF-2α是藏族與漢族多種基因頻率差別最大的基因,而且相關性分析發現HIF-2α與藏族血紅蛋白濃度呈顯著負相關,提示該基因參與了藏族的高原適應過程。綿羊的HIF-2α基因位于3號染色體上,有15個外顯子,全長44 252 bp,編碼843個氨基酸;山羊的HIF-2α基因位于11號染色體上,有15個外顯子,全長42 250 bp,編碼858個氨基酸。吳曉云[7]分析牦牛HIF-2α基因的SNPs及在組織中的表達,結果表明3個牦牛群體中的3個SNPs位點均處于高度連鎖狀態,形成5種基因型;HIF-2α基因mRAN在牦牛和高原地區黃牛的7個組織中均有表達,且表達量差異極顯著(P<0.01)。
湖羊主要飼養在江蘇、浙江兩省交界的太湖流域一帶,氣候濕潤,雨量充沛,年平均氣溫為15~16℃,海拔1 000 m以下。甘肅高山細毛羊屬毛肉兼用細毛羊品種,分布在高寒地區。海拔高,干旱少雨,年平均氣溫為1.9℃,海拔2 400~4 070 m;甘肅高山細毛羊能在高原寒冷、低氧的嚴酷環境中正常的生長發育。目前對HIF-2α基因的研究主要集中于該基因與人類癌癥的相關性,而對綿羊HIF-2α基因的研究報道相對較少。因此,本試驗以生活在低海拔的湖羊為對照,甘肅高山細毛羊為研究對象,采用PCR-SSCP方法,研究HIF-2α基因第11外顯子和內含子部分區段的SNPs和遺傳特征,為分析綿羊HIF-2α基因與高原適應性提供基礎理論數據。
1 材料和方法
1.1 試驗材料
選取2個綿羊品種的540個樣本,分別是湖羊340只,甘肅高山細毛羊200只。頸靜脈采血,酸性檸檬酸葡萄糖溶液(ACD)抗凝,-20℃冷凍保存,部分血樣滴到FTA卡(Whatman BioScience,M iddlesex,UK)上干燥保存。
1.2 試驗方法
1.2.1 基因組DNA的提取 用Zhou等[8]描述的兩步法提取綿羊血樣基因組DNA,冷凍保存的血樣用常規苯酚-氯仿抽提法提取基因組DNA,用于PCR擴增。
1.2.2 引物設計及PCR擴增 參照GenBank中綿羊HIF-2α基因第11外顯子和內含子部分區段的核苷酸序列(登錄號:NC_019460.1),用DNAMAN軟件設計特異引物,預計擴增目的片段約為470 bp。上游引物序列:5′-GAGAGGACTTCCAGCTTAGC-3′,下游引物序列:5′-TCCCCTATGCAGCCCAGC-3′,引物由北京六合華大基因科技股份有限公司合成。
PCR擴增采用20μL的體系:Taq預混酶10μL(北京百泰克生物技術有限公司),上下游引物各0.4μL,模板DNA 1μL(或1.2 mm FTA卡小圓片1個),ddH2O 8.2μL。退火溫度為61℃。PCR產物用1%的瓊脂糖凝膠電泳檢測。
1.2.3 SSCP檢測 取2μL的PCR產物加入9μL的變性上樣緩沖液(980 m L/L去離子甲酰胺,0.25 g/L溴酚藍,0.25 g/L二甲苯青,pH值8.0,0.01 mol/L EDTA),98℃變性10 m in,立即冰浴10 m in。用膠聯度39∶1、濃度14%的非變性聚丙烯酰胺凝膠,220 V、27℃電泳20 h,根據Byun等[9]描述的方法銀染聚丙烯酰氨凝膠,直至條帶清晰后對基因型判型,并用干膠儀干燥于A4紙保存。
1.2.4 序列分析與數據統計 將不同基因型樣品的PCR產物送生工生物工程(上海)股份有限公司進行測序。純合子直接測序,雜合子按照Gong等[10]描述的方法切膠測序。利用MEGA 5.0軟件進行等位基因核苷酸序列比對及分析,采用Popgene32軟件計算等位基因頻率、基因型頻率、純合度(Homozygosity,Ho)、雜合度(Heterozygosity,He),有效等位基因數(Effective number of alleles,Ne)和χ2檢驗,采用PIC軟件計算多態性信息含量(Polymorphism information content,PIC)。
2 結果與分析
2.1 PCR擴增結果
分別擴增甘肅高山細毛羊和湖羊HIF-2α基因的第11外顯子和內含子部分序列,并用1%的瓊脂糖凝膠檢測(圖1),目的條帶清晰無雜帶,與預期擴增片段長度相符,可直接用于SSCP分析。

圖1 綿羊H IF-2α基因外顯子11 PCR產物瓊脂糖檢測Fig.1 Agarose gel check of the PCR am p licons of H IF-2αgene for the exon 11 in sheep
2.2 SSCP檢測結果
甘肅高山細毛羊和湖羊群體HIF-2α基因第11外顯子和內含子部分區段的PCR擴增產物SSCP電泳帶型見圖2。2個綿羊群體540份樣品共檢測出10種SSCP帶型,分別代表4種等位基因:A、B、C、D,形成10種基因型:AA、BB、CC、DD、AB、AC、BC、AD、BD和CD。其中甘肅高山細毛羊未檢測出DD基因型。
2.3 H IF-2α基因不同等位基因序列比對

圖2 綿羊H IF-2α基因外顯子11 PCR-SSCP檢測Fig.2 Detection of H IF-2αgene for the exon 11 in sheep by PCR-SSCP

圖3 綿羊H IF-2α基因目的片段等位基因核苷酸序列比對Fig.3 Nucleotide sequences alignm ent the aim gene fragm ent of H IF-2αgene in sheep
以GenBank提供的HIF-2α基因序列(NC-019460.1)為模板,用MEGA 5.0軟件對擴增區域的各等位基因進行比對(圖3)。結果表明,該引物擴增區域等位基因B序列與原序列NC-019460.1相同,等位基因A、D各檢測到1個SNPs位點,分別是221位C>T的轉換,38位G>A的轉換;等位基因C檢測到5個SNPs位點,分別是38位G>A的轉換,71位G>C的轉換,213位T>C的轉換,425和450位的G>A的轉換。
2.4 H IF-2α基因遺傳多態性
2.4.1 基因頻率和基因型頻率 2個綿羊品種HIF-2α基因第11外顯子和內含子擴增區域的基因頻率和基因型頻率見表1,2。由表1可知,2個綿羊品種中均檢測出A、B、C和D 4種等位基因,其中甘肅高山細毛羊的等位基因頻率從大到小依次為A(43.5%)、B(41.00%)、C(12.50%)和D(3.00%),湖羊的等位基因頻率從大到小依次為B(32.15%)、A(27.59%)、D(23.89%)和C(16.37%);經χ2檢驗顯示,HIF-2α基因第11外顯子和內含子擴增片段的基因頻率在甘肅高山細毛羊和湖羊間存在明顯差異(P=0.001)。由表2可知,甘肅高山細毛羊檢測到AA、BB、CC、AB、AC、BC、AD、BD和CD 9種基因型,未檢測到DD基因型,而湖羊檢測到包括DD基因型在內的10種基因型。2個綿羊品種的優勢基因型均為AB,基因型頻率分別為34.50%和16.47%。經χ2適應性檢驗,2個綿羊品種均達到Hardy-Weinberg平衡狀態(P>0.05)。

表1 綿羊H IF-2α基因第11外顯子的等位基因頻率Tab.1 A llele and genotype frequencies of H IF-2αfor the exon 11 in sheep
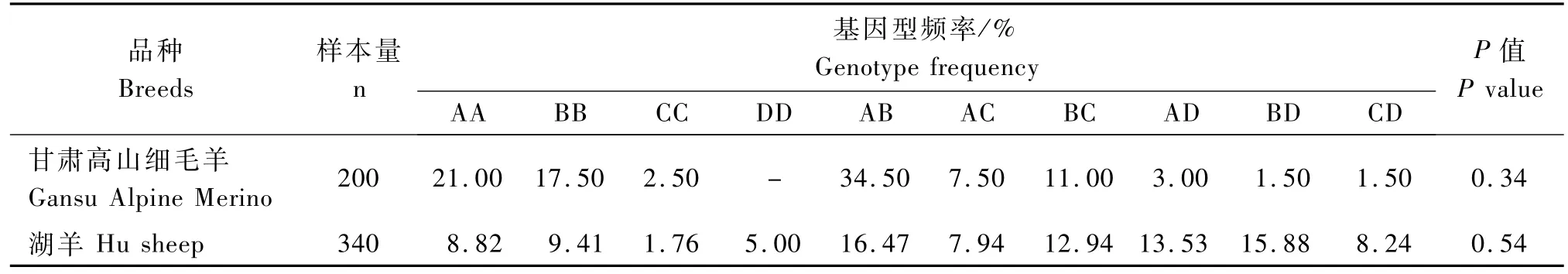
表2 綿羊H IF-2α基因第11外顯子的基因型頻率Tab.2 Genotype frequencies of H IF-2αfor the exon 11 in sheep
2.4.2 基因的純合度、雜合度、有效等位基因數和多態性信息含量 遺傳純合度(Ho)、有效等位基因數(Ne)和多態性信息含量(PIC)都是度量群體內遺傳變異的參數,群體間不同遺傳變異參數體現各群體遺傳差別,期望雜合度(He)越高,遺傳純合度(Ho)越低,說明遺傳變異越大,對環境有較強的適應能力。甘肅高山細毛羊和湖羊HIF-2α基因第11外顯子和內含子擴增區域的遺傳參數見表3。由表3知,2個綿羊品種的雜合度(He)分別為0.59和0.75,遺傳純合度(Ho)分別為0.41和0.25,表明遺傳多樣性豐富,遺傳變異程度最高,具有較大的遺傳潛力。甘肅高山細毛羊和湖羊的PIC值分別為0.55和0.69,均大于0.5,均屬于高度多態,表明這2個綿羊品種遺傳變異大,可用于動物遺傳育種研究。

表3 不同綿羊品種基因純合度、雜合度、有效等位基因數和多態性信息含量Tab.3 Ho,He,Ne and PIC of different sheep breeds
3 討論
HIF-2α是缺氧誘導因子(HIFs)家族的重要成員之一,主要作用的靶基因(如促紅細胞生成素(EPO))中都含有缺氧反應原件(HRE),通過識別核心序列5′-TACGTGCT-3′與HRE結合,誘導靶基因的轉錄,介導細胞對缺氧的應答[11-12]。研究表明HIF-2α基因不僅對EPO有調節作用[13],對血管內皮生長因子(VEGF)的調節作用更強,有較強的轉錄激活VEGF的能力[14-15]。低氧時,HIF-2α亞基降解受阻而聚集增多,調節低氧組織VEGF的增加,促進血管生成,有利于機體適合低氧環境,保證氧氣和營養物質通過血管輸送給低氧細胞[16]。Beall等[17]采用全基因組等位基因分化掃描(GWADS)法在世居藏族人(海拔4 200 m)中發現了31個連鎖不平衡的HIF-2α位點,這些位點與高原世居藏族人低Hb濃度有關。
單核苷酸多態(SNPs)是指在基因水平上單個核苷酸變異引起的DNA序列多態性,包括堿基替換而不包括堿基插入和缺失[18]。本試驗以甘肅高山細毛羊和湖羊2個生活在完全不同生境中的綿羊品種為研究對象,采用PCR-SSCP技術檢測HIF-2α基因第11外顯子和內含子部分區域在這2個綿羊品種中的遺傳多態性,共檢測到4種等位基因和10種不同的基因型,有6個SNPs位點(G/A、G/C、C/T、T/C、G/A、G/A),SNPs位點數占分析總堿基數的1.28%,其中第221位C>T的轉換引起氨基酸的改變,由苯丙氨酸突變為亮氨酸,其他SNPs位點突變未引起氨基酸的改變,均屬于同義突變。根據PIC>0.5高度多態[19]可得:甘肅高山細毛羊和湖羊的PIC值分別為0.55和0.69,均大于0.5,均屬于高度多態,說明群體的變異性較大。2個綿羊品種豐富的多態性信息含量(PIC>0.05)和較高的期望雜合度(均大于0.6)表明HIF-2α基因多態性較為分散,即遺傳基礎比較廣泛。
甘肅高山細毛羊和湖羊所生存的環境有一定的差異,甘肅高山細毛羊主要分布在高寒地區,海拔高,干旱少雨,海拔2 400~4 070 m,而湖羊飼養在江蘇、浙江兩省交界的太湖流域一帶,海拔較低[20]。本試驗通過對HIF-2α基因在2個綿羊品種中的基因頻率進行卡方檢驗,發現HIF-2α基因擴增區域的基因頻率在這2個綿羊品種間具有種間差異,且差異具有統計學意義。結合等位基因和基因型頻率的大小,推測在高海拔低氧適應過程中,HIF-2α基因的基因型可能受到了選擇,隨著長期的高海拔低氧環境的選擇,有利于低氧適應的基因型在高海拔綿羊品種中受到選擇并富集,如甘肅高山細毛羊基因型AA、BB和AB頻率較高,頻率之和達73.00%,而湖羊相同基因型頻率之和為34.70%;不利于低氧適應的基因型在高海拔綿羊品種中頻率下降,如基因型DD、BD和CD,甘肅高山細毛羊未檢測到基因型DD,基因型BD和CD的基因型頻率均為1.50%,而湖羊基因型DD的基因型頻率為5.00%,基因型BD和CD的基因型頻率均分別為15.88%和8.24%。由此推測隨著海拔的升高,攜帶基因型AA、BB和AB的綿羊可能更加適應高海拔低氧環境,而攜帶基因型DD、BD和CD的綿羊對高海拔低氧環境適應的能力可能較差。由于2個綿羊品種均達到Hardy-Weinberg平衡狀態,說明所選樣本未受到或受到人工選擇的影響較小,所以這種現象可能與綿羊所生存的環境有關。Dong等[21]研究HIF-2α基因的第8、9、10和15外顯子共14個SNPs位點,發現藏豬與平原豬種間基因型頻率差異極顯著,且SNP1、SNP7、SNP10和SNP11可能與藏豬適應高海拔環境相關聯。Wu等[22]研究表明甘南牦牛和大通牦牛的CD基因型頻率顯著高于天祝白牦牛(P<0.05);Beall等[17]運用基因芯片技術證實HIF-2α基因內含子的3個SNPs位點基因頻率在世居高原藏族與漢族之間有很大差別,以上報道與本試驗研究結果一致,都說明高原低氧環境是影響HIF-2α基因等位基因及基因型頻率顯著差異的重要因素,這種顯著差異可能與動物適應生存環境的能力即適應性有關。
[1] Lakkisto P,Kyt V,Forsten H,et al.Heme oxygenase-1 and Carbon monoxide promote neovascularization after myocardial infarction by modulating the expression of HIF-1alpha,SDF-1alpha and VEGF-B[J].European Journal of Pharmacology,2010,635(1/3):156-164.
[2] Semenza G L,Wang G-L.A nuclear factor induced by hypoxia via de novo protein synthesis binds to the human erythropoietin gene enhancer at a site required for transcriptional activation[J].Molecular and Cellular Biology,1992,12(12):5447-5454.
[3] Semenza G L.Hydroxylation of HIF-1:Oxygen sensing at the molecular level[J].Physiology,2004,19(19):176-182.
[4] 何 凌.HIF-1α/HIF-2α在惡性上皮性卵巢腫瘤中的表達及其對耐藥機制影響的初步探究[D].長沙:中南大學,2011.
[5] Tian H,Mcknight S L,Russell DW.Endothelial PAS domain protein 1(EPAS1),a transcription factor selectively expressed in endothelial cells[J].Genes&Development,1997,11(1):72-82.
[6] Yi Xin,Liang Yu,Huerta-sanchez Emilia,et al.Sequencing of 50 human exomes reveals adaptation to high altitude[J].Science,2010,329(5987):75-78.
[7] 吳曉云.牦牛EPAS1和VEGF-A基因的克隆、SNPs檢測及其表達分析[D].蘭州:甘肅農業大學,2012.
[8] Zhou H,Hickford JG,Fang Q.A two-step procedure for extracting genom ic DNA from dried blood spots on filter paper for polymerase chain reaction amplification[J].Analytical Biochem istry,2006,354(1):159-161.
[9] Byun SO,Fang Q,Zhou H,et al.An effective method for silver-staining DNA in large numbers of polyacrylamide gels[J].Analytical Biochemistry,2009,385(1):174-175.
[10] Gong Hua,Zhou Huitong,Hickford J G.Diversity of the glycine/tyrosine-rich keratin-associated protein 6 gene(KAP6)fam ily in sheep[J].Molecular Biology Reports,2011,38(1):31-35.
[11] Tissot van Patot M C,Gassmann M.Hypoxia:adapting to high altitude by mutating EPAS-1,the gene encoding HIF-2a[J].High Altitude Medicine&Biology,2011,12(2):157-167.
[12] Haase V H.Regulation of erythropoiesis by hypoxia-inducible factors[J].Blood Reviews,2013,27(1):41-53.
[13] Mastrogiannaki M,Matak P,Mathieu J R,et al.Hepatic hypoxia-inducible factor-2 down-regulates hepcidin expression in mice through an erythropoietin-mediated increase in erythropoiesis[J].Haematologica,2012,97(6):827-834.
[14] Greer SN,Metcalf JL,Wang Yi,et al.The updated biology of hypoxia-inducible factor[J].The EMBO Journal,2012,31(11):2448-2460.
[15] Compernolle V,Brusselmans K,Acker T,et al.Loss of HIF-2alpha and inhibition of VEGF impair fetal lung maturation,whereas treatment with VEGF prevents fatal respiratory distress in premature mice[J].Nature Medicine,2002,8(7):702-710.
[16] Partovian C,Adnot S,Eddahibi S,et al.Heart and lung VEGF mRNA expression in rats with monocrotaline-or hypoxia-induced pulmonary hypertension[J].The American Journal of Physiology,1998,275(6 Pt 2):H1948-H1956.
[17] Beall C M,Cavalleri G L,Deng Libin,et al.Natural selection on EPAS1(HIF2alpha)associated with low hemoglobin concentration in Tibetan highlanders[J].Proceedings of the National Academy of Sciences of the U-nited States of America,2010,107(25):11459-11464.
[18] 羅懷容,施 鵬,張亞平.單核苷酸多態性的研究技術[J].遺傳,2001,23(5):471-476.
[19] Vaiman D,Mercier D,Goudarzi K M,et al.A set of 99 cattle microsatellites:characterization synteny mapping[J].Mammalian Genome,1994,5(5):288-297.
[20] 趙有璋.中國養羊學[M].北京:中國農業出版社,2013:127.
[21] Dong Kunzhe,Kang Ye,Yao Na,et al.Genetic variation of EPAS1 gene in tibetan Pigs and three Low-Altitude pig breeds in China[J].Journal of Integrative Agriculture,2014,13(9):1990-1998.
[22] Wu Xiaoyun,Ding X Z,Chu Min,et al.Novel SNP of EPAS1 gene associated with higher hemoglobin concentration revealed the hypoxia adaptation of yak(Bos grunniens)[J].Journal of Integrative Agriculture,2015,14(4):741-748.
The Analysis of Gansu A lpine M erino and Hu Sheep H IF-2αGene Genetic Characteristics and Adap tability to H igh A ltitude Hypoxia
DANG Pengju1,LIShaobin1,WANG Jiqing1,LIU Xiu1,HONGWei2,HU Jiang1,LUO Yuzhu1
(1.Gansu Key Laboratory of Herbivorous Animal Biotechnology,Gansu Engineering Lab of Genetic Improvement in Runminants,Faculty of Animal Science and Technology,Gansu Agricultural University,Lanzhou 730070,China;2.Zhejiang Sainuo Ecological Agriculture Lim ited Company,Lin′an 311311,China)
To study the relationship between the variations of HIF-2a gene and adaption to plateau in sheep,we detected the 540 sheep including 200 Gansu Alpin Merino and 340 Hu sheep,and used the PCR-SSCP to analyze the SNPs of part of the eleventh exon and intron of HIF-2a gene.The results showed that 6 SNPs corresponding to 4 alleles(A,B,C and D)and 10 kinds of genotypes(AA,BB,CC,DD,AB,AC,BC,AD,BD and CD)were detected in HIF-2a gene in 2 sheep breed.The 5 SNPs existed in the eleventh exon and the one SNPs laid in the eleventh intron and the genotype DD was not detected in Gansu Alpine Merino.The advantaged genotype AB with frequency of 0.345 0 and 0.164 7 was detected in both sheep breed.The HIF-2a gene has abundant polymorphism in 2 sheep breed and the frequency of alleles has significantly difference in two popul ations(P<0.01).As the selecting of environment with p lateau and low oxygen,the HIF-2a gene was selected and clustered in p lateu sheep.The sheep withthe genetype AA,BB and AB can adapt in plateau environmentmore better and the adaptive capacity of other sheep with genetype CD,BD and DD was poor.
HIF-2αgene;Gansu Alpine Merino;Adaptability;Polymorphism
Q78;S826 文獻標識碼:A 文章編號:1000-7091(2016)03-0088-06
10.7668/hbnxb.2016.03.013
2016-03-11
甘肅省國際科技合作專項(1304WCGA178);甘肅農業大學盛彤笙科技創新基金項目(GSAU-STS-134);甘肅省青年科技基金計劃項目(1308RJYA025);甘肅省高等學校科研項目(2015B-052)
黨鵬舉(1987-),男,甘肅定西人,碩士,主要從事生物技術與動物育種研究。
羅玉柱(1962-),男,甘肅景泰人,教授,博士,博士生導師,主要從事現代生物技術與動物育種應用研究。

