生物大數據中的聚類方法分析
路東方,許俊富,項超娟,謝 江(上海大學計算機工程與科學學院,上海200444)
生物大數據中的聚類方法分析
路東方,許俊富,項超娟,謝江
(上海大學計算機工程與科學學院,上海200444)
隨著人類基因組計劃的實施和完成,生物實驗技術快速發展,生物數據呈現爆發式增長并不斷積累,生命科學迎來了大數據時代.在后基因組時代,單一的統計模式逐漸被智能化與綜合分析相結合的方式所取代,聚類分析便是核心的數據挖掘方式.描述了生物信息學領域中的大數據現狀,總結基因表達譜分析和生物網絡分析中常用的聚類方法,并對小鼠胚胎成纖維細胞的時間序列數據進行實驗對比.實驗結果表明,不同的聚類方法生成了不同的實驗結果,面臨高噪聲的生物大數據,選擇或結合合適的聚類方法進行綜合分析將有助于獲得更可靠的分析結果.
生物大數據;數據分析;聚類方法
1 概述
生物信息學是一門交叉學科,包含了對生物信息數據的獲取、處理、儲存、轉發、分析和解釋等方方面面.生物信息學綜合運用數學、計算機科學和生物學的各種工具,來闡明和理解生物信息數據所包含的生物學意義[1].生物信息學發展呈現出兩個主要特征:一是伴隨著海量生物數據的產生;二是相關科研活動逐步從傳統的手工統計方式轉向智能化與綜合分析相結合的方式.
2000年6月26日,被譽為生命“阿波羅計劃”的人類基因組計劃工作草圖的完成,是生物信息學發展史上又一個里程碑式的事件,它預示著完成人類基因組計劃已經指日可待.迄今已完成了約40多種生物的全基因組測序工作,其中人類基因組已完成了約3×109個堿基對的測序任務.尤其是第二代測序技術[2]的發展,使得基因組學每天都產生數以“T”計的海量數據.
在數據爆炸時代,根據數據類型進行有效整合是非常重要的.截至2012年[3],僅記錄在美國GenBank數據庫(美國國家生物技術信息中心的DNA序列總數據庫)中的DNA序列總量就已超過了70億個堿基對[3-4].在這樣一個生物信息的浪潮之巔,生物大數據的積累無疑會隨著時間的演進而愈發增多.基于cDNA序列測序建立起來的reptiledatabase[5](爬行動物數據庫)中已經存儲了超過1萬個基因的數據.美國國家生物技術信息中心(National Center for Biotechnology Information,NCBI)下的基因表達數據庫(Gene Expression Omnibus,GEO)[6]中已存儲了3 848個數據集,包括1 618 438個樣本的基因表達、基因芯片、蛋白質結構信息等數據.在這些數據的基礎上派生、整理出來的數據庫已超過500個,這一切構成了生物學數據的海洋.這些科學數據的急速增長和海量積累[7],在人類的科學研究歷史上是空前的.圖1統計了GEO中從2000年到2015年存儲樣本數據的變化情況.

圖1 GEO樣本數據增長趨勢Fig.1 Trends of GEO samples
常用的生物數據庫分為以下三類:一是基于DNA序列的數據庫,包括與DNA的復制、轉錄、修復等密切相關的蛋白質因子數據庫,有GenBank、EMBL(European Molecular Biology Laboratory,歐洲分子生物學實驗室的DNA和RNA序列庫)、RepBase(真核生物DNA中重復序列數據庫)等;二是基于核酸序列的數據庫,主要有INSD(International Nucleotide Sequence Database,國際核酸序列數據庫)、DDBJ(DNA Data Bank of Japan,日本核酸數據庫)等;三是基于蛋白質建立的數據庫,主要有BIOGRID(Biological General Repository for Interaction Datasets,蛋白質-蛋白質以及基因相互作用的數據庫)、DIP(Database of Interacting Proteins,蛋白質功能和蛋白質-蛋白質關系數據庫),STRING(Search Tool for the Retrieval of Interacting Genes/Proteins,已知和預測蛋白質相互作用數據庫)、Uniprot(Universal Protein Resource,蛋白質序列功能信息數據庫)等.
在當前的大數據時代,通常這些數據表現出以下四個特征[8-11]:①數據量巨大;②數據類別多樣;③數據潛在價值高;④增長速度驚人.人類基因組計劃的實施和完成,以及下一代測試技術(又稱為高通量測序技術)的不斷發展,不僅解決了生命科學發展中的經濟障礙,而且產生了更多的生物數據,推進了生命科學乃至醫學水平的發展.常見的生物數據類型包括基因組學、轉錄組學、蛋白質組學、代謝組學等,具體表現形式有基因的表達值、蛋白質相互作用、蛋白質結構等.生命科學的巨量數據正以多種類型和不同表現形式急劇膨脹,生命科學迎來了大數據時代[12-13].毫無疑問,這些生物大數據蘊涵著巨大價值,然而數據不等于信息和知識,而只是信息和知識的源泉.生物學家們需要將看似雜亂的數據轉變成可用的資源以后才能充分利用這些數據.計算科學的發展在伴隨著一定挑戰的同時,也給生命科學的“大數據時代”帶來了曙光.一方面,通過足夠的技術保障,可以保存這些具有巨大價值的生物大數據;另一方面,通過一些特定的方法充分挖掘這些大數據中隱藏的信息,將給生命科學研究帶來無限可能.
生物數據挖掘是當前生物數據研究的重點內容之一.常用的統計學方法已無法適應當前的大數據時代,而作為機器學習領域的無監督算法——聚類,卻備受青睞.在生物信息學研究領域中,從基本的序列分析、分子進化和比較基因組學,到蛋白質結構比對和預測,再到計算機輔助藥物設計等,聚類分析都顯示出極大的優勢.合適的聚類方法有助于發現相似的DNA,RNA或蛋白質功能組.一方面,是基于基因表達譜的聚類,其主要研究方式分為3種[14-15]:一是基于時間序列的分析,也就是測定基因在多個時間點的表達值,通過聚類和主成分分析等手段尋找共表達基因;二是基因表達差異的顯著性分析;三是蛋白質調控功能的研究.另一方面,是基于生物網絡的聚類,如蛋白質相互作用網絡的分析等.本研究結合生物大數據的研究背景,綜合概述常用的聚類方法在生物數據分析中的應用.
2 聚類分析
2.1聚類分析概念
聚類是現階段計算機科學領域最熱門的研究課題之一,在數據挖掘、生物信息學、圖像處理、復雜網絡(如社交網絡)等方面得到了成功應用.對生物大數據的分析和預測是生物信息學的兩個重要任務,聚類分析便是該領域中一個非常活躍的研究課題.聚類與分類不同,它不依賴于預先定義的類和帶標號的訓練實例,也不確定會產生幾個不同的簇.
聚類分析作為一種探索性的數據分析方法[16-18],根據所要研究對象(個體)的屬性值特征,采用合適的計算方法對其進行整理,將相似度較高的對象劃分為一類;再對同一類內個體的共性及不同類間個體的差異性作進一步歸納,從而得出新的規律.聚類方法建立在多元統計基礎之上,是模式識別、數據挖掘等領域中常用的基礎方法,尤其適用于分析模式類別數目不確定的情況.合理運用聚類分析方法,將復雜生物數據根據某些相似性度量規則(如基于歐式距離的相似矩陣)進行有效挖掘,對于相似基因表達模塊、蛋白質功能組等生物內容的研究大有裨益.
在生物信息學中,可以針對不同的數據集進行聚類分析.通過基因表達譜的聚類[19],分析聚在一個簇中的基因,有助于找到表達模式相近的共表達基因組、探索未知基因的功能、研究基因的調控以及細胞分化的過程等,相比分析單個基因其可靠性更高.通過基于網絡的聚類分析,可以發現功能相近的家族蛋白,充分了解細胞內部的工作機理.基于網絡的數據主要指蛋白質相互作用網絡、基因調控網絡和代謝網絡三種.
傳統的聚類方法包括基于劃分的聚類和基于層次的聚類.劃分聚類包括K-均值聚類、K-中心點聚類等;層次聚類包括基于距離的分層聚類和基于概率的分層聚類等.近年來,生物技術的突飛猛進和大數據的積累,產生了很多新型的聚類方法[20],如譜聚類、自組織映射神經網絡聚類、雙聚類、二次聚類、模糊聚類,以及基于生物大數據的并行聚類方法[21]等,這些聚類方法給大數據時代帶來了無限生機.
2.2聚類分析基本要求
聚類分析借助于不同的相似性度量規則,產生了不同的方法.對于不同的數據類型、不同的目的以及不同的應用領域,應該選擇合適的聚類方法加以分析.在生物信息發展領域,自1998年Eisen等[22]的基于距離度量的基因表達聚類分析,到2001年Hartmink等[23]的基于調控網絡拓撲結構的聚類分析,乃至如今對于大數據的挖掘,都體現了生物數據聚類的發展及挑戰.因此,聚類分析在生物領域的應用中也形成了一些基本的要求[24],主要有以下幾點.
(1)可伸縮性,指算法除了應用于小規模數據外,還要能夠適應大數據背景下的要求,因此要保證算法的時間復雜度足夠低,占用空間足夠小.
(2)適應性,指算法對于目標數據類型的識別,除了能處理數值型數據(如基因的表達值),還要能處理非數值型數據(如網絡類型、時間序列類型、生成樹類型等).
(3)可用性.目前數據庫中存儲的數據可能是任意形狀的,因此要求算法具備能夠發現任意形狀的聚類能力.
(4)弱依賴性.很多聚類方法都要求用戶輸入一些參數(如聚類數目、支持度等),這些參數的值都可能對聚類分析的結果產生很大影響.
(5)對高維數據分析的能力.傳統的數據分析對低維數據效果良好,卻不一定適合大數據背景下的高維數據,因此聚類分析應該具有降維分析或直接分析的能力.
3 常用的聚類方法
3.1基于基因表達譜的聚類
3.1.1K-均值聚類
K-均值聚類[25]的中心用各類別中所有數據的平均值表示,首先選k個初始點放入簇中計算平均值,然后重新將每個點放入與平均值距離最近的簇,重新計算平均值,直到均值不變或變化小于某一閾值.因此,這種方法一般被稱為基于“質心”的技術.需注意的是,在一般情況下,這些質心不是X中的點,雖然它們屬于同一個空間.K均值常常被稱為Lloyd's算法,其目標是在盡量減小簇內誤差平方和的基礎上選擇質心,即一組N個樣本集合X分為不相交的K個簇,各由簇中的樣本均值μj表示:

K-均值聚類的過程[26]如圖2所示.

圖2 K-均值聚類的過程Fig.2 Process of K-means clustering
2014年,Marco等[27]的基于分岔理論的單細胞分析聚類實驗中,從小鼠胚胎早期階段分離出438個體細胞,根據其基因的表達水平,使用高通量的反轉錄聚合酶鏈式反應(reverse transcription polymerase chain reaction,RT-PCR)定量選擇48個基因,其中包括發育過程中的27個關鍵轉錄因子.從單細胞到64細胞階段,在7個不同的時間點提取細胞,每一個時間點對應一個細胞加倍階段.對這些數據運用K-均值聚類方法和間距統計方法進行聚類.在每一個時間點,將每個細胞根據其基因表達譜分配給一個父類簇.為了確定某一分岔事件是否發生,將每個父類簇的子簇根據K-均值聚類進一步分為2個不同的簇,并采用間隙統計來選擇單簇或雙簇模型.重復上述過程,直到最后的時間點.這樣就可以創建細胞層次的二叉樹初始估計模型.
下面定義描述最佳全局基因表達模式二叉樹的結構.通過懲罰似然函數評估每個參數的性能,即

式中,θ表示所有參與定義樹結構中的參數;x為觀測數據;μc和μa(c)分別為簇c和a(c)的中心,其中a(c)為c的父類簇;λ為事先定義的常量,本實驗中設置λ=1.將最終的聚類結果[27]投影到空間上(見圖3).

圖3 細胞階段聚類結果Fig. 3 Clustering results of cell stages
圖3顯示了兩個分岔方向上跨平面的整體結構,其中X32和X64不完全正交,每種顏色代表不同的集群,父子集群對由直線連接.圖3進一步證明了細胞分化過程中的兩個重要階段,32細胞階段和64細胞階段,這對于細胞分化的動力學研究非常有益.
3.1.2K-中心點聚類
K-中心點聚類[28]不同于K-均值聚類,而是選取一個到其他所有點距離之和最小的點作為中心點.這種聚類方法能處理任意類型數據的屬性,對異常數據不敏感,因此這些看似異常的數據可能會導致聚類結果的均值變得“扭曲”.與K-均值聚類一樣,K-中心點聚類也可以采用歐幾里得距離來衡量樣本點之間的相似性,終止條件是當所有的類簇的質點都不再發生變化時,認為聚類結束.這里,距離的定義如下:

K-中心點聚類在小型數據集上運行良好,但不適合大數據集,其算法復雜度為O(k(nk)),相對較高.K-中心點聚類的過程[25]如圖4所示.
2014年,Trapnell等[29]的關于細胞擬時間動力學分析的研究中,提出對單細胞進行聚類,將具有相似基因表達的基因聚類為一組.該實驗首先將具有相似表達趨勢的基因分為一組,因為這些細胞可能共享某些生物學功能和調控.一旦每個基因具有廣義相加模型(generalized additive models,GAM)值,這些模型就可以用來預測擬時間功能的平滑響應曲線.規范這些曲線,允許所有基因的數據跨擬時間設置有效的K-中心點聚類.根據方向和時間,基因x和y之間的成對距離為

式中,ρx,y表示響應曲線的Pearson相關系數,簇對應的基因遵循相同的相對動力學趨勢.在實驗中,聚類對象不是原始數據,而是基于GAM響應曲線.相對于通常采用的中心點,這種聚類方法具有較小的均方根誤差,更符合動力學原理;同時,對于模式分析的支持也更多樣化.在這項研究中,聚類分析了全部可檢測的基因表達,無需考慮擬時間序列調控的意義.K-中心點聚類在基因的log轉換和標準化后對基因擬時間的GAM進行聚類.聚類應用R語言中的PAM包進行.實驗產生了6個區別明顯的簇,并最終根據聚類結果對6個簇進行順式調控分析.

圖4 K-中心點聚類的過程Fig.4 Process of K-mediods clustering
3.1.3層次聚類
層次聚類[25]是一種將數據結構化為組群對象,通過自上而下(或自下而上)的迭代,構建相似嵌套集群的聚類方法.層次聚類的結構常用樹狀圖來表示,樹根即為所選樣本的集合,葉子節點是單一的樣本數據.自下而上(凝聚)的聚類[20]是由每個單一樣本構成初始簇,根據歐式距離(見式(3))或余弦距離等度量標準,逐層合并相似簇;自上而下(分裂)的聚類是由所有樣本構成初始簇,逐層分裂為相似度較低的集合.層次聚類還衍生出了基于密度和基于網格[30]等的聚類方法,特別適用于中等規模的數據集,具有簡單、直接的優點.自下而上的層次聚類過程如圖5所示.
2015年,Levine等[31]的關于數據驅動下急性骨髓白血病的表現型研究中,對16個小兒急性髓細胞白血病(acute myelocytic leukemia,AML)患者以及5個正常人總共1.5億個高維數據定義的表型細胞作關聯性分析.對細胞表面的16個信號特征和內部的14個抗體信號特征進行觀察和數據采樣,以Jaccard系數作為相似度的衡量標準,對一組干細胞和相關分化細胞進行分層聚類,這些病人的層次聚類描述顯示一些遺傳異質性與遺傳標志物相關,細胞表面標志物并不能作為細胞表型變化的衡量標準.因此,在研究細胞變化的過程中,通過細胞內的信號分泌物才能得到比較準確的信息.

圖5 自下而上的層次聚類過程Fig.5 Process of bottom-up hierarchical clustering
隨著生物技術的逐漸成熟,大量生物數據不斷產生,數據維度也越來越高.在一般情況下,對高維數據的聚類主要分為子空間聚類法和維歸約法.子空間聚類法又大致分為子空間搜索法、基于相關性聚類法和雙聚類法.雙聚類[32]是指通過二維空間上兩個維度同時聚類的方法,比如在基因表達上的應用中,通常所說的基因表達數據或DNA微陣列數據是一個基因-樣本/條件矩陣,其中每行表示一個基因,每列表示一個樣本或條件,從聚類的角度看即是對兩個維度的分析.2012年,Gerstein等[33]的關于人類調控網絡的DNA元件百科全書(encyclopedia of DNA elements,ENCODE)數據研究中,通過雙聚類對基因表達譜進行分析,最終發現了特定背景下幾種共關聯的表達基因.當然,面對高維大數據也可以采用譜聚類等方法,或者借助于MapReduce工具來實現.
3.2基于蛋白質網絡的聚類
分子生物學是一門研究生物本身組成成分的科學,主要包括核酸、蛋白質和酶的結構和合成、功能和代謝、表達和調控以及它們之間的相互作用等,以此來揭示生命的本質,是目前生物發展進程中的基礎學科.而研究蛋白質的相互作用[34](protein-protein interaction,PPI)則是生命科學研究中的重要一環.蛋白質作為生命功能的執行者,尤其是隨著后基因組時代的到來,蛋白質相互作用網絡的研究更是成為系統生物學研究中的重要內容之一.比較成熟的研究理論認為,蛋白質并不是單獨發揮作用的,而是通過與相關蛋白質的相互作用,在空間和時間上協調一致,共同調控、維持細胞的特定功能.通過對蛋白質相互作用網絡的聚類分析,可以找到感興趣的蛋白質功能組,這對于生物體的行為研究、未知蛋白功能的預測以及藥物的設計都具有重要的作用.
利用聚類方法獲取蛋白質網絡中的生物相關功能模塊是目前的研究熱點之一[35].根據采取方式的不同,主要有基于圖論的方法(graph-theoretic approach)、基于模擬流的方法(flow simulation-based approaches)、基于譜聚類的方法(spectral clustering-based approaches)、基于監督學習的方法(supervised clustering approaches)、基于依賴核心的方法(core attachmentbased approaches)、基于群體智能的方法(swarm intelligence-based approaches)等.具體劃分[36]如表1所示.

表1 蛋白質網絡聚類方法分類Table 1 Classification of protein network clustering
2008年,Blondel等[37]基于層次聚類和模塊性最優化提出了Louvain方法.該方法實現了在模塊性最優化的基礎上,快速得到網絡分層的效果.此后,Xie等[38]將Louvain方法應用在蛋白質模塊比對的可視化分析中,利用Louvain層次化的網絡聚類結果,用可視化形式分析生物分子網絡比對的結果.層次聚類之后的網絡能很好地以整體和局部兩種視圖分析生物分子網絡的特點(見圖6).同時,結合蛋白質功能模塊與蛋白質相互作用網絡的比對結果,有助于發現生物相關功能模塊的比對結果.為便于進行相關功能蛋白的研究,還可以使用主成分分析(principal component analysis,PCA)方法[39].

圖6 Louvain層次化的蛋白質網絡聚類可視化效果[38]Fig.6 Visualization effect of Louvain hierarchical clustering protein network[38]
4 實驗對比
眾所周知,基因表達數據的聚類分析為生物研究提供了強有力的技術支持,但是不同的聚類方法卻會導致不同的實驗結果.本研究對GEO中小鼠胚胎成纖維細胞體外低溫下基因表達差異性[40]數據進行聚類,數據由Affymetrix公司提供[41],總共包括不同時間點的13組樣本數據.這種基于時間序列[42]的聚類可廣泛應用于不同領域,并且已被證明是非常有效的.將胚胎成纖維細胞體外暴露在輕度低溫(32°C)環境或常溫(37°C)環境下長達18 h,在不同時間點對數據進行采樣.低溫是一種臨床上有效的治療各種缺氧和缺血的手段之一.37和32°C取樣時間點分別為0,0.5,1.0,2.0,4.0,8.0,18.0 h和0.5,1.0,2.0,4.0,8.0,18.0 h,這些樣本分別編號為GSM1310500~GSM1310512,共計13組.正常溫度下樣本包括GSM1310500~GSM1310506,低溫下樣本包括GSM1310507~GSM1310512.聚類方法采用K-均值、K-中心點和分層聚類的方法,相似性度量選擇歐氏距離,初始選擇k=4.以熱圖(heatmap)的形式展現部分聚類結果如圖7~9所示[41].
實驗聚類的具體數據可參照文獻[41],圖7~9中顏色接近程度表示基因之間的相似表達程度.根據不同時間點的基因聚類結果可以得出同一時間點的共表達基因組,這為細胞分化的階段性分析提供了強有力的證據.
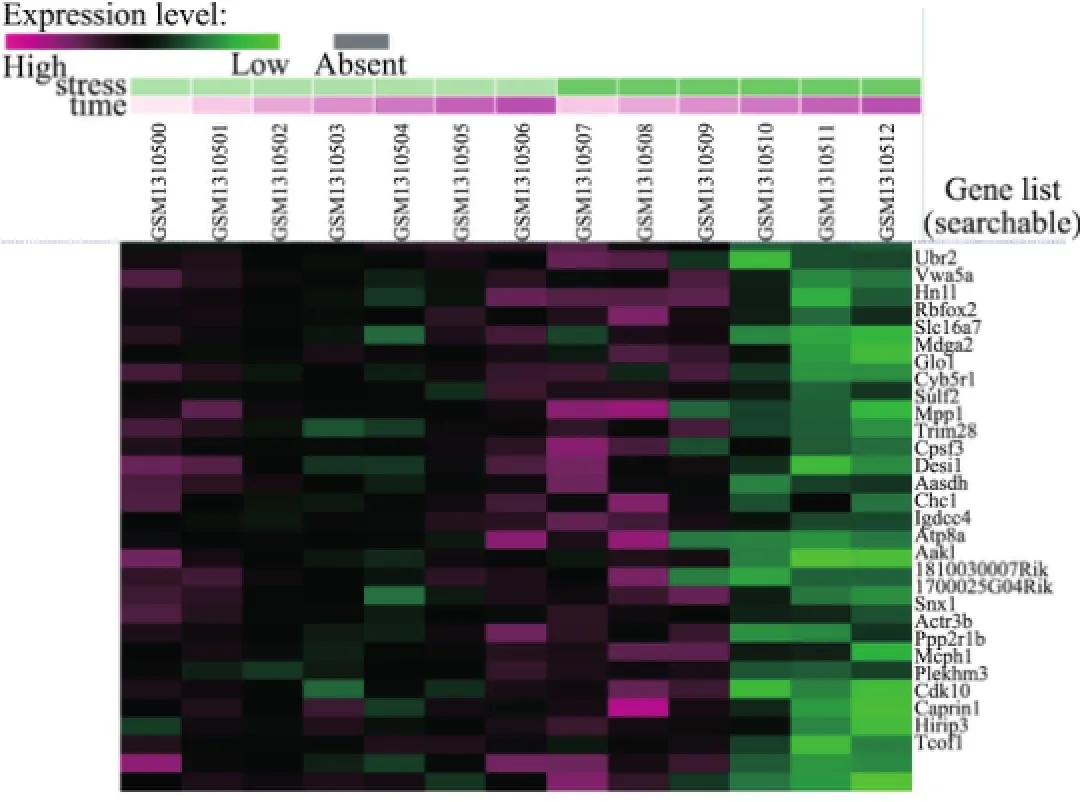
圖7 K-均值聚類結果Fig.7 Results of K-means clustering

圖8 K-中心點聚類結果Fig.8 Results of K-mediods clustering

圖9 層次聚類結果Fig.9 Results of hierarchical clustering

表2 三種聚類方法結果Table 2 Results of the three clustering methods
從3組聚類結果的基因表達譜可以看出:①從整體上而言,正常溫度下基因的表達值相對較高;②某些基因在低溫下0.5~1.5 h時表達增強.表2列出了三種聚類方法的結果,通過對比可以發現,K-均值和K-中心點聚類結果顯示一致,卻與層次聚類結果有所差異.也就是說,當采用不同的聚類方法時,得到的聚類結果將受到影響.表2中三種算法都聚在一個簇的樣本分別以下劃線、星號(*)、井號(#)及梅花符號(?)標記,這些樣本的聚類具有更高的可信度,也是在進一步分析中需要著重關注的內容.
5 結束語
生物信息學是一個成長中的跨學科領域[43-44],生物數據日新月異、逐年增長.聚類是數據挖掘的基本方法之一,在基因表達和生物網絡等數據挖掘過程中起到了舉足輕重的作用.后基因組時代伴隨著二代甚至三代測序技術的發展,單一的聚類方法已經無法滿足當前的大數據要求.另外,高噪聲數據的干擾也是亟待解決的問題.綜上所述,選擇和結合不同的聚類方法,針對不同的生物數據進行綜合分析與比較,進一步提出新的適用于大數據挖掘的方法,將有助于揭曉更多的生物奧秘.
[1]趙屹,谷瑞升,杜生明.生物信息學研究現狀及發展趨勢[J].醫學信息學雜志,2012,33(5):2-6.
[2]KOBOLDT D C,STEINBERG K M,LARSON D E,et al.The next-generation sequencing revolution and its impact on genomics[J].Cell,2013,155(1):27-38.
[3]任艷姣.生物信息學數據整合的應用研究[D].長春:吉林大學,2012.
[4]BENSON D A,KARSCH-MIZRACHI I,LIPMAN D J,et al.GenBank[J].Nucleic Acids Research,2000,28(1):15-18.
[5]UETZ P,ETZOLD T.The EMBL/EBI reptile database[J].Herpetological Review,1996,27(4):174-175.
[6]BARRETT T,WILHITE S E,LEDOUx P,et al.NCBI GEO:archive for functional genomics data sets-update[J].Nucleic Acids Res,2013,41:D1005-D1010.
[7]王洪昌,丁立軍,黃宇.生物信息學中模式識別技術應用與發展[J].醫學信息學雜志,2013(11):7-10.
[8]LI Y,CHEN L.Big biological data:challenges and opportunities[J].Genomics,Proteomics and Bioinformatics,2014,12(5):187-189.
[9]MARx V.Biology:the big challenges of big data[J].Nature,2013,498(7453):255-260.
[10]SCHUSTER S C.Next-generation sequencing transforms today's biology[J].Nature,2007,200(8):16-18.
[11]REIS-FILHO J S.Next-generation sequencing[J].Breast Cancer Res,2009,11(S3):S12.
[12]MARCOTTE E M,DATE S V.Exploiting big biology:integrating large-scale biological data for function inference[J].Briefings in Bioinformatics,2001,2(4):363-374.
[13]ARONOVA E,BAKER K S,ORESKES N.Big science and big data in biology:from the international geophysical year through the International Biological Program to the Long Term Ecological Research(LTER)Network,1957—present[J].Historical Studies in the Natural Sciences,2010,40(2):183-224.
[14]MADEIRA S C,OLIVEIRA A L.Biclustering algorithms for biological data analysis:a survey[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics(TCBB),2004,1(1):24-45.
[15]楊春梅,萬柏坤,高曉峰.基因表達聚類分析技術的現狀與發展[J].生物化學與生物物理進展,2003,30(6):974-979.
[16]黃金.聚類和分類技術在生物信息學中的應用[D].哈爾濱:黑龍江大學,2005.
[17]陳亮.聚類算法及其在生物信息學中的應用[D].無錫:江南大學,2012.
[18]REDDY C K,AL HASAN M,ZAKI M J.Clustering biological data[M]//Data clustering:algorithms and applications.London:Chapman and Hall/CRC,2013:381-414.
[19]ERCIYES K.Clustering of biological sequences[M]//ERCIYES K.Distributed and sequential algorithms for bioinformatics.Berlin:Springer International Publishing,2015:135-160.
[20]AGGARWAL C C,REDDY C K.Data clustering:algorithms and applications[M].Boca Raton:CRC Press,2014.
[21]WANG M,ZHANG W,DING W,et al.Parallel clustering algorithm for large-scale biological data sets[J].PLoS ONE,2014,9(4):e91315.
[22]EISEN M B,SPELLMAN P T,BROWN P O,et al.Cluster analysis and display of genome-wide expression patterns[J].Proceedings of the National Academy of Sciences,1998,95(25):14863-14868.
[23]HARTEMINK A J,GIFFORD D K,JAAKKOLA T,et al.Using graphical models and genomic expression data to statistically validate models of genetic regulatory networks[C]//Pacific Symposium on Biocomputing.2001:422-433.
[24]蘇志中.聚類分析研究及其在生物數據分析中的應用[D].長沙:湖南大學,2009.
[25]周洋.基因表達譜數據聚類分析的研究[D].咸陽:西北農林科技大學,2014.
[26]HAN J,KAMBER M,PEI J.Data mining:concepts and techniques:concepts and techniques[M]. Amsterdam:Elsevier,2011.
[27]MARCO E,KARP R L,GUO G,et al.Bifurcation analysis of single-cell gene expression data reveals epigenetic landscape[J].Proceedings of the National Academy of Sciences,2014,111(52):E5643-E5650.
[28]張琛.生物信息學中的基因表達譜數據分析研究[D].長春:吉林大學,2008.
[29]TRAPNELL C,CACCHIARELLI D,GRIMSBY J,et al.The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells[J].Nature Biotechnology,2014,32(4):381-386.
[30]MURTAGH F,CONTRERAS P.Algorithms for hierarchical clustering:an overview[J].Wiley Interdisciplinary Reviews:Data Mining and Knowledge Discovery,2012,2(1):86-97.
[31]LEVINE J H,SIMONDS E F,BENDALL S C,et al.Data-driven phenotypic dissection of AML reveals progenitor-like cells that correlate with prognosis[J].Cell,2015,162(1):184-197.
[32]安平.基因表達數據的雙聚類分析方法研究[D].蘇州:蘇州大學,2013.
[33]GERSTEIN M B,KUNDAjE A,HARIHARAN M,et al.Architecture of the human regulatory network derived from ENCODE data[J].Nature,2012,489(7414):91-100.
[34]王正華,董蘊源,王勇獻.蛋白質相互作用網絡的幾種聚類方法綜述[J].國防科技大學學報,2009,31(004):81-86.
[35]劉昊,廖波,彭利紅.基于蛋白質相互作用網絡的聚類算法研究[J].計算機工程與應用,2009,44(30):142-144.
[36]JI J Z,ZHANG A D,LIU C N,et al.Survey:functional module detection from protein-protein interaction networks[J].IEEE Transactions on Knowledge and Data Engineering,2014,26(2):261-277.
[37]BLONDEL V D,GUILLAUME J L,LAMBIOTTE R,et al.Fast unfolding of communities in large networks[J].Journal of Statistical Mechanics:Theory and Experiment,2008,DOI:10.1088/1742-5468/2008/10/P10008.
[38]XIANG C J,XIE J,GU Y L,et al.Visualization of module alignment discovery[C]//Control Conference(CCC).2015:8545-8549.
[39]ASUR S,UCAR D,PARTHASARATHY S.An ensemble framework for clustering protein-protein interaction networks[J].Bioinformatics,2007,23(13):i29-i40.
[40]TIBSHIRANI R,HASTIE T,EISEN M,et al.Clustering methods for the analysis of DNA microarray data[R].Stanford:Stanford University,1999.
[41]STEN I,ANSGAR H C,RIIN R,et al.Estimating differential expression from multiple indicators[J].Nucleic Acids Research,2014,42(8):e72.
[42]LIAO T W.Clustering of time series data—a survey[J].Pattern Recognition,2005,38(11):1857-1874.
[43]TORARINSSON E,HAVGAARD J H,GORODKIN J.Multiple structural alignment and clustering of RNA sequences[J].Bioinformatics,2007,23(8):926-932.
[44]FITZGERALD P C,SHLYAKHTENKO A,MIR A A,et al.Clustering of DNA sequences in human promoters[J].Genome Research,2004,14(8):1562-1574.
Survey of clustering methods for big data in biology
LU Dongfang,XU Junfu,XIANG Chaojuan,XIE Jiang
(School of Computer Engineering and Science,Shanghai University,Shanghai 200444,China)
With the implementation of the Human Genome Project and the rapid development of biological experiment technology,biological data sharply grow and continuous accumulate.Age of big data in biology is coming.In the post genomic era,single statistical models are gradually replaced with combination of intelligent and comprehensive analyses. Clustering is the core of data mining.This paper describes the state-of-the-art technology of big data in bioinformatics,and summarizes several popular clustering methods on gene expression profiling and biological networks.Furthermore,some experiments are made to compare different clustering methods on the time series data of mouse embryonic fibroblasts,showing that different clustering methods have different results.To achieve more reliable conclusions for highly noisy biological data,it is necessary for investigators to do comprehensive analyses by selecting and combining proper clustering methods.
big data in biology;data analysis;clustering method
TP 39
A
1007-2861(2016)01-0045-13
10.3969/j.issn.1007-2861.2015.04.018
2015-11-30
國家自然科學基金重大研究計劃項目(91330116);教育部留學回國人員科研啟動基金資助項目
謝江(1971—),女,副教授,博士,研究方向為生物信息學、高性能計算.E-mail:jiangxsh@shu.edu.cn

