梅花PmSOC1-like基因的克隆與表達分析
李玉舒,楊煒茹,程堂仁,王 佳,張啟翔
(1.花卉種質創新與分子育種北京市重點實驗室,國家花卉工程技術研究中心,城鄉生態環境北京實驗室,北京林業大學 園林學院,北京 100083;2.北京農業職業學院,北京 102442)
?
梅花PmSOC1-like基因的克隆與表達分析
李玉舒1,2,楊煒茹1,程堂仁1,王 佳1,張啟翔1
(1.花卉種質創新與分子育種北京市重點實驗室,國家花卉工程技術研究中心,城鄉生態環境北京實驗室,北京林業大學 園林學院,北京 100083;2.北京農業職業學院,北京 102442)
SOC1/TM3 (Suppressor of overexpression of constans 1/Tomato MADS-box gene 3)是 MADS-box家族一員,它能夠整合多條開花途徑的開花信號,調節植物由營養生長向生殖生長的轉變。為了解梅花成花轉變過程的分子機理,采用RT-PCR的方法從梅花長蕊綠萼中克隆到3個SOC1的同源基因,分別命名為PmSOC1-1、PmSOC1-2和PmSOC1-3,并采用熒光定量PCR對3個基因的表達模式進行了分析。序列分析表明,3個基因的編碼區長度分別為645 bp(PmSOC1-1)、654 bp(PmSOC1-2)和660 bp(PmSOC1-3);編碼的氨基酸長度分別為214,217,219 aa。系統進化結果顯示,PmSOC1-1、PmSOC1-2和PmSOC1-3分別與擬南芥SOC1/TM3亞家族中的SOC1、AGL42/71/72和AGL14/19聚為一組。實時熒光定量分析結果表明,3個PmSOC1-like基因均在莖、葉等營養器官中表達量較高,在花、果實和種子等生殖器官中表達量較低;在梅花花芽分化過程中,3個PmSOC1-like基因的表達量整體呈下降趨勢,推測PmSOC1-like基因可能在誘導梅花由營養生長向生殖生長轉變過程中起重要作用。
梅花;PmSOC1-like;基因克隆;表達分析
開花是高等植物由營養生長向生殖生長轉變的重要標志,由內在因子和外在因素共同控制。這一過程受一系列極其復雜的網絡信號傳導途徑的調控。到目前為止,發現了4條植物開花調控途徑,即光周期途徑、春化途徑、自主途徑和赤霉素途徑[1-3]。此外,周邊的環境溫度和植物自身的年齡也對開花時間產生一定的影響。在對擬南芥等模式植物的研究中發現,當植物感受到成花信號后,開花時間調控途徑即被啟動,各個途徑的主效基因開始介導開花信號的傳導并作用于下游的花序分生組織特異基因和花器官基因,最終完成營養生長向生殖生長的轉變過程。研究表明,MADS-box基因家族在控制開花時間方面起著非常重要的作用[4-7]。其中SOC1(Suppressor of overexpression of constans 1)基因屬于MADS-box基因家族中的SOC1/Tomato MADS-box gene 3(TM3)亞家族,它是作用于開花時間調控路徑的關鍵整合子,能夠整合來自光周期途徑、春化途徑、自主途徑和赤霉素途徑的多種開花信號[8-9],促進植物的營養分生組織向花分生組織轉變,進而促進開花。
目前已從矮牽牛(Petuniahybrida)[10]、石斛蘭(Dendrobium)[11]、美洲山楊(Populustremuloides)[12]、葡萄(Vitisvinifera)[13]和紫斑牡丹(Paeoniarockii)[14]等多個物種中克隆到SOC1的同源基因,該基因的功能也得以進一步研究,然而,在木本植物中關于SOC1同源基因功能的研究卻鮮有報道。
梅花(PrunusmumeSieb.et Zucc.)是薔薇科(Rosaceae)李屬(Prunus)重要的觀賞花木與果樹,為我國十大傳統名花之一。培育高產、抗寒、芳香的梅花新品種一直是梅花的主要育種目標,然而童期是梅花雜交育種的必經階段,多年童期一直是縮短梅花有性雜交育種周期最大的制約要素。鑒于SOC1基因在植物成花誘導中的重要作用,本研究克隆了梅花SOC1的同源基因,研究了該基因在梅花中的時空表達模式,為以后開展梅花分子育種,加速梅花育種進程提供理論依據。
1 材料和方法
1.1 植物材料與生長條件
試驗材料為梅花長蕊綠萼(P.mumeChangrui Lve)。該品種花瓣白色,萼片淡綠色,雄蕊發達,略短于花瓣,輻射后聚集中心。5年生成年樹取自鷲峰國際精品梅園;1月齡幼苗種于溫室中,溫度為16~25 ℃,相對濕度為60%。以葉片為試材,提取總RNA用于基因克隆;取長蕊綠萼成年樹的莖、幼嫩葉片、成熟葉片、葉芽、花芽、完全開放花朵萼片、花瓣、雄蕊、雌蕊、種子、幼果(花后45 d)、熟果(花后95 d)和1月齡播種苗的根、莖、葉片等材料進行基因的組織特異性表達分析。以未分化期(S1),花原基形成期(S2),萼片分化期(S3),花瓣分化期(S4),雄蕊分化期(S5),雌蕊分化期(S6),雌蕊伸長期(S7),胚珠、花藥形成期(S8)8個花芽分化時期的花芽為試材,研究其基因在花芽分化過程中的表達差異。所有試材液氮速凍后-80 ℃保存。
1.2 RNA的提取及cDNA合成
采用TRIzol試劑盒(Invitrogen)提取梅花不同組織中的總RNA。經瓊脂糖凝膠電泳和紫外分光光度法檢測RNA質量后,以2 μg總RNA為模板,采用TIANScript cDNA第一鏈合成試劑盒合成第一鏈cDNA。操作方法參照試劑盒說明書進行。
1.3 基因的克隆
以擬南芥(Arabidopsisthaliana)SOC1蛋白(GenBank ID:AEC10583.1)的氨基酸序列為參考序列,在梅花基因組蛋白庫中進行本地Blast搜索[15](梅花基因組蛋白庫數據的DNA來源為西藏野生種),將同源性高的3個序列(PmSOC1-1、PmSOC1-2和PmSOC1-3)作為候選基因。根據基因核苷酸序列設計特異引物(表1),以梅花葉片cDNA為模板擴增基因的編碼區(CDS)。PCR反應體系為:cDNA 1 μL,Taq酶0.5 μL,上、下游引物(10 μmol/L)各1 μL,1×PCR Buffer 5 μL,25 mmol/L MgCl23 μL,10 mmol/L dNTP 1 μL,ddH2O補足至25 μL。PCR擴增程序為:94 ℃預變性5 min;94 ℃變性30 s,57 ℃退火30 s,72 ℃延伸1 min,30個循環;72 ℃延伸7 min。PCR產物經1%瓊脂糖凝膠電泳后,采用Promega Gel extraction kit DNA凝膠回收試劑盒回收目的片段,連接到pMD18-T(TaKaRa)載體上,轉化大腸桿菌Top10,挑取陽性克隆經PCR驗證后,交由中美泰和生物技術有限公司測序。確定序列后,將序列提交到GenBank數據庫獲得登錄號。其中PmSOC1-2和PmSOC1-3的登錄號分別為KP938964和KP938965,而PmSOC1-1的序列則與登錄號為JF806632.1的序列完全一致。
1.4 序列的生物信息學分析
利用ExPASy提供的ProtParam程序(http://web.expasy.org/protparam/)分析梅花SOC1蛋白的相對分子量、理論等電點;利用NCBI的Protein Blast進行相似蛋白搜索,并下載相似性>50%的蛋白序列;利用DNAMAN 5.2.2軟件對梅花SOC1蛋白與其他物種SOC1蛋白進行比對分析;利用ClustalX1.83進行多序列比對,利用MEGA 5.1軟件采用Neighbor-joining(NJ)算法構建系統進化樹,Bootstrap值設為1 000次。
1.5 Southern雜交
采用改進的CTAB法提取梅花的基因組DNA[16],用PCR反應法制備探針,引物序列見表1。選用BamHⅠ,EcoRⅠ和XhoⅠ(購自TaKaRa公司)分別酶切基因組DNA。電泳轉膜按分子克隆手冊進行。雜交的具體步驟按DIG DNA Labeling and Detection Starter Kit Ⅱ試劑盒(購自Roche公司)的說明進行。
1.6 梅花花芽分化時期的確定
采用石蠟切片的方法確定梅花花芽分化的不同時期。每隔5~7 d取樣1次,取樣時取2份外觀基本一致的樣品,1份用FAA固定液(70%酒精∶福爾馬林∶冰醋酸=90∶5∶5)保存,常規石蠟切片法制片確定花芽分化時期;另1份液氮速凍,用于RNA的提取。根據石蠟切片,梅花花芽分化過程分為8個時期:未分化期(S1),花原基形成期(S2),萼片分化期(S3),花瓣分化期(S4),雄蕊分化期(S5),雌蕊分化期(S6),雌蕊伸長期(S7),胚珠、花藥形成期(S8)。

表1 引物序列
1.7 基因表達特性分析
采用實時熒光定量PCR檢測梅花PmSOC1-like基因在不同組織和花芽發育階段的時空表達模式。分別提取樣品的總RNA,反轉錄成cDNA,作為實時熒光定量PCR的模板。使用Primer3 v0.4.0軟件設計PmSOC1-like基因特異引物,以梅花PP2A基因作為內參基因[17],引物序列見表1。反應在Thermo實時定量PCR儀上進行,采用20 μL體系,即cDNA 1 μL,SYBR Premix Ex Taq Ⅱ(TaKaRa)10 μL,上下游引物(10 μmol/L)各0.4 μL,ddH2O 7.2 μL。反應程序采用2步法:95 ℃預變性30 s;95 ℃ 5 s,60 ℃ 30 s,40個循環;接下來步驟是60~95 ℃的溶解曲線,升溫0.5 ℃/s。擴增產物特異性通過溶解曲線進行確定,試驗設置3次生物學重復和3次技術重復。利用PikoReal Software以及Excel進行數據分析,采用2-ΔΔCt法計算基因相對表達量。
2 結果與分析
2.1 梅花PmSOC1-like基因的克隆與序列分析
用特異性引物以梅花葉片cDNA為模板進行PCR擴增,經連接、轉化、測序后,獲得3個CDS序列,分別命名為PmSOC1-1、PmSOC1-2和PmSOC1-3。PmSOC1-1的編碼區長度為645 bp,其開放閱讀框(ORF)編碼214個氨基酸組成的蛋白質。PmSOC1-2的編碼區長度為654 bp,其ORF編碼217個氨基酸組成的蛋白質。PmSOC1-3的編碼區長度為660 bp,其ORF編碼219個氨基酸組成的蛋白質。3個基因的相對分子質量分別為24.6,24.91,24.95 kDa;理論等電點分別為9.46,8.68,9.05。
為了了解PmSOC1基因在梅花基因組中的拷貝數情況,我們還分別利用BamH Ⅰ,EcoR Ⅰ和XhoⅠ 3種限制性內切酶對3個梅花PmSOC1-like基因進行了Southern雜交分析,結果發現3個基因的探針雜交均呈現單一條帶,表明這些同源基因在梅花基因組中均以單拷貝形式存在(圖1)。
2.2 梅花PmSOC1-like蛋白的同源性分析
將梅花PmSOC1-like基因推導的氨基酸序列和其他物種SOC1蛋白的氨基酸序列進行同源比對(圖2),結果發現PmSOC1-1與擬南芥SOC1蛋白序列的同源性為68%,與薔薇科李屬植物的同源性更高,例如,PmSOC1-1與杏(Prunusarmeniaca)SOC1蛋白的相似性最高,達98%,與桃(Prunuspersica)SOC1蛋白的相似性為95%。而PmSOC1-2和PmSOC1-3的2個基因與薔薇科李屬植物SOC1基因的相似性則較低,如PmSOC1-2與杏和桃SOC1蛋白相似性分別為59%和57%;PmSOC1-3與杏和桃SOC1蛋白相似性分別為55%和54%。

圖1 梅花PmSOC1-like基因的Southern雜交分析
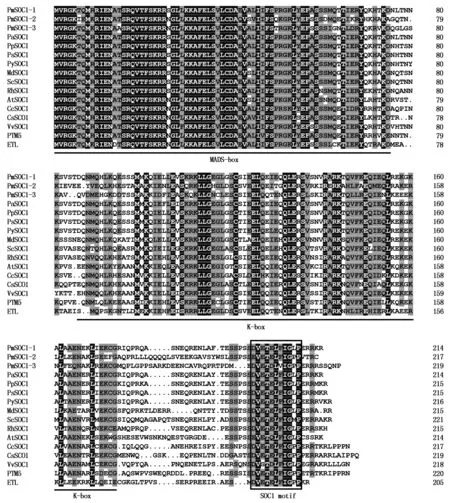
PaSOC1.杏; PpSOC1.桃;PsSOC1.李;PySOC1.櫻花;MdSOC1.蘋果;ScSOC1.麻葉繡線菊;RhSOC1.月季;AtSOC1.擬南芥;CcSOC1.山核桃;CsSOC1.柑橘;VvSOC1.葡萄;PTM.歐洲山楊;ETL.桉樹。
PaSOC1.Prunusarmeniaca; PpSOC1.Prunuspersica;PsSOC1.Prunussalicina;PySOC1.Prunus×yedoensis;MdSOC1.Malusdomestica;ScSOC1.Spiraeacantoniensis;RhSOC1.Rosahybrida;AtSOC1.Arabidopsisthaliana;CcSOC1.Caryacathayensis;CsSOC1.Citrussinensis;VvSOC1.Vitisvinifera;PTM.Populustremuloides;ETL.Eucalyptusglobulus.
圖2 梅花與其他物種SOC1基因的氨基酸多序列比對
Fig.2 Alignments of thePmSOC1-like deduced amino acid sequences with other SOC1 proteins from plants
氨基酸比對進一步發現,PmSOC1-like蛋白含有高度保守的MADS結構域、中度保守的K結構域和多變的C-端(圖2),屬于MADS-box基因家族中的Type-Ⅱ型[18],其MADS結構域和K結構域的下游還分別含有非保守的Ⅰ區和半保守的C區,因此又稱其為MIKC基因[19],也稱為MEF-2轉錄因子。Nakamura等[20]發現SOC1/TM3亞家族基因在C-端具有1個保守性很高的基序,即SOC1/MOTIF,這些基序在蛋白復合物的形成和轉錄中起重要的作用[21-22]。比對發現在PmSOC1-like基因的C-端也具有這個SOC1特有的基序(圖2)。因此,將PmSOC1-like基因劃入MADS-box基因家族SOC1/TM3亞家族中,推斷PmSOC1-like是具有生物功能的SOC1基因。
2.3 梅花PmSOC1-like的系統進化分析
為進一步了解梅花與其他物種SOC1蛋白的系統進化關系,選取了MADS家族的不同成員構建SOC1蛋白的系統進化樹(圖3)。根據系統進化樹,不同植物的SOC1蛋白可以分為2組,2種單子葉植物(玉米和小麥)單獨聚為1組;其余雙子葉植物的SOC1蛋白聚為1組。前人研究顯示,擬南芥MADS-box基因家族SOC1/TM3亞家族中包含AGL14、AGL19、AGL20(SOC1)、AGL42、AGL71和AGL72等6個基因[23]。在本研究中,PmSOC1-1與另外2種李屬植物桃和杏的親緣關系最近,先聚為1組,然后與擬南芥等其他雙子葉植物SOC1同源基因聚為1組。PmSOC1-2和PmSOC1-3則分別與研究較少的擬南芥SOC1/TM3亞家族中的AGL42/71/72和AGL14/19基因聚為1組。值得注意的是,在桃基因組中存在分別與梅花3個PmSOC1-like基因相對應的同源基因(圖3),且這些基因在梅花和桃基因組中具有相似的分布模式,都是2個基因位于同一染色體上,而另1個基因位于其他染色體上[24-25]。這種相似性表明,SOC1-like基因在梅花和桃中具有保守性。
2.4 梅花PmSOC1基因的表達模式分析
以梅花PP2A基因為內參[17],采用實時熒光定量PCR的方法分析3個PmSOC1-like基因在梅花不同組織中的表達情況。結果如圖4所示,在成年樹中,3個PmSOC1-like基因在根、莖、葉、葉芽或花芽等營養器官中有較高表達,而在萼片、花瓣、雄蕊、雌蕊、果實和種子等生殖器官中表達量很低;在1月齡播種苗中,PmSOC1-1和PmSOC1-3在根、莖和葉中均有表達,而PmSOC1-2則只在根中略有微弱表達。
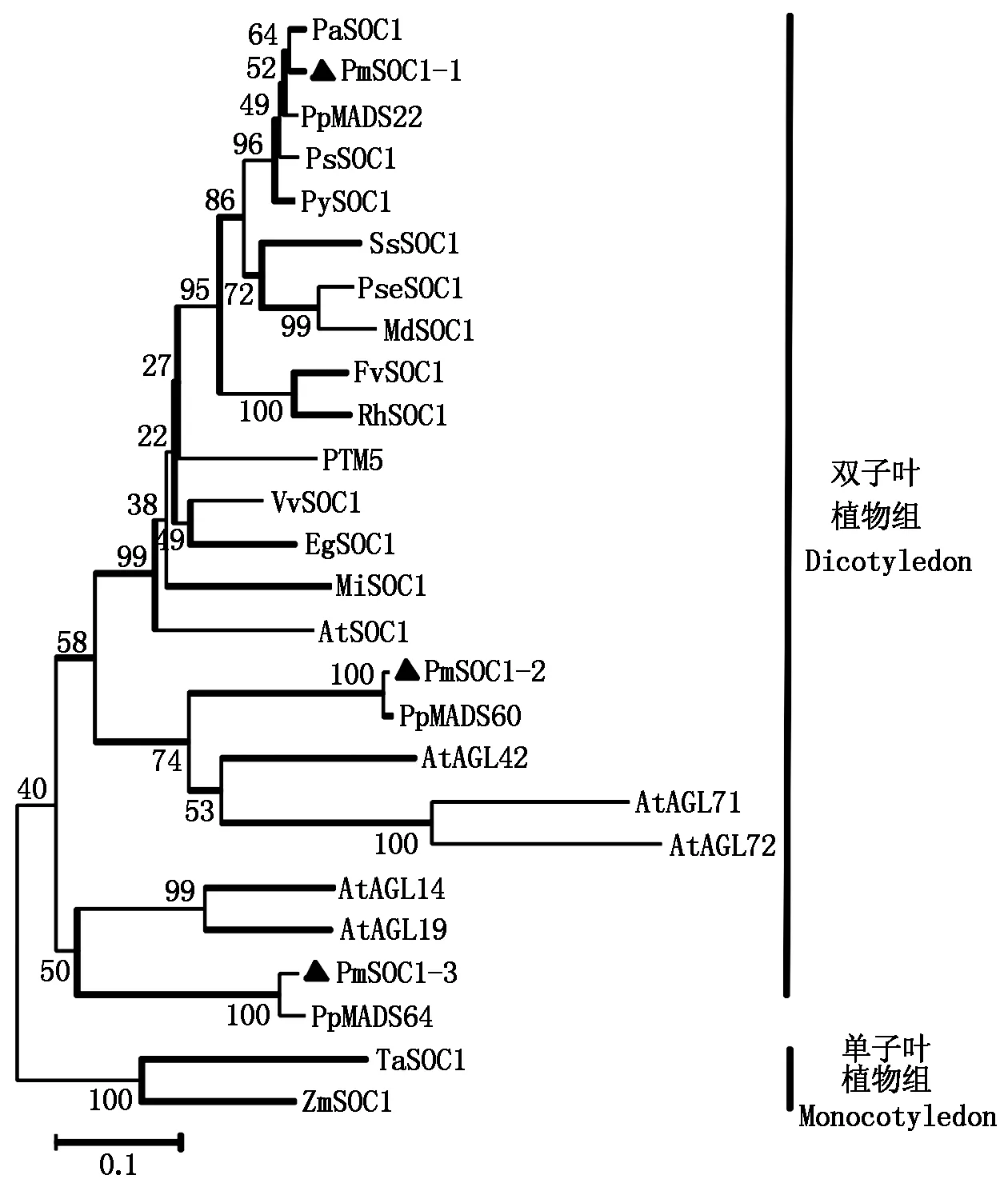
系統發育樹中基因的序列號如下:杏.PaSOC1(AGD88524);梅花.PmSOC1-1(AEQ20229),PmSOC1-2(KP938964),PmSOC1-3(KP938965);李.PsSOC1(AGD88523);櫻花.PySOC1(AEO20233);麻葉繡線菊.ScSOC1(AEO20234);石楠.PseSOC1(AEO20232);蘋果.MdSOC1(BAI49495);野草莓.FvSOC1(AEO20231);月季.RhSOC1(AEO20230);歐洲山楊.PTM5(AF377868);葡萄.VvSOC1(ACZ26527);巨桉.EgSOC1(XP_010053874);芒果.MiSOC1(ADX97324);擬南芥.AtSOC1(AEC10583),AtAGL42(AED97572),AtAGL71(AED96138),AtAGL72(AED96136),AtAGL14(AEE83062),AtAGL19(AEE84684);小麥.TaSOC1(BAF56968);玉米.ZmSOC1(ACG32892);桃.PpMADS22位于桃染色體scaffold 2:20,600,270…20,606,939,PpMADS60位于桃染色體scaffold 2:28,315,091…28,320,984,PpMADS64位于桃染色體scaffold 5:9,862,429…9,867,768。
The accession number for each protein in phylogenetic tree are as follows:Prunusarmeniaca.PaSOC1(AGD88524);Prunusmume.PmSOC1-1(AEQ20229),PmSOC1-2(KP938964),PmSOC1-3(KP938965);Prunussalicina.PsSOC1(AGD88523);Prunus×yedoensis.PySOC1(AEO20233);Spiraeacantoniensis.ScSOC1(AEO20234);Photiniaserratifolia.PseSOC1(AEO20232);Malusdomestica.MdSOC1(BAI49495);Fragariavesca.FvSOC1(AEO20231);Rosahybrid.RhSOC1(AEO20230);Populustremuloides.PTM5(AF377868);Vitisvinifera.VvSOC1(ACZ26527);Eucalyptusgrandis.EgSOC1(XP_010053874);Mangiferaindica.MiSOC1(ADX97324);Arabidopsisthaliana.AtSOC1(AEC10583),AtAGL42(AED97572),AtAGL71(AED96138),AtAGL72(AED96136),AtAGL14(AEE83062),AtAGL19(AEE84684);Triticumaestivum.TaSOC1(BAF56968);Zeamays.ZmSOC1(ACG32892);Prunuspersica.PpMADS22 is positioned on the peach genome map on scaffold 2:20,600,270…20,606,939 reverse,PpMADS60 is positioned on the peach genome map on scaffold 2:28,315,091…28,320,984 reverse,and PpMADS64 is positioned on the peach genome map on scaffold 5:9,862,429…9,867,768.
圖3 梅花PmSOC1-like與其他物種SOC1蛋白的系統進化分析
Fig.3 Phylogenetic evolutionary analysis of PmSOC1-like and SOC1 proteins
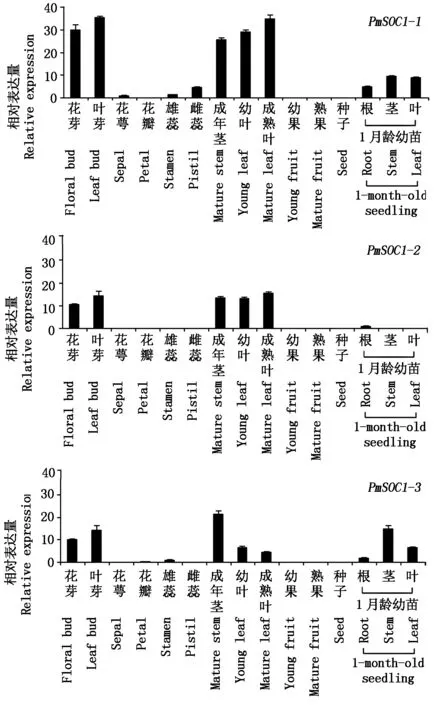
圖4 PmSOC1-like基因在不同組織器官中的相對表達量
為了進一步驗證PmSOC1-like基因在梅花開花轉變中是否與擬南芥中的AtSOC1基因一樣扮演相同的重要角色,筆者用石蠟切片的方法確定了梅花花芽分化過程中的8個時期,并檢測了3個基因在每個分化時期的表達情況(圖5)。在整個花芽分化過程中,3個PmSOC1-like基因的表達水平整體都呈現下降趨勢。在花芽未分化期(S1)表達量最高,一旦花芽被誘導分化表達量即開始下降,其中PmSOC1-2在3個基因中下降最為明顯(圖5)。
3 討論
開花是植物從營養生長到生殖生長的過渡,比較梅花等多年生木本植物與擬南芥等草本植物開花過程,發現最大的差異在于木本植物實生苗具備開花能力之前通常需要經歷一段很長的營養生長期(童期)。了解木本植物的開花轉變過程對縮短童期、加速品種改良進程具有十分重要的意義。SOC1是MADS-box基因家族中控制開花時間的基因,廣泛存在于單子葉植物和雙子葉植物中[26]。目前,在其他物種中鑒定到的SOC1同源基因在擬南芥中的過表達都一致地表現出了早花表型[27-30]。近期研究還發現,SOC1參與了冷響應信號和開花調節之間的反饋回路。SOC1蛋白可與抗寒基因CBF的啟動子直接結合以抑制CBF表達,從而促進開花;而CBF的過表達可以增加FLC的轉錄水平,進而實現對春化途徑中FLC的下游靶基因SOC1的負調控,以延遲開花[31]。這說明在冷響應信號和開花調節之間存在著1個反饋回路,以適應不斷變化的外界環境。
A.梅花不同花芽分化時期的石蠟切片圖;B.PmSOC1-like基因在梅花不同花芽分化時期的表達情況。S1.未分化期;S2.花原基形成期;S3.萼片分化期;S4.花瓣分化期;S5.雄蕊分化期;S6.雌蕊分化期;S7.雌蕊伸長期;S8.胚珠、花藥形成期。
A.Images of paraffin sections of floral bud at different developmental stages;B.Relative expression ofPmSOC1-like genes in the floral bud at different developmental stages.S1.Pre-differentiation stage;S2.Flower primordium differentiation stage;S3.Sepal differentiation stage;S4.Petal differentiation stage;S5.Stamen differentiation stage;S6.Pistil differentiation stage;S7.Pistil elongation stage;S8.Ovule and anther formation stage.
圖5PmSOC1-like基因在花芽分化時期的表達分析
Fig.5 Expression analyses ofPmSOC1-like genes duringP.mumefloral bud differentiation
本試驗從梅花中成功分離得到了3個成花調控網絡中的PmSOC1-like基因。序列同源性分析表明這些基因屬于MADS-box基因家族中的SOC1/TM3亞家族。系統進化分析顯示,PmSOC1-1、PmSOC1-2和PmSOC1-3分別與擬南芥的SOC1、AGL42/71/72和AGL14/19聚為一組。有研究表明,擬南芥SOC1/TM3亞家族中的AGL42、AGL71和AGL72 3個基因是由于SOC1基因在進化過程中的基因復制產生的[32]。因此,筆者推測梅花3個PmSOC1-like基因具有相同的祖先起源,但在進化過程中產生了分化,并最終在開花和營養器官發育中發揮著不同的作用。此外,與擬南芥中存在3個基因(AGL42/71/72)不同的是,在梅花和桃的基因組中都只有1個相應的同源基因(梅花:PmSOC1-2,桃:PpMADS60)。而且,AGL42、AGL71和AGL72在控制擬南芥開花中的功能被證明是冗余的[31]。因此,擬南芥SOC1/TM3亞家族中由于基因復制產生的基因多樣性可能是在擬南芥與李屬植物分化后發生的。
對PmSOC1-like基因進行不同組織部位的表達分析表明,3個基因均在營養器官中表達較高,而在生殖器官中表達量很低。此外,相對于1月齡幼苗,PmSOC1-1和PmSOC1-2在成年樹葉片和莖中的表達量更高,這與擬南芥AtSOC1的表達一致。在擬南芥中,AtSOC1的表達量隨著植株的生長而不斷提高,尤其在成熟葉片中的表達量最高[9,33-35]。PmSOC1-1和PmSOC1-2基因在梅花成年樹和幼苗中的不同表達水平可能反映植物的成熟度。在對梅花花芽分化過程中PmSOC1-like基因的表達研究發現,PmSOC1-like基因的表達水平整體都呈現下降趨勢。在擬南芥中,SOC1基因是調控植物從營養生長向生殖生長轉變的重要基因[36]。由此推測,PmSOC1-like基因在誘導梅花由營養生長向生殖生長轉變過程中起著重要作用。
近幾年的研究顯示,在其他一些木本植物中,SOC1同源基因存在不同的表達模式。如美洲山楊(Populustremuloides)的SOC1同源基因PTM5在維管組織中有特異表達,暗示該基因可能與美洲山楊木材的形成有關[12];紫斑牡丹PrSOC1在花芽中的表達量最高,在根、莖、葉中表達量略低[23]。月季RhSOC1則在頂芽中的表達量最高[37]。這些研究結果表明,SOC1在不同物種之間的表達存在一定的差異,推測SOC1基因在植物開花轉變的調控網絡中具有多種功能。
作為1個集成多個開花信號的整合子,SOC1已經被深入研究了十幾年。盡管人們對模式植物擬南芥AtSOC1已經有了比較深入的了解,但是對木本植物中SOC1同源基因的研究卻鮮見報道。目前,國家花卉工程技術中心得到了3條梅花PmSOC1-like基因,并對其進行了時空表達模式分析,為進一步深入研究SOC1基因在梅花開花過程中的功能奠定了理論基礎。但是,梅花SOC1與其他MADS-box開花相關基因、蛋白的作用關系還有待進一步探討研究;梅花SOC1基因的上游調控序列和下游調控區域也有待進一步深入研究。這會為從整體上揭示梅花的開花之謎奠定理論基礎依據,為今后研究SOC1在梅花其他生命活動中的可能作用提供技術支撐。
[1] Fornara F,de Montaigu A,Coupland G.SnapShot:control of flowering inArabidopsis[J].Cell,2010,141(3):550,550.e1-2.
[2] Simpson G G,Dean C.Arabidopsis,the rosetta stone of flowering time?[J].Science,2002,296(5566):285-289.
[3] Srikanth A,Schmid M.Regulation of flowering time:all roads lead to Rome[J].Cellular and Molecular Life Sciences,2011,68(12):2013-2037.
[4] Distelfeld A,Li C,Dubcovsky J.Regulation of flowering in temperate cereals[J].Current Opinion in Plant Biology,2009,12(2):178-184.
[5] Greenup A,Peacock W J,Dennis E S,et al.The molecular biology of seasonal flowering-responses inArabidopsisand the cereals[J].Annals of Botany,2009,103(8):1165-1172.
[6] Trevaskis B,Hemming M N,Dennis E S,et al.The molecular basis of vernalization-induced flowering in cereals[J].Trends in Plant Science,2007,12(8):352-357.
[7] 胡麗芳,金志強,徐碧玉.MADS-box基因對花的發育及開花早晚的影響[J].生命科學研究,2004,8(4):7-12.
[8] Moon J,Suh S S,Lee H,et al.TheSOC1 MADS-box gene integrates vernalization and gibberellin signals for flowering inArabidopsis[J].The Plant Journal,2003,35(4):613-623.
[9] Samach A,Onouchi H,Gold S E,et al.Distinct roles ofCONSTANStarget genes in reproductive development ofArabidopsis[J].Science,2000,288(5471):1613-1616.
[10] Ferrario S,Busscher J,Franken J,et al.Ectopic expression of the petunia MADS box geneUNSHAVENaccelerates flowering and confers leaf-like characteristics to floral organs in a dominant-negative manner[J].The Plant Cell,2004,16(6):1490-1505.
[11] Ding L,Wang Y,Yu H.Overexpression ofDOSOC1,an ortholog ofArabidopsisSOC1,promotes flowering in theOrchiddendrobiumChao Parya Smile[J].Plant and Cell Physiology,2013,54(4):595-608.
[12] Cseke L J,Zheng J,Podila G K.Characterization ofPTM5 in aspen trees:a MADS-box gene expressed during woody vascular development[J].Gene,2003,318(1):55-67.
[13] Sreekantan L,Thomas M R.VvFTandVvMADS8,the grapevine homologues of the floral integratorsFTandSOC1,have unique expression patterns in grapevine and hasten flowering inArabidopsis[J].Functional Plant Biology,2006,33(12):1129-1139.
[14] 劉傳嬌,王順利,薛璟祺,等.張秀新牡丹開花調控轉錄因子基因PrSOC1的克隆與表達分析[J].園藝學報,2014,41(11):2259-2267.
[15] Zhang Q X,Chen W B,Sun L D,et al.The genome ofPrunusmume[J].Nature Commun,2012,3(4):187-190.
[16] Porebski S,Bailey L G,Baum B R.Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components[J].Plant Mol Biol Rep,1997,15(1):8-15.
[17] Wang T,Hao R J,Pan H T,et al.Selection of suitable reference genes for quantitative Real-time polymerase chain reaction inPrunusmumeduring flowering stages and under different abiotic stress conditions[J].Journal of the American Society for Horticultural Science,2014,139(2):113-122.
[18] Alvarez-Buylla E R,Pelaz S,Liljegren S J,et al.An ancestral MADS-box gene duplication occurred before the divergence of plants and animals[J].Proceedings of the National Academy of Sciences,2000,97(10):5328-5333.
[19] Kaufmann K,Melzer R,Thei en G.MIKC-type MADS-domain proteins:structural modularity,protein interactions and network evolution in land plants[J].Gene,2005,347(2):183-198.
[20] Nakamura T,Song I J,Fukuda T,et al.Characterization ofTrcMADS1 gene ofTrilliumcamtschatcense(Trilliaceae) reveals functional evolution of theSOC1/TM3-like gene family[J].Journal of Plant Research,2005,118(3):229-234.
[21] Honma T,Goto K.Complexes of MADS-box proteins are sufficient to convert leaves into floral organs[J].Nature,2001,409(6819):525-529.
[22] Kramer E M,Irish V F.Evolution of genetic mechanisms controlling petal development[J].Nature,1999,399(6732):144-148.
[23] Becker A,Theiβen G.The major clades of MADS-box genes and their role in the development and evolution of flowering plants[J].Mol Phylogen Evol,2003,29:464-489.
[24] Wells C E,Vendramin E,Tarodo S J,et al.A genome-wide analysis of MADS-box genes in peach (Prunuspersica(L.) Batsch)[J].BMC Plant Biol,2015,15:41.
[25] Xu Z D,Zhang Q X,Sun L D,et al.Genome-wide identification,characterisation and expression analysis of the MADS-box gene family inPrunusmume[J].Mol Genet Genomics,2014,289:903-920.
[26] Smaczniak C,Immink R G,Angenent G C,et al.Developmental and evolutionary diversity of plant MADS-domain factors:insights from recent studies[J].Development,2012,139(17):3081-3098.
[27] Fu J X,Qi S,Yang L W,et al.Characterization of chrysanthemumClSOC1-1 andClSOC1-2,homologous genes ofSOC1[J].Plant Molecular Biology Reporter,2013,32(3):740-749.
[28] Nakano Y,Kawashima H,Kinoshita T,et al.Characterization ofFLC,SOC1 andFThomologs inEustomagrandiflorum:effects of vernalization and post-vernalization conditions on flowering and gene expression[J].Physiologia Plantarum,2011,141(4):383-393.
[29] Sreekantan L,Thomas M R.VvFTandVvMADS8,the grapevine homologues of the floral integratorsFTandSOC1,have unique expression patterns in grapevine and hasten flowering inArabidopsis[J].Functional Plant Biology,2006,33(12):1129-1139.
[30] Tan F C,Swain S M.Functional characterization ofAP3,SOC1 andWUShomologues from citrus(Citrussinensis)[J].Physiologia plantarum,2007,131(3):481-495.
[31] Seo E,Lee H,Jeon J,et al.Crosstalk between cold response and flowering inArabidopsisis mediated through the flowering-time geneSOC1 and its upstream negative regulatorFLC[J].The Plant Cell,2009,21(10):3185-3197.
[32] Dorca-Fornell C,Gregis V,Grandi V,et al.TheArabidopsisSOC1-like genesAGL42,AGL71 andAGL72 promote flowering in the shoot apical and axillary meristems[J].The Plant Journal,2011,67(6):1006-1017.
[33] Borner R,Kampmann G,Chandler J,et al.A MADS domain gene involved in the transition to flowering inArabidopsis[J].The Plant Journal,2000,24(5):591-599.
[34] Lee H,Shu S S,Park E,et al.TheAGAMOUS-LIKE20 MADS domain protein integrates floral inductive pathways inArabidopsis[J].Genes and Development,2000,14(18):2366-2376.
[35] Wang J W,Czech B,Weigel D.miR156-regulated SPL transcription factors define an endogenous flowering pathway inArabidopsisthaliana[J].Cell,2009,138(4):738-749.
[36] Liu C,Zhou J,Bracha-Drori K,et al.Specification ofArabidopsisfloral meristem identity by repression of flowering time genes[J].Development,2007,134(10):1901-1910.
[37] 李晶晶.薔薇科植物TFL1和SOC1基因的克隆及表達分析[D].武漢:華中農業大學,2011.
Molecular Cloning and Expression Analysis of Flowering-regulating Transcription FactorPmSOC1-like Gene inPrunusmume
LI Yushu1,2,YANG Weiru1,CHENG Tangren1,WANG Jia1,ZHANG Qixiang1
(1.Beijing Key laboratory of Ornamental Plants Germplasm Innovation and Molecular Breeding,National Engineering Research Center for Floriculture,Beijing Laboratory of Urban and Rural Ecological Environment,Landscape Architecture School,Beijing Forestry University,Beijing 100083,China;2.Beijing Vocational College of Agriculture,Beijing 102442,China)
The MADS-box geneSOC1/TM3 (Suppressor of overexpression of constans 1/Tomato MADS-box gene 3) integrates multiple flowering signals to regulate the transition from vegetative to reproductive development in plants. To better understand the molecular regulation of flower development inPrunusmume,three putative orthologs ofSOC1,includingPmSOC1-1,PmSOC1-2,andPmSOC1-3,were isolated.The expression patterns ofPmSOC1-1,PmSOC1-2 andPmSOC1-3 were determined by Real-time RT-PCR.EachPmSOC1-like gene has a single long open reading frame(ORF) encoding a putative protein of 214,217 and 219 amino acids respectively.Phylogenetic analysis suggested thatPmSOC1-1,PmSOC1-2,andPmSOC1-3 genes might be orthologous ofArabidopsisSOC1,AGL42/71/72,andAGL14/19,respectively.By Real-time quantitative RT-PCR experiment,threePmSOC1-like genes were mainly expressed in vegetative organs and expressed at low level in reproductive organs such as flowers,fruit,seed and so on.The expression of the threePmSOC1-like genes showed a decreasing trend.Above results suggest that thePmSOC1-like gene plays an important role in the transition from the vegetative growth to the reproductive growth phase.
Prunusmume;PmSOC1-like;Gene cloning;Expression analysis
2016-05-13
國家“863”計劃項目(2013AA102607);北京市共建項目專項
李玉舒(1982-),女,黑龍江伊春人,講師,在讀博士,主要從事園林植物栽培與應用研究。
張啟翔(1956-),男,湖北黃岡人,教授,博士,博士生導師,主要從事花卉資源與育種研究。
S685.17
A
1000-7091(2016)05-0078-08
10.7668/hbnxb.2016.05.012

