雞NLRC5啟動子區轉錄調控序列的初步研究
張 莘,郭曉敏,常國斌,朱鵬飛,徐 璐,仇玲玲,張 揚,徐 琪,陳國宏
(揚州大學 動物科學與技術學院,江蘇 揚州 225009)
雞NLRC5啟動子區轉錄調控序列的初步研究
張 莘,郭曉敏,常國斌*,朱鵬飛,徐 璐,仇玲玲,張 揚,徐 琪,陳國宏
(揚州大學 動物科學與技術學院,江蘇 揚州 225009)
為了探討NLRC5啟動子的潛在調控機制,將NLRC5基因啟動子系列缺失片段插入到pGL3-basic載體,構建重組質粒,并轉染DF1細胞系。通過雙熒光素酶實驗尋找核心調控區,然后用目標捕獲測序檢測27個雞品種的NLRC5核心啟動子區的SNPs,并利用TRANSFAC,JASPAR 和 MethPrimer預測該區域的轉錄因子結合位點和CpG島。結果顯示,NLRC5啟動子有2個核心區域,分別是1—617和1448—2108。第一個核心區域內存在3個SNPs,其中SNP1影響轉錄因子Hic1的結合序列,但SNPs對CpG島不產生影響,表明NLRC5基因的啟動子區的調控可能受不同因素的影響,但SNPs并不在甲基化的水平上影響啟動子的活性。
雞;NLRC5啟動子;SNPs;轉錄因子結合位點;CpG島
先天免疫和適應性免疫是動物宿主防御病毒、細菌和寄生蟲等病原體的2種免疫防御形式,多種模式識別受體識別病原體進而激活相應的免疫反應[1-2]來進行防御。目前,已發現4種模式識別受體家族,分別是 TLRs(Toll-like receptors)、CLRs(C-type lectin receptors)、RLRs(RIG-I-like receptors) 和 NLRs(NOD-like receptors)[1,3]。NLRC5蛋白屬于NLR家族[4-5],包含N端CARD、中心NACHT、C端LRRs 3個結構域。文獻報道NLRC5表達于多種細胞和組織,免疫組織和器官表達水平較高[3,6-7]。有大量研究表明,NLRC5在先天免疫和適應性免疫中都發揮著重要的作用,但本身的調控機制還不清楚,而啟動子的調控在轉錄環節中占據著至關重要的地位,國內關于NLRC5啟動子的結構和調控等的研究較少,本研究克隆了NLRC5的啟動子區,即起始密碼子前4 372 bp,并將不同長度的啟動子片段插入pGL3-basic載體中構建系列缺失載體,轉染DF1細胞,利用雙熒光素酶實驗來反映不同長度啟動子片段的活性來確定核心區域,檢測核心區域內SNPs,并進行轉錄因子結合位點和CpG島預測,以期通過對NLRC5啟動子區的初步分析,為NLRC5調控等提供信息和參考依據。
1 材料與方法
1.1 材料
1.1.1 動物和細胞系
本研究使用的細胞系和27個雞品種分別是:DF-1細胞系、紅色原雞(RJ)、茶花雞(CH)、藏雞(ZC)、白耳雞(BE)、仙居雞(XJ)、綠殼蛋雞(GS)、太湖雞(TH)、如皋雞(RG)、蕭山雞(XC)、北京油雞(BY)、固始雞(GS)、狼山雞(LS)、大骨雞(BB)、青腳麻雞(QJ)、廣西黃雞(GY)、文昌雞(WC)、絲羽烏骨雞(SC)、雪山草雞(XS)、鹿苑雞(LY)、溧陽雞(LC)、斗雞(DJ)、斗雞與羅斯雞雜交后代(DR)、來航雞(LH)、羅斯雞(RC)、AA雞(AA)、安卡雞(AK)、隱形白洛克(RW)。其中DF-1細胞購自北京北納創聯生物技術研究院,用添加10%胎牛血清的DMEM培養基培養。27個雞品種中XJ、CH、LY、BE、ZC、GS、BB、DJ、LS、SC、XC、BY等12個地方雞品種來自中國農業科學研究院家禽科學研究所國家地方禽種資源基因庫,RJ亞種采自云南省野生動物救護中心,另外14個雞品種來自揚州大學動物科學與技術學院遺傳資源實驗室禽種資源基因庫。
1.1.2 主要試劑
SYBR?PreniExTaqTMⅡ(Tli RNaseH Plus)、Prime STAR?GXL DNA Polymerase、MiniBEST Agarose Gel DNA Extraction Kit、DNA Ligation Kit Ver.2.1、DH5α感受態細胞購自TaKaRa公司;雙熒光素酶報告基因載體pGL3-basic和pRL-SV40、FuGENE?HD Transfection Reagent、Dual-Luciferase?Reporter Assay System購自Promega公司;無內毒素質粒大提試劑盒、FastQuant RT Kit、離心柱型普通DNA產物純化試劑盒、質粒小提試劑盒購自天根公司;胎牛血清、DMEM培養基、胰酶來自Hyclone;限制性內切酶KpnⅠ和NheⅠ來自Thermo。
1.1.3 主要儀器
AG-2233Mini式小型高速離心機和普通PCR儀為Eppendorf公司,Nano Drop 1000分光光度計、CO2培養箱來自Thermo公司,凝膠成像系統Gel Doc TM EZ imager、自動細胞計數儀購自BIO-RAD,DK-600 恒溫水槽和隔水式恒溫培養箱購自上海精宏,還有7500熒光定量PCR儀(ABI)、AllegraTMX-22R離心機(Beckman)、超低溫冰箱(海爾)、恒溫振蕩器(培英)、瓊脂糖凝膠電泳系統(北京六一)、IX71 熒光倒置顯微鏡(OLYMPUS)、Synergy 2 化學發光檢測儀(Bio Tek)、SW-CJ-1F型無菌超凈工作臺(蘇州凈化設備有限公司)。
1.2 DNA提取
每個雞品種收集10個樣本,翅靜脈采血1 mL,置于盛有0.1 mL肝素鈉抗凝劑的2 mL離心管中用于提取基因組DNA,選擇完整性好(無拖尾現象),D260/D280介于1.8~2.0之間的DNA樣品稀釋至50 ng·μL-1。
1.3 系列缺失表達載體的構建
根據GenBank上紅色原雞NLRC5基因序列(Gene ID: 100857413),采用系統缺失的方法設計引物(表1),運用PCR技術擴增NLRC5啟動子,使用KpnⅠ和NheⅠ限制性內切酶雙酶切PCR純化產物和pGL3-Basic質粒,切膠回收后進行連接,然后轉化DH5α感受態細胞,經菌液PCR、酶切、測序鑒定正確后,無內毒素質粒大提試劑盒提取,-20 ℃保存、備用。
將構建的系列缺失表達載體轉染DF1細胞系,轉染前將細胞以5×105的密度接種在24孔板中,待細胞完全貼壁生長至80%融合度時,參照FuGENE?HD Transfection Reagent轉染試劑的使用說明書,分別將各目的質粒與內參質粒共轉染到細胞中。24 h后檢測其雙熒光素酶活性。
表1 NLRC5啟動子系列缺失表達載體構建引物
Table 1 Primers for deletion plasmids construction of chickenNLRC5 promoter

名稱Name引物序列Primersequence(5'-3')產物Product/bp作用FunctionP1-FGGGGTACCATGAGAGGAAGTGCAAGTATCTGTTCAGGCAGCAGG4372構建P1ConstructingP1P1-RCGGCTAGCTGCTGTGTGGTCAGCCTCCCTTCAGTCGP2-FGGGGTACCGCGGCGCCGCGCTTTCACTTTTG3756構建P2ConstructingP2P2-RCGGCTAGCTTGCTGTGTGGTCAGCCTCCCTTCAGTCP3-FGGGGTACCGAACCCCCATCCACCCTCTGTGG2925構建P3ConstructingP3P3-RCGGCTAGCTTGCTGTGTGGTCAGCCTCCCTTCP4-FGGGGTACCAGCAGTAAGTTGTGAATTCATGAGAGAGGACG2265構建P4ConstructingP4P4-RCGGCTAGCTTGCTGTGTGGTCAGCCTCCCTTC
下劃線為酶切位點;KpnⅠ識別序列GGTACC,NheⅠ識別序列GCTAGC。
Underline indicates restriction enzyme recognition sites;KpnⅠrecognizes GGTACC;NheⅠrecognizes GCTAGC.
1.4 SNP、轉錄因子及CpG島檢測
運用目標捕獲測序檢測27個雞品種NLRC5啟動子區的SNPs。利用在線軟件TRANSFAC和JASPAR對轉錄因子結合位點進行預測,并運用MethPrimer預測目標序列CpG島(GC含量大于50%,Obs/Exp比值>0.6和CpG島范圍大于100 bp為判定標準)。
1.5 數據分析
對所有的實驗數據使用SPSS 21.0軟件(SPSS, Chicago, IL, USA)進行分析。用鄧肯氏法進行多重比較,DNAstar軟件比對DNA測序結果。利用Microsoft Excel 2010對以上分析所得數據進行整理歸納。
2 結果與分析
2.1 系列缺失載體雙熒光素酶活性實驗結果
為了尋找NLRC5啟動子的核心區域,構建了4個缺失載體,經酶KpnⅠ單酶切、酶KpnⅠ和NheⅠ雙酶切后瓊脂糖凝膠電泳,結果如圖1,單酶切后條帶單一,雙酶切后得到線性載體片段和NLRC5啟動子目的片段,大小和設計預期相同,載體構建正確,可用于后續實驗。
上述4個缺失載體的雙熒光素酶實驗結果(圖2)顯示片段P1的活性是片段P3的83%,并且兩者的活性都大于片段P2;片段P4的活性幾乎為0,說明刪除1—617和1 448—2 109后,活性顯著下降,而刪除617—1 448后活性上升,2 109—4 372幾乎沒有啟動子活性,初步推測NLRC5啟動子存在2個活性區域,分別為1—617和1 448—2 109。
2.2 不同雞種的SNP
本研究利用目標捕獲測序檢測27個雞種NLRC5啟動子區SNPs,將測序結果中reads超過50%的低質量堿基去除[quality value≤5 (E)]后的結果如表2、表3所示,共發現3個SNPs位點,其中SNP1位點和NCBI上dbSNP數據庫不匹配。表3顯示了各雞種NLRC5基因啟動子SNPs分布情況,27個雞種中仙居雞、大骨雞、文昌雞、絲羽烏骨雞、安卡雞、隱形白洛克6種雞種沒有SNPs,其他雞種中SNP1出現的比例最高,SNP3僅存在于茶花雞、太湖雞、斗雞以及斗雞和羅斯雞雜交后代中。除茶花雞和太湖雞中含有3個SNPs外,其余雞種SNPs大致相同。這些SNPs與NLRC5的表達調控和雞的性狀之間的關系還需要進一步驗證。
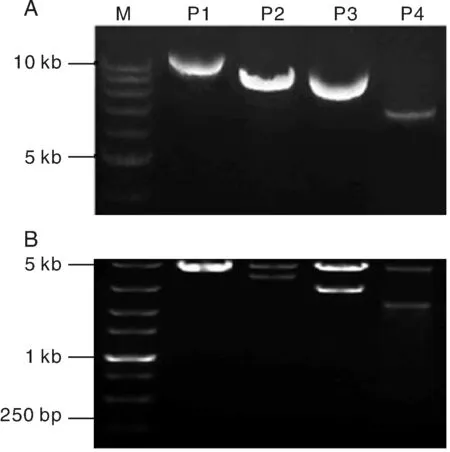
M為DNA相對分子質量標準;P1~P4為4個缺失載體Letter M represented DNA relative molecular mass standard; Letter P1 to P4 represented 4 deletion plasmids圖1 雞NLRC5啟動子系列缺失載體的單酶切(A)和雙酶切(B)瓊脂電泳結果Fig.1 Single and double restriction enzyme digestion products of plasmids of NLRC5 promoter
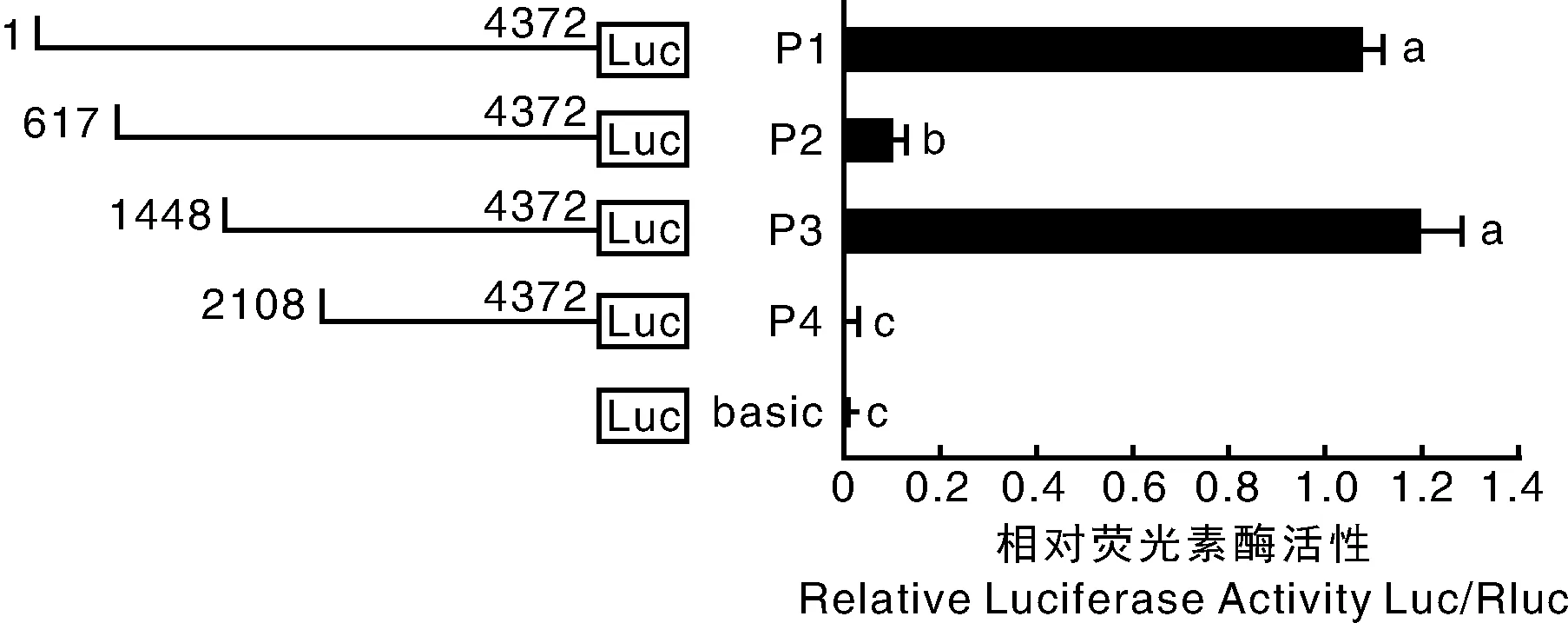
左邊顯示插入pGL3-basic載體的不同區域啟動子片段,右邊顯示缺失載體的相對熒光素酶活性,數據表示為3個重復的平均數±標準差,不同的小寫字母表示數據間差異顯著(P<0.05)The left showed the different length promoter fragments inserted into pGL3-basic vector. The right showed the relative luciferase expression activities of corresponding constructs. Data were expressed as mean±SD of 3 repeats. Different lowercase letters represented significant difference(P<0.05)圖2 雞NLRC5啟動子系列缺失載體的活性Fig.2 Activity of different length of NLRC5 promoter of chicken
2.3NLRC5啟動子區轉錄因子結合位點預測
本研究利用在線軟件TRANSFAC和JASPAR對轉錄因子結合位點進行預測,發現SNP1處于轉錄因子結合位點,結合轉錄因子Hic1(圖3),基序為GGGTGGCAT,此處結合序列發生變化,藏雞、仙居雞、固始雞、狼山雞、大骨雞、青腳麻雞、文昌雞、絲羽烏骨雞、斗雞、安卡雞、隱形白洛克沒有這個SNP,NLRC5啟動子區SNPs通過影響轉錄因子結合位點來影響啟動子的活性,從而影響基因的表達調控,使機體表現不同的抗病力。
2.4NLRC5啟動子區CpG島預測
本研究利用MethPrimer預測1-617序列CpG島(圖4),發現存在1個CpG島,其范圍是312~562 bp,大小為251 bp,有2個SNP位點(SNP2和SNP3)位于CpG島內,將突變后的序列重新提交,發現CpG島數量和長度均未發生改變。推斷SNP位點可能不通過影響啟動子區甲基化水平而調控基因表達。
表2 NLRC5啟動子第一個核心區域SNP信息
Table 2 SNP in the first core region ofNLRC5 promoter

多態位點信息PolymorphicsiteinformationSNP1SNP2SNP3位點坐標Location/bp217408551突變MutationA/GT/YG/R數據庫一致性Databaseconsistency011
0,不匹配;1,匹配;簡并堿基Y(C+T)、R(A+G)。
0, Mismatching; 1, Match; Y (C+T); R (A+G).
表3 不同雞種SNP分布情況
Table 3 SNP status in different chicken breeds
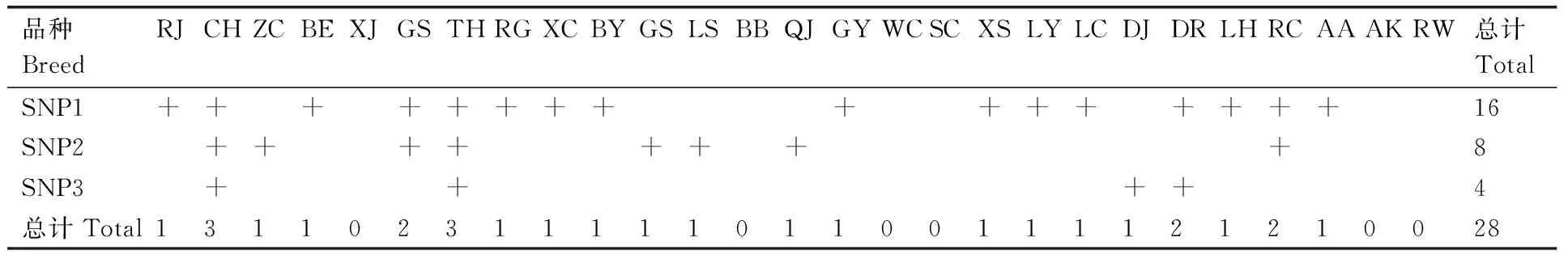
品種BreedRJCHZCBEXJGSTHRGXCBYGSLSBBQJGYWCSCXSLYLCDJDRLHRCAAAKRW總計TotalSNP1++++++++++++++++16SNP2++++++++8SNP3++++4總計Total13110231111101100111121210028
+,存在;空白,不存在。
+, Existence; Blank, Non-existent.
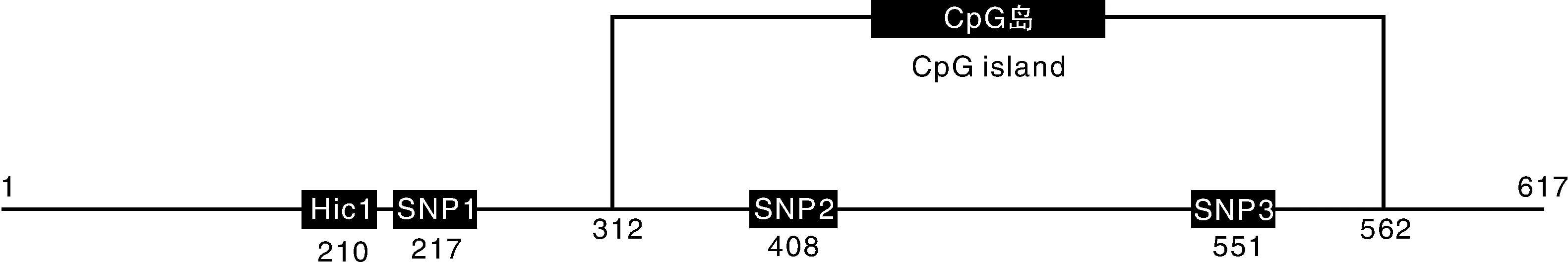
圖3 NLRC5啟動子第一個核心區域的示意圖Fig.3 The first core region of NLRC5 promoter
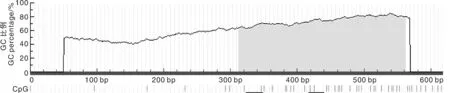
圖4 SNP2和SNP3突變前、后NLRC5啟動子第一個核心區域CpG島預測Fig.4 CpG islands predicted by MethPrimer in NLRC5 promoter
3 討論
NLRC5可調控NF-κB(nuclear factor-k-gene binding)[8-9]、I型干擾素[6,10]、炎性體信號路徑及MHC-I(major histocompatibility complex class I)基因表達[3,11-12], 表明其在免疫過程中有重要作用。雙熒光素酶實驗結果顯示NLRC5啟動子具有1—617和1 448—2 108兩個核心區域,本研究主要研究NLRC5啟動子1—617核心區域,通過對27個雞種的測序發現了3個SNPs位點,SNP3位點只在茶花雞、太湖雞、斗雞以及斗雞和羅斯雞雜交后代4個雞種中檢測到,仙居雞、大骨雞、文昌雞、絲羽烏骨雞、安卡雞、隱形白洛克6種雞中未發現SNPs,造成這種現象的可能原因是:雞種受外界環境和遺傳條件的影響,不同類型雞種有不同的表現型,從而導致雞種的抗病力不同,所以不同類型雞種的SNPs位點也不相同。到目前為止,大量文獻報道了很多基因的啟動子區域呈現相對豐富的多態性,并使基因的表達調控發生改變,從而對動物的多種性狀產生影響[13-18],但是這些SNPs位點是否影響雞種的抗病力需要進一步作缺失來縮短核心啟動子區,擴大樣本量來尋找核心啟動子區的不同單倍型來驗證。
有報道稱Hic1是抑癌候選基因,在正常組織中廣泛表達,但在多種腫瘤中表達缺失[14]。一系列的研究表明Hic1是調節細胞生長及凋亡關鍵基因的中心轉錄調節因子,抑制Hedgehog信號通路[18]和調節Wnt信號通路[16],并且能夠調節phrin-A1基因的直接轉錄,從而抑制上皮腫瘤的形成[17],所以Hic1應該與機體的免疫有關。有研究表明,NLRC5可正調控Wnt信號通路,進而誘導下游靶基因轉錄和蛋白表達,從而影響肝癌細胞的增殖、遷移和侵襲特性[19]。由此可以看出,Hic1和NLRC5之間可能存在一定聯系。本研究預測到NLRC5啟動子存在該轉錄因子的結合位點,可能影響NLRC5的轉錄。另外,SNP1位點位于轉錄因子結合位點,通過影響轉錄因子與調控序列的結合來調控基因表達,說明NLRC5啟動子區SNPs對啟動子調控有影響。有文獻報道,DNA甲基化可影響靶基因表達水平[20],本研究預測到NLRC5啟動子區有范圍較近的1個CpG島,并且發現有2個SNPs位點處于預測的CpG島區,說明DNA甲基化水平對NLRC5表達調控有重要作用。但是SNPs不對CpG島產生影響,該原因可能是本研究所采用的序列位于核心啟動子區,其保守性較強,SNPs位點可能不在甲基化水平上影響NLRC5的表達水平,NLRC5啟動子功能和NLRC5的調控機制還需進一步研究探討。
[1] KUMAR H, KAWAI T, AKIRA S. Pathogen recognition in the innate immune response[J].BiochemicalJournal, 2009, 420(1):1-16.
[2] LAMKANFI M, KANNEGANTI T D. Regulation of immune pathways by the NOD-like receptorNLRC5[J].Immunobiology, 2012, 217(1):13-16.
[3] NEERINCX A, LAUTZ K, MENNING M, et al. A role for the human nucleotide-binding domain, leucine-rich repeat-containing family memberNLRC5 in antiviral responses[J].JournalofBiologicalChemistry, 2010, 285(34):26223-26232.
[4] WILMANSK J M, PETNICKI-OCWIEJA T, KOBAYASHI K S. NLR proteins: integral 394 members of innate immunity and mediators of inflammatory diseases[J].JournalofLeukocyteBiology, 2008, 83(1):13-30.
[5] TING J P, LOVERING R C, ALNEMRI E S, et al, The NLR gene family: a standard nomenclature[J].Immunity, 2008, 28(3):285-287.
[6] CUI J, ZHU L, XIA X J, et al. NLRC5 negatively regulates the NF-kappa B and type I interferon signaling pathways[J].Cell, 2010, 141(3):483-496.
[7] DAVIS B K, ROBERTS R A, HUANG M T, et al. Cutting edge:NLRC5-dependent activation of the inflammasome[J].JournalofImmunobiology, 2011, 186(3):1333-1337.
[8] LI HY, LIU H, WANG C H, et al. Deactivation of the kinase IKK by CUEDC2 though recruitment of the phosphatase PP1[J].NatureImmunolbiology, 2008, 9(5):533-541.
[9] BISWAS A, MEISSNER T B, KAWAI T, et al. Cutting edge: impaired MHC class I expression in mice deficient for Nlrc5/class I transactivator[J].JournalofImmunolbiology, 2012, 189(2):516-520.
[10] WANG L, LI K, XU Q, et al. Potential synergy between SNP and CpG-A or IL-1 beta in regulating transcriptional activity of IL-20 promoter[J].JournalofInvestigativeDermatology, 2014, 134(2):389-395.
[11] KUENZEL S, TILL A, WINKLER M, et al. The nucleotide-binding oligomerization domain-like receptorNLRC5 is involved in IFN-dependent antiviral immune responses[J].JournalofImmunology, 2010, 184(4):1990-2000.
[12] 常國斌,陳蓉,欒德琴,等. 細菌內毒素對雞NLRC5受體及IFN基因轉錄的影響[J]. 畜牧獸醫學報,2011,42(11):1598-1604. CHANG G B, CHEN R, LUAN D Q, et al. Transcription change of chickenNLRC5 receptor and IFN genes initiated by bacterial endotoxins[J].ChineseJournalofAnimalandVet-
erinarySciences, 2011, 42(11):1598-1604. (In Chinese with English abstract)
[13] JENAL M, BRITSCHGI C, FEY M F, et al. Inactivation of the hypermethylated in cancer 1 tumour suppressor-not just a question of promoter hypermethylation[J].SwissMedicalWeekly, 2010, 140:w13106.
[14] DI MARCOTULLIO L, FERRETTI E, DE SMAELE E, et al. REN(KCTD11) is a suppressor of Hedgehog signaling and is deleted in human medulloblastoma[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica, 2004, 101(29):10833-10838.
[15] VALENTA T, LUKAS J, DOUBRAVSKA L, et al. HIC1 attenuates Wnt signaling by recruitment of TCF-4 and β-catenin to the nuclear bodies[J].TheEMBOJournal, 2006, 25 (11):2326-2337.
[16] ZHANG W,ZENG X, BRIGGS K J, et al. A potential tumor suppressor role for Hicl in breast cancer through transcriptional repression of ephrin-A1[J].Oncogene, 2010, 29(17):2467-2476.
[17] PAN S, WANG Z, CHEU Y, et al. Inactivation of tumor suppressor gene HIC1 in gastric cancer is reversed via small activating RNAs[J].Gene, 2013, 527(1):102-108.
[18] YANG Z H, ZHOU C L, ZHU H, et al. A functional SNP in the MDM2 promoter mediates E2F1 affinity to modulate cyclin D1 expression in tumor cell proliferation[J].AsianPacificJournalofCancerPrevention, 2014, 15(8):3817-3823.
[19] 彭云云. NLRC5靶向Wnt/β-catenin信號通路調控肝癌細胞增殖、遷移和侵襲的功能及機制研究[D]. 合肥:安徽醫科大學,2015. PENG Y Y. Mechanisms of NLRC5 in the cell proliferation, migration and invasion of hepatocellular carcinoma by targeting the Wnt/ρ-catenin signaling pathway [D]. Hefei: Anhui Medical University, 2015. (in Chinese with English abstract)
[20] TAMURA G, YIN J, WANG S, et al. E-cadherin gene promoter hypermethylation in primary human gastric carcinomas[J].JournaloftheNationalCancerInstitute, 2000, 92(7):569-573.
(責任編輯 盧福莊)
Preliminary study on transcriptional regulation sequence of chickenNLRC5 promoter
ZHANG Xin, GUO Xiaomin, CHANG Guobin*, ZHU Pengfei, XU Lu, QIU Lingling, ZHANG Yang, XU Qi, CHEN Guohong
(CollegeofAnimalScienceandTechnology,YangzhouUniversity,Yangzhou225009,China)
In order to explore the potential regulation mechanism of chickenNLRC5 promoter, deletion fragments of chickenNLRC5 promoter was cloned and inserted into pGL3-basic vector to construct recombinant plasmids for transfecting DF1 cells. Dual-luciferase assay was carried out to find the core regulation region. Then target sequence capture assay was used to detect SNPs in the core region ofNLRC5 promoter of 27 chicken breeds. Finally, transcription factor binding sites and CpG islands were predicted by using online softwares TRANSFAC, JASPAR and MethPrimer. The results showed thatNLRC5 promoter had 2 core regions, 1 to 617 and 1 448 to 2 108, respectively. There were 3 SNPs loci totally in the first core region in all breeds. SNP1 was found to influence the transcription factor Hic1 binding site. However, SNPs in the first core region did not affect CpG island. The results above suggested thatNLRC5 promoter activity might be regulated by different factors, but SNPs didn’t influence the promoter activity at methylation level.
chicken;NLRC5 promoter; SNPs; transcription factor binding site; CpG island
10.3969/j.issn.1004-1524.2017.03.07
2016-10-01
國家科技支撐計劃(2015BAD03B03);江蘇省六大人才高峰項目(2015-NY-019)
張莘(1995—),女,江蘇邳州人,本科在讀,E-mail: 768655881@qq.com
*通訊作者,常國斌,E-mail: passioncgb@163.com
S852.4
A
1004-1524(2017)03-0395-06
浙江農業學報ActaAgriculturaeZhejiangensis, 2017,29(3): 395-400
http://www.zjnyxb.cn
張莘,郭曉敏,常國斌,等. 雞NLRC5啟動子區轉錄調控序列的初步研究[J].浙江農業學報,2017,29(3): 395-400.

