番茄熱激轉錄因子HSF家族的系統進化分析
王婷婷 仇有文 王沛文 陳宏宇 楊俊穎 王竇逗 伍淼 王傲雪

摘要:熱激轉錄因子(heat Shock factors)普遍存在于整個生物界中,在調控植物生長發育以及對環境的響應中起重要作用。目前,已經對多個物種的HSF基因進行生物信息學分析,但未見對番茄中HSF基因家族的分析報道。通過番茄基因組數據庫,鑒定和分析番茄HSF轉錄因子家族,獲得24個HSF基因家族成員。多重序列比對發現番茄HSF基因具有保守的DBD結構域和廣泛的保守基序。根據與擬南芥基因系統進化分析將這些基因分類,分成Ⅰ、Ⅱ、Ⅲ支,又將1支分成A、B、C 3個亞支,并且存在5對旁系同源蛋白和10對直系同源蛋白。染色體分布和遺傳分析結果表明,番茄HSF基因存在于10條染色體上,呈不均勻分布。
關鍵詞:番茄;熱激轉錄因子(HSF);基因家族;生物信息學;系統進化
中圖分類號:S641.203 文獻標志碼:A 文章編號:1002—1302(2016)01—0048—05
轉錄因子又稱反式作用因子,其主要功能是激活或抑制基因的轉錄,在調控植物生長發育及對環境的響應中起重要作用。熱轉錄因子(heat factor HSFs)是近年來在植物中發現的一類重要的轉錄因子,廣泛分布于植物細胞內,在熱脅迫響應基因的中心元件,在植物熱脅迫信號轉導以及耐熱性調控中起著關鍵作用。熱激轉錄因子(heat shock factor,HSF)在本質上是具有轉錄調節活性的蛋白質,植物遭受高溫脅迫的時候熱激基因迅速增加,導致熱激蛋白快速累積,熱激蛋白作為分子伴侶幫助相關蛋白重新折疊、組裝、分配和降解,對受損蛋白進行修復起著極其重要的作用。熱激轉錄因子是信號轉導途徑的末端組分,通過調節基因活性對熱激和其他脅迫作出響應。這些經熱激而活化的熱激轉錄因子可以識別并特異性結合在熱激蛋白(heat shock protein,HSP)基因啟動子區熱激元件(heat shock element,HSE)的保守基序上,從而調控熱激蛋白基因的開啟與關閉,誘導HSPs的轉錄,完成其相應的生物學功能。20世紀80年代,自研究者首次在酵母中克隆HSF基因以來,多種哺乳動物HSF基因相繼被克隆。然而植物的第1個HSF基因是在番茄中克隆得到的,隨著基因組測序工作的不斷進行,在擬南芥和水稻中也克隆得到相應的HSFs基因,隨后研究者在大豆,玉米等植物分別發現至少具有52,30個HSFs基因,由此可知,植物HSFs基因是一個大的基因家族,而且對植物耐熱具有重要意義。
典型的熱激轉錄因子一般包括4個部分:N端的DNA結合域(DNA binding domain,DBD)、寡聚化結構域(HR-A/B)、細胞核定位信號(nuclear localizationsignal,NLS)、細胞核輸出信號(nuclear export signal,NES),少數還具有1個C端激活域(C-terminal activation domain,CTAD)。植物熱激因子通過形成回文發卡結構,特異地結合高度保守的熱激元件,從而控制熱激蛋白的表達。根據保守DBD和HR-A/B區的結構特點,熱激轉錄因子又可以分為A、B、C 3類。這3類基因主要區別表現為:B類基因HR-A/B結構域中A、B結構域之問只有7個氨基酸殘基,在A類、C類中,除了這7個氨基酸,還分別有21個和7個氨基酸的插入;另外,CTAD和NES的結合區域是A類HSFs所特有的結構,B類、C類均不包含CTAD結構域。
番茄是一種重要的蔬菜作物,在夏季栽培或保護地生產中,高溫是影響其產量和品質的主要非生物脅迫因素之一。番茄全基因組測序工作的完成,為其遺傳育種及相關基因的生物功能鑒定提供重要的信息參考。本研究利用生物信息學方法,在番茄基因組數據庫中搜索HSFs基因,分析這些基因的數量、序列特征、染色體定位以及進化關系等,研究結果不僅有助于鑒定番茄HSFs基因家族的功能,還可進一步為培育番茄耐熱新品種提供理論基礎和基因信息。
1材料與方法
1.1番茄bZIP家族成員的確定
從擬南芥基因組數據庫TAIR(http:∥www.arabidopsis.org/)獲得已經鑒定的21個擬南芥HSF蛋白序列,將其在番茄基因組數據庫SGN(hnp:∥www.sgn.comell.edu/)上進行同源性搜索,E值設定為1×10-10;以關鍵詞“HSF"在SGN數據庫中進行搜索,合并2次搜索結果,去除重復,下載候選番茄HSF核苷酸序列、氨基酸序列及其內含子一外顯子等信息。通過CELLO(http:∥cello.1ife.nctu.edu.tw/)進行亞細胞定位分析。利用在線工具Pfam(http:∥pfam.janelia.org/)和SMART(http:∥smart.embl-heidelberg.de/)對獲得的候選番茄HSF蛋白家族成員的氨基酸序列進行保守HSF蛋白結構域預測。利用ExPASy Proteomic Server(ht-tp:∥expasy.org/tools/protparam.html)對所有番茄HSF蛋白氨基酸序列進行分子質量、等電點預測。
1.2系統進化樹的構建及保守motif的分析
利用擬南芥和番茄的HSF氨基酸全序列構建系統發育樹,用以研究HSF基因家族成員的進化關系。使用ClustalX2.0程序對氨基酸序列進行多序列聯配,將結果輸出到MEGA 6.0軟件中,使用鄰接法(Neighbor-Joiningmethod)構建擬南芥和番茄的HSF基因家族的系統發育樹,Pairwise Deletion處理缺失數據,P-distance模型,Boot-strap檢驗1 000次,去除Bootstrap支持率低于50%的節點。通過MEME(http:∥meme.nbcr.net/meme/)在線分析番茄HSF基因的保守motif,motif的長度設置2~200 bp,最多檢測25個motif。
1.3番茄保守基序分析
通過MEME工具(http:∥meme.nbcr.net/meme/)在線分析番茄HSF基因的保守motif,保守基序最小長度為6,最大長度為50。最大發現數目為15個,其他為默認數值。
1.4染色體定位分析
根據下載的番茄基因組數據庫的染色體數據,繪制番茄染色體圖;在利用獲得的番茄HSF基因信息通過MapDrawV2.1繪制HSF基因在染色體的定位圖。
2結果與分析
2.1番茄HSF基因家族的鑒定
通過對番茄基因組的比對分析,獲得24條番茄HSF基因家族成員(HSF01~HSF80)(表1),以蛋白質生化屬性分析結果表示。
2.2番茄熱激轉錄因子基因家族的鑒定
為了全面獲得番茄HSF基因及其蛋白序列分別對番茄基因組數據庫SGN進行了搜索,將得到的候選基因在Pfam和SMART數據庫中檢測其是否含有HSF結構域,同時為了驗證獲得HSF基因的準確性將其蛋白質序列在數據庫MAR-COIL(http:∥toolkit.tuebingen.mpg,de/sections/seqanal)進行HSF基因專有的卷曲結構檢測,最后鑒定獲得了24個番茄HSF基因分別將其命名為solyHSF01~solyHSF24.番茄HSF基因蛋白質編碼的長度介于142(solyHSFl4)~527(SolyHSF01)氨基酸之間;理論等電點位于4.68~9.44之間,最高的是SolyHSF14,高達9.44,最低等電點為SolyHSF07,只有4.68,這些蛋白從堿性到酸性分布都有。分子量位于7.7 ku(SolyHSF01)~16.62 ku(SolyHSF14)之間。通過亞細胞定位軟件分析HSF基因分布在細胞外、細胞周質、細胞膜外及細胞質(表1)。
2.3番茄HSF基因的多重序列比對分析
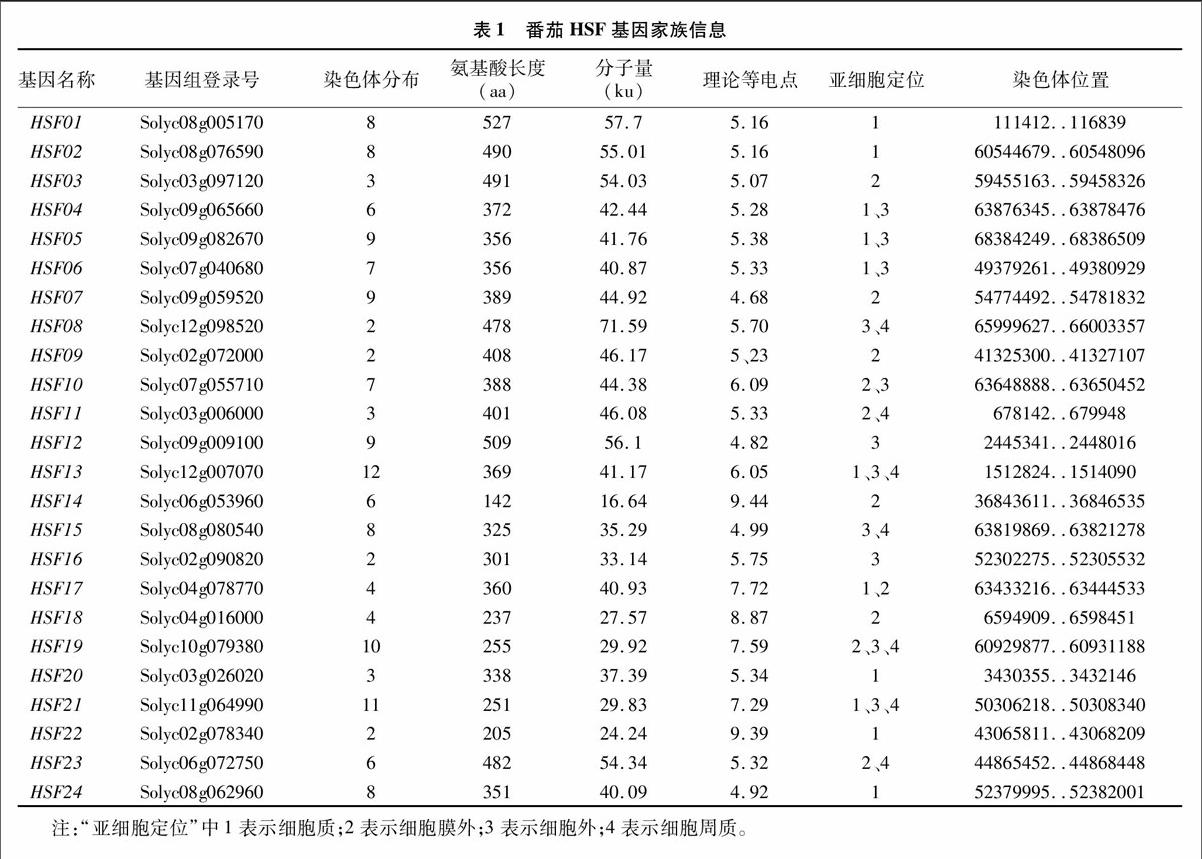
為了揭示番茄HSF基因家族的成員之間序列保守性特征,本研究將獲得的24條HSF基因的氨基酸序列進行多序列比對,結果發現在所有參加比對的氨基酸序列中包含高度保守的DNA結構域(DNA bing-domain)。DBD結構域位于HSF基因的Ⅳ端,包含3個α螺旋(α1~α3,)和4個β折疊(β1~β4)具有特異性識別和精確定位熱應激元件的功能,在其他植物中均發現類似現象。采用ClustalW在線比對對番茄DBD結構域進行多重序列比對,結果(圖1)表明,24個番茄HSF蛋白都具有高度保守的DBD結構域。氨基酸殘基在75~81之間,最多的是Soly02g078340。但是也有少數基因發生了堿基的缺失,其中除了Solyc02g078340、Solyc09g009100外其他基因在β1和β3均有缺失,這種缺失可能是番茄HSF基因的遺傳多樣化的原因。
2.4番茄HSF基因系統發育樹的構建及分析
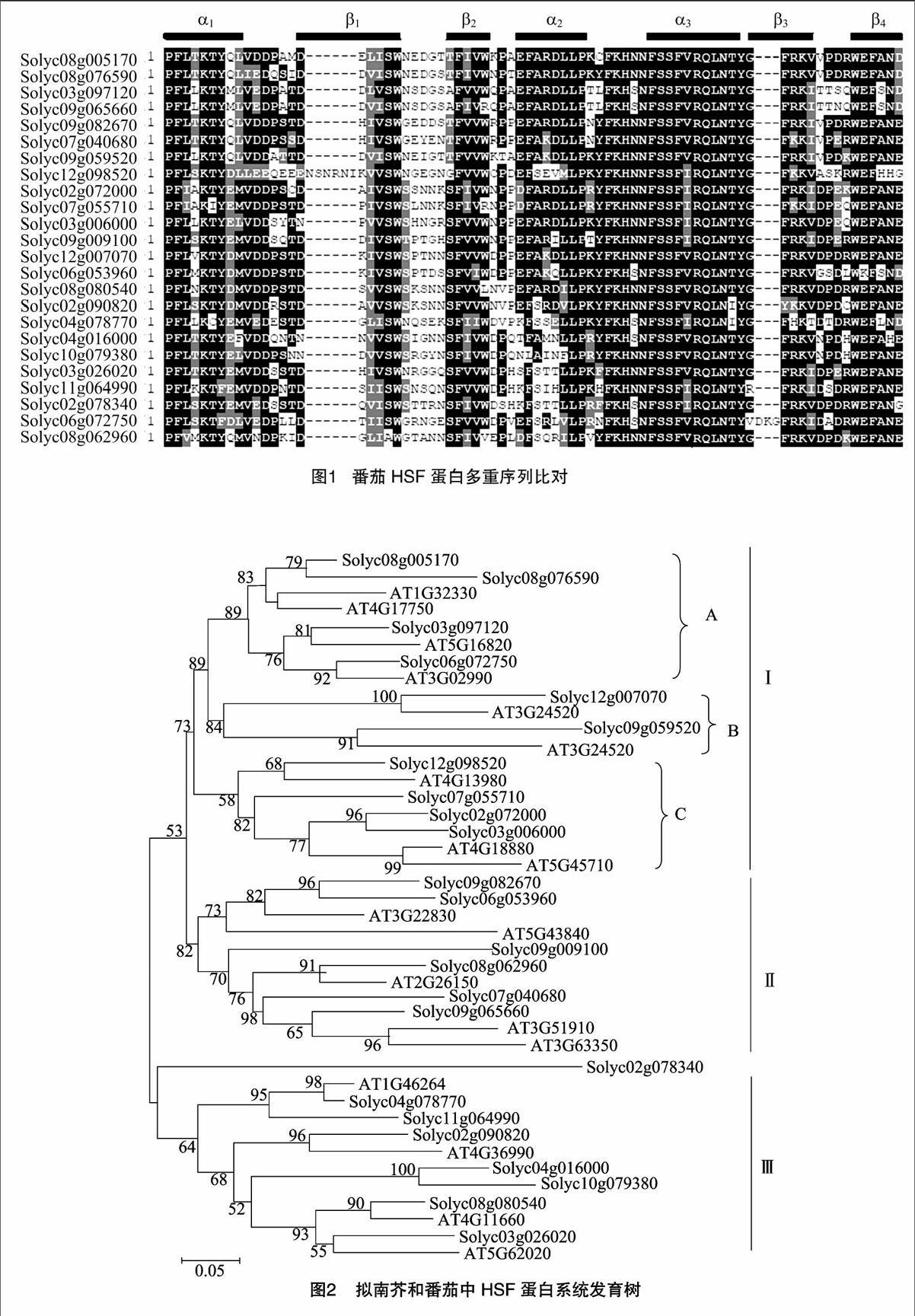
為了揭示番茄HSF基因在進化過程中的同源關系,將番茄的24條HSF氨基酸序列與模式植物擬南芥的條氨基酸序列進行聚類分析。結果(圖2)表明,所有的HSF基因可以分為3大類(Ⅰ、Ⅱ、Ⅲ),其中1分支可分為3個亞類(A、B、C)。在A類中番茄的Solye08g005170、Solye08g076590與擬南芥中的AT1G32330、AT4G17750具有較高的同源性而聚集在一起,其中Solyc08g005170、Solye08g076590為旁系同源蛋白,而Solyc03g097120、Solye06g072750分別與擬南芥AT5G16820、AT3G02990為直系同源蛋白。在B類和C類中Solyc12g007070、Solyc09g059520、Solyc12g098520分別與擬南芥AT3G24520、AT3G24520、AT4G13980為直系同源蛋白,Solyc02g072000與Solyc03g006000為旁系同源蛋白,番茄Solyc07g055710與Solye03g006000、Solyc02g072000,擬南芥AT4G18880、AT5G45710有很高的同源性聚集在一起。在Ⅱ分支中,Solye09g082670、AT3G51910分別與Solyc06g053960、AT3G63350為旁系同源蛋白,AT2G26150與Solyc08g062960為直系同源蛋白。在Ⅲ分支中,Solyc04g078770、Solyc02g090820、Solyc08g080540、Solyc03g026020分別與AT1G46264、AT4G36990、AT4G11660、AT5G62020為直系同源蛋白。番茄Solyclog079380與Solyc04g016000為旁系同源蛋白。在番茄的24個HSF基因有10對直系同源蛋白,5對旁系同源蛋白。通過進化關系可以說明HSF基因在2個物種中經歷了不同的進化過程。
2.5番茄HSF基因的保守基序分析
對番茄24個HSF保守基序的分析結果(圖3)表明,24個HSF基因具有15個保守基序,長度為6~50個氨基酸(表2);進一步分析了15個保守基序在番茄HSF蛋白中的分布情況,結果無一包含所有15個保守基序,所有基因中均不同程度存在保守基序的缺失,每個基因保守基序為4~13個。其中,基因Solyc06g053960.2.1保守基序缺失最多,只包含4個保守基序,Solyc08g005170.2.1、Solyc08g076590.2.1、Solyc03g097120.2.1含有保守基序最多,包含13個保守基序。發現4個基序(Motif1、Motif2、Motif3、Motif4)在所有HSF基因中是完全保守的,這些HSF基因家族之間具有較高保守型。
2.6番茄HSF基因染色體定位
根據番茄基因組測序的數據信息,分析24個HSF基因在染色體上的分布情況。結果(圖4)表明,24個HSF基因可以定位在10條染色體上,24條HSF基因在染色體上的分布并不均勻,其中8、9號染色體分布最多均為4個,10、11號染色體分布最少均為1個,2、3號染色體上有3個HSF基因,4、6、7、12號染色體均有2個HSF基因。
3討論與結論
隨著基因組的深入研究,轉錄因子調控植物基因表達的研究成為現今植物基因功能研究的熱點,隨著全球變暖氣溫上升,高溫成為影響植物生長及產量的重要因素之一。高溫脅迫也為番茄的周年生產帶來了困難,選育耐熱品種是解決這一難題的主要途徑,深入了解和挖掘植物耐熱基因是途徑之一。近年來,番茄基因組的測序完成為我們在基因組水平上分析熱激轉錄因子奠定了基礎。
通過對番茄基因組進行Blast搜索共鑒定出24個具有典型HSF結構域的熱激轉錄因子,分為3個分支,分別包含10個、6個、8個HSF基因,每個組中的基因數目都是有差異的,表明番茄HSF基因家族成員分布不均勻,他們之間存在著廣泛的多樣性。這些基因的差異性與多樣性可能造成HSF基因功能的多樣性,從而為番茄HSF基因研究提供了資源。
HSF轉錄因子N端的DBD結構域主要負責HSF識別熱激元件并與其正確結合,番茄HSF基因的DBD結構域具有高度的保守性,二級結構具有3個α結構和4個B折疊組成。從擬南芥與番茄HSF基因的系統發育關系發現存在10對直系同源蛋白和5對旁系同源蛋白,表明這些基因家族大部分成員在番茄和擬南芥基因組中按照它們的物種特異性的方式進行了擴展,這種現象在植物其他基因家族中也普遍存在。研究發現番茄HSF基因以分散的形式存在于10條染色體上,表明它們在進化過程中存在片段重復。
近年來,相關研究表明,HSF不僅參與熱脅迫,還參與其他逆境脅迫,本研究通過對番茄HSF蛋白基因家族24個成員進行生物信息學分析,及與擬南芥HSF基因家族聚類分析,為進一步揭示其功能奠定了基礎。

