高分辨率熔解曲線法在GLP1R基因多態性位點rs3765467檢測中的應用Δ
張 遠,何 霞,鐘 磊,串俊蘭,龍恩武(四川省醫學科學院/四川省人民醫院藥學部,成都 610072)
·精準醫療·
高分辨率熔解曲線法在GLP1R基因多態性位點rs3765467檢測中的應用Δ
張 遠*,何 霞,鐘 磊,串俊蘭,龍恩武#(四川省醫學科學院/四川省人民醫院藥學部,成都 610072)
目的:建立檢測腸促胰島素樣肽-1受體(GLP1R)基因已知突變位點rs3765467(NT_007592.16的第39 065 819位)的方法,并評價其準確性與實用性。方法:收集我院體檢中心2015年10月-2016年2月72例健康體檢者的外周靜脈血樣本,采用柱提法提取其全血DNA,經降落聚合酶鏈反應擴增后,采用高分辨率熔解曲線(HRM)法對產物進行分析;同時選取其中38例受試樣本進行雙脫氧鏈終止法(Sanger測序法)測序驗證,比較2種方法的結果。結果:突變掃描結果顯示,擴增片段中存在39 065 817和39 065 819兩個多態性位點。HRM法只檢測出了4種基因型[GCG/GCG、GCA/GCG或ACG/GCG、GCA/GCA或ACG/ACG、A(G)CA(G)];而Sanger測序法共檢測出6種基因型[GCG/GCG、ACG/GCG、ACG/ACG、A(G)CA(G)、GCA/GCG、GCA/GCA]。結論:HRM法可區分GCG/GCG和A(G)CA(G)基因型,但無法區分GCA/GCG與ACG/GCG雜合突變、GCA/GCA與ACG/ACG純合突變。該方法并不適用于多個鄰近位點的單核苷酸多態性檢測。在進行單核苷酸突變檢測時,應對序列進行綜合分析后再選取經濟、簡便的方法。
GLP1R基因;高分辨率熔解曲線;單核苷酸多態性;基因檢測
腸促胰島素效應是指口服葡萄糖對胰島素分泌的促進作用明顯強于靜脈注射的現象。該效應所產生的胰島素占進食后胰島素總量的60%左右,且有研究發現,這種腸促胰島素效應在2型糖尿患者中有所減退,因此,基于腸促胰島素樣肽1(Glucagon-like peptide 1,GLP-1)作用機制的藥物應運而生,為2型糖尿病的治療開辟了新的途徑[1]。GLP-1由腸道L細胞分泌,與胰島B細胞表面GLP-1受體(Glucagon-like peptide 1 receptor,GLP1R)結合后,通過G蛋白偶聯途徑調控胰島素的分泌[2]。近幾年有研究發現,2型糖尿病患者雖然餐后GLP-1水平未見改變,但胰島素分泌功能卻明顯受損;此外,一些2型糖尿病患者對外源性GLP-1刺激不敏感,提示可能與GLP1R功能異常有關[3-4]。Sathananthan A等[4]發現,GLP1R基因多態性位點rs3765467(NT_ 007592.16的第39 065 819位)可顯著影響健康人輸注GLP-1后胰島B細胞的應答,故在2型糖尿病患者中檢測該多態性位點可能有助于闡明個體差異的原因和指導疾病治療。
高分辨率熔解曲線(High resolution melting,HRM)是近年來國內外興起的一種靈敏度高、特異性好、通量高、檢測成本低、快速、方便的單核苷酸多態性(Single nucleotide polymorphism,SNP)檢測方法。該方法通過在DNA雙鏈中嵌入飽和染料、監控DNA熔解曲線的變化來進行分析,不僅可檢測已知突變位點,還可以同時掃描目標片段中的未知突變位點[5]。本研究通過建立GLP1R基因多態性位點rs3765467(G>A)的HRM分析方法,對72例健康受試者進行該突變位點的檢測及其前后共68 bp片段的突變掃描,并采用雙脫氧鏈終止法(Sanger測序法)對其中38例樣本進行驗證,評價HRM法用于SNP檢測的準確性與實用性。
1 資料與方法
1.1 研究對象
收集我院體檢中心2015年10月-2016年2月72例健康體檢者的外周靜脈血樣本。其中,男性48例,平均年齡(52.00±5.27)歲;女性24例,平均年齡(50.79± 6.01)歲。所有標本的采集均獲得醫院醫學倫理委員會的批準,所有受試者均知情同意。
1.2 DNA的提取
采用柱提法提取受試者的全血DNA。所有受試者均采集靜脈血1~2 mL,經乙二胺四乙酸(EDTA)抗凝后,采用柱式全血基因組提取試劑盒(上海百傲科技股份有限公司)提取全血DNA。采用NanoDrop 2000型超微量分光光度計(美國Thermo公司)測得其DNA質量濃度為40~120 ng/μL;以不同波長下光密度(OD)比值計算其純度分別為1.6~2.0(OD260nm/OD280nm)、1.7~2.2(OD260nm/OD230nm)。
1.3 引物合成
根據美國國立生物技術信息中心(National Center for Biotechnology Information,NCBI)人類GLP1R的參考序列NT_007592.16,設計針對rs3765467(G>A)突變位點的上、下游引物,詳見表1。引物由生工生物工程(上海)股份有限公司合成,并采用高效液相色譜(HPLC)法純化。
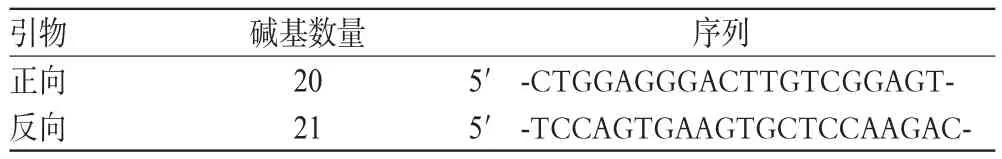
表1 GLP1R基因多態性位點rs3765467的引物序列Tab 1 Primer sequences for GLP1R gene polymorphism site rs3765467
1.4 擴增及HRM分析
擴增及HRM分析均在Light Cycler 480型實時熒光定量聚合酶鏈反應(PCR)儀(羅氏診斷)中完成,試劑盒采用預混有Taq酶、dNTP、高分辨率熔解染料的Light Cycler 480 HRM試劑盒(羅氏診斷)。
采用降落PCR法對受試者全血DNA進行擴增。擴增體系共20 μL,包括2 ng DNA模版、150 nmol/L引物、1.5 μmol/L Mg2+。PCR反應條件:95℃預變性10 min,95℃擴增10 s;隨后,退火溫度由66℃降至58℃,保持10 s,每1℃為1個循環,延遲2個循環開始,共擴增50個循環;72℃再延伸10 s。擴增產物長度為109 bp。
HRM反應條件:95℃熔解1 min,降至40℃保持90 s,以0.04℃/s的速率從75℃上升到95℃,完成熔解曲線分析,探測次數為15次/℃。采用Light Cycler 480基因掃描軟件(R 1.5.1.62)(羅氏診斷),設置“溫度偏移(Temperature shift)”一欄下的“溫度變化水平(Threshold)”為0,其他參數為默認,每個待測樣本均設置復孔,進行掃描與分析。
1.5 Sanger測序
為進一步驗證HRM法檢測結果,從各基因型樣本中隨機抽取7~13例樣本,共38例,采用Sanger測序法進行驗證。該試驗交由生工生物工程(上海)股份有限公司完成。
2 結果
2.1 HRM法的檢測結果
突變掃描結果顯示,擴增片段中存在39 065 817和39 065 819兩個多態性位點,共檢出基因型4種。其中,野生型(GCG/GCG)23例,雜合型(GCA/GCG或ACG/ GCG)28例,突變型(GCA/GCA或ACG/ACG)11例,多位點突變型[A(G)CA(G)]10例,詳見圖1、圖2(因涉及兩個位點的突變,故為明確表達,結果均采用NT_ 007592.16第39 065 817~39 065 819位的3個堿基表示)。序列第39 065 817位也存在G>A的突變,而HRM法無法區分這兩個位點間的雜合突變(GCA/GCG和ACG/GCG)和純合突變(GCA/GCA和ACG/ACG)。
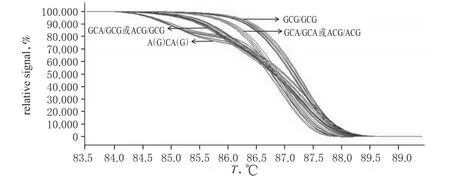
圖1 樣本標化熔解曲線Fig 1 The normalized melting curves of samples
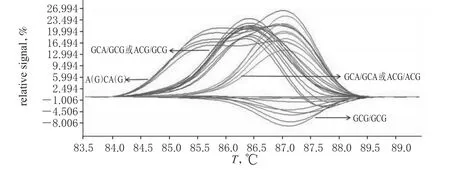
圖2 樣本差異作圖Fig 2 The difference plot for samples
2.2 Sanger測序法的檢測結果
共檢出基因型6種。其中,GCG/GCG型14例,ACG/GCG型2例,ACG/ACG型1例,A(G)CA(G)型9例,GCA/GCG型6例,GCA/GCA型6例,詳見圖3。由于HRM法無法區分5′-ACG-3′與5′-GCA-3′的純合和雜合型,故無法判斷HRM法的檢測結果是否與Sanger測序一致,但HRM法能夠區分GCG/GCG與A(G)CA(G)這兩種基因型,其檢測結果與Sanger測序是一致的。
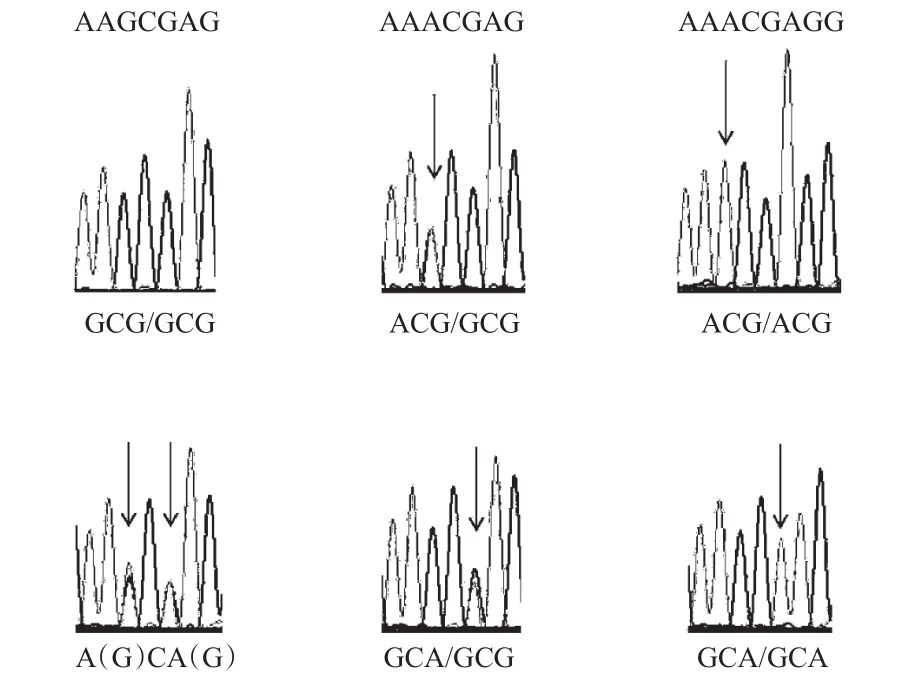
圖3 GLP1R rs3765467位點的Sanger測序圖Fig 3 Sanger sequencing for GLP1R rs3765467
3 討論
在倡導精準醫療的大背景下,基于遺傳多態性的個體化用藥被越來越多的醫務工作者所重視。現有多種SNP篩查技術如測序、DNA芯片、Tagman探針、突變擴增阻滯系統(Amplification refractory mutation system,ARMS)等已逐漸應用于臨床檢測,并已覆蓋到心腦血管疾病、精神類疾病及腫瘤等方面[6-7]。本研究通過檢測GLP1R基因中的已知SNP位點,探討HRM法用于突變檢測的優缺點,為使用該技術的同行提供參考。
PCR-HRM的分析原理是在普通PCR的基礎上加入可與雙鏈DNA結合的飽和熒光染料,擴增結束后采取逐步升溫的過程將產物解鏈,熒光染料因此脫落,熒光信號逐漸減弱,形成一個隨溫度升高而熒光信號減弱的曲線圖(如圖1)。當50%的雙鏈解鏈成單鏈時所對應的溫度稱為熔解溫度(Tm),由于各片段的堿基(GC)含量不同,所對應的Tm也有所不同。HRM法至少可分辨0.5℃Tm的差異,其精密的溫控設計使單個堿基的突變(一般是C/T或G/A突變分辨效果較好)分型成為可能[5]。
由于HRM法不需要設計探針,且PCR擴增過程完成后只需運行一個升溫程序即可完成檢測,因此具有成本低、簡便、閉管操作等優勢。但在有些情況下(比如C>G、A>T突變,突變基因豐度很低,或待測位點鄰近區域有其他突變堿基),則需進行條件優化,優化方法包括:摻雜一定比例的野生型樣本、使用低變性溫度下共擴增PCR或設計多重PCR及各式探針等[8-10]。雖然這些方法可以擴大HRM法的檢測范圍,但探針引物的設計及PCR反應體系的探索更耗時、耗力,甚至有時候需要在擴增后進行開管操作,難以體現HRM法成本低、簡便等特點。
本研究共檢測了72例樣本,為避免人力、物力不必要的浪費,根據統計學理論和Janavicius R等[11]的對比研究,抽取了超過16例樣本(本研究抽取的38例樣本)進行驗證[12],采用的方法為基因檢測的“金標準”——Sanger測序法(檢測成本高)。驗證結果顯示,與HRM法只檢出了4種基因型比較,Sanger測序法共檢出了6種基因型,表明HRM法的分辨率不如Sanger測序法高。其原因可能為:本研究的待測位點鄰近區域恰巧也存在1個G>A的突變,使得包括待測位點在內的3個堿基呈對稱排列[5′-GCG-3′(野生型)或5′-ACA-3′(突變型)],這種排列造成5′-ACG-3′與5′-GCA-3′的純合型(ACG/ACG和GCA/GCA)和雜合型(ACG/GCG和GCA/GCG)無法被區分。因此對于此類位點的檢測,HRM法也許不是一個適當的選擇。Tindall EA等[13]和Chen C等[14]在各自的研究中也得出與本研究一致的結論,即當待測片段中含有多個SNP位點時會影響HRM法分析結果的準確性。
綜上所述,當檢測范圍有2個以上鄰近突變位點時不宜采用HRM法;若是對突變片段進行掃描,必須結合測序結果才能作出準確判定,否則會影響結果的準確性。如此一來,HRM法成本低、簡便等優勢也許就不那么明顯了。本研究為HRM法直接檢測SNP位點提供了一定的方法學參考,但如何全面地評價HRM法在SNP檢測中的應用地位還需要參考更多的方法,如加入探針或改變試驗條件,或與Taqman探針、DNA芯片等檢測方法進行比對,以及進行經濟學方面的比較等。
[1] Nauck MA,Holst JJ,Willms B,et al.Glucagon-like peptide 1(GLP-1)as a new therapeutic approach for type 2-diabetes[J].Exp Clin Endocrinol Diabetes,1997,105(4):187-195.
[2] Salehi M,Aulinger B,Prigeon RL,et al.Effect of endogenous GLP-1 on insulin secretion in type 2 diabetes[J].Diabetes,2010,59(6):1330-1337.
[3] Shu L,Matveyenko AV,Kerr-Conte J,et al.Decreased TCF7L2 protein levels in type 2 diabetes mellitus correlate with downregulation of GIP-and GLP-1 receptors and impaired beta-cell function[J].Hum Mol Genet,2015,24(10):3004.
[4] Sathananthan A,Man CD,Micheletto F,et al.Common genetic variation in GLP1R and insulin secretion in response to exogenous GLP-1 in nondiabetic subjects:a pilot study[J].Diabetes Care,2010,33(9):2074-2076.
[5] Chambliss AB,Resnick M,Petrides AK,et al.Rapid screening for targeted genetic variants via high-resolution melting curve analysis[J].Clini Chem Lab Med,2017,55(4):507-516.
[6] Howe R,Miron-Shatz T,Hanoch Y,et al.Personalized medicine through SNP testing for breast cancer risk:clinical implementation[J].J Genet Couns,2015,24(5):744-751.
[7] Rai AJ,Yee J,Fleisher M.Biomarkers in the era of personalized medicine:a multiplexed SNP assay using capillary electrophoresis for assessing drug metabolism capacity[J]. Scand J Clin Lab Invest Suppt,2010,doi:10.3109/ 00365513.2010.493355.
[8]Reed GH,Kent JO,Wittwer CT.High-resolution DNA melting analysis for simple and efficient molecular diagnostics[J].Pharmacogenomics,2007,8(6):597-608.
[9] Poon KS,Liu TC,Tan KM,et al.One-step molecular detection of the MYD88 L265P mutation by unlabeled probe genotyping analysis[J].Mol Cell Probes,2015,29(1):74-77.
[10] Wu J,Zhou Y,Zhang CY,et al.Co-amplification at lower denaturation-temperature PCR combined with unlabledprobe high-resolution melting to detect KRAS codon 12 and 13 mutations in plasma-circulating DNA of pancreatic adenocarcinoma cases[J].Asian Pac J Cancer Prev,2014,15(24):10647-10652.
[11] Janavicius R,Matiukaite D,Jakubauskas A,et al.Microsatellite instability detection by high-resolution melting analysis[J].Clini Chem,2010,56(11):1750-1757.
[12] 羅森,李立,安偉.多種檢測目標下樣本容量設計的比較[J].統計與決策,2013,1(1):35-37.
[13] Tindall EA,Petersen DC,Woodbridge P,et al.Assessing high-resolution melt curve analysis for accurate detection of gene variants in complex DNA fragments[J].Hum Mutat,2009,30(6):876-883.
[14] Chen C,Li S,Lu X,et al.High resolution melting method to detect single nucleotide polymorphism of VKORC1 and CYP2C9[J].Int J Clin Exp Pathol,2014,7(5):2558-2564.
Application of High Resolution Melting Method in the Detection of GLP1R Gene Polymorphism Site rs3765467
OBJECTIVE:To establish the method for the detection of the known glucagon-like peptide 1 receptor(GLP1R)gene mutation site rs3765467(NT_007592.16,position:39 065 819),and to evaluate its accuracy and practicability.METHODS:Peripheral venous blood samples of 72 healthy subjects were collected in medical examination center of our hospital during Oct. 2015-Feb.2016.The whole blood DNA was extracted by column extraction method.After amplified by touch down PCR,high resolution melting(HRM)method was adopted to analyze amplified product.Sequencing verification by double stranded chain termination method(Sanger sequencing method)was performed for 38 test samples.The results of 2 methods were compared.RESULTS:The results of mutation scanning showed that there were 39 065 817 and 39 065 819 polymorphism sites in amplified segments. Four types of mutations were detected by HRM method[GCG/GCG,GCA/GCG or ACG/GCG,GCA/GCA or ACG/ACG,A(G)CA(G)],but 6 types of mutations was detected by Sanger sequencing method[GCG/GCG,ACG/GCG,ACG/ACG,A(G)CA(G),GCA/GCG,GCA/GCA].CONCLUSIONS:HRM method can identify GCG/GCG and A(G)CA(G)genotype,but can not identify GCA/GCG and ACG/GCG heterozygous mutation,GCA/GCA and ACG/ACG homozygous mutation.The method is not suitable for the detection of single nucleotide polymorphism for multiple neighboring sites.In the detection of single nucleotide mutation,economical and simple method should be selected after comprehensive analysis of sequence.
GLP1R gene;High resolution melting;Single nucleotide polymorphism;Gene detection
R394
A
1001-0408(2017)17-2305-04
2016-08-08
2017-02-07)
(編輯:張元媛)
四川省科技支撐計劃項目(No.2015SZ0182)
*主管藥師,碩士。研究方向:藥物基因組學。電話:028-87393405。E-mail:447415054@qq.com
#通信作者:副主任藥師。研究方向:內分泌臨床藥學、藥事管理學、藥物經濟學評價。電話:028-87393316。E-mail:dragon984169@ 126.com
DOI10.6039/j.issn.1001-0408.2017.17.01
ZHANG Yuan,HE Xia,ZHONG Lei,CHUAN Junlan,LONG Enwu(Dept.of Pharmacy,Sichuan Academy of Medical Sciences/Sichuan Provincial People’s Hospital,Chengdu 610072,China)

