藏靈菇發酵乳中醋酸菌的多樣性分析
張靖晞,趙欣,索化夷
(1.西南大學食品科學學院,重慶400715;2.西南大學重慶市特色食品工程技術研究中心,重慶 400715;3.重慶第二師范學院重慶市功能性食品協同創新中心,重慶 400067)
藏靈菇發酵乳中醋酸菌的多樣性分析
張靖晞1,2,趙欣3,索化夷1,2
(1.西南大學食品科學學院,重慶400715;2.西南大學重慶市特色食品工程技術研究中心,重慶 400715;3.重慶第二師范學院重慶市功能性食品協同創新中心,重慶 400067)
對國內不同地區采集的8份藏靈菇發酵乳樣品中醋酸菌進行分離鑒定,通過16S rDNA同源性分析和系統發育樹分析來探究藏靈菇中醋酸菌的組成和遺傳多樣性。結果表明,共分離得到44株醋桿菌,分別為Acetobacter fabarum(20株)、Acetobacter orientalis(5株)和Acetobacter syzygii(19株);Acetobacter fabarum和Acetobacter syzygii為藏靈菇發酵乳中的優勢醋桿菌,且二者親緣關系較近。序列分析顯示Acetobacter fabarum菌株表現出良好的穩定性;而在Acetobacter syzygii中,共發現4個變異位點,在系統發育樹中進化程度高;在Aceto?bacter orientalis中,共發現5個變異位點。
藏靈菇;醋桿菌;鑒定;多樣性
0 引 言
藏靈菇發酵乳風味獨特,帶有輕微的醇香和起泡性,具有增強免疫力等,該發酵乳中醋酸菌對其品質形成具有重要作用[1]。醋酸菌主要分布于藏靈菇的表層,能維持藏靈菇中微生物共生體系的穩定[2]。
Gao研究發現,藏靈菇中微生物分屬11個屬[3]。李劍從藏靈菇發酵乳中分離出巴氏醋桿菌(Acetobacter pasteurianus)、醋化醋桿菌(Acetobacter aceti)和過氧化醋桿菌(Acetobacterperoxydans)[5];曹宜從其中篩出 Aceto?bacter orientalis[6];董健從其中分離出Acetobacter orientalis和Acetobacter okinawensis[7];周龍霞從其中分離出Aceto?bacter peroxide和Acetobacter aceti[8]。以上研究表明,醋酸菌是藏靈菇發酵乳中微生物的重要組成之一。
本實驗以8份藏靈菇樣品為研究對象,對其分離、純化所得菌株進行16S rDNA序列分析和系統發育樹分析,以期為藏靈菇發酵乳中醋酸菌的組成及其遺傳多樣性的研究提供參考。
1 實驗
1.1 材料與儀器
1.1.1 材料與試劑
8份藏靈菇樣品采自西藏不同地區,保藏于西南大學食品科學學院,用ZLG01-ZLG08表示。實驗中分離菌株編號說明,如ZLG01-1表示第一份藏靈菇樣品發酵乳中分離出的第一株菌。
λDNA/HindⅢ,6× DNA Loading Buffer,100 bp DNA Ladder,溶菌酶,細菌基因組DNA提取試劑盒(DP302),2×Taq PCR MasterMix;上游引物 27F(5-AGAGTTTGATCCTGGCTCA-3),下 游 引 物1495R(5-CTACGGCTACCTTGTTACGA-3);MRS肉湯,瓊脂,瓊脂糖,安佳脫脂乳粉。
1.1.2 儀器設備
潔凈工作臺(SW-CJ-2F),水浴恒溫振蕩器(DSHZ-300A),隔水式恒溫培養箱(GHP-9160),生物顯微鏡(OLYMPUS-BX43),離心機(5810),梯度PCR儀(S1000 Thermal Cycler),小型水平電泳槽(Mini-Sub Cell GT),Gene Genius凝膠成像系統。
1.2 方法
1.2.1 藏靈菇發酵乳中醋酸菌的分離與純化
參照曹宜等[8]的方法并稍作修改,吸取相應稀釋液100 μL涂布于MRS平板,30℃培養48 h。選擇菌落數在30~300之間的平板,從中挑取菌落接種于MRS液體培養基,水浴30℃轉速為150 r/min培養24 h,通過2~5次劃線培養后獲得純菌落。
1.2.2 醋酸菌形態特征的觀察
觀察純化菌株在MRS平板上的菌落形態。參照BENSALAH F等[9]的方法進行革蘭氏染色、鏡檢。
1.2.3 16S rDNA序列擴增與系統發育樹構建
參照華鶴良[10]的方法并稍作修改,PCR反應采用25 μL體系:模板 1 μL,27F(10 μmol/L)1 μL,1495R(10 μmol/L)1 μL,2× Taq PCR MasterMix 12.5 μL,ddH2O 9.5 μL。PCR反應條件:94 ℃(5 min);94 ℃(40 s),55 ℃(40 s),72 ℃(1 min),共35個循環;72 ℃(10 min)。反應結束后,取5 μL PCR產物用1.5%瓊脂糖凝膠電泳進行檢測,同時PCR產物測序、比對分析。使用Clustalx 1.83軟件進行序列匹配比對,MEGA 5.0軟件構建分離菌株的系統發育樹,置信度檢測采用自舉法,自舉數據集為1 000[11-13]。
2 結果與分析
2.1 醋酸菌的純化與鏡檢
由圖1可知,菌落呈淡黃色,表面濕潤,不透明,菌落小而凸起,邊緣整齊。由圖2可知,菌株經革蘭氏染色后,在顯微鏡下為紅色,為革蘭氏陰性菌,符合醋桿菌特征。細胞形態結構均一,表明為純化菌株。

圖1 菌株菌落形態

圖2 分離菌株革蘭氏染色結果(×1 000)
2.2 醋酸菌16S rDNA PCR擴增結果
分離菌株16S rDNA按照PCR反應條件擴增和瓊脂糖凝膠電泳檢測后,在凝膠成像系統中觀察結果如圖3,分離菌株16S rDNA基因擴增產物在1500 bp附近出現一條清晰、明亮的條帶,無異常,陰性對照無條帶,與預期結果一致,PCR擴增成功。

圖3 部分分離菌株PCR產物瓊脂糖凝膠電泳結果
2.3 醋酸菌16S rDNA同源性比對與系統發育樹分析
將44株分離菌株測序結果與GenBank中已有序列進行比對分析,結果表明分離菌株同源性均在99%以上,大于分類研究閾值97%[14],其中Acetobacter fabarum 20株、Acetobacter orientalis 5株和Acetobacter syzy?gii 19株。圖4為8個樣品中分離菌株種類分布及數量情況,由圖可知ZLG08分出菌株最多,包括3個種的醋桿菌,分別為Acetobacter fabarum、Acetobacter orientalis和Acetobacter syzygii,是本實驗采集樣品中唯一分出這3種菌種的樣品。ZLG05和ZLG07樣品分出的醋桿菌種類單一,只含Acetobacter syzygii;其中,ZLG07樣品分出數量最少,只有2株。而Acetobacter syzygii在87.5%(7份)的樣品中被發現,僅ZLG02中未被發現。Aceto?bacter fabarum在62.5%(5份)的樣品中被發現,其中Ace?tobacter fabarum和Acetobacter syzygii同時存在于50%(4份)的樣品中,在所有樣品中該組合出現頻率較高。Acetobacter orientalis僅在37.5%(3份)的樣品中被發現,分別為ZLG02、ZLG03和ZLG08。
基于分離菌株16S rDNA序列構建系統發育樹結果如圖5所示,由圖可知,44株分離菌株與參考菌株以非常高的自舉支持率聚在同一分支上,共包括醋桿菌屬中的3個種,分別為Acetobacter fabarum(20株)、Ace?tobacter orientalis(5株)和 Acetobacter syzygii(19株),與16S rDNA序列比對分析結果一致。圖中各分支長度的大小代表物種進化程度的高低,3個種屬中Aceto?bacter orientalis的進化程度高于Acetobacter fabarum和Ace?tobacter syzygii,與 Acetobacter orientalis相比,Acetobacter fabarum和Acetobacter syzygii的進化程度接近,親緣關系較近。此外,進化樹分析表明Acetobacter fabarum與Ace?tobacter lovaniensis和Acetobacter ghanensis的親緣關系近;Acetobacter syzygii與Acetobacter okinawensis的親緣關系近;而Acetobacter orientalis與Acetobacter cibinongensis的親緣關系近。序列匹配分析結果顯示Acetobacter fabarum菌株表現出良好的穩定性,而Acetobacter syzygii中的ZLG03-4在第641位點上堿基由G突變為T;ZLG07-2在第226位點上堿基由A突變為C、第227位點上堿基由A突變為T和第465位點上堿基由G突變為T,在系統發育樹中二者的進化程度高。Aceto?bacter orientalis中的ZLG02-7在第158位點上堿基由T突變為C;ZLG03-1在第1350位點上堿基由C突變為G;ZLG08-5在第154位點上堿基由A突變為C、第158位點上堿基由T突變為C、第1352位點上堿基由A突變為G。
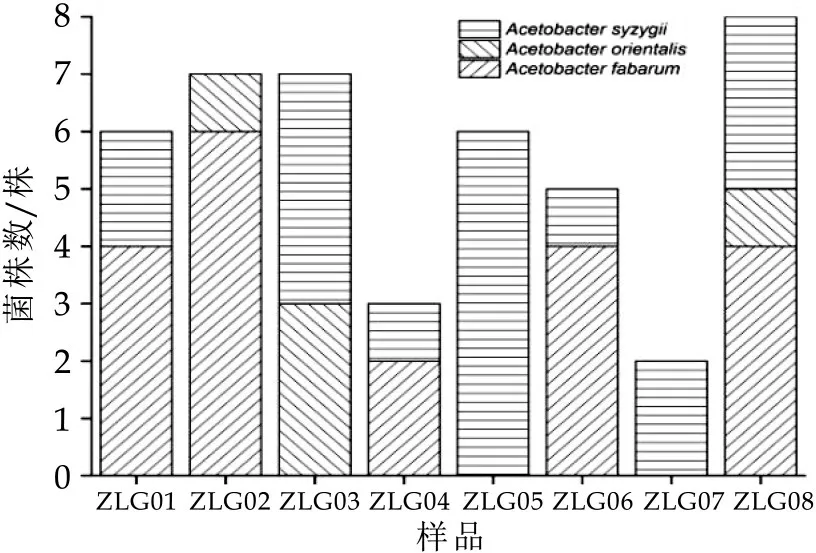
圖4 各樣品中分離菌株的情況
4 討 論
(1)本研究從8份西藏不同地區藏靈菇的發酵乳中均篩選出醋酸桿菌,共44株,分屬Acetobacter orienta?lis,Acetobacter fabarum 和Acetobacter syzygii,而醋酸菌在發酵過程中具有很強的活性和酸化作用,其能利用多種糖類產生有機酸,如乙酸、檸檬酸、乳酸、蘋果酸、丙酮酸和琥珀酸等,在發酵產品品質的形成中發揮重要作用。研究表明,Acetobacter orientalis是參與乳糖酸生產的主要微生物,在以牛乳為原料的發酵過程中,Ace?tobacter orientalis主要表現為氧化乳糖作用,在發酵乳表層10mm范圍內可積累大量乳糖酸[15-17]。乳糖酸不僅具有很強的香味,而且可以縮短成熟時間和促進風味產生;因其自身結構中帶有8組氫氧水基,因此可抓住大量的水,從而在發酵乳品質形成中發揮重要作用[18];此外,其能促進腸道對礦物質的吸收和抗氧化等功能[19]。因此Acetobacter orientalis可能對研究藏靈菇發酵乳的品質和功能的形成息息相關。
(2)16S rDNA在結構與功能上具有高度保守性,但在特殊條件的作用下,其基因序列也會發生一定程度的突變。本實驗中Acetobacter fabarum表現出良好的穩定性;在Acetobacter syzygii中,共發現4個變異位點;在Acetobacter orientalis中,共發現5個變異位點。這可能是由于青藏高原極端的氣候條件、地理位置和長期選擇進化而成。而堿基位點變異菌株的存在可能引起藏靈菇發酵乳品質與風味的變化;如發酵乳中有堿基變異位點的Acetobacter syzygii和Acetobacter orientalis組織形態良好,乳清不易析出,風味較佳或者產乳糖酸能力強等特點,則可考慮將這些變異位點作為特殊的遺傳標記。以上推測還需要進一步實驗用更多分離菌株加以驗證,但本實驗結果可為研究藏靈菇發酵乳中醋酸桿菌的遺傳多樣性提供一定參考。
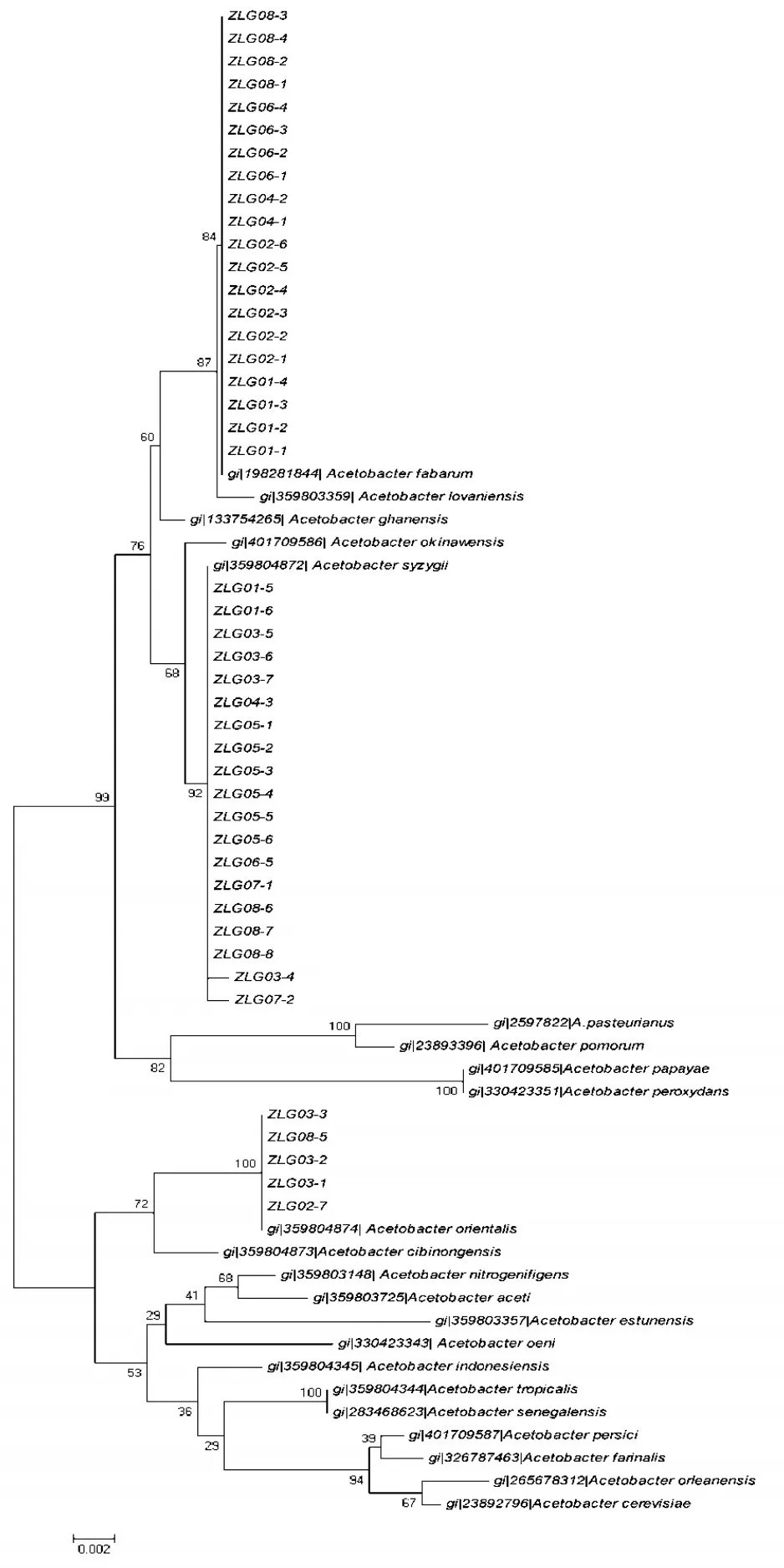
圖5 分離菌株16S rDNA的系統發育樹
(3)在開菲爾粒及其發酵乳研究中,Miguel[20]應用培養和非培養方法相結合分析不同地區開菲爾乳中細菌的組成,從中發現巴西(圣卡塔琳娜州)樣品中存在Acetobacter syzygii;Korsak[21]從開菲爾乳中還發現Ace?tobacter orientalis和Acetobacter lovaniensis的存在。此外,相關研究表明醋酸菌在發酵乳品質形成和功能特性中具有重要作用。在開菲爾粒中乳酸菌、酵母菌和醋酸菌為主要菌群,三者相互共生。具體為乳酸菌利用乳糖產生乳酸使奶酸化,同時促進酵母菌生長;酵母菌的生長為乳酸菌和醋酸菌提供發育促進物質;而醋酸菌合成的維生素對酵母菌和乳酸菌的生長也起促進作用;乳酸菌利用乳糖分解產生的葡萄糖形成莢膜多糖,促進開菲爾乳品質的形成。藏靈菇雖與國外開菲爾粒有所不同,但屬于開菲爾品系,其微生物組成存在一定相似性。因此,可借鑒國外對開菲乳粒的相關研究方法解析我國藏靈菇中微生物的多樣性,促進藏靈菇中微生物的開發和功能特性的研究。
5 結 論
通過8份西藏不同地區藏靈菇發酵乳中醋酸菌篩選結果可知,Acetobacter fabarum和Acetobacter syzygii為主要優勢菌種,在少數樣品中分離到Acetobacter orienta?lis,這可能與藏靈菇形成中的發酵條件和區域差異有關。序列分析結果表明,雖然受青藏高原極端的氣候條件和自然長期的選擇與馴化,Acetobacter fabarum菌株仍然表現出良好的穩定性。而Acetobacter syzygii中ZLG03-4在第641位點上堿基由G突變為T;ZLG07-2在第226位點上堿基由A突變為C、第227位點上堿基由A突變為T和第465位點上堿基由G突變為T,在系統發育樹中二者的進化程度較其他Aceto?bacter syzygii高;此外,Acetobacter orientalis中ZLG02-7在第158位點上堿基由T突變為C;ZLG03-1在第1350位點上堿基由C突變為G;ZLG08-5在第154位點上堿基由A突變為C、第158位點上堿基由T突變為C、第1352位點上堿基由A突變為G。這可為研究西藏不同地區藏靈菇發酵乳的風味和品質差異提供參考。
[1]李輝,卜祥龍,呂永通.藏靈菇酸奶發酵工藝優化及抑菌特性研究[J].中國釀造,2013,01:165-168.
[2]劉宇峰,王金英,曲曉軍,等.西藏靈菇菌的菌相菌學的研究[J].中國乳品工業,2005,33(9):35-39.
[3]GAO J,GU F Y,HE J,et al,Metagenome analysis of bacterial diversity in Tibetan kefir grains.European Food Research and Technology,2013.236(3):p.549-556.
[5]李劍,周劍忠,孫宇輝,等.藏靈菇中有益微生物的分離與鑒定[J].江蘇農業科學,2009(1):242-244.
[6]曹宜,劉蕓,劉波,朱育菁,陳倩倩.藏靈菇發酵乳中醋酸菌的分離純化與鑒定[J].福建農業學報,2012,12:1339-1342.
[7]董健,陳歷俊,姜鐵民,等.西藏靈菇顆粒中菌種的分離鑒定[J].食品科技,2015(1):6-9.
[8]周龍霞,鄭學云,薛鋒,等.天山雪蓮菌酸奶中菌株的分離及生理生化鑒定[J].安徽農業科學,2013,41(18):7963-7964.
[9]BENSALAH F,DELORME C,RENAULT P.Characterisation of ther?motolerant cocci from indigenous flora of‘leben’in algerianarid area and DNA identification of atypical Lactococcus lactis strains[J].Current Microbiology,2009,59(2):139-146.
[10]華鶴良.乳酸菌的分離鑒定及其抗菌肽與發酵性能研究[D].揚州大學,2014.
[11]ENNAHAR S,CAI Y,FUJITA Y.Phylogenetic diversity of lactic acid bacteria associated with paddy rice silage as determined by 16S ribo?somal DNA analysis[J].Applied and Environmental Microbiology,2003,69(1):444-451.
[12]EI-NAGGAR N E A,HAROUN S A,OWEIS E A,et al.Identifica?tion of newly isolated Talaromyces pinophilus and statistical optimiza?tion of β-glucosidase production under solid-state fermentation[J].Preparative Biochemistry&Biotechnology,2015,45(7):712-729
[13]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:Molec?ular evolutionary genetics analysis using maximum likelihood,evolu?tionary distance,and maximum parsimony methods[J].Molecular Biol?ogy and Evolution,2011,28(10):2 731-2 739.
[14]HAGHSHENAS B,NAMI Y,ABDULLAH N,et al.Potentially probi?otic acetic acid bacteria isolation and identification from traditional dairies microbiota[J].International Journal of Food Science&Tech?nology,2015,50(4):1056-1064.
[15]NAKANO H,KIRYU T,KISO T,et al.Lactobionic acid and its bio?catalytic production[J].Foods and Food Ingredients Journal of Japan,2006,211(10):874.
[16]KIRYU T,KISO T,NAKANOakano H,et al.Involvement of Aceto?bacter orientalis in the production of lactobionic acid in Caucasian yogurt(“Caspian Sea yogurt”)in Japan[J].Journal of dairy science,2009,92(1):25-34.
[17]KIRYU T,YAMAUCHI K,MASUYAMA A,et al.Optimization of lactobionic acid production by Acetobacter orientalis isolated from Caucasian fermented milk,“Caspian Sea yogurt”[J].Bioscience,Bio?technology,and Biochemistry,2012,76(2):361-363.
[18]HARLOUX C,PAUL M,LOISANCE D,et al.Inhibition of hydroxyl radical production by lactobionate,adenine,and tempol[J].Free Radi?cal Biology and Medicine,1995,19(5):699-704.
[19]白會釵,繆銘,江波,等.乳糖酸的研究進展[J].食品工業科技,2012(2):430-432.
[20]MIGUEL M G C P,CARDOSO P G,LAGO L A,et al.Diversity of bacteria present in milk kefir grains using culture-dependent and cul?ture-independent methods.Food Research International,2010.43(5):p.1523-1528.
[21]KORSAK N,TAMINIAU B,LECLERCQ M,et al.Short communi?cation:Evaluation of the microbiota of kefir samples using metagenet?ic analysis targeting the 16S and 26S ribosomal DNA fragments[J].Journal of Dairy Science,2015,98(6):3684-3689.
Diversity analysis of acetic acid bacteria isolated from Tibetan kefir dairy
ZHANG Jingxi1,2,ZHAO Xin3,SUO Huayi1,2
(1.Food Science Department,Southwest University,Chongqing 400715,China;2.Chongqing Engineering Research Center of Regional Food,Chongqing 400715,China;3.Chongqing Collaborative Innovation Center for Functional Food,Chongqing University of Education,Chongqing 400067,China)
Acetic acid bacteria from the eight samples of Tibetan kefir dairy,which collected from different regions in China were isolated and identified by 16S rDNA sequence analysis and phylogenetic tree analysis,aimed to explore the acetobacter composition and genetic diversity in Tibetan kefir.The results of the identification included Acetobacter fabarum(20 strains),Acetobacter orientalis(5 strains)and Acetobacter syzygii(19 strains).Acetobacter fabarum and Acetobacter syzygii,which hadclose relationship,were themain Acetobacter in Tibetan kefir dairy.Sequence analysis shown that Acetobacter fabarum has a good characteristic of stability.Four mutations were tested and the strains with mutations had highdegreeof evolution in Acetobacter syzygii.In addition,5 mutations were tested inAcetobacter orientalis.
Tibetan kefir;Acetobacter;Identification;Diversity
Q93-331
A
1001-2230(2017)08-0004-05
2017-01-13
國家公益性行業(農業)科研專項(201303085);重慶市社會民生科技創新專項(cstc2015shmszx80021);中央高校基本業務費項目(xdjk2016A018)。
張靖晞(1996-),女,本科,研究方向為食品科學。
索化夷

