鼻息肉的全基因表達譜
白偉良,宋巖,王梟維,李婷,項清華,周倩,李進興
(中國醫科大學附屬盛京醫院耳鼻咽喉科,沈陽 110004)
鼻息肉的全基因表達譜
白偉良,宋巖,王梟維,李婷,項清華,周倩,李進興
(中國醫科大學附屬盛京醫院耳鼻咽喉科,沈陽 110004)
目的研究鼻息肉的全基因表達譜情況,探尋其與正常鼻腔黏膜的基因表達差異。方法提取鼻息肉中的RNA,將RNA合成雙鏈的cDNA,將標記的cDNA在Roche NimbleGen基因芯片上進行雜交。應用Axon GenePix 4000B scanner進行掃描,應用NimbleScan軟件進行圖像分析,探針及基因水平標準化后進行計算。結果與正常鼻腔黏膜相比,鼻息肉中有1 000個基因表達上調,占2.22% (1 000/45 033),有1 123個基因表達下調,占2.49%(1 123/45 033)。經過信號通路分析,發現在核糖體、蛋白酶體、檸檬酸循環、膀胱癌、非小細胞肺癌、神經膠質瘤、子宮內膜癌、內質網蛋白合成、慢性髓樣白血病、谷胱甘肽代謝等信號通路中相關基因表達上調,在嗅覺傳導、自然殺傷細胞介導的細胞毒性、細胞色素P450的化合物代謝、細胞色素P450的藥物代謝、抗原加工和呈遞、瘧疾、移植物抗宿主疾病、視黃醇代謝、亞油酸代謝、戊糖和葡萄糖醛酸轉換等信號通路中相關基因表達下調。結論鼻息肉的發生、發展與多個基因及信號通路的表達升高或降低有關,鼻息肉全基因表達譜的研究為探求鼻息肉的發生機制
提供了有益的線索。
鼻息肉; 基因芯片; 基因表達; 信號通路
Abstract ObjectiveTo analyze the gene expression profiles of nasal polyp and gene expression differences between the nasal polyp and normal nasal mucosa.MethodsTotal RNA from nasal polyp tissues was purified and synthesized into double-stranded cDNA.The cDNA was labeled and hybridized in a NimbleGen hybridization chamber. The slides were scanned using the Axon GenePix 4000B microarray scanner. Scanned images were analyzed using NimbleScan software. The probes and gene levels were standardized and calculated.ResultsCompared with normal nasal mucosa tissues,expression of 2.22% (1 000/45 033) of genes was up-regulated in all cases of nasal polyp tissues,while 2.49% (1 123/45 033) of genes were down-regulated in all cases of nasal polyp tissues. We found genes related to the ribosome,proteasome,citrate cycle (TCA cycle),bladder cancer,non-small cell lung cancer,glioma,endometrial cancer,protein processing in the endoplasmic reticulum,chronic myeloid leukemia,and glutathione metabolism were up-regulated.Genes related to olfactory transduction,natural killer cell-mediated cytotoxicity,metabolism of xenobiotics by cytochrome P450,drug metabolism-cytochrome P450,antigen processing and presentation,malaria,graft-versus-host disease,retinol metabolism,linoleic acid metabolism,and pentose and glucuronate interconversions were down-regulated.ConclusionMultiple genes or pathways may be involved in the occurrence and development of nasal polyp. Gene expression profiling provides insight into the mechanism of nasal polyp development.
Keywordsnasal polyp; gene chip; gene expression; signaling pathway
鼻息肉本質上是一種炎癥疾病,是一個多步驟、多因素、序貫的復雜過程,目前已發現涉及到多種基因及產物的改變,多種信號通路相互調節和拮抗[1]。基因表達譜芯片是目前應用最為廣泛的基因芯片,具有快速、高效、大規模、高通量及平行性獲取相關生物信息的特點,可以同時分析多個基因的表達情況[2]。目前有關鼻息肉基因表達譜的研究較少,本研究應用基因芯片技術研究了鼻息肉全基因表達譜的情況。
1 材料與方法
1.1 臨床資料
取臨床經病理證實的鼻息肉3例,正常鼻腔下鼻甲黏膜組織2例,迅速放入液氮中凍存備用,所有患者術前均未接受激素等特殊治療。
1.2 總RNA提取
按TRIzol試劑說明書提取每例組織標本的總的RNA,紫外分光光度儀測量提取的RNA的純度,要求A260/A280為1.9~2.1。
1.3 基因表達譜芯片
應用Roche NimbleGen公司的12×135K基因芯片,可同時檢測人類45 033個基因的表達。
1.4 RNA標記和雜交
應用美國Invitrogen公司的SuperScript ds-cDNA試劑盒,將5 μg 的RNA合成雙鏈的cDNA。雙鏈cDNA應用美國NimbleGen公司的基因表達分析方法進行標記和清洗,雙鏈cDNA與4 μg RNase A在37 ℃孵育 10 min,用苯酚∶氯仿∶異戊醇(25∶24∶1)清洗,然后用低溫乙醇沉淀。NimbleGen單色DNA標記試劑盒標記cDNA。1 μg的雙鏈cDNA與1單位的Cy3-9引物在98℃孵育10 min,然后加入100 pmol的脫氧核苷三磷酸和100單位的克列諾片段(美國New England Biolabs),37 ℃孵育2 h。加入0.1 μL的0.5 mol/L EDTA停止反應,異丙醇/乙醇將標記的雙鏈cDNA提純,將4 μg Cy3標記的雙鏈cDNA在基因芯片上進行雜交,42 ℃孵育16~20 h。然后應用NimbleGen Wash緩沖液沖洗。
1.5 圖像與數據的分析
基因芯片應用Axon GenePix 4000B scanner(美國Molecular Devices公司)進行掃描,數據應用NimbleScan軟件進行分析,探針及基因水平標準化后進行計算,數據應用Agilent GeneSpring GX軟件進行分析,篩選出比值>2或<0.5的基因。
2 結果
研究鼻息肉的基因表達譜,發現與正常鼻腔黏膜相比,3例鼻息肉中同時表達上調的基因有1 000個,占2.22% (1 000/45 033),同時表達下調的基因有1 123個,占2.49% (1 123/45 033)。見圖1。
圖1 鼻息肉的基因芯片Fig.1 Gene chip of nasal polyp
經過信號通路分析,發現在核糖體、蛋白酶體、檸檬酸循環、膀胱癌、非小細胞肺癌、神經膠質瘤、子宮內膜癌、內質網蛋白合成、慢性髓樣白血病、谷胱甘肽代謝等信號通路中有基因表達上調(圖2),在嗅覺傳導、自然殺傷細胞介導的細胞毒性、細胞色素P450的化合物代謝、細胞色素P450的藥物代謝、抗原加工和呈遞、瘧疾、移植物抗宿主疾病、視黃醇代謝、亞油酸代謝、戊糖和葡萄糖醛酸轉換等信號通路中有基因表達下調(圖3)。

圖2 鼻息肉基因比正常鼻腔黏膜表達高的相關信號通路Fig.2 Higher gene expression in the pathway of nasal polyp compared to normal nasal mucosa
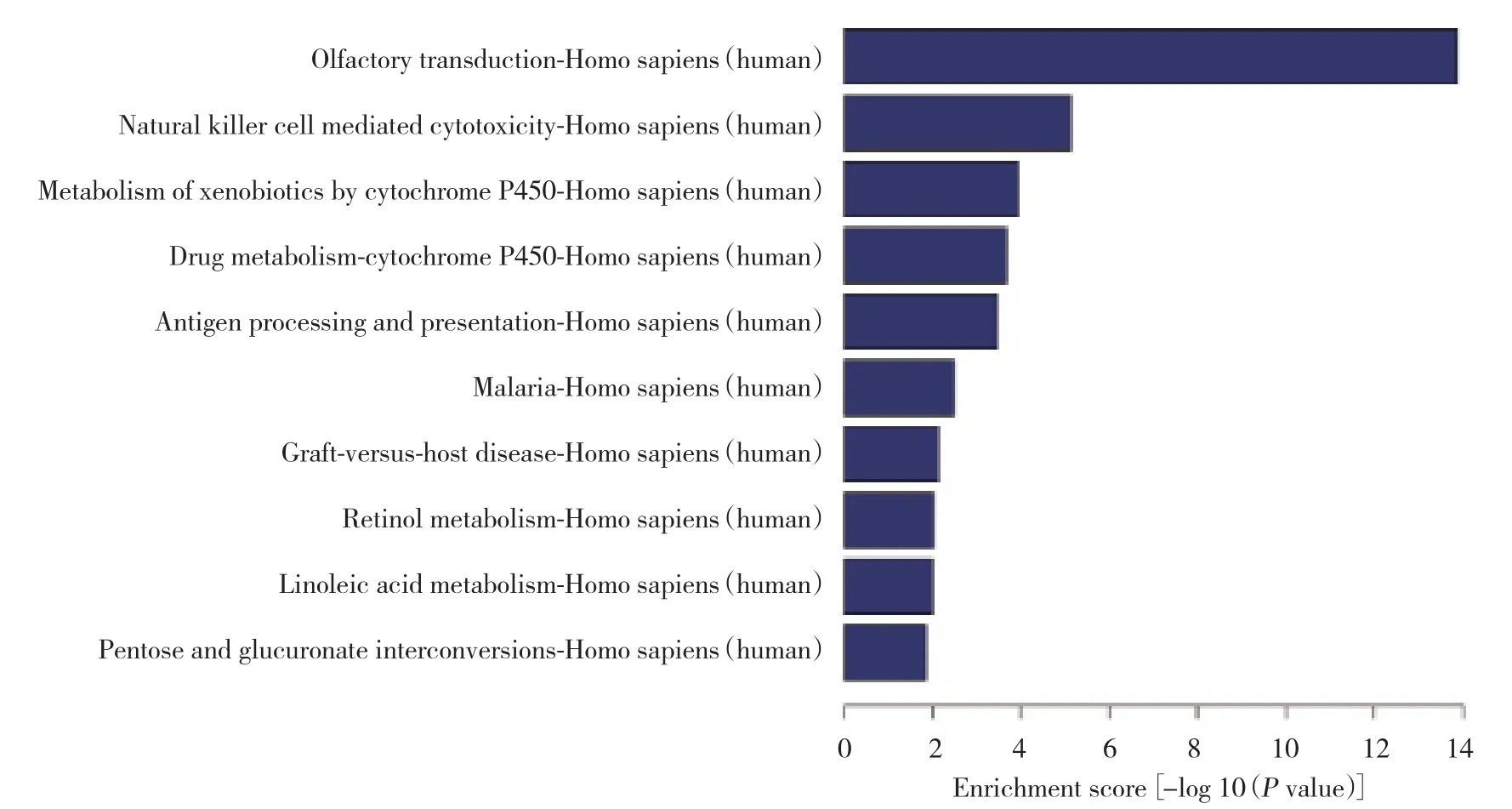
圖3 鼻息肉基因比正常鼻腔黏膜表達低的相關信號通路Fig.3 Lower gene expression in the pathway of nasal polyp compared to normal nasal mucosa
3 討論
鼻息肉是鼻腔黏膜的慢性炎癥疾病,組織病理學研究發現,鼻息肉為呼吸上皮覆蓋的高度水腫的疏松纖維組織,間質中有大量炎癥細胞浸潤,多以嗜酸性粒細胞浸潤為主。鼻息肉的發病機制和病因仍不十分清楚[3],已發現有多個基因、多個信號通路與鼻息肉發生有關,BENITO等[4]研究了LTC4S、CYSLTR1、PTGDR和NOS2A基因在鼻息肉中多形態分析,發現這些基因與鼻息肉臨床關系密切,在鼻息肉發展中起重要作用。FIGUEIREDO等[5]發現細胞因子、趨化因子、生長因子基因、金屬基質蛋白酶參與了鼻息肉的炎癥及局部組織的重塑。有研究[6]報道,鼻息肉發生與干擾素α、轉錄因子、核因子κB、AP-1、p38MAPK、ERK 1/2、JNK、AKT等多個信號通路有關。基因芯片技術使得同時檢測多個基因的表達成為可能,能夠篩選和發現疾病新的基因或信號通路,FR CZEK等[7]應用基因芯片技術發現NOS2A、SERPINA1、UCP2、OXTR和IL-8在鼻息肉中表達升高,而LTF、KRT6B、LYZ、SD11B2和MMP-3表達降低。基因芯片有多種,本研究采用的是Roche NimbleGen的人全基因組表達芯片,可同時檢測45 033個基因的表達。本研究發現,與正常鼻腔黏膜組織相比,鼻息肉組織中表達上調的基因有1 000個,占2.22%;表達下調的基因有1 123個,占2.49%,可見鼻息肉的發生、發展與多個基因的表達失調有關。
本研究進一步對表達失調的基因進行信號通路分析,發現鼻息肉組織中在核糖體、蛋白酶體、檸檬酸循環、膀胱癌、非小細胞肺癌、神經膠質瘤、子宮內膜癌、內質網蛋白合成、慢性髓樣白血病、谷胱甘肽代謝等信號通路中有基因表達上調。其中核糖體、蛋白酶體、檸檬酸循環等信號通路中的基因與鼻息肉的關系未見文獻報道,而在膀胱癌、非小細胞肺癌、神經膠質瘤、子宮內膜癌信號通路中有共同的CCND1和EGFR基因,在鼻息肉中表達均升高。LIN等[8]發現CCND1在鼻息肉組織中表達升高,DUAN等[9]認為EGFR等基因在鼻息肉中表達升高,在鼻息肉發生、發展、組織重塑過程中起重要作用。這些研究結果與本研究基因芯片結果相符合。而在內質網蛋白合成及谷胱甘肽代謝信號通路出現的基因,國內外未見與鼻息肉相關報道,但是其中GPX-1與上呼吸道感染及哮喘有一定關系,WON等[10]認為缺乏GPX-1表達的小鼠中卵清蛋白不能誘發哮喘,鼻息肉與哮喘關系密切,可見GPX-1有可能是研究鼻息肉新的候選基因。
本研究發現,在嗅覺傳導、自然殺傷細胞介導的細胞毒性、細胞色素P450的化合物代謝等多個信號通路中,有基因表達下調。其中尤其值得關注的是嗅覺傳導通路,嗅覺傳導通路中的嗅覺受體在嗅神經軸中起重要作用[11]。鼻息肉不僅阻塞溴素到達嗅區,影響嗅覺,本研究顯示鼻息肉組織嗅覺受體基因表達也下降。當鼻息肉出現在嗅區時,會對嗅覺黏膜有一定影響,這也可能是鼻息肉引起嗅覺障礙的另一個機制,目前這方面研究未見報道。本研究發現,自然殺傷細胞介導的細胞毒性信號通路中的基因在鼻息肉中表達下降,其中ITGB2基因突變會導致呼吸道反復感染以及傷口不愈合[12],鼻息肉中ITGB2基因表達下降無疑會促進鼻息肉的發生和發展,以往并無這方面的研究。其他細胞色素P450的化合物代謝、瘧疾、抗原加工和呈遞等信號通路中的基因與鼻息肉關系研究較少。通過基因芯片技術,可以對鼻息肉的基因表達譜有一個全新的認識,本研究篩選到了一些有價值的基因,通過信號通路分析,進一步明確了研究的方向,為鼻息肉基礎研究提供了思路。
[1] YAO Y,XIE S,YANG C,et al. Biomarkers in the evaluation and management of chronic rhinosinusitis with nasal polyposis[J]. Eur Arch Otorhinolaryngol,2017,274(10):3559-3566. DOI:10.1007/s00405-017-4547-2.
[2] JAKSIK R,IWANASZKO M,RZESZOWSKA-WOLNY J,et al. Microarray experiments and factors which affect their reliability [J].Biol Direct,2015,10:46. DOI:10.1186/s13062-015-0077-2.
[3] STEVENS WW,LEE RJ,SCHLEIMER RP,et al. Chronic rhinosinusitis pathogenesis[J]. J Allergy Clin Immunol,2015,136(6):1442-1453. DOI:10.1016/j.jaci.2015.10.009.
[4] BENITO PESCADOR D,ISIDORO-GARC A M,GARC A-SOLAESA V,et al. Genetic association study in nasal polyposis[J]. J Investig Allergol Clin Immunol,2012,22(5):331-340.
[5] FIGUEIREDO CR,SILVA ID,WECKX LL. Inflammatory genes in nasal polyposis [J]. Curr Opin Otolaryngol Head Neck Surg,2008,16(1):18-21. DOI:10.1097/MOO.0B013E3282F363F1.
[6] PLATT MP,SOLER Z,METSON R,et al. Pathways analysis of molecular markers in chronic sinusitis with polyps [J].Otolaryngol Head Neck Surg,2011,144(5):802-808. DOI:10.1177/0194599810395091.
[7] FR CZEK M,ROSTKOWSKA-NADOLSKA B,KAPRAL M,et al.Microarray analysis of NF-κ B-dependent genes in chronic rhinosinusitis with nasal polyps [J]. Adv Clin Exp Med,2013,22(2):209-217.
[8] LIN H,LIN D,XIONG XS,et al. Downregulation of caveolin-1 in chronic rhinosinusitis with and without nasal polyps[J]. Eur Arch Otorhinolaryngol,2015,272(3):613-618. DOI:10.1007/S00405-014- 3146-8.
[9] DUAN C,LI CW,ZHAO L,et al. Differential expression patterns of EGF,EGFR,and ERBB4 in nasal polyp epithelium [J]. PLoS One,2016,11(6):e0156949. DOI:10.1371/JOURNAL.PONE.0156949.
[10] WON HY,SOHN JH,MIN HJ,et al. Glutathione peroxidase 1 deficiency attenuates allergen-induced airway inflammation by suppressing Th2 and Th17 cell development [J]. Antioxid Redox Signal,2010,13(5):575-587. DOI:10.1089/ARS.2009.2989.
[11] HSIEH YW,ALQADAH A,CHUANG CF,et al. Mechanisms controlling diversification of olfactory sensory neuron classes[J].Cell Mol Life Sci,2017,74(18):3263-3274. DOI:10.1007/S00018-017-2512-2.
[12] TAGHIZADE MORTEZAEE F,ESMAELI B,BADALZADEH M,et al. Investigation of ITGB2 gene in 12 new cases of leukocyte adhesion deficiency-type Ⅰ revealed four novel mutations from Iran [J].Arch Iran Med,2015,18(11):760-764. DOI:0151811/AIM.006.
(編輯 陳 姜)
Gene Expression Profiling in Nasal Polyp
BAI Weiliang,SONG Yan,WANG Xiaowei,LI Ting,XIANG Qinghua,ZHOU Qian,LI Jinxing
(Department of Otorhinolaryngology,Shengjing Hospital,China Medical University,Shenyang 110004,China)
R765.2
A
0258-4646(2017)10-0895-04
http://kns.cnki.net/kcms/detail/21.1227.R.20170927.1452.016.html
10.12007/j.issn.0258‐4646.2017.10.008
遼寧省科學事業公益研究基金(2013001010);沈陽市科學技術計劃(F11-264-1-07,F13-220-9-21)
白偉良(1972 -),男,教授,博士.
白偉良,E-mail:bweiliangcmu@163.com
2017-04-21
網絡出版時間:2017-09-27 14:52

