利用改良SOE-PCR技術定點突變TuMV C4-VPg基因
李國亮 錢 偉 張淑江 李 菲 章時蕃 張 慧 謝露露 武 劍王曉武 孫日飛
利用改良SOE-PCR技術定點突變TuMV C4-VPg基因
李國亮 錢 偉 張淑江 李 菲 章時蕃 張 慧 謝露露 武 劍王曉武 孫日飛*
(中國農業科學院蔬菜花卉研究所,北京 100081)
蕪菁花葉病毒(Turnip mosaic virus,TuMV)是白菜類蔬菜生產上的重要病害,其中TuMV VPg基因在TuMV侵染白菜類蔬菜的過程中起著至關重要的作用,且不同TuMV株系的致病性不同。本試驗采用改良的重疊延伸PCR(SOE-PCR)技術,以TuMV C4-VPg基因序列為模板,定點突變TuMV C4-VPg與TuMV CDN1-VPg基因間5個差異核苷酸位點(決定5個氨基酸差異)。結果表明:通過SOE-PCR技術獲得了5個TuMV C4-VPg基因的單點突變體,即TuMV C4-VPg-Ⅰ、TuMV C4-VPg-Ⅱ、TuMV C4-VPg-Ⅲ、TuMV C4-VPg-Ⅳ和TuMV C4-VPg-Ⅴ。
白菜類蔬菜;蕪菁花葉病毒;VPg基因;重疊延伸PCR技術;單點突變
蕪菁花葉病毒(Turnip mosaic virus,TuMV)屬于馬鈴薯Y病毒科Y病毒屬,是白菜類蔬菜生產上的主要病害之一。TuMV VPg蛋白是一種多功能蛋白,共價連接在病毒RNA的5′末端,類似帽子結構,在病毒復制的過程中起重要作用(Miyoshi et al.,2008),可與寄主擬南芥的翻譯起始因子eIF(iso)4E互作,調控病毒RNA翻譯的起始(Duprat et al.,2002)。Gallois等(2010)通過酵母雙雜交試驗發現寄主翻譯起始因子eIF(iso)4E可以與VPg蛋白互作,通過調控病毒翻譯的起始,從而對病毒的侵染產生干擾(Wittmann et al.,1997),在擬南芥eIF(iso)4E基因的帽結合蛋白中,將第46位和第92位氨基酸Trp突變為Leu后,eIF(iso)4E就不能與病毒VPg互作(Miyoshi et al.,2006);Dinkova等(2016)發現辣椒eIF4E的第94位和第107位氨基酸突變后,將影響TuMV VPg與eIF4E的互作,但eIF4E其他位置的突變對二者的互作沒有影響。由此可知,關鍵氨基酸位點的變化對其功能的表達具有重要的影響,通過重疊延伸PCR技術(splicing by overlap extension PCR,SOE-PCR),可以快速、高效地獲得單點突變體,為驗證關鍵氨基酸位點的功能奠定基礎。
重疊延伸PCR技術利用具有互補末端的特定引物,將兩條PCR產物形成重疊鏈,在下一輪的擴增反應中,通過重疊鏈延伸來實現不同片段的拼接(熊愛生 等,2000;帥勇 等,2009)。該技術不需要限制性內切酶和連接酶的處理(Horton et al.,1989;Senanayake & Brian,1995),也不受突變位置及突變類型的限制,可以在DNA的任何位點插入突變(Urban et al.,1997;Gang et al.,2011)。重疊延伸PCR技術在生命科學領域已有廣泛應用。翟玲等(2012)利用重疊延伸PCR技術,對LacZ報告基因的一些位點隨機插入目標內含子,再對LacZ報告基因的活性進行比較分析,結果表明該方法與In-Fusion克隆相比有一定的優越性;李諾敏等(2013)利用重疊延伸PCR技術,對人tau基因進行定點突變,獲得tau基因的6種同工異構體的克隆,進而在蛋白水平上驗證了tau蛋白表達。該技術無需利用限制性內切酶,可以對基因的多個位置進行剪切和連接,具有很大的靈活性和簡便性。
重疊延伸PCR技術即在體外定點突變基因位點,是一種獲得單點突變基因的技術。蕪菁花葉病毒(TuMV)侵染白菜類蔬菜后,TuMV VPg蛋白通過與白菜翻譯起始因子eIF(iso)4E互作,促進病毒自身的增殖。為了進一步闡明不同TuMV株系間VPg基因的差異對TuMV致病性的影響,本試驗采用改良的重疊延伸PCR技術,以TuMV C4-VPg基因為模板,定點突變核苷酸,克隆了5個單點突變基因,以期為揭示特定氨基酸位點對病毒致病機制的影響奠定基礎。
1 材料與方法
1.1 試驗材料
試驗于2016年9月在中國農業科學院蔬菜花卉研究所分子遺傳實驗室進行。TuMV C4株系、大腸桿菌trans 5α菌株由中國農業科學院蔬菜花卉研究所白菜育種課題保存。質粒小提試劑盒、DNA Marker、DNA Polymerase、DNA 回收試劑盒購自天根生化科技(北京)有限公司;克隆載體pEASY-T1 Cloning Kit購自全式金(北京)有限公司;卡那霉素購自西格瑪奧德里奇(上海)貿易有限公司。
1.2 重疊延伸PCR引物設計及第1輪PCR反應體系
重疊延伸PCR引物設計遵循常規引物設計原則,突變堿基位于引物中央偏3′端,引物長度30 bp左右為佳,引物過短不利于2個長片段的融合。在每個差異核苷酸位點處分別設計正、反向引物。TuMV C4-VPg和TuMV CDN1-VPg氨基酸序列存在5個差異位點,分別標記為Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ,分別設計5對引物,即bioC4-Ⅰ-R/F、bioC4-Ⅱ-R/F、bioC4-Ⅲ-R/F、bioC4-Ⅳ-R/F和bioC4-Ⅴ-R/F(表1),TuMV C4-VPg基因的兩端引物分別為bio120213/bio120214。引物由生工生物工程(上海)有限公司合成。第1輪重疊延伸PCR反應體系為50 μL,其中包括10×PCR緩沖液5 μL,0.8 mmol·L-1dNTPs 4 μL,1.0 μmol·L-1上、下游引物各 1 μL,50 ng·L-1模板 DNA 5 μL,1 U Taq 酶1 μL,ddH2O補齊50 μL。PCR反應程序:94 ℃變性 5 min;94 ℃變性 30 s,55 ℃退火 30 s,72 ℃延伸30 s,35個循環。利用2%瓊脂糖凝膠電泳對PCR擴增產物進行鑒定,利用TIANGEN膠回收試劑盒對目標長度條帶進行切膠回收。

表1 重疊延伸PCR引物
1.3 重疊延伸PCR技術過程解析
重疊延伸PCR技術利用變異位點處正、反向引物以及該基因兩端引物進行第1輪PCR擴增,PCR產物形成包含overlap的重疊鏈;在第2輪PCR擴增中,通過overlap重疊鏈的延伸,可以將重疊鏈連接起來,如圖1所示。以TuMV C4-VPg質粒為模板,進行第1輪PCR擴增。TuMV C4-VPg和TuMV CDN1-VPg氨基酸序列存在5個差異位點,將VPg基因序列分為6個片段,分別為P1、P2、P3、P4、P5、P6,以 TuMV C4-VPg質粒為模板,以 bio120213/bioC4-Ⅰ-R和 bioC4-Ⅰ-F/bio120214組合為引物,分別擴增短片段P1和P23456。
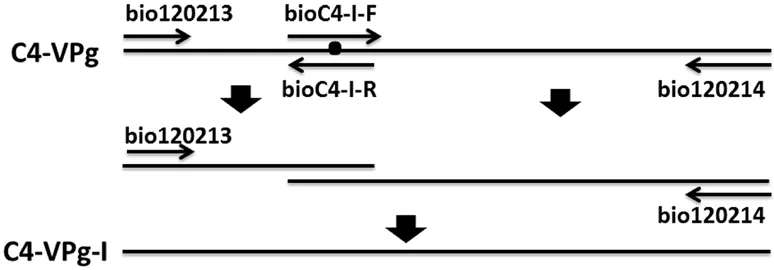
圖1 重疊延伸PCR擴增示意圖
利用2%瓊脂糖凝膠電泳分別對P1和P23456的PCR擴增產物進行檢測鑒定,并利用TIANGEN膠回收試劑盒回收目標條帶DNA,等比例混合。取上述混合DNA為模板,以bio120213/bio120214為引物,擴增包含第1個突變位點的P1-23456片段。第2輪PCR反應體系亦為50 μL,其中包括10×PCR緩 沖 液 5 μL,0.8 mmol·L-1dNTPs 4 μL,1.0 μmol·L-1上、下游引物各1 μL,兩端片段PCR產物都調至100 ng·L-1,各取5 μL,1 U Taq酶1 μL,ddH2O補齊50 μL。PCR反應程序:94 ℃變性5 min;94 ℃變性30 s,50~65 ℃退火(當兩條片段融合時,通過設置退火溫度梯度,尋找最合適的退火溫度)30 s,72 ℃延伸40 s,35個循環。對PCR擴增產物進行切膠回收,回收的DNA片段作為目標基因與pEASY-T1載體重組,均勻涂抹在固體LB培養基上,37 ℃過夜培養12 h。挑取若干單菌落,重懸在1 mL液體LB培養基中,在37 ℃、200 r·min-1的搖床上搖菌4 h。取3 μL菌液為模板,以bio120213/bio120214為引物,進行PCR鑒定。克隆載體過程參考全式金(北京)有限公司關于克隆載體pEASY-T1 Cloning Kit的操作指南。
2 結果與分析
2.1 TuMV CDN1-VPg與TuMV C4-VPg氨基酸差異位點分析
利用DNAMAN 5.0軟件對TuMV C4-VPg和TuMV CDN1-VPg氨基酸序列進行比對,發現二者存在5個差異氨基酸位點,即第52位異亮氨酸I/亮氨酸L,二者同為非極性疏水性氨基酸;第97位谷氨酸E/賴氨酸K,前者為酸性氨基酸,后者為堿性氨基酸;第101位和第105位天冬酰胺N/天冬氨酸D,前者為極性氨基酸,后者為酸性氨基酸;第117位異亮氨酸I/纈氨酸V,二者都為非極性疏水性氨基酸(圖2)。
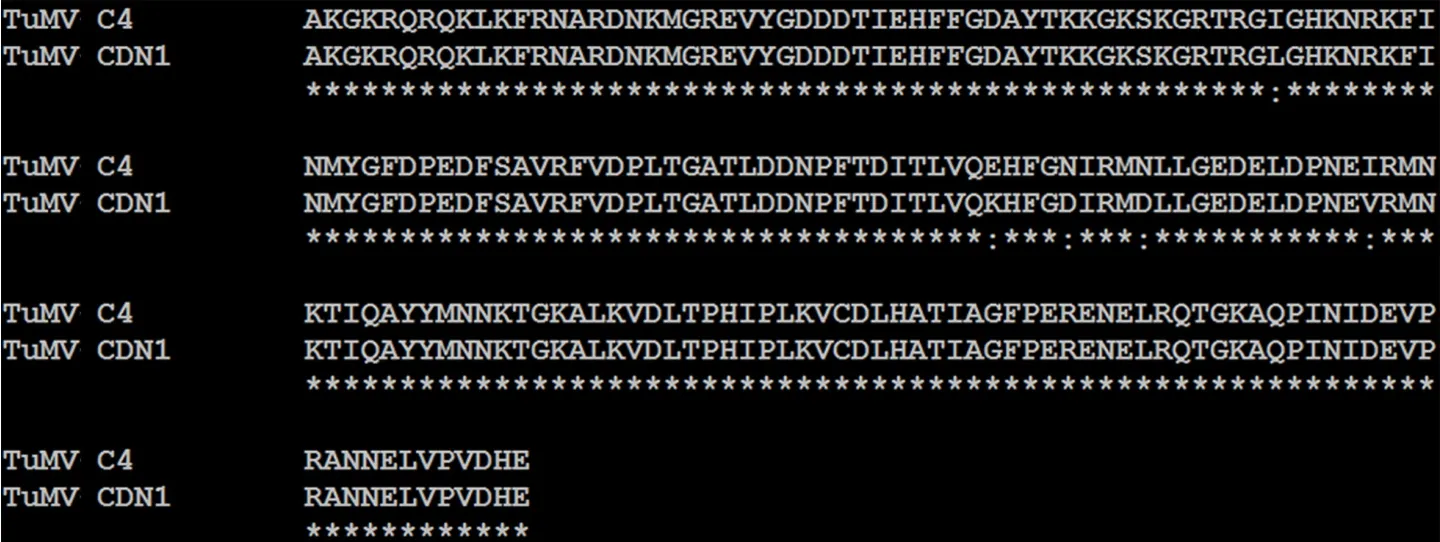
圖2 TuMV CDN1/C4-VPg氨基酸序列比對結果
2.2 利用改良SOE-PCR技術定點突變TuMV C4-VPg-Ⅰ基因
經過序列比對,TuMV C4-VPg與TuMV CDN1-VPg蛋白間存在5個氨基酸差異位點,在這5個差異氨基酸位點所對應的核苷酸位點處,設計正、反向引物(表1),即bioC4-Ⅰ-R/F、bioC4-Ⅱ-R/F、bioC4-Ⅲ-R/F、bioC4-Ⅳ-R/F和bioC4-Ⅴ-R/F。第1輪PCR擴增獲得P1(160 bp)和P23456(448 bp),第 2輪 PCR 擴增獲得 P1-23456,即 C4 VPg-Ⅰ(576 bp)。兩輪PCR擴增后,將獲得的PCR產物通過2%瓊脂糖凝膠電泳檢測,確認在退火溫度為53 ℃的PCR反應體系中,可以獲得目標片段長度的產物(圖3)。同時,通過梯度PCR儀,分別找到適于TuMV C4-VPg另外4個差異位點的退火溫度,分別為58、50、55、55 ℃。
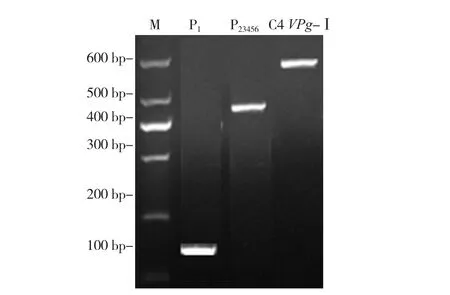
圖3 P1、P23456以及C4 VPg-Ⅰ 目標片段電泳檢測結果M,MarkⅠ。
第2輪PCR擴增后,將擴增產物通過2%瓊脂糖凝膠電泳檢測鑒定,采用天根膠回收試劑盒對目標長度的條帶進行切膠回收純化(由于第2輪PCR反應中會出現非特異性擴增,所以此時不宜用PCR產物純化試劑盒進行純化,應該通過瓊脂糖凝膠電泳鑒定后,切取目標長度的條帶進行回收),純化產物連接到pEASY-T1載體,轉化到大腸桿菌trans 5α。挑取2個陽性克隆菌液送美吉生物科技有限公司進行Sanger測序檢測。通過上述方法,分別對TuMV C4/CDN1-VPg基因序列存在的5個核苷酸差異位點進行定點突變,經過Sanger測序檢測,5個突變位點均達到預期要求,獲得5個單點突變體即TuMV C4-VPg-Ⅰ、TuMV C4-VPg-Ⅱ、TuMV C4-VPg-Ⅲ、TuMV C4-VPg-Ⅳ和TuMV C4-VPg-Ⅴ(圖 4)。
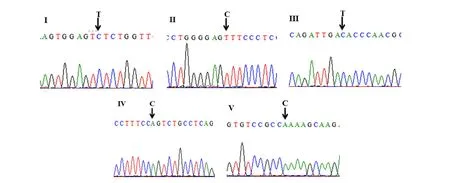
圖4 5個單點突變體基因的測序檢測Ⅰ,TuMV C4-VPg-Ⅰ;Ⅱ,TuMV C4-VPg-Ⅱ;Ⅲ,TuMV C4-VPg-Ⅲ;Ⅳ,TuMV C4-VPg-Ⅳ;Ⅴ,TuMV C4-VPg-Ⅴ。
3 結論與討論
Rusholme等(2007)利用大白菜抗/感病親本,定位克隆了retr01與ConTR01基因,其中retr01為隱性抗病基因,ConTR01為顯性抗病基因,這2個基因分別位于R4和R8染色體上,共同控制TuMV CDN1株系;Qian等(2013)以大白菜感病材料極早春和抗病材料BP8407為親本,采用BSA法,通過篩選SSR和InDel標記,最終定位克隆retr02基因,該基因為隱性抗病基因,具有廣譜抗性,尤其對TuMV C4株系表現高度抗性。白菜類蔬菜中不同的抗病毒基因對TuMV不同株系的抗性也不一樣,通過探究TuMV CDN1與C4株系間的差異,可以為揭示白菜類蔬菜抗病毒基因的抗病機制奠定基礎。
李國亮等(2017)通過酵母雙雜交試驗和雙分子熒光互補試驗表明:TuMV-C4與白菜eIF(iso)4E.a可以相互作用,而與白菜eIF(iso)4E.c不能相互作用;TuMV-UK1與白菜eIF(iso)4E.c可以相互作用,而與白菜eIF(iso)4E.a不能相互作用,通過對TuMV-UK1 VPg與TuMV-C4 VPg氨基酸序列進行比對,發現二者間存在4個氨基酸差異位點,且相對集中,分別是第89位(F/L)、第105位(N/D)、第 114位(P/S)和第119位(M/V)氨基酸,因此推測,某個關鍵氨基酸位點的變化可以影響其功能的表達。重疊延伸PCR技術可以在體外定點突變核苷酸,制備單點突變體用于其功能研究,該技術已有很多報道,但其反應體系尚待優化。雒麗娜等(2012)采用重疊延伸PCR技術,以重組的人組織型纖溶酶原激活劑rPA基因為模板,成功實現3個位點的定點突變,為rPA基因的進一步克隆和功能研究奠定了基礎。孟祥平等(2013)通過優化中間引物互補長度、SOE-PCR體系的模板濃度、起始模板的純度以及循環次數等條件,成功獲得特異性強和保真度高的BTRCP-Cyp A融合基因。
常規獲得一段DNA的方法包括限制性內切酶直接分離法、文庫篩選法、體外擴增法和人工合成法等。基因功能的研究往往集中于其中的若干個堿基變化,重疊延伸PCR技術對于研究體外基因突變的功能,提供了一種有利的方法。在該技術過程中經常出現擴增條帶彌散和非特異性擴增等情況,因此需要整理一個優化的技術流程,來快速、準確地定點突變基因。重疊延伸PCR依據兩條模板鏈中間的overlap進行重疊,互為模板進行延伸,因此overlap長度對第2輪PCR擴增比較重要,建議中間引物長度為30 bp左右,引物過短容易出現擴增條帶彌散現象;引物中GC含量在55%以上,有利于第2輪PCR擴增過程中兩段DNA片段融合;第2輪PCR擴增前,應將第1輪PCR擴增產物進行純化,避免第1輪PCR擴增過程中剩余的中間引物對第2輪PCR擴增產生影響;在第2輪PCR擴增過程中,模板過多或過少都易產生彌散或非特異擴增,應將第1輪PCR擴增的兩組產物等量混合(各取500 ng左右,50 μL擴增體系),作為第2輪PCR擴增的模板;另外,在第2輪PCR擴增過程中,通過設置退火溫度梯度,可以快速找到擴增該位點的適宜退火溫度,節約時間。本試驗通過梯度PCR儀,找到適于TuMV C4-VPg 5個差異位點的退火溫度,分別為53、58、50、55、55 ℃。
隨著植物基因定位、克隆的不斷發展,科學家們發現野生型個體和突變型個體往往只在某個基因上有若干個堿基的變化,最終導致其表型的巨大變化,但目前植物體內基因的定點突變技術受轉基因技術及植物組織培養技術的影響,推廣比較困難。重疊延伸PCR(SOE-PCR)技術是在植物體外對目標基因定點突變,成本低、簡單易操作,可以高效用于驗證目標氨基酸位點的功能。隨著二代、三代高通量測序的成熟,對目標基因上的SNP位點不斷深入挖掘,通過重疊延伸PCR技術,以SNP位點為橋梁,可以將基因定向轉化為單點突變體,為深入研究氨基酸位點的功能奠定基礎。
李國亮,錢偉,張淑江,李菲,章時藩,張慧,謝露露,武劍,王曉武,孫日飛.2017.白菜翻譯起始因子eIF(iso)4E.a/c與蕪菁花葉病毒TuMV-C4/UK1蛋白互作分析.園藝學報,44(7):1299-1308.
李諾敏,劉可夫,李靈蕓,何放,顧文彬,董一名,慶宏.2013.改進重疊延伸PCR克隆tau基因6種同工異構體.北京理工大學學報,33(88):871-875.
雒麗娜,王盛,王玉炯.2012.利用重疊延伸PCR技術進行定點突變研究.安徽農業科學,40(4):719-722.
孟祥平,楊建英,喬曉嵐,梁玲霞,席守民.2013.優化重疊延伸PCR條件構建BTRCP-CypA融合基因.生物技術,23(6):51-54.
帥勇,胡興旺,易朵,王成,趙曉蒙,周暢.2009.重疊延伸PCR法構建小鼠Sumf1基因的定點突變真核表達載體.生命科學研究,13(5):430-436.
熊愛生,彭日荷,陳建民,李賢,姚泉洪.2000.透明顫菌血紅蛋白(VHb)基因合成及原核生物中的效應.上海農業學報,16(3):19-24.
翟玲,董娜,張雅慧,黃勇,周岳華.2012.不需酶切位點的重疊延伸PCR在克隆表達中的應用.南昌大學學報:理科版,36(3):273-276.
Dinkova T D,Martinez-Castilla L,Cruz-Esp í ndola M A.2016.The diversification of eIF4E family members in plants and their role in the Plant-Virus interaction//Hern á ndez G,Rosemary J.Evolution of the Protein Synthesis Machinery and Its Regulation.〔S.l.〕:Springer International Publishing:187-205.
Duprat A,Caranta C,Revers F,Menand B,Browning K S,Robaglia C.2002.The Arabidopsis eukaryotic initiation factor(iso)4E is dispensable for plant growth but required for susceptibility to potyviruses.The Plant Journal,32(6):927-934.
Gallois J L,Charron C,S á nchez F,Pagny G,Houvenaghel M C,Moretti A.2010.Single amino acid changes in the Turnip mosaic virus viral genome-linked protein(VPg)confer virulence towards Arabidopsis thaliana mutants knocked out for eukaryotic initiation factors eIF(iso)4E and eIF(iso)4G.Journal of General Virology,91(1):288-293.
Gang H,Wen Q,Gao Q,Zhang F,Bai Y.2011.An efficient and rapid method for cDNA cloning from difficult templates using codon optimization and SOE-PCR:with human RANK and TIMP2 gene as examples.Biotechnology Letters,33(10):1939-1947.
Horton R M,Hunt H D,Ho S N,Pullen J K,Pease L R.1989.Engineering hybrid genes without the use of restriction enzymes:gene splicing by overlap extension.Gene,77(1):61-68.
Miyoshi H,Suehiro N,Tomoo K,Muto S,Takahashi T,Tsukamoto T,Ohmori T,Natsuaki T.2006.Binding analyses for the interaction between plant virus genome-linked protein(VPg)and plant translational initiation factors.Biochimie,88(3):329-340.
Miyoshi H,Okade H,Muto S,Suehiro N,Nakashima H,Tomoo K,Natsuaki T.2008.Turnip mosaic virus VPg interacts with Arabidopsis thaliana eIF(iso)4E and inhibits in vitro translation.Biochimie,90(10):1427-1434.
Qian W,Zhang S,Zhang S,Li F,Zhang H,Wu J,Wang X,Sun R.2013.Mapping and candidate-gene screening of the novel Turnip mosaic virus resistance gene retr02 in Chinese cabbage(Brassica rapa L.).Theoretical and Applied Genetics,126(1):179-188.Rusholme R L,Higgins E E,Walsh J A,Lydiate D J.2007.Genetic control of broad-spectrum resistance to Turnip mosaic virus in Brassica rapa(Chinese cabbage).Journal of General Virology,88(11):3177-3186.
Senanayake S D,Brian D A.1995.Precise large deletions by the PCR-based overlap extension method.Molecular Biotechnology,4(1):13.
Urban A,Neukirchen S,Jaeger K E.1997.A rapid and efficient method for site-directed mutagenesis using one-step overlap extension PCR.Nucleic Acids Research,25(11):2227.
Wittmann S,Chatel H,Fortin M G,Laliberte J.1997.Interaction of the viral protein genome linked of Turnip mosaic virus with the translational eukaryotic initiation Factor(iso)4E of Arabidopsis thaliana using the Yeast Two-Hybrid system.Virology,234(1):84-92.
Abstract:Turnip mosaic virus(TuMV)was an important disease for Brassica rapa L. ssp. pekinensis production,and the TuMV VPg gene had played a vital role infecting production process.While,the pathogenicity of different TuMV strains was diverse.This experiment adopted improved splicing by overlap extension PCR(SOE-PCR)technology,took TuMV-C4 VPg gene sequence as template,carried out sitedirected mutagenesis on 5 different single-nucleotides between TuMV C4-VPg and TuMV CDN1-VPg genes.The results indicated that 5 single point mutants of TuMV C4-VPg were obtained by the optimized SOE-PCR,i.e.TuMV C4-VPg-Ⅰ,TuMV C4-VPg-Ⅱ,TuMV C4-VPg-Ⅲ,TuMV C4-VPg-Ⅳ and TuMV C4-VPg-Ⅴ.
Key words:Brassica rapa L. ssp. pekinensis;Turnip mosaic virus(TuMV);VPg gene;Splicing by overlap extension PCR(SOE-PCR);Single point mutant
Site-directed Mutagenesis of TuMV C4-VPg Gene by Optimized Splicing by Overlap Extension PCR
LI Guo-liang,QIAN Wei,ZHANG Shu-jiang,LI Fei,ZHANG Shi-fan,ZHANG Hui,XIE Lu-lu,WU Jian,WANG Xiao-wu,SUN Ri-fei*
(Institute of Vegetables and Flowers,Chinese Academy of Agricultural Sciences,Beijing 100081,China)
李國亮,博士研究生,專業方向:蔬菜遺傳育種,E-mail:li_guo_liang@126.com
*通訊作者(Corresponding author):孫日飛,研究員,博士生導師,專業方向:蔬菜遺傳育種,E-mail:sunrifei@caas.cn
2017-07-11;接受日期:2017-08-30
國家自然科學基金項目(31372069),“十二五”國家科技支撐計劃項目(2013BAD01B04-03),農業部園藝作物生物學與種質創制重點實驗室項目

