孝感鳳窩酒曲真菌多樣性評價
王丹丹,沈 馨,董 蘊,鐘小丹,張振東,郭 壯*
(1.湖北文理學院 化學工程與食品科學學院鄂西北傳統發酵食品研究所,湖北 襄陽 441053;2.湖北米婆婆生物科技股份有限公司,湖北 孝感 432003)
孝感鳳窩酒曲真菌多樣性評價
王丹丹1,沈 馨1,董 蘊1,鐘小丹2,張振東1,郭 壯1*
(1.湖北文理學院 化學工程與食品科學學院鄂西北傳統發酵食品研究所,湖北 襄陽 441053;2.湖北米婆婆生物科技股份有限公司,湖北 孝感 432003)
采集了3個孝感鳳窩米酒曲樣品,使用Miseq高通量測序技術對其真菌微生物多樣性進行了評價。在屬水平上,相對含量>0.5%的屬分別為隸屬于毛霉亞門(Mucoromycotina)的淀粉霉(Amylomyces)、小克銀漢霉屬(Cunninghamella)和毛霉屬(Mucor)及隸屬于子囊菌門(Ascomycota)的復膜孢酵母屬(Saccharomycopsis)、曲霉屬(Aspergillus)、念珠菌(Candida)、擬威爾酵母(Cyberlindnera)和接合酵母(Zygosaccharomyces)。在分類操作單元(OUT)水平上,發現26個核心OTU,包含的序列數占所有質控后合格序列數的94.32%。由此可見,孝感鳳窩酒曲中的真菌微生物主要由隸屬于毛霉亞門和子囊菌門的若干個真菌屬構成,且米酒曲樣品共有大量的核心真菌菌群。
孝感;鳳窩酒曲;真菌;高通量測序
作為中國地理標識產品和湖北省非物質文化遺產代表性項目,孝感傳統釀造米酒已有千年的歷史,其以鳳窩酒曲發酵釀制而成,并因制法獨特而暢銷海內外[1]。鳳窩酒曲是在特定的生態環境內,用成曲作為母種培養制作而成,含有包括細菌和真菌在內的大量微生物,對米酒品質的形成起著決定性作用[2]。孝感地區現有米酒生產企業30余家,然而多數企業還是手工作坊式的生產方式,產品存在質量不穩定等諸多問題。由于發酵食品的制作多采用傳統制作工藝,制作環境相對開放,所以大部分發酵食品中的微生物群落信息要比以前想象的豐富而復雜[3]。由此可見,由于米酒曲微生物群落結構的復雜性,加之發酵溫度的不恒定,可能是導致孝感多數米酒企業產品質量不穩定的原因。因此,深入研究孝感鳳窩酒曲中微生物的組成,對其微生物多樣性信息進行全面系統的解析已成為當務之急。
眾所周知,純培養技術在微生物多樣性研究方面顯示出很大的片面性和局限性,變性梯度凝膠電泳(denaturing gradient gel electrophoresis,DGGE)等指紋圖譜技術雖然為認識微生物生態學提供了新的視角,然而其并不能讓研究人員在一次試驗中全面、平行分析多個樣本中的微生物群落信息[4]。以Illumina MiSeq為代表的第二代高通量測序技術彌補了上述技術的不足,具有操作簡單、成本低和結果可行度高的優勢,目前已在酸奶[5]、臘腸[6]、窖泥[7]、泡菜[8]、黑茶[9]和葡萄酒[10]等微生物多樣性研究中有了廣泛的應用。
本項目以孝感鳳窩酒曲為研究對象,在提取樣品宏基因組脫氧核糖核酸(deoxyribonucleic acid,DNA)的基礎上,使用引物對真菌18S rRNA V4-V5區進行聚合酶鏈反應(polymerase chain reaction,PCR)擴增,在使用Miseq高通量測序平臺進行序列測序的基礎上,結合生物信息學手段對其真菌多樣性進行揭示,以期為后續孝感米酒品質的提升及米酒用復合發酵劑的制備提供參考。
1 材料與方法
1.1 材料與試劑
5×TransStartTM、脫氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)Mix、FastPfu Buffer、FastPfu Fly DNA聚合酶:北京全式金生物技術有限公司;DNA 1000試劑盒:美國Agilent公司;QIAGENDNeasymericonFoodKit:德國QIAGEN公司。
1.2 儀器與設備
2100芯片生物分析儀:美國Agilent公司;5810R臺式高速冷凍離心機:德國Eppendorf公司;DYY-12電泳儀:北京六一儀器廠;Miseq高通量測序平臺:美國Illumina公司;ND-2000C微量紫外分光光度計:美國Nano Drop公司;UVPCDS8000凝膠成像分析系統:美國BIO-RAD公司;Vetiri梯度基因擴增儀:美國AB公司。
1.3 試驗方法
1.3.1 樣品采集
從湖北省孝感市孝南區九真和大東門農貿市場購買3個鳳窩酒曲,樣品采集后置于含有冰袋的采樣箱中冷鏈運送回實驗室。
1.3.2 DNA提取
取3g采集的米酒曲粉碎后加入27 mL0.01 mol/LpH值為7.4的磷酸鹽緩沖溶液,置于4℃冰箱中放置2 h后400×g離心10 min后取上清液,上清液于3 000×g離心10 min后收集菌體,采用QIAGEN DNeasy mericon Food Kit試劑盒進行微生物宏基因組DNA提取,濃度和純度均符合要求的DNA樣品置于-20℃保存備用。
1.3.3 PCR擴增及Miseq高通量測序
18S rRNA PCR擴增體系為:4 μL 5×PCR緩沖液,2 μL 2.5mmol/LdNTP,0.8μL5μmol/L正向引物,0.8μL5μmol/L反向引物,0.4 μL 5 U/μL DNA聚合酶,10 ng DNA模板,體系用ddH2O補充至20 μL。正向引物為SSU0817F(5′-TTAGCATGGAATAATRRAATAGGA-3′),反向引物為SSU1196R(5′-TCTGGACCTGGTGAGTTTCC-3′),引物對18S rRNA V4-V5區進行擴增,擴增長度為379 bp,其中在正向引物中加入7個核苷酸標簽(barcode)。
擴增條件為:95℃、3 min;95℃、30 s,55℃、30 s,72℃、45 s,30個循環;72℃、10 min,經1.0%瓊脂糖凝膠電泳檢驗合格的PCR擴增產物稀釋至100 nmol/L,并寄往上海美吉生物醫藥科技有限公司使用Miseq平臺進行高通量測序。
1.3.4 序列的拼接及質控
(1)Miseq數據下機后,依據序列之間的重疊關系將兩條對應的雙端序列拼接成一條序列,在拼接過程中若重疊區的堿基數<10 bp或最大錯配比率>0.2,或barcode堿基發生錯配,或引物堿基錯配數>2 bp,則該序列予以剔除;(2)根據barcode信息將每條拼接好的序列劃分到每個樣品并對序列方向進行校正;(3)將拼接好的序列中的barcode和引物切除,若切掉barcode和引物后序列堿基數<50 bp,則該拼接好的序列亦予以去除。
1.3.5 生物信息學分析
將3個樣品經過拼接和質控的序列合并成一個序列文件,使用QIIME(v1.7.0)分析平臺[11]進行物種鑒定和相對含量分析。使用PyNAST[12]軟件將所有序列對齊后,采用兩步UCLUST算法[13]分別以100%和97%的相似度進行序列劃分并建立分類操作單元(operationaltaxonomicunits,OTU),繼而從每個OTU中選取1條代表性序列,使用SILVA[14-15]數據庫進行比對,繼而獲得樣品中真菌微生物各分類地位學的信息,并使用FastTree軟件[16]繪制基于代表序列的系統發育進化樹,最終對米酒曲中真菌微生物的香農指數(Shannonindex)、發現物種數(ObservedSpecies)和超1指數(Chao1 index)等α多樣性指標進行計算。
1.3.6 優勢和核心菌群的定義
若隸屬于某一真菌門、真菌屬或OTU的微生物類群在3個孝感鳳窩酒曲樣品中均存在,則定義為核心菌群;若該真菌類群平均含量>0.5%,則將其定義為優勢菌群;若該類群在3個樣品中均存在且平均含量>0.5%,則定義為優勢核心菌群。
1.3.7 核酸登錄號
本研究中所有高通量測序數據均已提交至MG-RAST數據庫,ID號為mgp 80403。
1.3.8 數據處理
采用在線工具Venny 2.1(http://bioinfogp.cnb.csic.es/tools/venny/index.html)繪制維恩(Venn)圖;采用Mega5.0軟件繪制核心OTU系統發育樹;采用R軟件作圖繪制熱圖;其他圖采用Origin 8.6軟件繪制。
2 結果與分析
2.1 序列豐富度和多樣性分析
本研究的3個孝感鳳窩酒曲樣品18S rRNA測序情況及各分類地位數量如表1所示。

表1 樣品18S rRNA測序情況及各分類地位數量Table 1 18S rRNA sequencing conditions of samples and number of classifications at different taxonomical levels
由表1可知,本研究采集的3個孝感鳳窩酒曲樣品共產生了83 198條高質量18S rRNA序列,平均每個樣品產生27 733條。根據100%的相似性進行序列劃分共得到13 445條代表性序列,根據序列的97%相似性進行分類操作單元(OTU)劃分后,共得到126個OTU,每個樣品平均66個。通過稀疏曲線和香農指數曲線圖對產生的數據量是否滿足后續生物信息學分析進行了評價,其結果如圖1所示。

圖1 稀疏曲線圖(A)和香農指數曲線圖(B)Fig.1 Rarefaction curve(A)and Shannon diversity index curve(B)
由圖1A可知,隨著測序量的增加孝感鳳窩米酒曲中發現真菌物種的數量也增加,這說明隨著測序深度的加大,新的真菌種系型可能會被發現。由圖1B可知,在測序量到1 000條的時候,香農指數曲線就已經進入了平臺期,這說明隨著測序深度的加大,物種的多樣性不再隨之變化了。由此可見,本研究中每個樣品產生27 733條18S rRNA序列的測序深度是可以滿足后續生物信息學分析要求的。
2.2 基于不同分類地位核心真菌菌群相對含量的分析
在采用UCLUST進行序列兩步劃分的基礎上,本研究進一步使用SILVA數據庫對每個OTU包含的代表性序列進行了同源性比對,并對各OTU包含序列數的相對含量進行了計算。納入本研究的83 198條序列,所有的序列鑒定為3個門,7個綱,7個目,17個科和27個屬,僅有0.76%的序列不能鑒定到屬水平。在門水平上,所有的序列鑒定為子囊菌門(Ascomycota)、擔子菌亞門(Basidiomycota)和毛霉亞門(Mucoromycotina),其平均含量分別為46.90%、0.03%和53.08%,其中子囊菌門(Ascomycota)和毛霉亞門(Mucoromycotina)在3個樣品中均存在。由此可見,孝感鳳窩酒曲中真菌主要由隸屬于子囊菌門(Ascomycota)和毛霉亞門(Mucoromycotina)的菌群構成。值得一提的是,3個樣品中隸屬于子囊菌門(Ascomycota)的真菌相對含量分別為60.45%、4.87%和75.38%,隸屬于毛霉亞門(Mucoromycotina)的真菌相對含量分別為39.47%、95.13%和24.62%,因而在門水平上不同鳳窩酒曲樣品中真菌微生物的構成存在較大的差異。基于屬水平3個鳳窩酒曲樣品中優勢真菌的相對含量如圖2所示。

圖2 孝感鳳窩酒曲中優勢真菌屬相對含量的比較分析Fig.2 Comparative analysis on the relative contents of the dominant fungal genera in Xiaogan Fengwo rice wine koji
由圖2可知,納入本研究的3個孝感鳳窩酒曲樣品中優勢真菌屬分別為隸屬于毛霉亞門(Mucoromycotina)的淀粉霉(Amylomyces)、小克銀漢霉屬(Cunninghamella)和毛霉屬(Mucor),其平均相對含量分別為49.06%、1.44%和1.14%;及隸屬于子囊菌門(Ascomycota)的復膜孢酵母屬(Saccharomycopsis)、曲霉屬(Aspergillus)、念珠菌(Candida)、擬威爾酵母(Cyberlindnera)和接合酵母(Zygosaccharomyces),其平均相對含量分別為32.12%、10.07%、1.44%、1.37%和1.16%。上述8個優勢真菌屬的累計平均相對含量為97.80%,其他19個屬的平均相對含量累計為1.44%。由此可見,孝感鳳窩酒曲中的真菌微生物主要由隸屬于毛霉亞門(Mucoromycotina)和子囊菌門(Ascomycota)的若干個屬構成。
近些年來,國內學者對我國信陽[17]、廣東[18]、黑龍江[19]以及陜北地區[20]米酒或者米酒曲中微生物的多樣性開展了多項卓有成效的研究,均認為霉菌和酵母菌是其中的優勢菌。部分學者以孝感地區米酒或鳳窩酒曲為研究對象進行了相關研究工作:況啟生等[21]采用傳統分離方法對孝感民間米酒的微生物多樣性進行了研究,共分離出包括霉菌、酵母菌、乳酸菌和芽孢桿菌在內的13個屬、36個種的微生物菌株,這在一定程度上也說明了孝感米酒具有較高的微生物多樣性;采用傳統分離鑒定和平板菌落計數法,王艷萍等[22]對來自孝感的米酒醪中的微生物進行了研究,發現霉菌和酵母菌是米酒發酵過程中間的優勢菌;王小紅等[2-3]對鳳窩酒曲中酵母菌和根霉進行了分離鑒定,研究發現,釀酒酵母和假絲酵母為其中的優勢酵母菌屬;蔡麗等[23]亦對鳳窩酒曲中根霉進行了分離鑒定,分離到6株米根霉(Rhizopus oryzae)和7株華根霉(Rhizopus chinesis)。
本研究進一步對平均相對含量>0.5%且在3個樣品中均存在的核心真菌屬進行了統計分析,其結果如圖3所示。
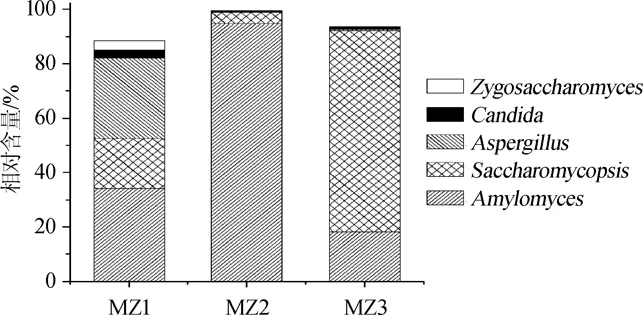
圖3 孝感鳳窩酒曲中優勢核心真菌屬相對含量的比較分析Fig.3 Comparative analysis on the relative contents of the core fungal genera in Xiaogan Fengwo rice wine koji
由圖3可知,隸屬于毛霉亞門(Mucoromycotina)的淀粉霉(Amylomyces),隸屬于子囊菌門(Ascomycota)的復膜孢酵母屬(Saccharomycopsis)、曲霉屬(Aspergillus)、接合酵母(Zygosaccharomyces)和念珠菌(Candida)為納入本研究的3個孝感鳳窩酒曲中優勢核心真菌菌群,上述5個真菌屬在MZ1、MZ2和MZ3中的累計相對含量分別為88.50%、99.50%和93.60%。這也進一步證實了,孝感鳳窩酒曲共有大量的核心真菌菌群。不僅在中國,在韓國、日本、印度尼西亞和越南等亞洲國家也有飲用米酒的習慣,雖然在不同的國家,米酒的名字及原料可能不同,但其發酵工藝流程是相似的。SUJAYA I N等[24]采用純培養的方法發現釀酒酵母(Saccharomyces cerevisiae)是印尼地區米酒Brem發酵過程中的優勢酵母菌株。采用相同的方法,DUNG N T P等[25]對越南的米酒Men進行了研究,認為酵母菌和霉菌是其中的優勢菌群。本研究進一步統計了OTU在3個樣品中出現的次數,其結果如圖4所示。
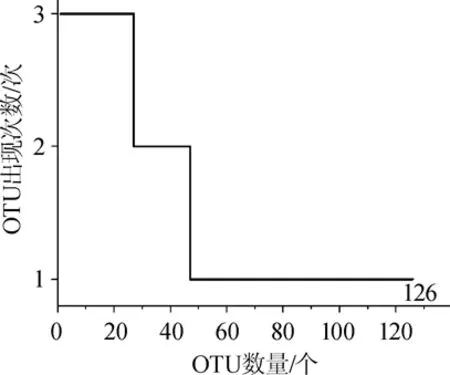
圖4 OTU在孝感鳳窩酒曲樣品中出現次數統計Fig.4 Occurrence frequency statistics of OTU in Xiaogan Fengwo rice wine koji
由圖4可知,3個孝感鳳窩酒曲樣品共產生126個真菌OTU,其中核心OTU有26個,占OTU總數的20.64%,分別出現2次和1次的OTU分別有20個和80個,分別占OTU總數的15.87%和63.49%。基于OTU水平的維恩圖如圖5所示。
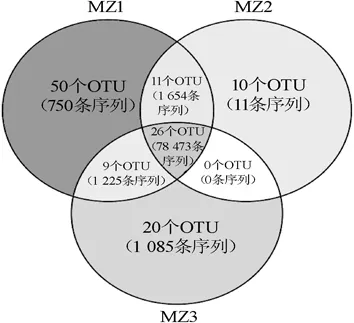
圖5 基于OTU水平的維恩圖Fig.5 Venn diagram based on OTU level
由圖5可知,雖然納入本研究的3個孝感鳳窩酒曲樣品中共發現了26個真菌的核心OTU,僅占OTU總數的20.63%,但其包含了78473條序列,占所有質控后合格序列數的94.32%。MZ1、MZ2和MZ3特有的OTU數量分別為50個、10個和20個,包含的序列數分別為750條、11條和1 085條。在3個樣品中僅出現2次的OTU,其中MZ1和MZ2共有11個OTU和1 654條序列,MZ1和MZ3共有20個OTU和1 085條序列,MZ2和MZ3無共有OTU。綜上所述,在3個孝感鳳窩酒曲樣品中出現2次和1次的OTU所包含序列數分別為2 739條和1 846條,分別占所有質控后合格序列數的3.29%和2.22%。由此可見,納入本研究的3個孝感鳳窩酒曲共有大量的核心真菌菌群,雖然有的米酒曲中可能含有一些較為獨特的真菌種系型,但其相對含量是極低的。在26個核心OTU中相對含量>0.5%的OTU共有6個,依據OTU的進化關系建立了系統發育樹,結果如圖6所示。

圖6 相對含量大于0.5%的核心OTU在各孝感鳳窩酒曲樣品的系統發育樹Fig.6 Phylogenetic tree of the core OTU with relative abundance more than 0.5%in Xiaogan Fengwo rice wine koji
由圖6可知,6個相對含量>0.5%的核心OTU中,各有1個隸屬于復膜孢酵母屬(Saccharomycopsis)和念珠菌(Candida)、各有2個隸屬于曲霉屬(Aspergillus)和淀粉霉(Amylomyces)。在3個孝感鳳窩酒曲樣品中OTU102(隸屬于復膜孢酵母屬)的相對含量分別為17.88%、3.96%和72.99%,OTU100(隸屬于曲霉屬)的相對含量分別為24.83%、0.25%和0.28%,OTU57(隸屬于淀粉霉)的相對含量分別為33.09%、94.12%和17.50%。上述3個核心OTU的累計相對含量分別為75.80%、98.33%和90.78%。由此可見,雖然孝感鳳窩酒曲樣品共有大量的核心真菌菌群,但核心真菌菌群的多樣性相對較低,3個核心OTU的相對含量就占到了真菌類群的接近90%。本研究進一步對相對含量>0.5%的核心OTU之間的相關性進行了分析,其結果如圖7所示。

圖7 相對含量大于0.5%的核心OTU相關性的聚類樹Fig.7 Clustering tree of correlation among the core OTU with relative abundance more than 0.5%
由圖7可知,OTU116(隸屬于念珠菌屬)與OTU100(隸屬于曲霉屬)呈顯著正相關(R2=0.9997,P=0.011),與OTU83(隸屬于曲霉屬)呈非常顯著正相關(R2=0.999 9,P=0.007),OTU100(隸屬于曲霉屬)與OTU83(隸屬于曲霉屬)亦呈顯著正相關(R2=0.999 1,P=0.018)。除此之外,其他相對含量>0.5%的核心OTU之間無顯著相關性(P>0.05)。
3 結論
在提取樣品宏基因組DNA的基礎上,本研究使用高通量測序技術,以18S rRNA V4-V5區為測序靶點,對孝感鳳窩酒曲中真菌微生物多樣性進行了評價。結果發現,孝感鳳窩酒曲中的真菌微生物主要由隸屬于毛霉亞門和子囊菌門的淀粉霉屬、小克銀漢霉屬、毛霉屬、復膜孢酵母屬、曲霉屬、念珠菌、擬威爾酵母和接合酵母屬的8個屬組成,且酒曲樣品共有大量的核心真菌菌群。通過本研究的開展,可為后續孝感米酒品質的提升和米酒用復合發酵劑的制備提供一定理論參考。
[1]談生安.孝感米酒工藝技術[J].食品工業科技,1998,19(1):64-65.
[2]王小紅,徐 康,趙 山,等.孝感鳳窩酒曲中酵母菌的分離及特性研究[J].現代食品科技,2008,24(2):134-137.
[3]王小紅,王 瑩,趙 山,等.孝感鳳窩酒曲中根霉的分離及特性研究[J].中國釀造,2008,27(13):21-24.
[4]COCOLIN L,ALESSANDRIA V,DOLCI P,et al.Culture independent methods to assess the diversity and dynamics of microbiota during food fermentation[J].Int J Food Microbiol,2013,167(1):29-43.
[5]智楠楠.Illumina Miseq平臺深度測定酸奶中微生物多樣性[J].食品工業科技,2016,37(24):78-82.
[6]JUSTYNA P,ANNALISA R,VINCENZA P,et al.Bacterial diversity in typical Italian salami at different ripening stages as revealed by highthroughput of 16S rDNA amplicons[J].Food Microbiol,2015,46(4):342-356.
[7]HU X,DU H,REN C,et al.Illuminating anaerobic microbial community and cooccurrence patterns across a quality gradient in Chinese liquor fermentation pit muds[J].Appl Environm Microbiol,2016,82(8):2506-2515.
[8]YANG H,WU H,GAO L,et al.Effects ofLactobacillus curvatusand Leuconostoc mesenteroideson Suancai fermentation in northeast China[J]. J Microbiol Biot,2016,26(12):2148-2158.
[9]FU J,LV H,CHEN F.Diversity and variation of bacterial community revealed by MiSeq Sequencing in Chinese dark teas[J].PloS One,2016,11(9):e0162719.
[10]BOYNTON P J,GREIG D.Fungal diversity and ecosystem function data from wine fermentation vats and microcosms[J].Data Brief,2016,8(12):225-229.
[11]CAPORASOJ G,KUCZYNSKIJ,STOMBAUGHJ,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature Meth,2010,7(5):335-336.
[12]CAPORASO J G,BITTINGER K,BUSHMAN F D,et al.PyNAST:a flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[13]EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[14]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucleic Acid Res,2012,41(1):590-596.
[15]YILMAZP,PARFREYLW,YARZAP,etal.TheSILVAand"all-species living tree project(LTP)"taxonomic frameworks[J].Nucleic Acid Res,2014,42(D1):643-648.
[16]PRICE M N,DEHAL P S,ARKIN A P.Fasttree:computing large minimum evolution trees with profiles instead of a distance matrix[J].Mol Biol Evol,2009,26(7):1641-1650.
[17]李福榮.信陽米酒微生物群落組成[J].信陽師范學院學報:自然科學版,2005,18(1):52-53.
[18]李健容,蔡愛群.民間傳統酒曲主要微生物的分離及鑒定[J].釀酒科技,2007,28(5):111-115.
[19]焦晶凱.傳統釀造米酒微生物多樣性及優勢菌特性的研究[D].哈爾濱:哈爾濱工業大學,2012.
[20]郝 瑩,王衛衛,王莉娟,等.陜北傳統米酒曲中優勢菌種的分離,純化及鑒定[J].檢驗檢疫科學,2010,19(2):44-47.
[21]況啟生,李安明,戴余軍,等.孝感民間傳統米酒菌株的分離,鑒定與篩選,培養[J].食品科學,2003,24(9):94-96.
[22]王艷萍,程巧玲,張 陽,等.米酒醪中優勢微生物菌相組成的初步研究[J].中國釀造,2008,27(5):12-14.
[23]蔡 麗,易 響,陳福生,等.傳統米酒酒曲的評價及根霉的分類鑒定[J].中國釀造,2010,29(12):30-33.
[24]SUJAYA I N,ANTARA N S,SONE T,et al.Identification and characterization of yeasts inbrem,a traditional Balinese rice wine[J].World J Microbiol Biot,2004,20(2):143-150.
[25]DUNG N T P,ROMBOUTS F M,NOUT M J R.Characteristics of some traditional Vietnamese starch-based rice wine fermentation starters[J].LWT-Food Sci Technol,2007,40(1):130-135.

WANG Dandan1,SHEN Xin1,DONG Yun1,ZHONG Xiaodan2,ZHANG Zhendong1,GUO Zhuang1*
(1.Northwest Hubei Research Institute of Traditional Fermented Food,College of Chemical Engineering and Food Science,Hubei University of Arts and Science,Xiangyang 441053,China;2.Hubei Granny Mi Biotechnology Co.,Ltd.,Xiaogan 432003,China)
TS262.4
0254-5071(2017)11-0038-05
10.11882/j.issn.0254-5071.2017.11.009
2017-08-09
湖北省教育廳科學技術研究計劃中青年人才項目(Q20152603)
王丹丹(1996-),女,本科生,研究方向為食品生物技術。
*通訊作者:郭 壯(1984-),男,副教授,博士,研究方向為食品生物技術。

