海南省6個養殖羅非魚群體遺傳差異的微衛星分析
,,,,
(1.海南省海洋與漁業科學院,海南 海口 570206; 2.國家羅非魚產業技術體系海口綜合試驗站,海南 海口570100)
海南省羅非魚養殖開始于1956年,是國內極早開始養殖羅非魚的省份之一,養殖品種從最初的莫桑比克羅非魚到20世紀90年代的奧尼羅非魚、奧利亞羅非魚,再到現在的雜交品種吉富羅非魚等,尤其進入2000年以后,海南省羅非魚產業得到飛速發展,產量從1999年的5.8×104t增加到2010年的27×104t,而后,每年產量基本維持穩定[1]。
微衛星標記具有高度多態性、符合孟德爾遺傳、在基因組中廣泛分布等優點,已被廣泛應用于動植物種質鑒定和遺傳結構多樣性分析[2-6]。
近年來,多位學者已將微生物標記應用于許多與羅非魚相關的研究中,如Boris 等[7]利用微生物標記分析了6個紅羅非魚群體的遺傳變異,評估了紅羅非魚的育種計劃;強俊等[8]研究吉富羅非魚與奧利亞羅非魚自繁與雜交 F1代的遺傳特性與抗病力,發現雜交后代的抗病力明顯提高;李建林等[9]發現 2個吉富羅非魚群體的遺傳多樣性水平較高,存在雜合子過剩現象,且具有較強的環境適應能力,選育空間大。本研究擬通過篩選羅非魚微衛星標記,獲得海南省6個養殖羅非魚[尼羅羅非魚(Oreochromisniloticus)、奧尼羅非魚(Oreochromisniloticus×O.aureus)、莫桑比克羅非魚(Oreochromismossambica)、紅羅非魚(Red tilapia,Oreochromisniloticus×O.mossambica)、泰奧羅非魚(Oreochromisniloticus×O.aureus)、吉富羅非魚(GIFT strain ofOreochromisniloticus)]群體的等位基因,并用軟件Popgen3.2對每一個微衛星位點的等位基因的遺傳參數進行分析;此外根據各群體間的遺傳相似性指數和遺傳距離,采用MEGA3.0鄰接法(NJ)構建6個羅非魚群體的系統發育樹,以分析群體間的親緣關系。加之現在羅非魚種質資源退化嚴重,造成魚苗成活率下降,極大影響了產業的可持續發展,因此,研究羅非魚群體遺傳差異勢在必行,亦可為選育優良的羅非魚品種奠定理論基礎。
1 材料與方法
1.1 試驗材料
海南省6個羅非魚(尼羅羅非魚、奧尼羅非魚、莫桑比克羅非魚、紅羅非魚、泰奧羅非魚、吉富羅非魚)群體采集自海南省海口市和文昌市兩個地區,其中尼羅羅非魚、奧尼羅非魚等5個群體采集的規格約500 g,莫桑比克羅非魚采集規格為50~100 g,每個群體隨機抽取30尾,分別采集其尾鰭樣品,保存于無水乙醇中。
1.2 基因組DNA的提取
采用基因組DNA提取試劑盒(天根生物有限公司)提取尾鰭基因組DNA,1%瓊脂糖凝膠電泳檢測抽提DNA的質量,-20℃保存備用。
1.3 微衛星引物的篩選
在美國漢普郡大學Hubbard基因研究中心網站數據(http://hcgs.unh.edu/)和Carleton等報道的羅非魚微衛星引物序列中選取20對微衛星引物,對海南養殖的6個羅非魚群體(尼羅羅非魚、奧尼羅非魚、莫桑比克羅非魚、紅羅非魚、泰奧羅非魚、吉富羅非魚)的DNA樣本進行擴增,引物序列由艾基生物技術有限公司合成,引物序列見表1。

表1 微衛星擴增引物序列

續表1
1.4 PCR擴增體系及程序
反應總體積為20μL,其中10×PCR buffer 2.0 μL;MgCl2(25 mmol·L-1)0.8 μL;dNTP(10 mmol·L-1each)0.3 μL;Taq Polymerase(5 U·μL-1)0.2 μL;上下游引物(10 mmol·L-1)各0.5 μL;DNA模板(40~100 ng·μL-1)0.3 μL;滅菌雙蒸水14.4 μL。PCR反應程序為:94℃預變性3 min;94℃變性30 s;50~60℃退火30 s;72℃延伸45 s;共25個循環;最后72℃延伸7 min。根據擴增效果調節MgCl2濃度和退火溫度,MgCl2濃度為0.8~1.2 mmol·L-1。
1.5 電泳和銀染檢測
利用濃度為10%的非變性聚丙烯酰胺凝膠(每100 mL中ddH2O 35.0 mL;30% Acylamide/Bis 4.0 mL;10×TBE 9.33 mL;20% AP 3.27 mL;TEMED 117 μL)對微衛星PCR產物進行電泳,PCR產物上樣量均為4 μL,DNA Marker上樣量為0.5 μL, 200 V電泳2 h左右。然后進行銀染3 min,洗滌后進行顯色,記錄電泳結果。
1.6 等位基因統計與數據處理
對銀染后得到的譜帶進行整理并用軟件Popgen3.2進行分析,對每一個微衛星位點的等位基因的數量進行統計,并計算等位基因數、有效等位基因數、觀測雜合度、期望雜合度、Shannon’s 多態性指數等遺傳參數;此外根據各群體間的遺傳相似性指數和遺傳距離,采用MEGA3.0鄰接法(Neighbor-Joining,NJ)構建6個羅非魚群體的系統發生樹,以分析群體間的親緣關系。
2 結果與分析
2.1 微衛星電泳及等位基因分析結果
20對微衛星引物能夠在6個羅非魚群體樣本(尼羅羅非魚、奧尼羅非魚、莫桑比克羅非魚、紅羅非魚、泰奧羅非魚、吉富羅非魚)中穩定重復地擴增出特異片段,非變性聚丙稀酰胺凝膠電泳部分結果見圖1。20對微衛星引物共擴增出68個等位基因,各基因座的等位基因為2~5個,大小為100~300 bp,其中莫桑比克羅非魚平均有效等位基因數量最高(2.26),奧尼羅非魚平均等位基因數量最低(1.99),表2為微衛星引物的擴增條帶統計結果。
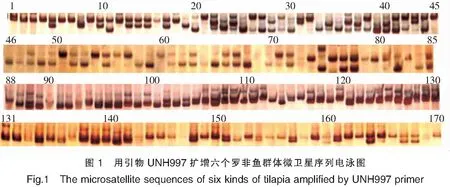
注:1~27為紅羅非魚;28~57為吉富羅非魚;58~85為莫桑比克羅非魚;86~112為泰奧羅非魚;113~141為尼羅羅非魚;142~170為奧尼羅非魚。
Notes:1~27 was red tilapia,Oreochromisniloticus×O.mossambica;28~57 was GIFT strain ofOreochromisniloticus;58~85 wasOreochromismossambica;86~112 was Thailand strain ofOreochromisniloticus×O.aureus;113~141 wasOreochromisniloticus;142~170 wasOreochromisniloticus×O.aureus.
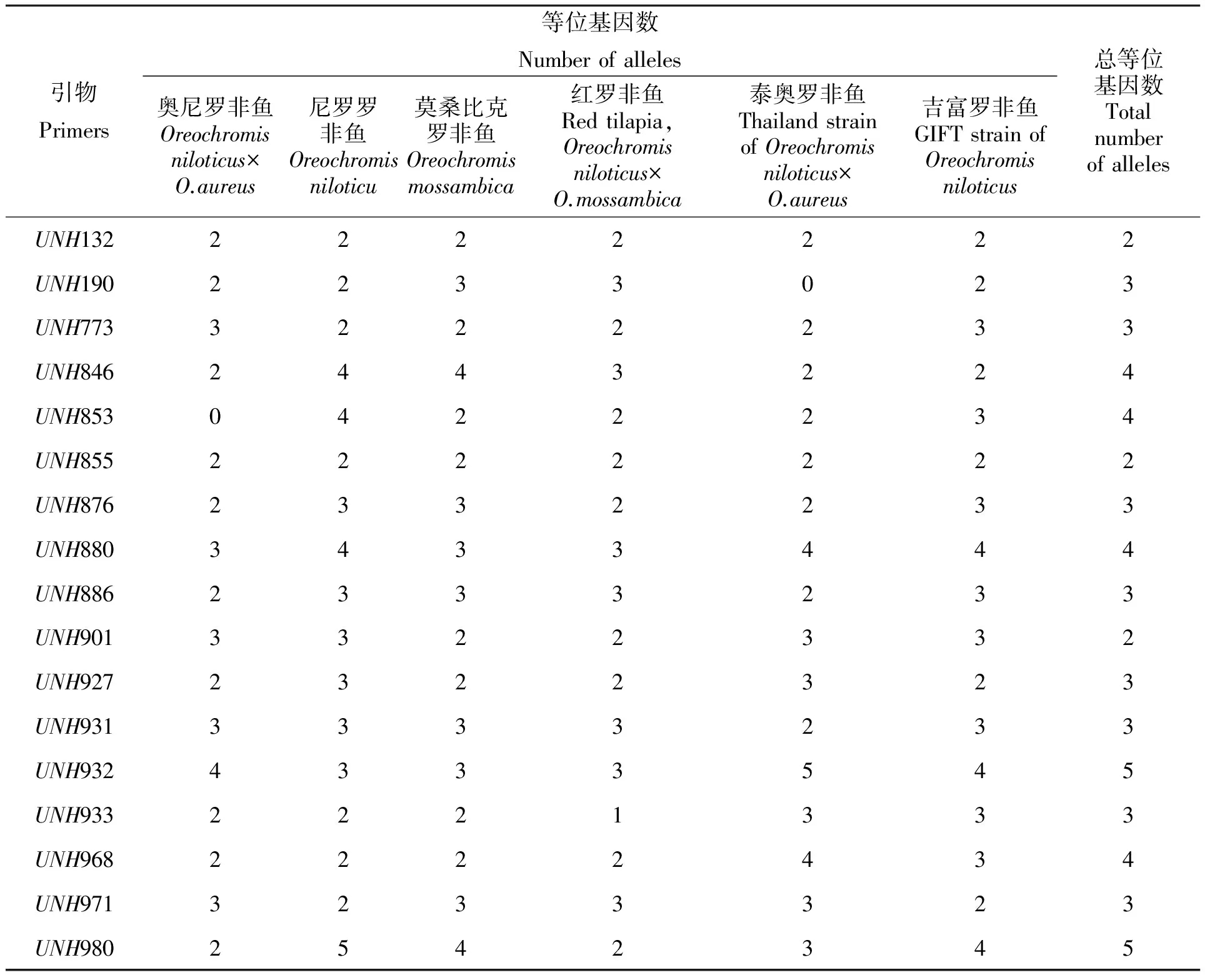
表2 6個羅非魚群體微衛星鑒定位點的等位基因數目
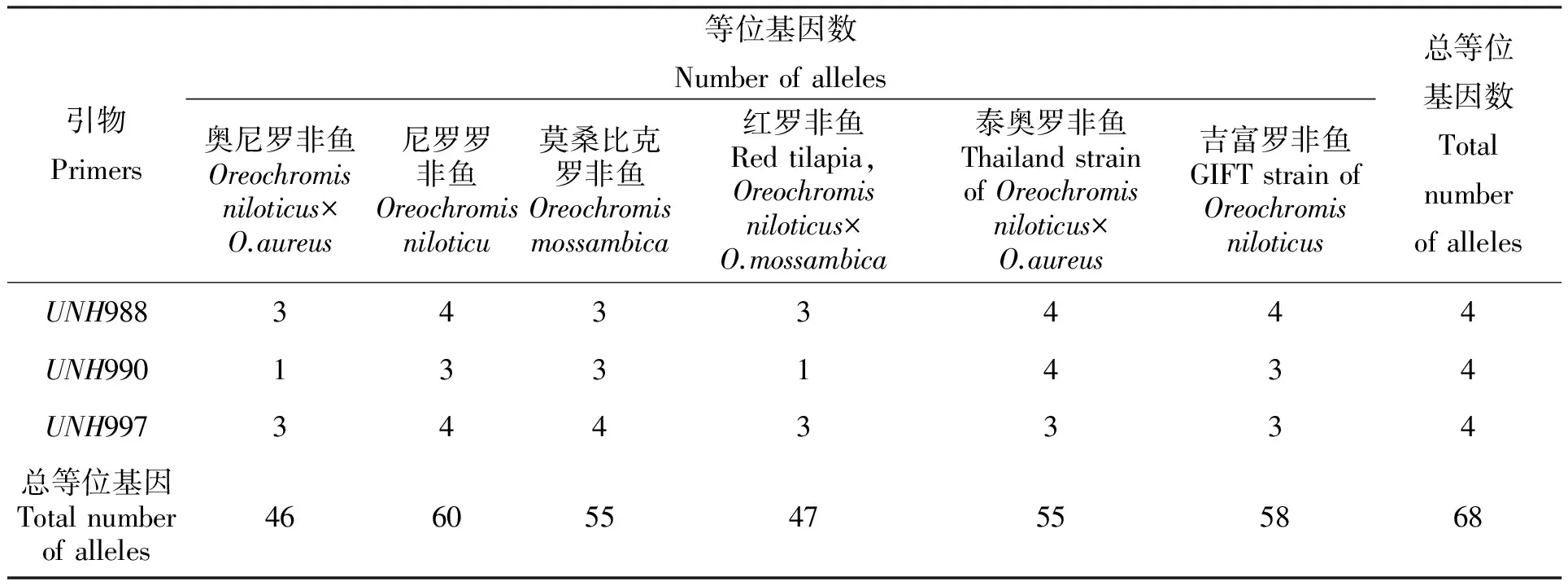
續表2
2.2 6個羅非魚群體的遺傳參數分析結果
表3為20個微衛星位點在6個羅非魚群體的遺傳參數分析結果,平均觀測雜合度在莫桑比克羅非魚中最高(0.75),在尼羅羅非魚中最低(0.57);平均期望雜合度和多態信息含量在莫桑比克羅非魚中最高(0.53,0.44),在奧尼羅非魚中最低(0.47,0.38)。Shannon’s 多樣性指數在莫桑比克羅非魚中最高(0.85),其次分別為泰奧羅非魚(0.83)、尼羅羅非魚(0.80)和吉富羅非魚(0.80),而奧尼羅非魚(0.72)最低。
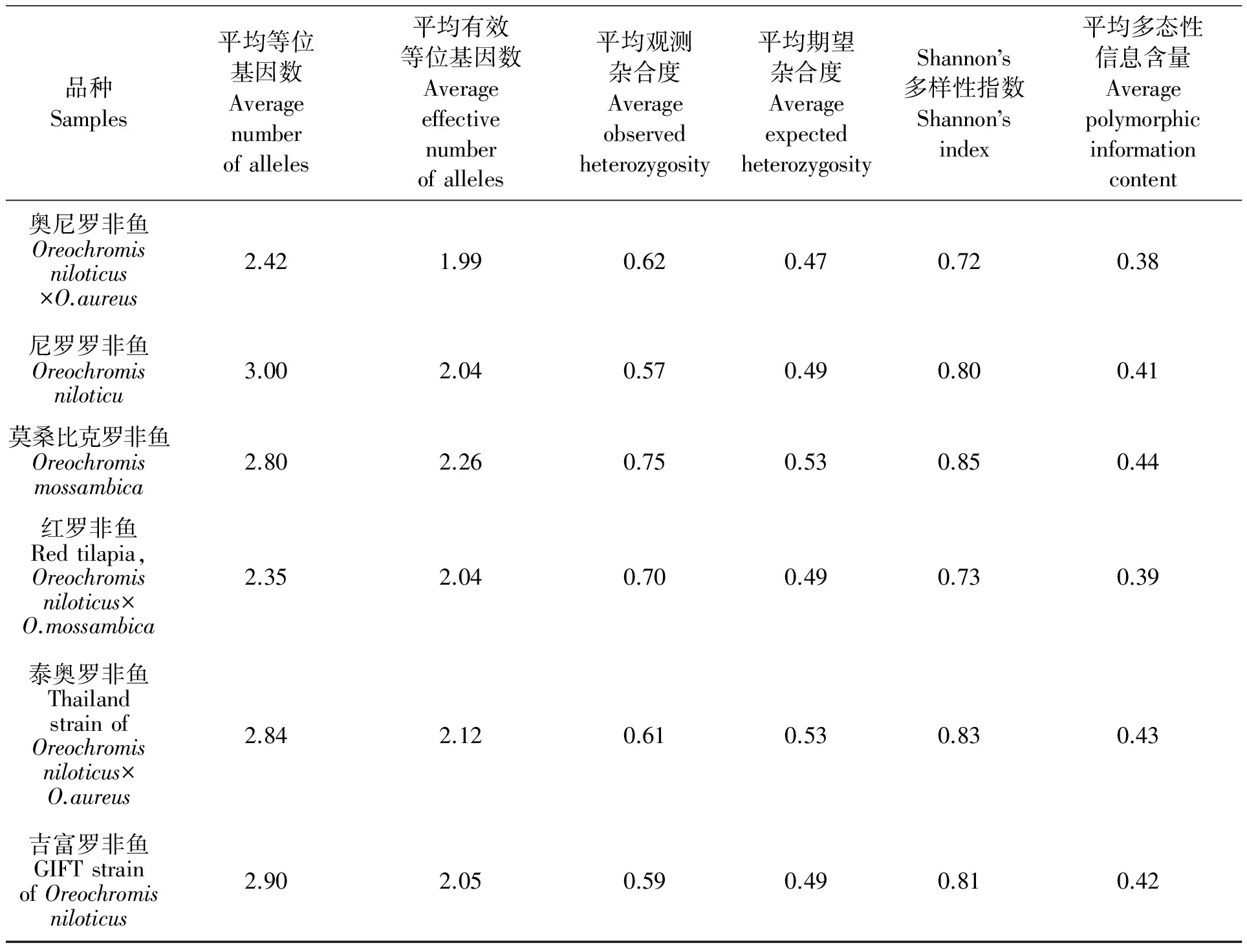
表3 20個微衛星位點在6個羅非魚群體的遺傳參數
2.3 6個羅非魚群體的遺傳距離
遺傳距離和遺傳相似度分析結果表明:尼羅羅非魚與泰奧羅非魚的遺傳距離最大(0.21),遺傳相似系數最小(0.81),說明兩者親緣關系最遠;吉富羅非魚和尼羅羅非魚的遺傳距離最小(0.08),遺傳相似系數最大(0.93),說明兩者親緣關系最近(表4)。

表4 6個羅非魚群體間遺傳距離(左下部分)和遺傳相似度(右上部分)
注:1為奧尼羅非魚;2為尼羅羅非魚; 3為莫桑比克羅非魚; 4為紅羅非魚;5為泰奧羅非魚; 6為吉富羅非魚; *為缺省。
Notes:1 wasOreochromisniloticus×O.aureus;2 wasOreochromisniloticu;3 wasOreochromismossambica;4 was red tilapia,Oreochromisniloticus×O.mossambica;5 was Thailand strain ofOreochromisniloticus×O.aureus;6 was GIFT strain ofOreochromisniloticus;* was default.
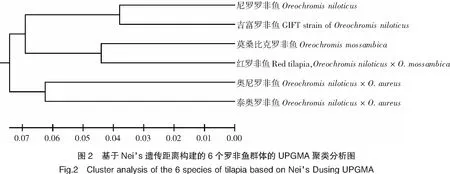
利用群體間的遺傳距離矩陣,在MEGA3.0 軟件中采用UPGMA 法繪制6個羅非魚群體的系統進化樹。結果表明,吉富羅非魚是尼羅羅非魚中的一個品系,它們屬于同一種,并聚成一支;紅羅非魚和莫桑比克羅非魚聚為一支,再與吉富羅非魚和尼羅羅非魚聚為一大支,奧尼羅非魚和泰奧羅非魚聚為一支(圖2)。
3 討論
本研究應用的微衛星標記技術是較理想的品種鑒定方法,不僅可準確鑒定同一群體中親本之間、親本與雜交種之間的不同種,也可鑒別不同雜交組合的雜種子代,目前,該技術已被廣泛應用于研究物種分類、進化及遺傳結構,其中Ho、He和PIC等是衡量遺傳多樣性的重要指標。
Ho是衡量種群遺傳變異程度的重要參數,與群體遺傳結構變異程度呈正相關,且Ho越高,遺傳多樣性越豐富[10]。本研究中,6個羅非魚群體的Ho分別為0.62、0.57、0.75、0.70、0.61和0.59,略高于Dewoody的研究結果[11],研究結果表明,6個養殖羅非魚群體的遺傳多樣性為中等水平,其中奧尼羅非魚和紅羅非遺傳多樣性指數較低,說明其存在種質退化現象。紅羅非魚雖由尼羅羅非魚和莫桑比克羅非魚雜交選育而來,但多態性指數卻低于莫桑比克羅非魚群體,因此可根據本文的研究結果,對其親本家系進行擴充,擴大繁殖群體規模,提高品種遺傳多樣性,進而提高該羅非魚品種的種質質量。泰奧羅非魚與奧尼羅非魚都為尼羅羅非魚與奧利亞羅非魚的雜交種,但其遺傳多樣性差異較大,這可能主要源于親本中本地尼羅羅非魚與泰國品系的尼羅羅非魚遺傳多樣性的差異。
PIC是衡量微衛星位點多態性水平的重要指標。一般情況,當PIC<0.25時微衛星位點低度多態性,當0.25
根據微衛星分析結果建立的系統進化樹顯示吉富羅非魚和尼羅羅非魚的親緣關系最近,其次是與莫桑比克羅非魚和紅羅非魚,而與泰奧羅非魚和奧尼羅非魚親緣關系最遠。此結果與群體的來源和種屬關系相符,也說明本實驗所選用的微衛星位點可用于分析6個羅非魚群體間的親緣關系。
[1]符思嘉.海南省羅非魚產業現狀分析[J].現代經濟信息,2014,(14):475.
[2]Rutten M J M,Komen H,Deerenberg R M,et al.Genetic characterization of four strains of Nile tilapia(OreochromisniloticusL.)using microsatellite markers[J].Animal Genetics,2004,35(2):93-97.
[3]Khan S R,Akter H,Sultana N,et al.Genetic diversity in three river populations of the giant freshwater praw(Macrobrachiumrosenbergii)in Bangladesh assessed by microsatellite DNA markers[J].International Journal of Agriculture & Biology,2014,16(1):195-200.
[4]Kocher T,Lee W,Sobolewska H,et al.A genetic linkage map of a cichlid fish,the tilapia(Oreochromisniloticus)[J].Genetics,1998,148(3):1225-1232.
[5]Xia J H,Liu F,Zhu Z Y,et al.A consensus linkage map of the grass carp(Ctenopharyngodoidella)based on microsatellites and SNPs[J].BMC Genomics,2010,11:135.
[6]Guyon R,Rakotomanga M,Azzouzi N,et al.A high- resolution map of the Nile tilapia genome:a resource for studying cichlids and other percomorphs[J].Bmc Genomics,2012,13(1):222.
[8]強俊,楊弘,馬昕羽,等.吉富羅非魚與奧利亞羅非魚自繁與雜交F1遺傳特性與抗病力分析[J].水產學報,2015,39(1):32-41.
[9]李建林,李紅霞,唐永凱,等.利用微衛星標記分析兩個吉富羅非魚群體的遺傳差異[J].南方農業學報,2015,46(1):138-143.
[10]Nei M.Estimation of average heterozygosity and genetic distance from a small number of individuals[J].Genetics,1978,89(3):583-590.
[11]Dewoody J A,Avise J C.Microsatellite variation in marine,freshwater and anadromous fishes compared with other animals[J].Journal of Fish Biology,2000,56(3):461-473.
[12]戰愛斌,包振民,陸維,等.仿刺參的微衛星標記[J].水產學報,2006,30(2):192-196.
[13]魯雙慶,劉臻,劉紅玉,等.鯽魚4群體基因組DNA遺傳多樣性及親緣關系的微衛星分析[J].中國水產科學,2005,12(4):371-376.
[14]梁利群,常玉梅,董崇智,等.微衛星DNA標記對烏蘇里江哲羅魚遺傳多樣性的分析[J].水產學報,2004,28(3):241-244.
[15]張庭,盧邁新,葉星,等.四個奧利亞羅非魚群體的微衛星分析[J].水生生物學報,2009,33(3):498-508.

