2種活菌飼料對金鯽幼魚腸道及水體微生態的影響
馬小康,吳小嫚,胡樂琴
( 上海海洋大學,水產科學國家級實驗教學示范中心,農業部淡水產種質資源重點實驗室, 農業部魚類營養與環境生態研究中心,上海 201306 )
魚類腸道含有大量微生物,部分微生物接受宿主腸道環境及本身遺傳特征等因素的定向選擇[1]而定殖下來,成為腸道固有菌群,與宿主及外界環境之間形成一個相對穩定的微生態系統[2];其余微生物變化是動態的,隨著魚類的生長階段、生活水環境及餌料組成等變化而發生改變[3]。研究表明,魚類腸道菌群結構與數量變化會直接影響魚體的營養與免疫,特別是對外源致病菌的拮抗競爭[4-5],而菌群失衡更會導致機體免疫下降及疾病爆發等[6],對魚類腸道菌群進行更好的調控和維持顯得極為重要。
目前水產養殖業多為集約化和高密度養殖模式,加上抗生素的濫用,對魚體腸道菌群穩態造成極大負擔,而益生菌作為一類通過改善宿主腸道微生態平衡而發揮有益作用的活菌物質,能夠改善腸道菌群結構,調節由抗生素或其他因素造成的腸道菌群紊亂,幫助恢復穩態[7-8]。在飼料中施用益生菌作為一種有效方法在水產養殖中廣泛應用[9],但多采用在飼料中直接添加益生菌,用益生菌發酵飼料較少。飼料發酵后除益生菌外,還有代謝產物[10],對畜禽類腸道形態及菌群結構有明顯改善[11],但在水生生物中應用稀少。筆者以金鯽(Carassiusauratusred var.)幼魚為試驗對象,采用16S rDNA高通量測序手段,一方面,利用復合益生菌制備2種活菌飼料,研究2種活菌飼料對金鯽幼魚腸道及養殖水體菌群結構的影響,鑒定核心菌群變化,為益生菌在魚類飼料應用中提供參考;另一方面,研究飼料、養殖水體及腸道三者菌群進化關系,分析腸道菌群關鍵影響因子,為微生態調控研究提供一定幫助。
1 材料與方法
1.1 活菌飼料制備
試驗所用菌種枯草芽孢桿菌(Bacillussubtilis)、嗜酸乳桿菌(Lactobacillusacidophilus)購自廣東微生物菌種保藏中心,分別進行菌種活化和小規模發酵培養,離心收集菌體,用無菌水制成菌懸液,1∶1比例混合備用。基礎飼料配方為魚粉40%,小麥粉20%,豆粕20%,玉米粉10%,麥麩6%,魚油2%,預混料2%。原料粉碎后過60目篩按配方比例稱量質量混勻,逐級擴大法添加微量組分,制備完成后分為3份:第1份用無菌水調節原料含水量40%,記為基礎飼料A1;第2份按3%比例加入復合菌懸液,用無菌水調節原料含水量40%(水和菌液),記為活菌飼料B1;第3份同樣按3%比例加入復合菌懸液,用無菌水調節原料含水量40%(水和菌液),以此為發酵底物裝入帶發酵呼吸膜袋密封,固體發酵3 d,溫度30 ℃,記為活菌發酵飼料C1。所有濕基飼料均通過小型手搖顆粒機制粒,陰干后4 ℃保存,取0.5 g飼料經無菌生理鹽水適當稀釋,取一定量稀釋液涂布到平板上,培養后統計菌落數,確保飼料中枯草芽孢桿菌和嗜酸乳桿菌活菌含量達到108cfu/g。
1.2 試驗魚及分組
試驗所用金鯽幼魚取自上海浦東觀賞魚養殖基地同一批魚種,平均體質量為(8.49±0.26) g。試驗魚暫養2周后隨機挑選規格整齊、體質健壯的魚270尾,分成3組,每組3個重復(每個重復30尾),放入圓形循環水桶(養殖水體積200 L)中,投喂3種飼料,即基礎飼料(基礎組A)、活菌飼料(活菌組B)和活菌發酵飼料(發酵組C)。試驗周期為8周,日投喂2次,日投飼量為魚體質量的2%(根據魚的攝食情況進行調節)。水源為曝氣自來水,水溫20~25 ℃,pH 7.4~8.0,溶解氧≥6 mg/L,單循環水充氣,定期排污。
1.3 樣品采集與處理
1.3.1 生長狀況測定
試驗前后,分別對基礎組和試驗組采樣,每組取30尾,計算平均體質量。
1.3.2 測序樣品采集與處理
試驗開始前,采集15尾魚腸道混合樣品,標記CK。試驗結束禁食后,分別對基礎組和試驗組采樣,每組采集5 g飼料,分別標記A1、B1、C1;3 L混合水樣,分別標記A2、B2、C2;15尾魚腸道混合樣品,分別標記A3、B3、C3。處理時稱取1 g飼料研缽磨碎,置于15 mL離心管中,加入1~3 mL TE緩沖液穩定細菌DNA;在水桶中心離水面15 cm處取水樣,用0.22 μm濾膜過濾,剪碎濾膜置于15 mL離心管中,加入1~3 mL TE緩沖液備用;在無菌操作臺中用75%酒精擦洗魚體表面,解剖魚體,用75%酒精棉球擦拭腸道外壁,剔除腸道外脂肪,分離出腸道,無菌生理鹽水沖洗數遍后置于研磨管中,加入1~3 mL TE緩沖液研磨至勻漿液,轉入15 mL離心管中備用。
1.4 樣品細菌總DNA提取
樣品細菌基因組總DNA選用OMEGA試劑盒E.Z.N.ATM Mag-Bind Soil DNA Kit提取,嚴格按照說明書操作,所有樣品總DNA均重復提取2次以上并混合在一起,以避免偏差。用1%瓊脂糖凝膠電泳檢測DNA完整性。利用Qubit 2.0 DNA檢測試劑盒對基因組DNA精確定量,確定PCR反應DNA量。PCR引物為融合Miseq測序平臺的V3~V4通用引物341F:CCCTACACGACGCTC-TTCCGATCTG(barcode)CCTACGGGNGGCWG-CAG,805R引物:GACTGGAGTTCCTTGGCACCCGAGAATTCCAGACT ACHVGGGTATCTAATCC,反應體系為2×Taq master Mix 15 μL,Bar-PCR primer F(1 μmol/L) 1 μL,Primer R (10 μmol/L) 1 μL,Genomic DNA 10~20 ng,H2O 30 μL。擴增體系為94 ℃預變性3 min,94 ℃變性30 s,69 ℃退火30 s,72 ℃延伸50 s,共30個循環,72 ℃延伸10 min。PCR結束后進行第2輪擴增,引入Illumina橋式PCR兼容引物,體系如上。每個樣品3個重復,將同一樣品PCR產物混合后進行瓊脂糖電泳檢測后純化回收。每個樣品DNA量取10 ng,按1∶1等量混合后測序。測序工作由生工生物工程(上海股份有限公司)完成,測序平臺為Illumina MiSeq測序系統。
1.5 數據處理與分析
用SPSS 19.0對生長數據進行計算,運用LSD法進行單因素方差分析,P<0.05表示顯著差異。菌群分析中,將樣本序列聚類,基于可操作分類單元在97%相似水平下進行Alpha多樣性分析,計算香儂指數、辛普森指數、物種豐富度指數及覆蓋率。采用RDP classifier軟件對樣品菌群門、屬進行分類,計算樣品群落結構比例。除去未分類的,篩選相對豐度大于0.5%的優勢菌屬,費希爾精確檢驗菌屬豐度差異顯著性(P<0.05)。通過UniFrac加權距離矩陣分析樣品Beta多樣性,進行進化樹分析、熱度圖分析和主成分分析。
2 結 果
2.1 2種活菌飼料對生長性能的影響
投喂2種活菌飼料8周后金鯽幼魚生長情況見表1。由表1可見,攝食活菌飼料(B)的魚,質量增加率顯著提高5.15%(P<0.05);攝食活菌發酵飼料(C)的魚,質量增加率顯著降低3.44%(P<0.05)。
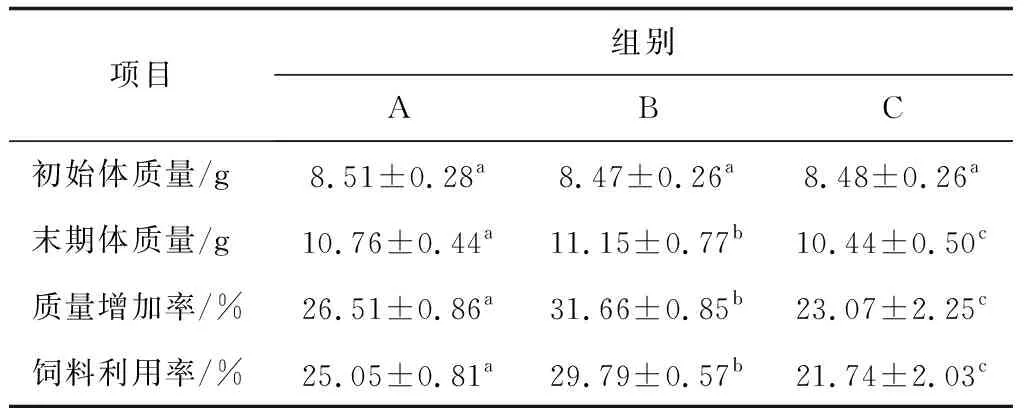
表1 活菌飼料對金鯽生長的影響
注:表中同行肩標不同小寫字母表示差異顯著(P<0.05).
2.2 2種活菌飼料對菌群多樣性指數的影響
在97%水平上對所有樣品進行Alpha多樣性分析(表2),與A組相比,試驗組對應樣品菌群物種豐度均出現不同程度下降,C組較明顯;B組飼料菌群多樣性變低,水體無明顯變化,腸道多樣性變高;C組3個樣品菌群多樣性均出現明顯降低。說明試驗組樣品菌群物種豐度和多樣性出現不同變化,2種活菌飼料均對腸道及水體菌群結構產生一定影響,C組飼料影響較大。
2.3 2種活菌飼料對菌群結構組成的影響
2.3.1 飼料菌群結構
飼料樣品中優勢菌門為變形菌門、厚壁菌門、擬桿菌門和放線菌門(表3),優勢菌屬(相對豐度>0.5%,下同)17種(圖1),主要為芽孢桿菌屬、立克次體屬(Rickettsia)、不動桿菌屬(Acinetobacter)、假單胞菌屬(Pseudomonas)及乳球菌屬(Lactococcus)等。與A1相比,B1、C1中嗜酸乳桿菌和枯草芽孢桿菌大量繁殖,菌屬相對豐度顯著增加(P<0.05),立克次體屬、不動桿菌屬及假單胞菌屬等相對豐度顯著降低(P<0.05),菌群結構明顯變化,3組飼料樣品菌群結構差異顯著。
2.3.2 養殖水體菌群結構
水體樣本中優勢菌門為變形菌門、擬桿菌門、放線菌門、厚壁菌門和梭桿菌門(表3),優勢菌屬21種(圖2),主要為多核桿菌屬(Polynucleobacter)、微小桿菌屬(Exiguobacterium)、黃桿菌屬(Flavobacterium)、假單胞菌屬、檸檬酸桿菌屬(Citrobacter)、不動桿菌屬及鯨桿菌屬(Cetobacterium)等。與A2相比,B2、C2中多核桿菌屬、微小桿菌屬等相對豐度明顯增加(P<0.05),黃桿菌屬等相對豐度明顯降低(P<0.05),其他菌屬相對豐度也出現不同變化,其中B2主要是物種豐度降低,如黃桿菌屬、分枝桿菌屬(Mycobacterium)、擬桿菌屬(Bacteroides)等幾乎消失,多樣性無明顯變化;而C2中物種豐度和多樣性均明顯降低,菌群結構得到簡化。
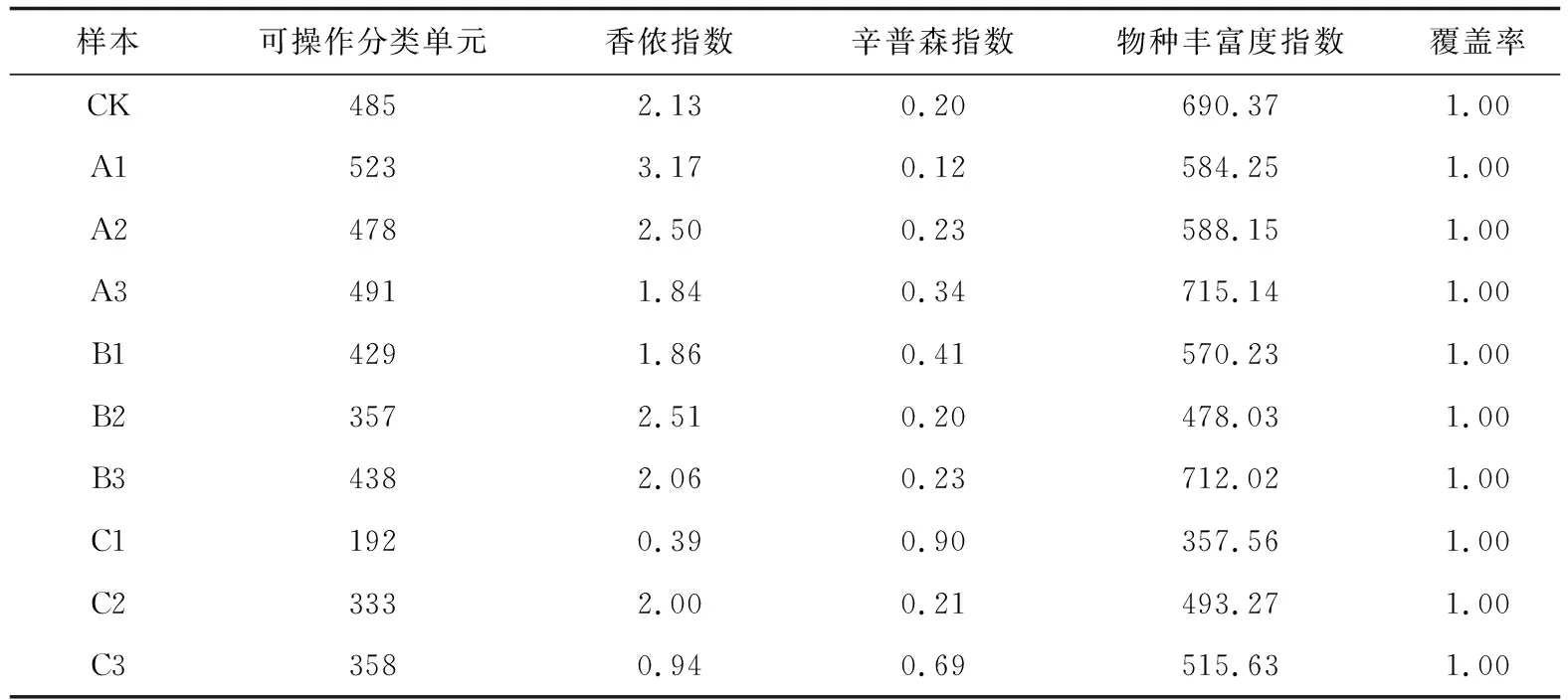
表2 金鯽幼魚腸道、飼料及水體樣品菌群多樣性指數
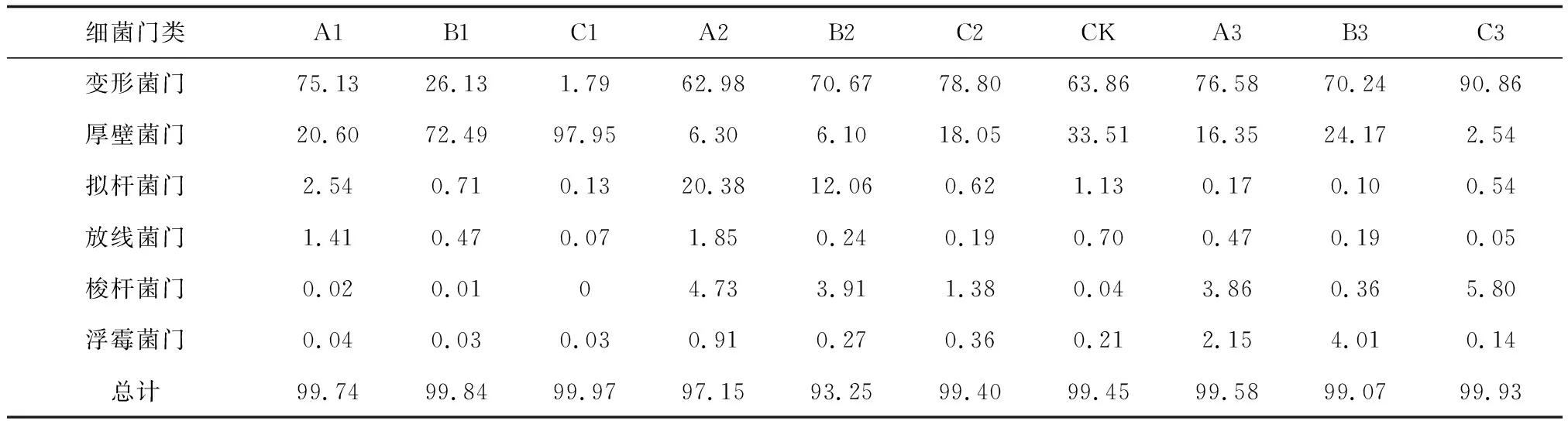
表3 金鯽幼魚腸道、飼料及水體優勢細菌門類及相對豐度 %
2.3.3 腸道菌群結構
腸道樣品中優勢菌門為變形菌門、厚壁菌門、擬桿菌門、浮霉菌門及梭桿菌門(表3),優勢菌屬14種(圖3),主要為氣單胞菌屬、微小桿菌屬、假單胞菌屬、檸檬酸桿菌屬、不動桿菌屬、鯨桿菌屬等。與CK相比,A3、B3及C3均發生不同變化,說明腸道菌群結構因飼料和水體菌群的不同而發生改變。與A3相比,B3中氣單胞菌屬相對豐度明顯降低(P<0.05),其他菌屬如微小桿菌屬、假單胞菌屬等相對豐度顯著增加(P<0.05),菌群多樣性升高,物種豐度無明顯變化;C3中假單胞菌屬、不動桿菌屬等幾乎消失,氣單胞菌屬、微小桿菌屬及檸檬酸桿菌屬等優勢地位得到加強,物種豐度和多樣性均明顯降低,菌群結構趨于簡化。
2.4 菌群進化關系分析
A組中CK與A3聚類,A1與A2聚類;B組中CK與B3聚類,B1與B2聚類;C組中C2與CK先聚類,之后依次與C1、C3聚類。A、B組中水體與飼料關系最近,C組中水體與初始腸道關系最近,與飼料及末期腸道關系較遠(圖4)。
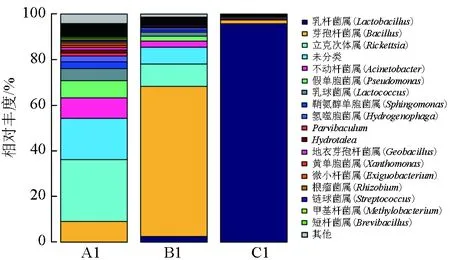
圖1 飼料樣品中屬水平細菌種群結構及分布
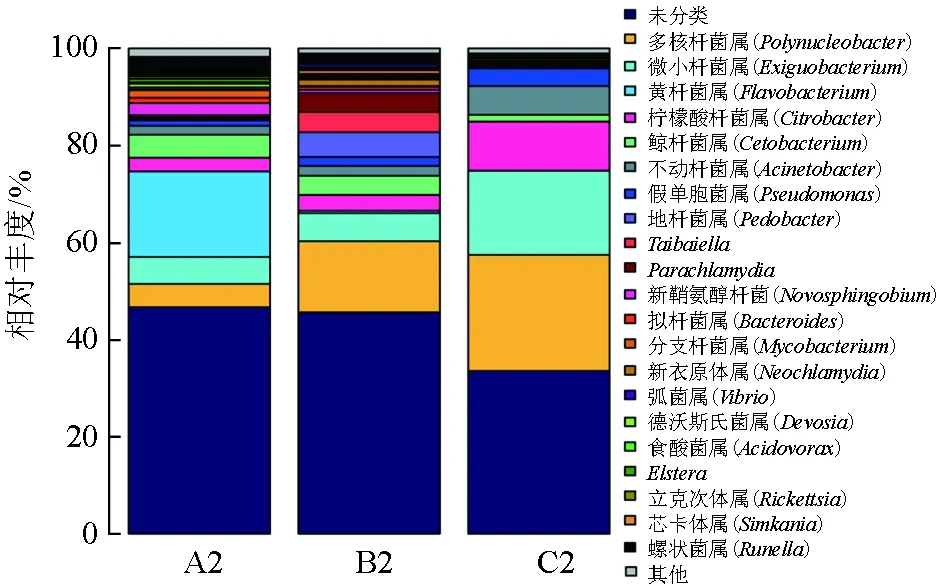
圖2 水體樣品中屬水平細菌種群結構及分布
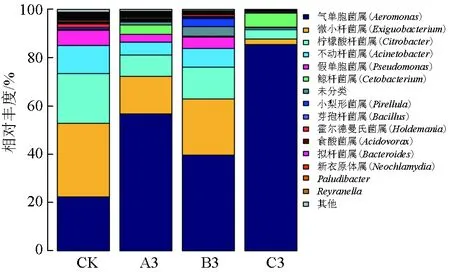
圖3 金鯽幼魚腸道樣品中屬水平細菌種群結構及分布

圖4 各組樣品之間聚類圖
A2、B2、C2顏色偏紅,與其他樣品顏色差別明顯,進化樹距離最短,說明不同組相同樣品中水體菌群之間相似性最高(圖5)。之后距離由近到遠依次為A1、B1、C1(顏色偏黃)、CK、B3、A3、C3(顏色偏藍),說明與水體距離由近到遠依次為飼料、腸道,但顏色顯示相似性均不高。圖中腸道與飼料距離較近,且顏色無明顯差別,說明腸道與飼料菌群間相似性較高。
同組不同樣品之間,腸道距離飼料較近,距離水樣較遠,說明腸道與飼料菌群主成分相似性比水體高(圖6)。綜合上述菌群結構與分布變化,B1、C1遠離A1,不在同一聚集區域,說明與A1相比,B1主成分發生變化,主要為菌群多樣性降低,物種豐度無明顯變化;C1距離A1較遠,說明C1主成分明顯變化,主要為菌群物種豐度和多樣性同時降低。同理,與A2相比,B2主要為菌群物種豐度降低,多樣性無明顯變化;C2主要為菌群物種豐度和多樣性共同降低。圖中CK、A3、B3及C3不同程度分散開來,與CK相比,其余腸道樣品主成分均發生變化。同理,與A3相比,B3主要為菌群多樣性增加,物種豐度無明顯變化;而C3偏離腸道樣品聚集區域,主要為菌群物種豐度和多樣性明顯降低。比較不同組相同樣品間距離長短,以物種豐度和多樣性為樣本菌群兩種變化水平,發現與A組相比,B組3個樣品均只發生單一水平明顯變化;而C組3個樣品均發生兩種水平明顯變化,說明同一微生態中飼料、水體及腸道三者菌群變化可能存在動態關聯。
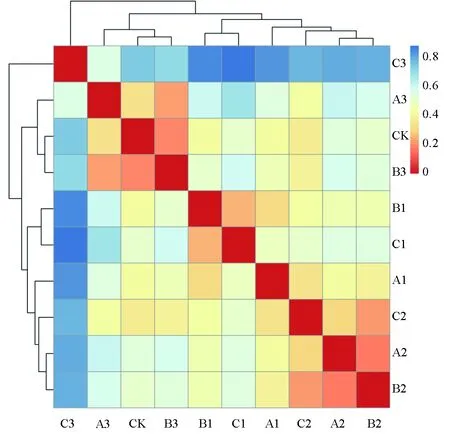
圖5 金鯽幼魚腸道、飼料及水體間距離熱度顏色塊代表距離值,顏色越紅表示樣本間距離越近,越藍則表示距離越遠.
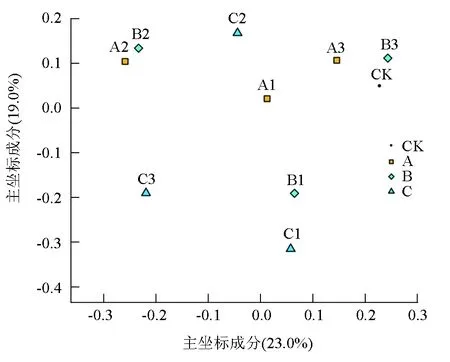
圖6 金鯽幼魚腸道、飼料及水體主成分分析
3 討 論
3.1 飼料菌群多樣性變化
芽孢桿菌和乳酸菌是水產養殖中應用較廣泛的微生態制劑菌屬,其中應用最多的有枯草芽孢桿菌和嗜酸乳桿菌,兩種菌促進魚類生長的研究較多[12-13],但將兩種菌一起復配的研究較少。本研究中試驗組飼料菌群結構發生明顯變化,這與兩種菌的加入和對其他細菌的競爭抑制有直接關系,其中活菌發酵飼料菌群物種豐度和多樣性明顯降低,芽孢桿菌屬與乳桿菌屬占絕對優勢地位,總量高達97.38%,這與飼料經過厭氧發酵有關,一方面枯草芽孢桿菌與嗜酸乳桿菌協同性高,大量繁殖創造厭氧酸環境,抑制其他細菌生長;另一方面代謝產物如乳酸能抑制和殺死一些細菌。本試驗中立克次體屬、不動桿菌屬及假單胞菌屬等相對豐度明顯變低,原因可能與此有關。立克次體屬是一類專性細胞內寄生的人畜共患病原菌屬[14],假單胞菌屬中許多菌株在水產養殖中被證實和病原菌有關[15-16],不動桿菌屬中多為病原菌[17],其中鮑氏不動桿菌(Acinetobacterbaumannii)曾導致鱖魚(Sinipercachuatsi)爆發性死亡[18];而嗜酸乳桿菌及枯草芽孢桿菌均為水產益生菌[19]。因此,益生菌處理一定程度上提高了飼料生態質量。本試驗中,活菌飼料促進了魚體生長,而活菌發酵飼料卻產生抑制效果,有研究[20]顯示,發酵飼料添加量控制在一定范圍內才會促進水產動物生長,添加量較高時,大部分水產動物生長都會受到一定程度的抑制,原因可能是發酵組全部使用發酵飼料投喂,導致腸道菌群多樣性顯著降低。
3.2 養殖水體菌群多樣性變化
養殖末期,試驗組水體中多核桿菌屬和微小桿菌屬等相對豐度明顯增加,黃桿菌屬等相對豐度明顯降低,其中黃桿菌屬是柱狀病、白頭白口病的病原體[21]。一方面可能與益生菌的抑菌作用有關,另一方面可能與益生菌對有機物的分解有關,增加了水體中無機氮磷含量,抑制了部分異養菌。與基礎組相比,活菌組水體菌群多樣性未發生明顯變化,主要是物種豐度減少,這與部分研究結果一致,直接施菌對水體菌群多樣性改變有限[22-23];而發酵組水體菌群物種豐度和多樣性均明顯降低,這可能與飼料經過發酵有關。目前施菌會明顯降低水體菌群多樣性的研究僅見于蛭弧菌(Bdellovibrio)[24],蛭弧菌具有較寬的裂解譜,一方面能大量裂解病原菌,甚至能攻擊產生耐藥性的致病菌株[25];另一方面,蛭弧菌能穿透生物膜對深藏在生物膜中的致病菌進行攻擊,克服抗生素不能有效控制生物膜內致病菌的缺點,高效控制病原菌的繁殖[26],推測活菌發酵飼料可能具有類似于蛭弧菌的控菌作用,能同時對水體菌群物種豐度和多樣性產生影響。
3.3 腸道菌群多樣性變化
在魚類腸道菌群研究中,多數學者將腸道內容物作為研究對象,對定殖在腸道黏膜層上的菌群研究較少。其實,腸道黏膜層與內容物菌群結構之間有較大差異,腸道黏膜層上的菌群結構較穩定,應作為腸道菌群研究的重點對象[27]。本研究對整個腸道菌群測序結果顯示,變形菌門和厚壁菌門在金鯽幼魚腸道中占絕對優勢地位,為常駐菌門。研究顯示厚壁菌門在腸道中主要是對碳水化合物和蛋白質的水解[28],與魚類營養吸收關系密切,值得深入研究。本試驗中金鯽幼魚腸道菌群結構因飼料和水體菌群的不同而發生改變,但飼料與水體優勢菌群、甚至兩種益生菌均未在腸道中大量出現,進一步說明魚類腸道菌群的定殖是基于宿主的定向選擇,不是隨機遷入的。與基礎組相比,養殖末期活菌組魚腸道菌群中氣單胞菌屬相對豐度明顯降低,部分菌屬相對豐度變高,物種豐度無明顯變化,推測活菌飼料能促進魚腸道菌群均勻度增加,提高多樣性,進而促進魚體生長。養殖末期發酵組腸道菌群中假單胞菌屬、不動桿菌屬等相對豐度明顯變低,優勢菌屬變為氣單胞菌屬、鯨桿菌屬、檸檬酸桿菌屬、微小桿菌屬等。有研究表明氣單胞菌屬是淡水魚類腸道有益菌[19];鯨桿菌屬能夠發酵多肽、碳水化合物,還能產生維生素B12,對營養吸收有重要作用[29];檸檬酸桿菌屬是一類纖維素降解菌屬[30],而微小桿菌屬是一類重要的產蛋白酶菌屬[31],推測活菌發酵飼料能增強部分腸道有益菌豐度優勢,一定程度上簡化腸道菌群結構。
3.4 飼料、水體及腸道菌群進化分析
本研究發現水體菌群結構受飼料和腸道菌群的一定影響,與飼料關系較近;而腸道菌群結構明顯受飼料菌群的影響,關系密切,這與Moran等[32]結論相一致,認為魚類腸道菌群結構與宿主對食物的選擇密切相關。而Romero等[33]卻認為水環境對魚類腸道菌群定殖起到關鍵作用,這可能與魚種、地域及環境因子的不同有關。本試驗條件下水環境受外界因素干擾較少,且無底泥影響,養殖環境相對穩定,金鯽幼魚腸道菌群與飼料關系密切,水體次之,同時飼料、水體及腸道三者菌群變化可能存在動態關聯,如試驗組飼料菌群發生明顯變化,造成腸道和水體菌群發生相同水平明顯變化,相互調節達到整體動態平衡,這可能為微生態調控研究提供一定幫助。
利用嗜酸乳桿菌和枯草芽孢桿菌對基礎飼料進行不同處理得到兩種活菌飼料,一定程度上提高了飼料生態質量。其中活菌飼料對金鯽幼魚腸道及水體菌群結構產生一定影響,提高了魚體生長性能;而活菌發酵飼料對金鯽幼魚腸道及水體菌群結構影響較顯著,但抑制了魚體生長,這可能與發酵飼料的使用比例有關,有待于進一步研究。
[1] Yan Q Y, Li J J, Yu Y H, et al. Environmental filtering decreases with fish development for the assembly of gut microbiota[J]. Environmental Microbiology, 2016, 18(12):4739-4754.
[2] Spanggaard B, Huber I, Nielsen J, et al. The microflora of rainbow trout intestine: a comparison of traditional and molecular identification[J]. Aquaculture, 2000, 182(1/2):1-15.
[3] Zhang M, Liu N, Qian C, et al. Phylogenetic and functional analysis of gut microbiota of a fungus-growing higher termite:Bacteroidetesfrom higher termites are a rich source β-glucosidase genes[J]. Microbial Ecology, 2014, 68(2):416-425.
[4] Fujimura K E, Slusher N A, Cabana M D et al. Role of the gut microbiota in defining human health[J]. Expert Review of Anti-Infective Therapy, 2010, 8(4):435-454.
[5] 李艷宇, 任盟, 張叢堯, 等. 紅鰭東方鲀稚魚腸道可培養細菌的多樣性[J]. 水產科學, 2015, 34(10):652-656.
[6] Dimitroglou A, Merrifield D L, Carnevali O, et al. Microbial manipulations to improve fish health and production—a mediterranean perspective[J]. Fish & Shellfish Immune, 2011, 30(1):1-16.
[7] Gomez-Gil B, Roque A, Turnbull J F. The use and selection of probiotic bacteria for use in the culture of larval aquatic organisms[J]. Aquaculture, 2000, 191(1/3):259-270.
[8] 張雯, 鐘雷, 李南充, 等. 微生態制劑對池養建鯉體成分、血清指標、消化酶活性以及腸道菌群組成的影響[J]. 水產科學, 2015, 34(12):769-777.
[9] Verschuere L, Rombaut G, Sorgeloos P, et al. Probiotic bacteria as biological control agents in aquaculture[J]. Microbiology and Molecular Biology Reviews, 2000, 64(4):655-671.
[10] Niba A, Beal J, Kudi A, et al. Bacterial fermentation in the gastrointestinal tract of non-ruminants: influence of fermented feeds and fermentable carbohydrates[J]. Tropical Animal Health and Production, 2009, 41(7):1393-1407.
[11] 張俊, 朱建津, 劉江英, 等. 益生菌發酵產物對肉雞腸道形態和腸道屏障功能的影響[J]. 浙江農業學報, 2016, 28(10):1657-1662.
[12] 程遠, 黃凱, 黃秀蕓, 等. 飼料中添加枯草芽孢桿菌對吉富羅非魚幼魚生長性能、免疫力和抗氧化功能的影響[J]. 動物營養學報, 2014, 26(6):1503-1512.
[13] 張羽. 乳酸桿菌對斑馬魚免疫力和繁殖性能的影響[D]. 楊凌:西北農林科技大學,2013.
[14] Raoult D, Roux V. Rickettsioses as paradigms of new or emerging infectious diseases[J]. Clinical Microbiology Reviews, 1997, 10(4):694-719.
[15] Driscoll J A, Brody S L, Kollef M H. The epidemiology, pathogenesis and treatment ofPseudomonasaeruginosainfections[J]. Drugs, 2007, 67(3):351-368.
[16] Cai J, Lin S, Wu B. Characterization ofPseudomonasaeruginosaassociated with diseased postlarval abalone in Shenzhen, China[J]. Aquaculture International, 2009, 17(5):449-458.
[17] Bergogne B E, Towner K J.Acinetobacterspp.as nosocomical pathogens: microbiological, clinical, and enidemiological features[J]. Clinical Microbiology Reviews, 1996, 9(2):148-165.
[18] 湯雅殊, 陸承平. 動物源不動桿菌的藥敏試驗[J]. 南京農業大學學報, 1997, 20(3):120-123.
[19] Nayak S K. Role of gastrointestinal microbiota in fish[J]. Aquaculture Research, 2010, 41(11):1553-1573.
[20] Refstie S, Sahlstrom S, Brathen E et al. Lactic acid fermentation eliminates indigestible carbohydrates and antinutritional factors in soybean meal for Atlantic salmon (Salmosalary)[J]. Aquaculture, 2005, 246(1/4):331-345.
[21] 李存玉, 徐永江, 柳學周, 等. 池塘和工廠化養殖牙鲆腸道菌群結構的比較分析[J]. 水產學報, 2015, 39(2):245-255.
[22] Wu D X, Zhao S M, Peng N, et al. Effects of a probiotic (BacillussubtilisFY99-01)on the bacterial community structure and composition of shrimp (Litopenaeusvannamei, Boone) culture water assessed by denaturing gradient gel electrophoresis and high-throughput sequencing[J]. Aquaculture Research, 2016, 47(3):857-869.
[23] Tamminen M, Karkman A, Corander J, et al. Differences in bacterial community composition in Baltic Sea sediment in response to fish farming[J]. Aquaculture, 2011, 313(1):15-23.
[24] Li H, Chen C, Sun Q, et al.Bdellovibrioand like organisms enhanced growth and survival ofPenaeusmonodonand altered bacterial community structures in its rearing water[J]. Applied and Environmental Microbiology, 2014, 80(20):6346-6354.
[25] Dwidar M, Monnappa A K, Mitchell R J. The dual probiotic and antibiotic nature ofBdellovibriobacteriovorus[J]. Bmb Reports, 2012, 45(2):71-78.
[26] Kadouri D, O′Toole G A. Susceptibility of biofilms toBdellovibriobacteriovorusattack[J]. Applied and Environmental Microbiology, 2005, 71(7):4044-4051.
[27] 周志剛, 石鵬君, 姚斌, 等. 基于PCR-DGGE指紋圖譜川紋笛鯛及圓白鯧消化道壁優勢菌群結構比較分析[J]. 水生生物學報, 2007, 31(5):682-688.
[28] Backhed F, Ley R E, Sonnenburg J L, et al. Host-bacterial mutualism in the human intestine[J]. Science, 2005, 307(5717):1915-1920.
[29] Tsuchiya C, Sakata T, Sugita H. Novel ecological niche ofCetobacteriumsomerae, an anaerobic bacterium in the intestinal tracts of freshwater fish[J]. Letters in Applied Microbiology, 2008, 46(1):43-48.
[30] Wu S, Wang G, Angert E R, et al. Composition, diversity, and origin of the bacterial community in grass carp intestine[J]. PloS One, 2012, 7(2):e30440.
[31] Kasana R C, Yadav S K. Isolation of a psychrotrophicExiguobacteriumsp. SKPB5 (MTCC7803) and characterization of its alkaline protease[J]. Current Microbiology, 2007, 54(3):224-229.
[32] Moran D, Turner S J, Clements K D. Ontogenetic development of the gastrointestinal microbiota in the marine herbivorous fishKyphosussydneyanu[J]. Microbial Ecology, 2005, 49(4):590-597.
[33] Romero J, Navarrete P. 16S rDNA-based analysis of dominant bacterial populations associated with early life stages of coho salmon (Oncorhynchuskisutch)[J]. Microbial Ecology, 2006, 51(4):422-430.

