DNA條形碼在廈門灣魚卵和仔稚魚分類鑒定中的應用
林君卓,吳 昊,陳 凱,杜慶紅,黃智偉,黃 昆,王鑫煌,陳 然,鄭連明
(1.福建海洋研究所,福建 廈門 361013; 2.福建省海陸界面生態環境重點實驗室,福建 廈門 361102)
魚卵、仔稚魚的數量將直接影響到魚類補充群體資源量的豐歉,因此,魚卵、仔稚魚具有非常重要的生態地位和研究價值。但魚卵、仔稚魚的準確鑒定是魚類浮游生物多樣性與生態學研究長期以來的瓶頸。我國已知海洋魚類已超過3000 種,約占世界海洋魚類種類的1/4,但是有資料可查的魚卵與仔稚魚種類不超過300 種[1]。作為種類甄別的形態特征較少,加上專業分類資料及人才的缺乏,使僅憑外部形態特征對魚卵、仔稚魚進行精確分類變得相當困難。
DNA條形碼(DNA barcoding)以其操作簡便性和高效性的特點,已經成為生物分類學中引人注目的新方向,越來越多的研究證明DNA條形碼是一個行之有效的生物種類鑒定手段[2]。全球共享的基因數據庫如DDBJ、ENBL、GenBank等所收藏魚類的各種基因及序列數量迅速增加,為魚卵、仔稚魚提供了大量可比對的成魚同源性DNA基因序列,研究者可以將DNA條形碼技術直接運用到魚卵、仔稚魚的鑒別中,保證了鑒定結果的準確性。本文以2017年4月廈門灣的魚卵、仔稚魚樣品為材料,采用形態學與DNA條形碼技術相結合的形式進行種類鑒定,并確定其形態特征,探索DNA條形碼在魚卵、仔稚魚樣品種類鑒定中的應用,旨在為魚卵、仔稚魚的鑒定提供參考。
1 材料與方法
1.1 調查海區及站位設置
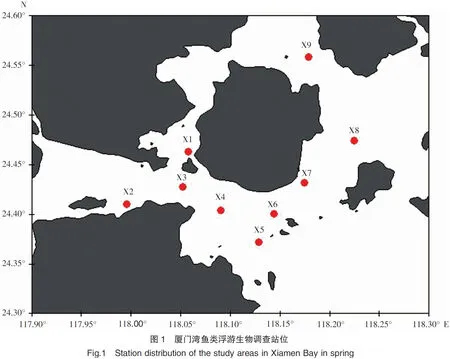
2017年4月(春季)于廈門灣及其鄰近海域(118°01′~118°13′E,24°22′~24°35′N)進行了魚類浮游生物調查。本次調查共設置9個站位,如圖1所示。
1.2 樣品采集與處理
魚類浮游生物海上采集根據《海洋調查規范 第6部分:海洋生物調查》(GB/T 12763.6—2007)進行。使用浮游生物淺水I型網(網口直徑50 cm,網長145 cm,網口面積0.2 m2,網衣篩絹孔徑0.505 mm)進行垂直樣品采集,使用浮游生物海水大型網(網口直徑80 cm,網長280 cm,網口面積0.5 m2,網衣篩絹孔徑0.505 mm)以2 kn的船速進行水平拖網10 min,采集表層樣品。所獲的網采樣品先保存于500 mL樣品瓶中,現場使用中性甲醛固定,加入量為樣品體積的5%,回實驗室后轉移到無水乙醇中避光保存。本研究僅分析各站位垂直拖網采集的定量樣品。
在實驗室中于Zeless Stemi 2000-C立體顯微鏡下進行魚類浮游生物的挑取和分離,參考各類魚類浮游生物的分類圖鑒[3-6],根據形態學特征將魚卵、仔稚魚先進行大致的分類并采集圖像,最后對挑選出的魚類浮游生物進行分子鑒定。
1.3 分子鑒定實驗方法
進行基因組DNA提取的魚卵與仔、稚魚,體長較小的仔魚取整尾,較大的稚魚取尾部或者背部組織;魚卵則取整粒。使用TaKaRa MiniBEST Universal Genomic DNA Extraction Kit Ver 5.0 試劑盒進行DNA提取,使用Ward等[7]針對魚類設計的FishCOI引物進行PCR擴增,引物序列如表1。

PCR反應體系(50 μL):10×buff 5 μL,Dntp 0.8 μL,FishF1與FishR1各0.5 μL,rTaq 0.3 μL,DNA 5.0 μL,滅菌超純水37.9 μL。
PCR程序為:94℃預變性5 min,94℃變性1 min,50℃退火50 s,72℃延伸1 min,循環30次,72℃延伸5 min,4℃保存。
PCR產物經0.8%瓊脂糖凝膠電泳(110 V,20 min)后,選取條帶明亮、無拖帶的樣品送往上海生工測序公司進行雙向測序。
1.4 數據處理
用Chromos 軟件對測序結果進行檢測,對序列兩端堿基峰型不合格的部分進行剪切,再將校正完畢的雙向序列用DNAMAN進行拼接,保留序列長度在600~700 bp之間。獲取序列在GenBank數據庫、FISH-BOL數據庫進行序列相似性比較,根據比對的結果來初步確定樣品所屬的魚類種類或魚類類群。在GenBank中收集所比對出的魚類種類同屬、同種的COI序列,使用MEGA 5.1利用鄰近法構建分子系統發育樹,基于Kimura-2-parameter 模型,采用N-J法(Neighbor joining method)構建系統進化樹來對比對結果進行驗證,最終確認魚卵與仔稚魚所屬的魚類種類或類群。
2 結果與分析
2.1 形態種初選
本研究在廈門灣共采集到魚卵44粒,仔稚魚31尾;經形態初步甄別,共分選出了5種形態的魚卵與5種形態的仔、稚魚。
2.2 測序結果
將經形態初選的5種魚卵樣品30份,5種仔稚魚樣品10份分別作分子鑒定實驗。由于部分樣未能成功提取基因組DNA,故無法擴增得到COI基因序列,最終獲得27條COI基因序列,其中魚卵樣品獲得19條序列,仔稚魚樣品獲得8條序列。序列長度為580 bp。
2.3 序列比對
本研究采用將遺傳相似度>99%的序列視為同一種類、90%~98% 為同一屬、80%~89% 為同一科的標準對廈門海域的魚卵、仔稚魚進行種類判定。經比對,實驗所獲序列與GenBank和FISH-BOL上的魚類基因相似度為99%~100%,且兩庫鑒定結果一致,共鑒定出了7種魚卵與5種仔稚魚,其中魚卵樣品比形態學分類鑒別的種類數多出了2種;所有樣品經分子鑒定,共計鑒定出11個種類,分別隸屬于鯡形目(Clupeiformes)、鱸形目(Perciformes)、鲉形目(Scorpaeniformes與鰈形目(Pleuronectiformes)(表2);其中,隸屬于鱸形目的魚類浮游生物種類數量最多,有6種,占所有魚類種類的55%,其次為鲉形目有2種,占所有種類的18%。
2.4 基于COI基因序列構建的N-J進化樹
根據獲得序列在GenBank和FISH-BOL數據庫中的比對結果,收集鯡形目的鯡科(Clupeidae),鱸形目的鯧科(Stromateidae)、鯛科(Sparidae)、鲾科(Leiognathidae)、石首魚科(Sciaenidae)、擬魚叚虎魚科亞科(Gobionellinae),鲉形目的毒鲉科(Synanceiidae)與鲬科(Platycephalidae),鰈形目的鰨科(Soleidae)等同屬同源序列,選取合適的序列作為外群,利用鄰接法構建NJ樹,如圖2所示。可以看出,各樣品序列與比對結果相似性最高的同源序列聚集成獨立分支,且在進化樹上基本都與其他同源序列聚集成科分類階元分支,說明其分子鑒定結果的有效性。
2.5 形態描述
采集經分子鑒定出的11種魚類浮游生物的高清形態圖(圖3),并參考相關文獻,對其甄別性的形態特征進行描述,詳見表3。
3 討論
3.1 DNA條形碼技術在魚類浮游生物鑒定中的適用性及局限性
早在2002年,Tautz等首先提出可以運用DNA序列的方法來進行物種鑒定和分類,隨后Hebert等[2]提出了基因條形碼的概念,同時指出線粒體細胞色素氧化酶亞基I基因中的一個片段可以有效地鑒別物種,并認為全球的生物均可進行條形編碼。這項技術很快在形態學方法遭遇瓶頸的魚卵仔稚魚分類鑒定領域得到應用。卞曉東等[8]采用mtCOI序列分析方法鑒定了在黃海海區海藻上采集到的魚卵,并最終確定為沙氏下鱵魚(Hyporhamphussajori)魚卵,而非魚卵形態相近、產卵時間、海域相同的太平洋頜針魚(Strongyluraanastomella)魚卵。Ko 等[9]采用DNA條形碼技術對臺灣100 種仔稚魚的分析中,將遺傳相似度>99%的兩個物種認定為同一種,遺傳相似度在92%~99% 之間的為同一屬物種、遺傳相似度在84%~91%之間的為同一科物種。
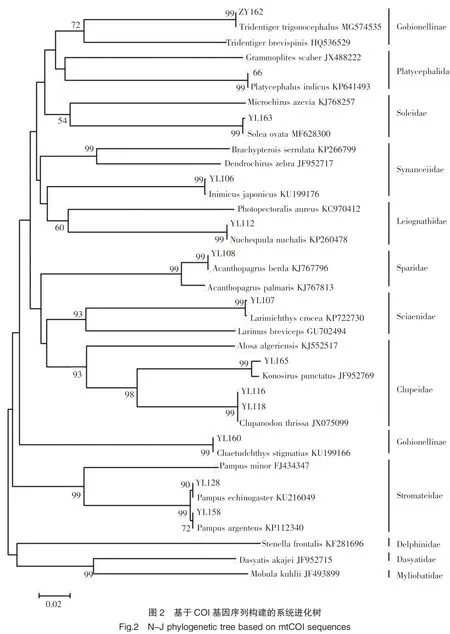

表 2 11種魚類浮游生物的COI 基因信息
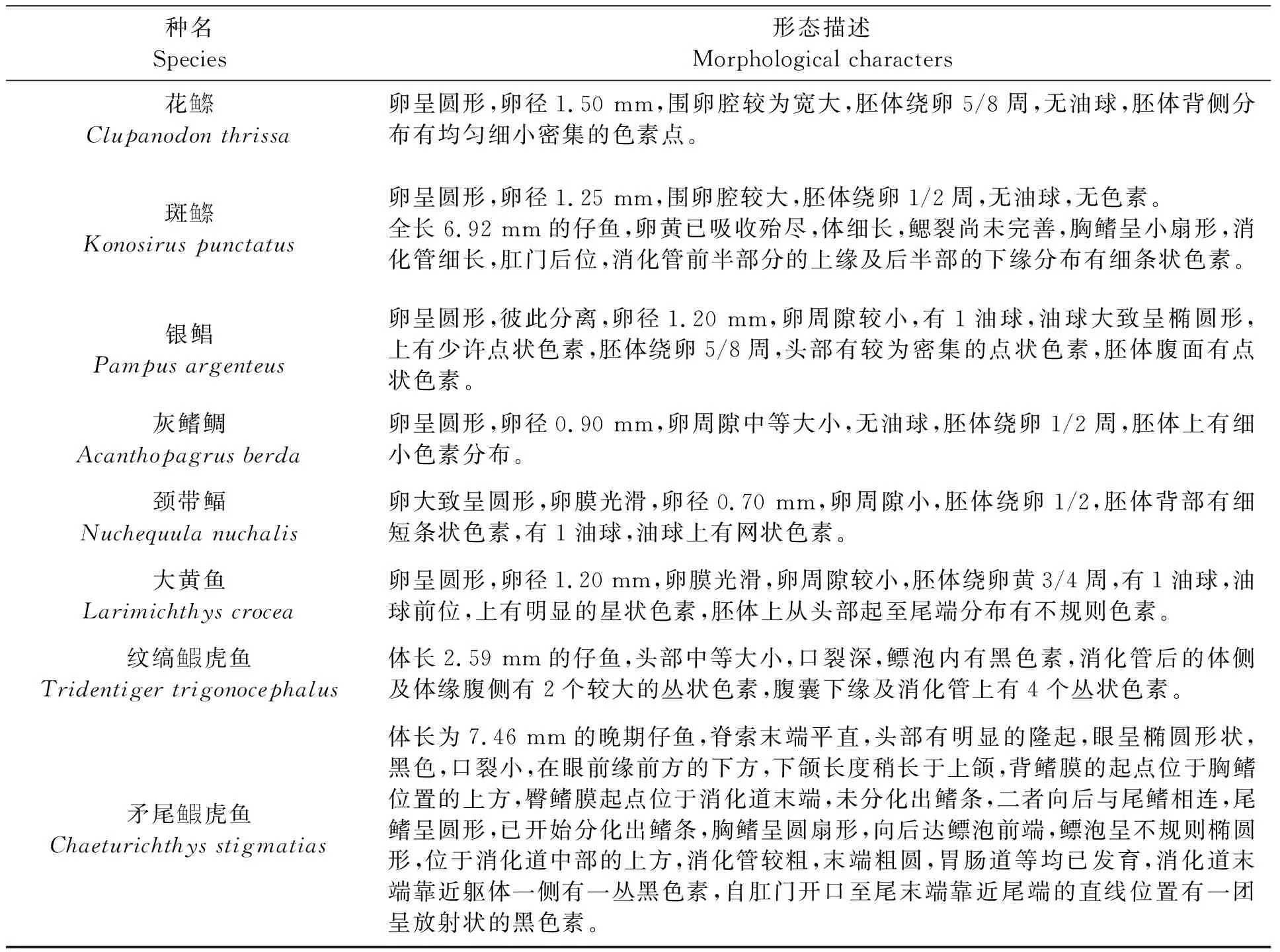
表3 魚類浮游生物形態描述

續表3

本研究采用將遺傳相似度>99%的序列視為同一種類、90%~98% 為同一屬、80%~89% 為同一科的標準對廈門海域的魚卵、仔稚魚進行種類判定。遵循以上標準,將本研究在廈門灣采集并獲得的魚卵、仔稚魚的DNA條形碼序列在GenBank數據庫中進行序列比對后,所得分子結果比傳統形態學鑒定結果更高效、更準確。如本研究根據DNA條形碼的鑒定結果,共鑒定出了7種魚卵與5種仔稚魚,全部鑒定到種,而形態學鑒別結果只有5種魚卵與5種仔稚魚,且只有5種仔稚魚能鑒定到種,魚卵依據形態特征僅能鑒定到屬或科水平。此外,本次研究中有部分魚卵還處于卵裂期,在形態方面,除了卵徑大小之外沒有其他可用于鑒定的特征,但是使用DNA條形碼技術卻能夠直接鑒定出種類,并且在處于同等大小的卵裂期魚卵中,鑒定出了銀鯧與日本鬼鲉2個種,在最初此2種魚卵被認定是同一種。
將DNA條形碼技術運用到魚類浮游生物的鑒定中,幫助解決了很多傳統分類無法解決的問題,在魚類分類學、漁業資源保護等方面都取得了不錯的成果,但該技術的應用時間僅有短短的十數年,目前仍然存在著一些不足與需要解決的問題。GenBank是一個開放的共享數據庫,雖然擁有龐大的數據資料,但是其中的基因序列質量卻良莠不齊,利用DNA條形碼技術雖然可以快速鑒別物種,但這項技術還是需要結合傳統的形態鑒定方法才能獲得正確的種類序列,從而為DNA條形碼鑒別工作累積有效的資料。雖然使用傳統的形態學分類方法需要較高的專業水準,并且具有局限性,但是只有以此為基礎,將傳統的形態學分類方法與DNA條形碼技術這一全新的分類方法結合起來,才能進一步對數據庫進行完善,才能更好地評估與保護魚類的多樣性。
3.2 廈門灣魚類浮游生物多樣性
廈門灣海域屬于亞熱帶性質的河口海灣,地形復雜,港闊水深,常年不凍,是一個條件十分優越的天然海港。此海區初級生產力水平較高[10],物種豐富多樣,同時是許多經濟魚類的產卵場及育幼場[11]。江素菲等[12]根據1987年3月至1988年2月采集的樣品,并結合1983年4月至1984年3月在九龍江口采集的樣品進行鑒定,得出共有魚卵14種,隸屬于4目7科11屬;仔稚魚49種,隸屬于9目29科47屬。林楠等[13]于2007年11月—2008年8月在九龍江口共鑒定出33種魚類浮游生物,隸屬于16屬15科。張躍平[14]于2007年8月至2009年10月期間在廈門海域共鑒定6種魚卵,其中斑魚祭(占總量的44.3%)和多鱗魚喜(占總量的20.2%)是主要優勢種;仔稚魚共鑒定19種,主要優勢種是康氏小公魚、多鱗魚喜和肩鰓鳚。徐春燕等[15]于2013 年4 月在廈門南部海域共鑒定魚卵和仔稚魚21 種,其中魚卵14 種,仔稚魚9 種,魚卵、仔稚魚數量主要以黃姑魚(Nibeaalbifora))、青鱗小沙丁魚(Sardinellazunasi)和魚叚虎魚科(Gobiidae sp.)為主。廈門海域魚資源豐富,但在有關魚卵、仔稚魚的調查研究中卻有很多種類都無法鑒定到種,甚至未能鑒定。例如,蔡秉及等[16]對廈門島區和大嶝島區海域進行4 個航次的浮性魚卵和仔稚魚的種類組成與數量分布進行報道,共記錄44種魚類,其中魚卵和仔、稚魚各28 種,但鑒定到種的分別為18 種和21 種。張躍平于2007年8月至2009年10月期間在廈門海域共鑒定22種魚類浮游生物,鑒定到種水平的僅11種[14]。
本研究采用DNA 條形碼技術并結合傳統形態甄別,對廈門海域采集到的魚卵、仔稚魚進行種類鑒定,共獲得有效序列27條,完成的數據分析中,共鑒定種類11種。本研究結果報道的種類數較少,主要原因在于本次DNA條形碼測序樣品僅選取垂直拖網的定量樣品,如果能全面分析水平拖網樣品,相信能夠得到更豐富的多樣性結果;此外,本次采樣主要集中在環廈門島周邊,未往九龍江口方向延伸采樣,而據以往報道,九龍江口是魚類浮游生物多樣性較高區域[12-13]。在本次鑒定出的魚類浮游生物中,雖大部分種類在廈門灣的魚類資源調查中都有被報道過[16-19],但之前的報道多見于仔、稚魚的樣品鑒定結果,而本研究中有7種魚卵經過DNA條形碼技術分析,全部鑒定到種,并對其形態特征做了詳細的描述,有效地補充了廈門灣魚類浮游生物多樣性的研究資料。在未來可以擴大采樣范圍,發現以往未能鑒定出的物種,更全面地反映廈門灣魚類浮游生物的多樣性。

