菊葉香藜轉錄組數據庫中FPPS基因的挖掘與生物信息學分析
付蘇宏 雷鳴 張勇群 施靜 郝豆豆


摘 要:為深入了解菊葉香藜法尼基焦磷酸合酶基因,該研究對菊葉香藜轉錄組數據庫進行挖掘,獲取了兩條FPPS基因序列(DsFPPS1和DsFPPS2),并對DsFPPS1和DsFPPS2編碼蛋白的理化性質、結構、功能、系統進化進行了分析。結果表明:DsFPPS1和DsFPPS2基因分別包含1 029 bp和969 bp的開放閱讀框,分別編碼342個(DsFPPS1)和322個(DsFPPS2)氨基酸。DsFPPS1和DsFPPS2位于線粒體內,未發現信號肽和跨膜結構,DsFPPS1為穩定蛋白,DsFPPS2為不穩定蛋白。氨基酸序列比對發現DsFPPS1和DsFPPS2序列相似性為60.53%,均含有5個保守結構域和2個天冬氨酸富集區域。DsFPPS1和DsFPPS2二級結構主要由α-螺旋構成,三級結構為由8個α-螺旋形成的α-螺旋束,但DsFPPS2的三級結構中缺少一個α-螺旋A。系統進化樹中DsFPPS1與藜科植物聚為一枝,與藜科植物遺傳距離較近,而DsFPPS2單獨聚為一枝。通過對菊葉香藜轉錄組數據庫中FPPS基因的挖掘與生物信息學分析,為菊葉香藜FPPS的功能研究及其倍半萜類化合物的生物合成研究奠定了一定的理論基礎。
關鍵詞:法尼基焦磷酸合酶, 轉錄組, 菊葉香藜, 基因挖掘, 生物信息學
中圖分類號:Q943.2
文獻標識碼:A
文章編號:1000-3142(2019)06-0831-12
Abstract:Dysphania schraderiana, in the Chenopodiaceae family, is widely distributed in Lhasa (Tibet, China) and used as a traditional medicine. The essential oil of Dysphania schraderiana? contains abundant sesquiterpenes compounds, appeared to possess potential medicinal value. Farnesyl pyrophosphate synthase (FPPS) is a key branch-point enzyme in biosynthesis of terpene. In order to reveal D. schraderiana FPPS gene, the transcriptome database of D. schraderiana was mined and two gene sequences (DsFPPS1 and DsFPPS2) were obtained in this research. Subsequential protein physicochemical property, architectural feature, function and phylogeny relationship analysis of DsFPPSs were also predicted and analyzed. The results showed that DsFPPS1 and DsFPPS2 sequences contained an ORF span of 1 029 bp and 969 bp respectively, encoding 342 (DsFPPS1) and 322 (DsFPPS2) amino acids respectively. The analysis of amino acid composition showed that the dominant components of DsFPPS1 and DsFPPS2 were both nonpolar amino acids. The molecular weight of DsFPPS1 and DsFPPS2 were 39.68 kD and 36.76 kD, respectively. Isoelectric point were 5.11 and 5.65 for DsFPPS1 and DsFPPS2, respectively. Besides, DsFPPS1 protein was predicted to be a stable protein, but DsFPPS2 protein was predicted to be an unstable protein. The amino acid sequence analysis showed that DsFPPS1 and DsFPPS2 had no signal peptide and transmembrane region. The possible localization of DsFPPS1 and DsFPPS2 was both in mitochondria. DsFPPS1 and DsFPPS2 protein exhibited 60.53% sequence identity, and possessed five conserved domain (Ⅰ-Ⅴ) and two characteristic Asp-rich motifs (DDXXD). The amino acid sequence of DsFPPS1 had higher homology with Chenopodium quinoa, Spinacia oleracea and Beta vulgaris than DsFPPS2. In addition, the secondary structure of DsFPPS proteins mainly consisted of α-helixes, which resulted in a bundle of 8 α-helices in tertiary structure. However, tertiary structure analysis showed that DsFPPS2 protein missed an α-helix compared to DsFPPS1 protein. The result of phylogenetic analysis indicates that phylogenetic relationships of DsFPPS1 protein are close to Chenopodiaceae plants consistent with sequence alignment results, while DsFPPS2 protein is clustered alone in phylogenetic tree. In general, these results provides a certain reference for an insight to molecular function of DsFPPS and the synthetic biology of sesquiterpenes in D. schraderiana.
Key words:farnesyl pyrophosphate synthase, transcriptome, Dysphania schraderiana, gene mining, bioinformatics
菊葉香藜(Dysphania schraderiana)系藜科刺藜屬植物,全草入藥,具有平喘解痙、解毒、解表、止痛、止癢等功效,是我國傳統的藥用植物。現代研究發現菊葉香藜精油具有廣泛的生物活性,本課題組在前期研究中發現菊葉香藜精油對大腸桿菌和枯草芽孢桿菌的生長具有顯著抑制作用,且呈現出一定的濃度依賴性;此外菊葉香藜精油可濃度依賴性地抑制昆蟲玉米象的活力(雷鳴等, 2015);現已有利用菊葉香藜精油治理植物螨蟲的專利(劉志龍等, 2015)。本課題組前期研究中利用GC-MS檢測了菊葉香藜精油的化學成分,除了精油中大量的脂肪族化合物(54.232%)外,倍半萜類化合物占有較大的比例(26.846%)。
倍半萜是一類包含有15個碳原子的天然化合物,即包含有3個異戊二烯單元,具有多種結構類型,是新藥研發的主要來源之一(王佳等, 2012)。植物萜類化合物主要是由細胞質中的甲羥戊酸(MVA)途徑和質體中的2-C-甲基-D-赤蘚糖醇-4-磷酸(MEP)途徑合成,這兩條途徑生成萜類化合物的共同前體異戊烯焦磷酸(isopentenyl pyrophosphate,IPP)和二甲基丙烯基二磷酸(dimethylallylpyrophosphate,DMAPP),隨后一分子的IPP和一分子的DMAPP結合生成香葉基二磷酸(geranyl diphosphate,GPP),一分子的GPP和一分子的IPP在法尼基焦磷酸合酶(farnesyl pyrophosphate synthase, FPPS)的作用下生成倍半萜類化合物的前體物質法尼基焦磷酸(farnesyl diphosphate, FPP),FPP再在多種倍半萜合酶的作用下生成結構多樣的倍半萜化合物(孫麗超等, 2017)。
植物FPPS由基因家族編碼,通常有多個FPPS亞型,如擬南芥含有FPPS1和FPPS2兩個基因,編碼3個FPPS亞型:FPPS1S、FPPS1L和FPPS2,同時敲除兩條FPPS基因會造成植物死亡,單個基因敲除不會對植物生長發育造成影響,這種多基因模式可保證某一基因受損時植物仍能正常生存,但不同的亞型仍然具有不同的表達模式與活性,擬南芥FPS1存在于植物整個生命周期,FPS2則主要在種子生長發育階段發揮作用(Closa et al., 2010;趙麗等,2018),且FPS2具有更強的催化活性和熱穩定性,對氯化鈉的抑制作用更敏感(Keim et al., 2012)。
FPPS是萜類化合物生物合成分支點的關鍵酶,催化生成重要的中間產物FPP進而合成倍半萜類化合物。鑒于FPPS在倍半萜類化合物生物合成途徑中的重要性,而目前尚未見菊葉香藜FPPS基因的研究報道。因此,為了深入探討菊葉香藜的FPPS,本研究對菊葉香藜花和葉轉錄組數據庫進行挖掘以獲取菊葉香藜FPPS (DsFPPS)基因,并利用生物信息學的方法對該基因所編碼蛋白的理化性質、亞細胞定位、結構特征、功能以及系統進化方面進行分析和預測,為今后深入研究該酶的結構和功能奠定理論基礎。
1 材料與方法
1.1 材料
菊葉香藜的兩條FPPS基因來自于實驗室前期菊葉香藜轉錄組研究,花組織轉錄組與葉組織轉錄組在SRA (Sequence Read Archive)數據庫中的登錄號分別為SRX3145242和SRX3145241,其余物種相關的FPPS蛋白序列均來自NCBI (National Center for Biotechnology Information)數據庫,物種及序列信息如下:藜麥(Chenopodium quinoa) (CqFPPS:XP_021740746.1)、菠菜(Spinacia oleracea) (SoFPPS:XP_021846881.1)、甜菜(Beta vulgaris) (BvFPPS:XP_010675977.1)、野茶樹(Camellia sinensis) (CsFPPS:ANA11766.1)、杧果(Mangifera indica) (MiFPPS:AFJ52720.1)、蓖麻(Ricinus communis)(RcFPPS:AMN82836.1)、橡膠樹(Hevea brasiliensis)(HbFPPS:ANJ77846.1)、大戟(Euphorbia pekinensis)(EpFPPS:ACN63187.1)、人參(Panax ginseng)(PgFPPS:AAY87903.1)、竹節參(P. japonicas)(PjFPPS:AKN52395.1)、三七(P. notoginseng)(PnFPPS:AGS79228.1)、常春藤(Hedera helix)(HhFPPS:APV45530.1)、西洋參(Panax quinquefolius)(PqFPPS:ADJ68004.1)、薄荷(Mentha x piperita)(MpFPPS:AAK63847.1)、撒爾維亞(Salvia officinalis)(SolFPPS:AQY54371.1)、丹參(S. miltiorrhiza)(SmFPPS:ABV08819.1)、狹葉薰衣草(Lavandula angustifolia)(LaFPPS:AGQ04160.1)、米團花(Leucosceptrum canum)(LcFPPS:ALT07952.1)、黃芪(Astragalus membranaceus)(AmFPPS:AID51444.1)、甘草(Glycyrrhiza uralensis)(GuFPPS:ADE18770.1)、紫苜蓿(MsFPPS:ADC32809.1)、里氏木霉菌(Trichoderma reesei)(TrFPPS:AFX82678.1)、新月金孢子菌(Emmonsia crescens)(EcFPPS:POS87336.1)、地衣芽孢桿菌(E. crescens)(BlFPPS:ARC74194.1)、莢膜紅細菌(Rhodobacter capsulatus)(RHcFPPS:ADE44162.1)、牛(Bos taurus)(BtFPPS:AAL58886.1)、黑猩猩(Pan troglodytes)(PtFPPS:JAA28793.1)。
1.2 分析方法
1.2.1 蛋白基本性質分析 開放閱讀框(ORF)通過GenBank的在線軟件ORF Finder (https://www.ncbi.nlm.nih.gov/orffinder/)進行查找。獲取基因所編碼的蛋白質氨基酸序列后,運用多種在線軟件分析、預測蛋白的相關信息:蛋白的理化性質利用ProtParam (http://web.expasy.org/protparam/)進行分析;蛋白的親/疏水性分析應用在線軟件ProtScale (http://web.expasy.org/protscale/);蛋白的信號肽分析應用在線工具SignalP 4.1 (http://www.cbs.dtu.dk/services/SignalP/);蛋白的跨膜結構域預測應用在線程序TMHMM (http://www.cbs.dtu.dk/services/TMHMM/);蛋白的亞細胞定位預測運用在線軟件Cell-PLoc 2.0 (http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)。
1.2.2 蛋白結構特征與功能分析 蛋白質的二級結構運用在線SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)軟件進行預測;蛋白質的三級結構運用Phyre2 (http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)在線軟件進行預測;利用在線數據庫CDD (Conserved Domain Database) (https://www.ncbi.nlm.nih.gov/cdd/)進行保守位點分析。
1.2.3 蛋白氨基酸序列比對及系統進化樹構建 用NCBI中的BLASTP (https://blast.ncbi.nlm.nih.gov/Blast.cgi)模塊進行不同物種FPPS氨基酸序列的在線搜索,用ClustalW進行多條FPPS氨基酸序列比對。采用鄰接法(Neighbor-Joining)構建FPPS氨基酸序列的系統進化樹,使用自展值(Bootstrap)重復檢驗1 000次,系統進化樹的構建和檢驗通過本地軟件MEGA 7.0.14實施。
2 結果與分析
2.1 菊葉香藜FPPS基因挖掘
通過對菊葉香藜轉錄組數據中核酸序列進行比對和功能注釋,挖掘得到兩條編號分別為c13053_g1和c12747_g1的FPPS候選基因,分別重命名為DsFPPS1和DsFPPS2。DsFPPS1長度為1 257 bp,其互補鏈轉錄翻譯為FPPS蛋白,開放閱讀框為1 029 bp,開放閱讀框對應到DsFPPS1的位置為1 185~155 bp,編碼342個氨基酸,編碼蛋白命名為DsFPPS1;DsFPPS2長度為1 667 bp,包含一個969 bp的開放閱讀框,位置從296~1 264 bp,編碼322個氨基酸,編碼蛋白命名為DsFPPS2(圖1,圖2)。
2.2 DsFPPS蛋白的理化性質分析
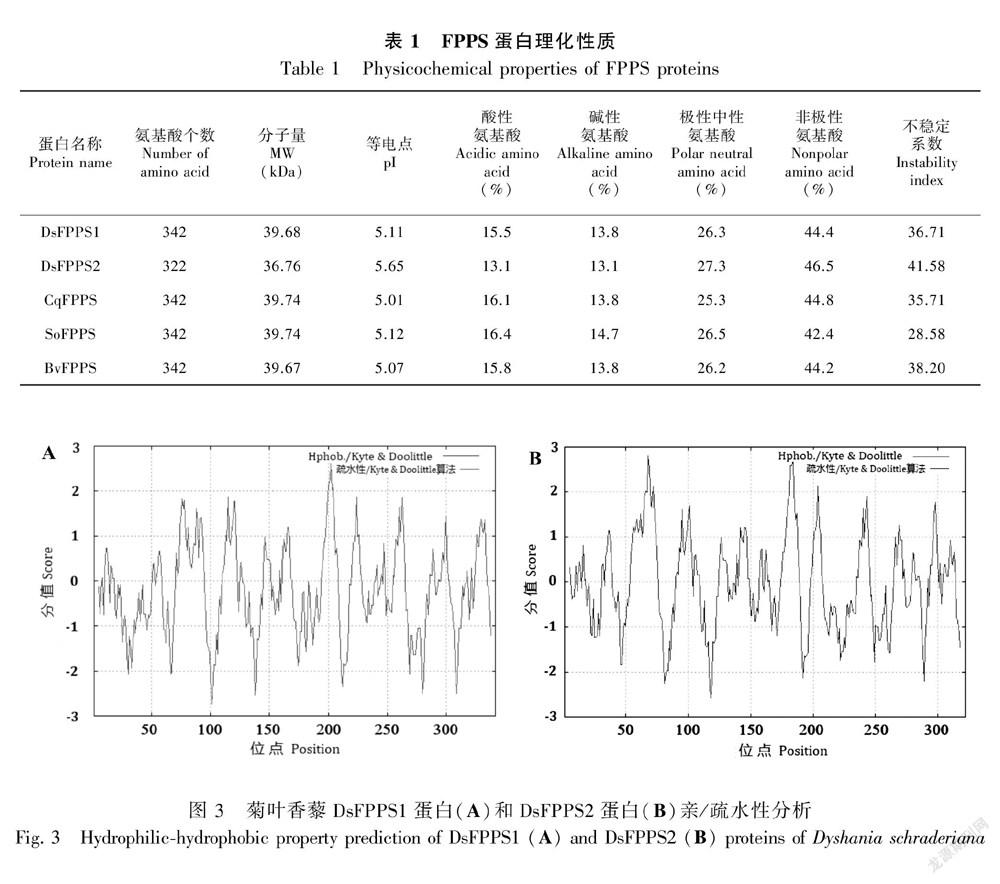
用在線軟件ProtParam分析DsFPPS1蛋白和DsFPPS2蛋白以及3種藜科植物的FPPS蛋白(藜麥CqFPPS;菠菜SoFPPS;甜菜BvFPPS)的理化性質,結果如表1所示。DsFPPS1蛋白與藜科植物FPPS蛋白較相似,氨基酸長度均為342 aa;分子量在39.67~39.74之間;等電點(pI值)在5.01~5.12;酸性氨基酸比例在15.5%~16.4%之間,堿性氨基酸比例在13.8%~14.7%之間,極性中性氨基酸比例在25.3%~26.5%之間,非極性氨基酸比例在42.4%~44.4%之間,差異均較小;不穩定系數值均小于40,屬于穩定蛋白。而DsFPPS2蛋白與DsFPPS1蛋白以及藜科植物FPPS蛋白差異較大,氨基酸個數為322 aa,長度短于其他FPPS蛋白;分子量為36.76;等電點(pI值)為5.65;堿性氨基酸和極性中性氨基酸的比例與其他FPPS蛋白差異較小,比例分別為13.1%和27.3%,酸性氨基酸的比例低于其他FPPS蛋白,為13.1%,非極性氨基酸的比例高于其他FPPS蛋白,為46.5%;不穩定系數值大于40,為不穩定蛋白。
運用在線軟件ProtScale分析DsFPPS蛋白的親/疏水性,蛋白的每一位氨基酸的親/疏水性用分值表示,正值則表示疏水,負值則表示親水,絕對值越大則表示其親/疏水性越強。DsFPPS1蛋白和DsFPPS2蛋白的親/疏水性預測結果如圖3所示,在DsFPPS1蛋白氨基酸序列的第202位處有最高分值2.611,表明其疏水性水性較強;第101位有最小分值-2.733,表明其親水性較強;在DsFPPS2蛋白氨基酸序列的第68位處有最高分值2.800,表明其疏水性水性較強;第118位有最小分值-2.578,表明其親水性較強。
2.3 DsFPPS蛋白的功能結構域分析
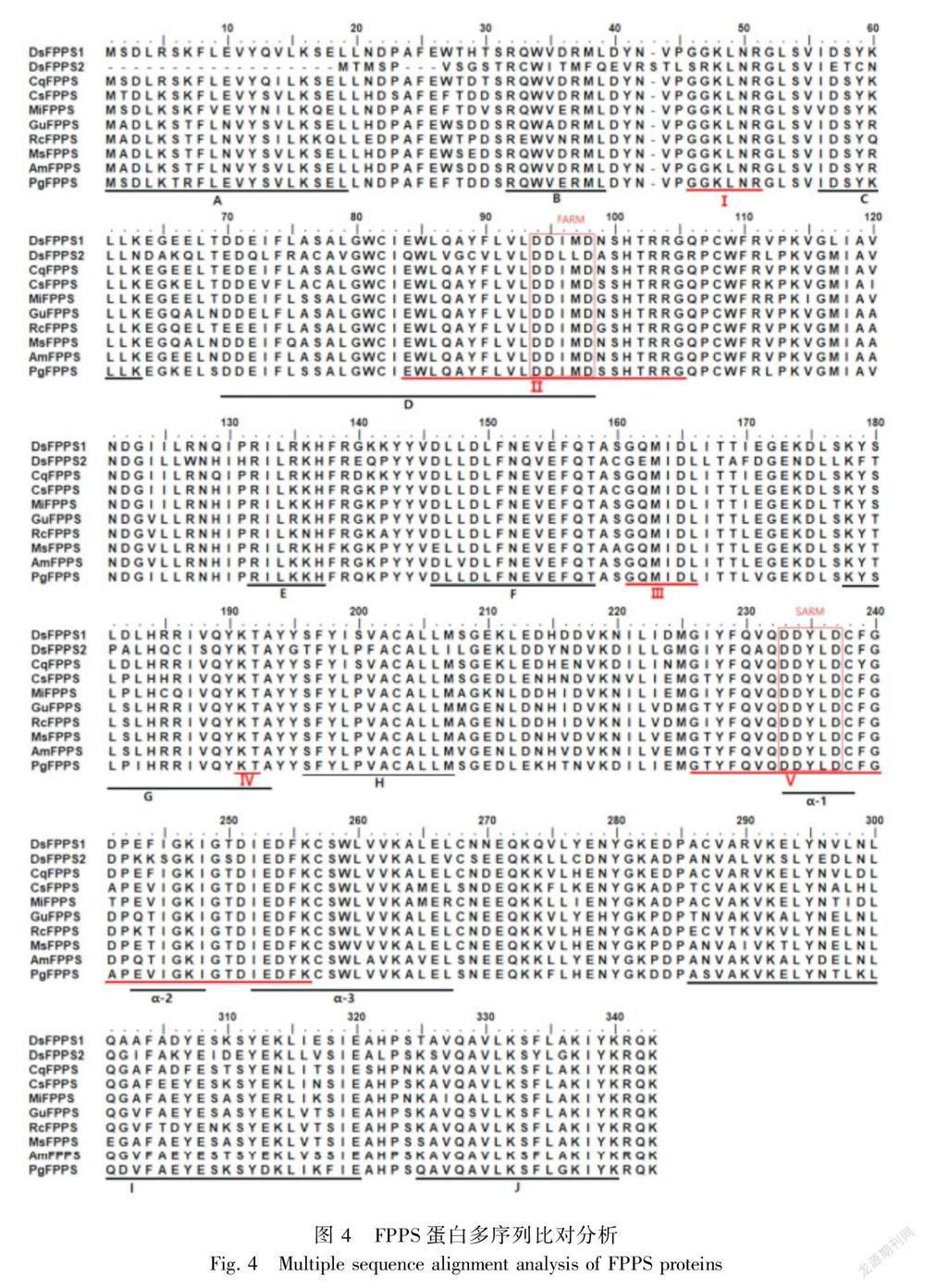
采用NCBI中的CDD工具、本地軟件ClustalW以及相關文獻(Chen et al., 1994; Guo et al., 2015; 李永波等, 2012)尋找FPPS蛋白的保守結構域,結果顯示FPPS家族蛋白共包含5個保守區域(Ⅰ~Ⅴ),以及分別位于保守區域Ⅱ和Ⅴ的兩個天冬氨酸富集區域(DDXXD,X為任意氨基酸):FARM (First Aspartic Rich Motif)和SARM (Second Aspartic Rich Motif),如圖4所示。DsFPPS1蛋白和DsFPPS2蛋白均屬于FPPS家族,序列比對結果顯示DsFPPS1蛋白和DsFPPS2蛋白具有60.53%的相似性,均具有FPPS家族蛋白特征的兩個天冬氨酸富集區域FARM和SARM,以及序列保守區域Ⅰ~Ⅴ。天冬氨酸富集區域(DDXXD)是金屬離子Mg2+結合位點,Mg2+再與底物的磷酸基團結合,從而發揮催化作用(Christianson, 2017)。DsFPPS1蛋白對應的五個保守區域序列分別為GGKLNR (45~50, Ⅰ)、EWLQAYFLVLDDIMDNSHTRRG (83~104,Ⅱ)、GQMIDL (160~165,Ⅲ)、KT (190~191,Ⅳ)、GIYFQVQDDYLDCFGDPEFIGKIGTDIEDFK (225~255,Ⅴ),FARM為DDIMD (93~97),SARM為DDYLD (232~236)。DsFPPS2蛋白對應的五個保守區域分別為SRKLNR (25~30,Ⅰ)、QWLVGCVLVLDDLLDASHTRRG (63~84,Ⅱ)、GEMIDL (140~145,Ⅲ)、KT (170~171,Ⅳ)、GIYFQAQDDYLDCFGDPKKSGKIGSDIEDFK (205~235,Ⅴ),FARM為DDLLD (73~77),SARM為DDYLD (212~216)。
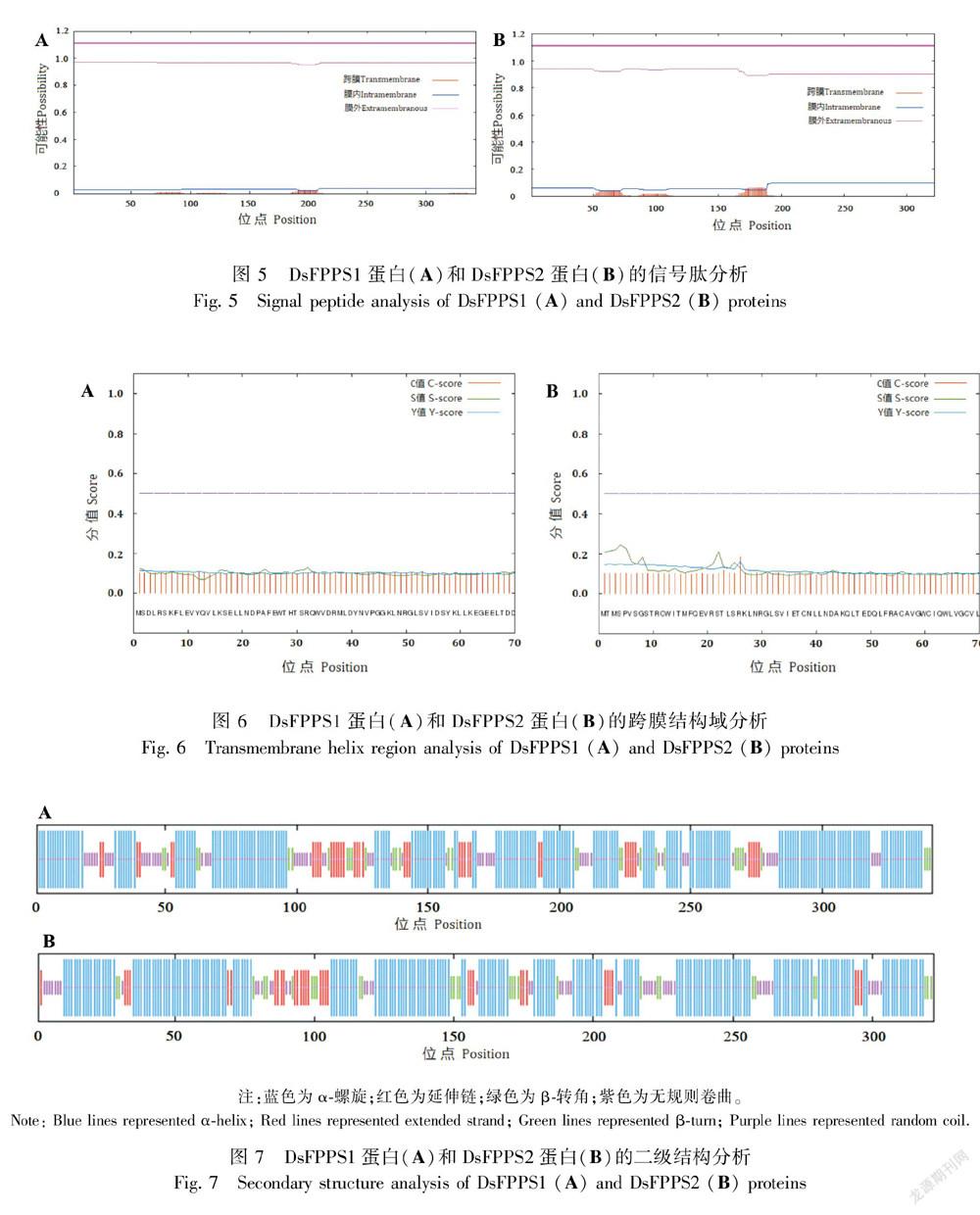
2.4 DsFPPS蛋白的信號肽、跨膜結構域與亞細胞定位分析個氨基酸殘基的短肽,在分泌蛋白合成結束后將其切除,因此可以通過分析蛋白的氨基酸序列是否具有分泌肽來判斷蛋白是否為分泌蛋白。利用在線軟件SignalP 4.1對DsFPPS基因所編碼的蛋白進行分析,結果顯示DsFPPS1蛋白和DsFPPS2蛋白均不具有信號肽,屬于非分泌蛋白(圖5:A,B)。然后,我們利用TMHMM在線軟件分析DsFPPS基因編碼蛋白的跨膜結構,結果顯示DsFPPS1蛋白和DsFPPS2蛋白均不具有跨膜結構(圖6:A,B)。此外,Cell-PLoc 2.0在線軟件的亞細胞定位分析表明,DsFPPS1蛋白和DsFPPS2蛋白均存在于細胞的線粒體內。
2.5 DsFPPS蛋白的結構特征分析
蛋白的二級結構進行分析和預測,結果表明(圖7) DsFPPS1蛋白和DsFPPS2蛋白的二級結構組成類似,都是主要由α-螺旋組成,其比例分別為59.36%和64.29%,此外,DsFPPS1蛋白的二級結構由19.88%的無規則卷曲、11.70%的延伸鏈以及9.06%的β-轉角組成,DsFPPS2蛋白的二級結構由15.53%的無規則卷曲、10.87%的延伸鏈以及9.32%的β-轉角組成。
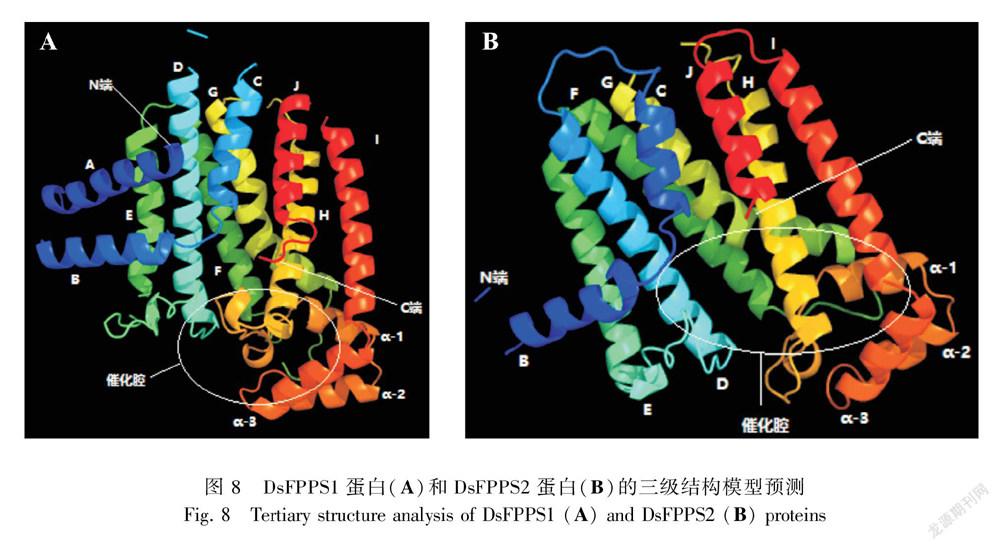
利用在線軟件Phyre2對DsFPPS基因所編碼的DsFPPS1蛋白和DsFPPS2蛋白的三級結構進行預測,以Artemisia spiciformis的FPPS蛋白(PDB ID:4KK2)為模板,采用線串法同源建模構建DsFPPS1蛋白(79%相似性)和DsFPPS2蛋白(63%相似性)的三級結構,結果如圖8所示。與大多數的FPPS蛋白類似,DsFPPS1蛋白的空間結構中總共包含有13個α-螺旋,其中有10個長α-螺旋(A~J),α-螺旋A和α-螺旋B形成一個發夾結構,α-螺旋C至α-螺旋J共8個α-螺旋則形成α-螺旋束,圍繞形成一個中央腔;此外還包含有3個小α-螺旋(α-1、α-2、α-3),嵌入到α-螺旋H和α-螺旋I之間,酶的催化反應則發生在α-螺旋腔底。DsFPPS2蛋白的三級結構與DsFPPS1蛋白類似,但只包含有12個α-螺旋,9個長α-螺旋(B~J)和3個小α-螺旋(α-1,α-2,α-3),不包含α-螺旋A,因此缺少α-螺旋A和α-螺旋B形成的發夾結構。
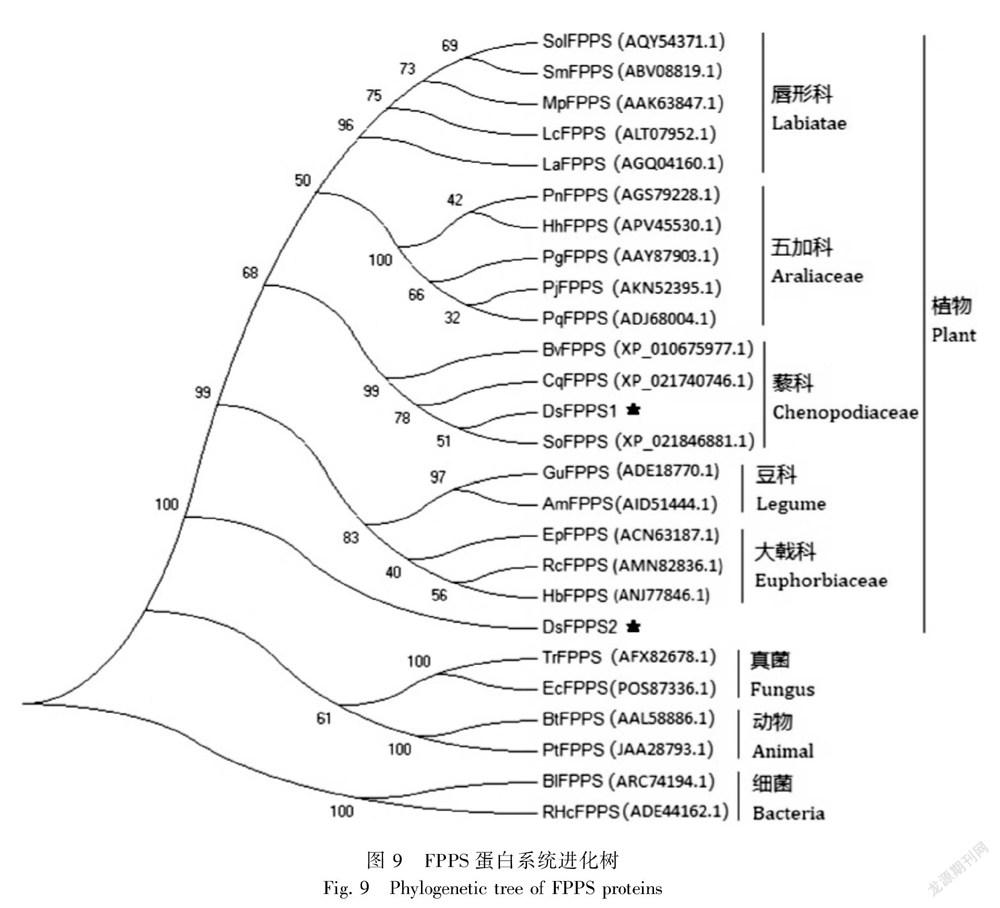
2.6 DsFPPS蛋白的系統進化分析
采用本地軟件MEGA 7.0.14對DsFPPS1蛋白、DsFPPS2蛋白和來自于GenBank數據庫的24條FPPS蛋白序列進行分子系統進化分析,N-J法(Neighbor-joining)構建系統進化樹。圖9結果表明,FPPS蛋白的系統進化樹聚類分為植物、真菌、動物和細菌4個類群,具有較顯著的種族特異性。細菌單獨聚為一大枝,動物、真菌和植物聚為一大枝,該分枝中植物單獨聚為一枝,另一枝又分化為動物和真菌兩大枝。在植物分枝中,唇形科、五加科、藜科、蝴蝶花科、大戟科植物各自聚為一枝,菊葉香藜的DsFPPS1蛋白分布于藜科植物分枝中,說明其與藜科植物的親緣關系較近,而菊葉香藜的DsFPPS2蛋白單獨聚為一枝。
3 討論與結論
倍半萜類化合物作為植物的次級代謝產物具有較強的生物活性(樸英花和樸惠順, 2012),例如青蒿素(Artemisinin, C15H22O5)為倍半萜內酯藥物,是有效的抗瘧疾特效藥(Tu, 2011);吉馬酮(Germacrone, C15H22O)具有抗潰瘍、抗腫瘤、抗炎、抗菌、止咳、利膽等多種功效(Wu et al., 2017);β-桉葉醇(β-Eudesmol, C15H26O)可通過作用于瞬態受體電位錨蛋白1 (TRPA1)和自主神經系統從而刺激食欲(Ohara et al., 2017);雪松醇(Cedrol, C15H26O)可劑量依賴性地加速纖維母細胞生長以及增加1型膠原蛋白和彈性蛋白的生成(Mu et al., 2012);纈草烯酸(Valerenic acid, C15H22O2)可變構調節GABA-A受體從而發揮抗焦慮活性(Becker et al., 2014)。
FPPS是植物萜類化合物生物合成途徑中分支點上的關鍵酶,經FPPS的催化可生產倍半萜類化合物的前體,菊葉香藜精油中倍半萜類的生物合成需要此酶的參與。目前,隨著轉錄組學研究的崛起,我們從包含有海量信息的轉錄組數據中挖掘FPPS候選基因,獲得了兩條FPPS基因:FPPS1和FPPS2,并對這兩條基因的開放閱讀框進行了預測并進行了進一步的生物信息學分析,結果顯示,FPPS1蛋白和FPPS2蛋白序列相似度達60.53%,均含有FPPS蛋白特征的5個保守結構域(Ⅰ~V)以及兩個天冬氨酸富集區域(FARM和SARM)。然而,FPPS1蛋白和FPPS2蛋白的穩定性存在差異,FPPS2蛋白為不穩定蛋白,且FPPS2蛋白結構中缺少α-螺旋A,因此其三級結構中缺少由α-螺旋A和α-螺旋B組成的發夾結構,該結構可能會影響其穩定性。在分子進化分析樹的植物分枝中,FPPS2蛋白與其他植物分開,單獨聚為一枝,FPPS2蛋白可能是一類新的FPPS亞型。植物FPPS通常由基因家族編碼,含有多個FPPS亞型,不同亞型在植物不同的生長階段和組織中發揮作用,擬南芥種子中FPS2的缺失可升高HMGR(3-hydroxy-3-methylglutaryl CoA reductase,3-羥基-3-甲基戊二酰CoA還原酶)活性(Closa et al., 2010),從而調節植物中“碳流”的流向,控制類異戊二烯種類的比例。
在前期的菊葉香藜轉錄組研究中發現,FPPS1在花和葉組織中的表達均高于FPPS2,且FPPS1蛋白的穩定性優于FPPS2蛋白。由此可推測,FPPS1蛋白廣泛且長期存在于菊葉香藜植物中,而FPPS2的出現則具有“時-空”特異性,但菊葉香藜FPPS各亞型的功能、活性、表達以及調節機制仍需要更進一步的研究。總的來說,該研究對菊葉香藜轉錄組數據庫中的FPPS基因進行了挖掘與生物信息學分析,為菊葉香藜FPPS基因今后的基礎研究和開發應用奠定了理論基礎。
參考文獻:
BECKER A, FELGENTREFF F, SCHRDER H, et al., 2014. The anxiolytic effects of a valerian extract is based on valerenic acid? [J]. BMC Complem Altern M, 14(1):1-5.
CHEN A, KROON PA, POULTER CD, 1994. Isoprenyl diphosphate synthases:Protein sequence comparisons, a phylogenetic tree, and predictions of secondary structure? [J]. Protein Sci, 3(4):600-607.
CHRISTIANSON DW, 2017. Structural and chemical biology of terpenoid cyclases? [J]. Chem Rev, 117(17):11570.
CLOSA M, VRANOV E, BORTOLOTTI C, et al., 2010. The Arabidopsis thaliana FPP synthase isozymes have overlapping and specific functions in isoprenoid biosynthesis, and complete loss of FPP synthase activity causes early developmental arrest? [J]. Plant J Cell Mol Biol, 63(3):512.
GUO D, LI HL, PENG SQ, 2015. Structure conservation and differential expression of farnesyl diphosphate synthase genes in Euphorbiaceous plants? [J]. Int J Mol Sci, 16(9):22402-22414.
KEIM V, MANZANO DA, DAVID, FERNNDEZ FJ, et al., 2012. Characterization of Arabidopsis FPS isozymes and FPS gene expression analysis provide insight into the biosynthesis of isoprenoid precursors in seeds? [J]. PLoS ONE, 7(11):e49109.
LEI M, HE H, ZHANG PF, et al., 2015. Study on the extraction of Chenopodium oetidum essential oil and its inhibition against insect activity? [J]. J Anhui Agric Sci, 43(28):64-66.? [雷鳴, 何花, 張鵬飛, 等, 2015. 菊葉香藜精油的提取及對昆蟲活力抑制的研究? [J]. 安徽農業科學, 43(28):64-66.]
LI YB, FAN QQ, WANG BL, et al., 2012. Advances in the study of plant farnesyl pyrophosphate synthase (FPPS) gene? [J]. Chin J Agric Biotechnol, 20(3):321-330.? [李永波, 樊慶琦, 王寶蓮, 等, 2012. 植物法呢基焦磷酸合酶基因(FPPS)研究進展? [J]. 農業生物技術學報, 20(3):321-330.]
LIU ZL, LIU XC, SHI WP, et al., 2015. Essential oil of Dysphania schraderiana in the prevention and control of plant mites:China, 104782667A [P]. 2015-07-22.? [劉志龍, 劉昕超, 石旺鵬, 等, 2015. 菊葉香藜植物揮發油在防治植物螨中的用途. 中國, 104782667A [P]. 2015-07-22.]
MU HJ, SUN GP, HWANG YL, et al., 2012. Cedrol enhances extracellular matrix production in dermal fibroblasts in a MAPK-Dependent manner? [J]. Ann Dermatol, 24(1):16-21.
OHARA K, FUKUDA T, ISHIDA Y, et al., 2017. β-Eudesmol, an oxygenized sesquiterpene, stimulates appetite via TRPA1 and the autonomic nervous system? [J]. Sci Rep, 7(1):15785-15081.
PIAO YH, PIAO HS, 2012. Research progress in biological activity of sesquiterpene compounds? [J]. Occup Health, 28(18):2291-2293.? [樸英花, 樸惠順, 2012. 倍半萜類化合物生物活性研究進展? [J]. 職業與健康, 28(18):2291-2293.]
SUN LC, LI SY, WANG FZ, et al., 2017. Research progresses in the synthetic biology of terpenoids? [J]. Biotechnol Bull, 33(1):64-75.? [孫麗超, 李淑英, 王鳳忠, 等, 2017. 萜類化合物的合成生物學研究進展? [J]. 生物技術通報, 33(1):64-75.]
TU YY, 2011. The discovery of artemisinin (qinghaosu) and gifts from Chinese medicine? [J]. Nat Med, 17(10):1217-1220.
WANG J, YOU S,ZHOU LN, 2012. Progress in research of sesquiterpenoids biotransformation? [J]. J Shenyang Pharm Univ, 29(2):76-84.? [王佳, 游松, 周麗娜, 2012. 倍半萜生物轉化的研究進展? [J]. 沈陽藥科大學學報, 29(2):76-84.]
WU J, FENG Y, HAN C, et al., 2017. Germacrone derivatives:synthesis, biological activity, molecular docking studies and molecular dynamics simulations? [J]. Oncotarget, 8(9):15149-15158.
ZHAO L, PAN CX, PANG KQ, et al., 2018. Gene cloning of PpGL2 transcription factor in peach and its bioinformatics analysis [J]. J Southern Agric, 49(10):1901-1908. [趙麗, 潘彩霞, 龐可, 等, 2018. 桃樹PpGL2轉錄因子基因克隆及生物信息學分析 [J]. 南方農業學報, 49(10):1901-1908. ]

