利用CRISPR/Cas9敲除OsNramp5基因創制低鎘秈稻
龍起樟 黃永蘭 唐秀英 王會民 蘆明 袁林峰 萬建林
利用CRISPR/Cas9敲除基因創制低鎘秈稻
龍起樟 黃永蘭 唐秀英 王會民 蘆明 袁林峰 萬建林*
[江西省超級水稻研究發展中心/水稻國家工程實驗室(南昌)/江西省綠色水稻種質重點實驗室,南昌 330200;*通訊聯系人,E-mail: ncwanjl@163.com]
為了嘗試快速培育低鎘秈稻,選取廣泛應用的雜交水稻親本華占和五豐B以及常規品種五山絲苗和中早35為材料,通過CRISPR/Cas9技術創制基因突變株系,在鎘污染及正常土壤中種植并測定突變株系籽粒(糙米)鎘含量,其他相關元素含量亦同時在鎘污染土壤種植條件下測定,在非鎘污染土壤種植條件下考查基因敲除對農藝性狀的影響。成功獲得了前述品種的基因敲除株系;非鎘污染條件下種植的4個品種基因敲除株系籽粒鎘含量低于0.02 mg/kg,平均較野生型降低85.5%;而在鎘污染土壤種植時,不同品種基因敲除株系籽粒鎘含量低于0.1 mg/kg,平均比野生型降低94.8%;錳含量也降低52.7%,鉻含量增加59.5%,鉛含量在華占中增加79.1%,而在其他品種中無變化;銅、鐵、鋅、鈣、硒和砷含量(后4種元素只在華占及衍生品系中檢測)受影響較小或不受影響;基因敲除株系株高、結實率和千粒重較野生型小幅降低,而有效分蘗略微增加,產量平均減少6.9%。通過基因敲除,可以顯著降低鎘積累,但在某些種植條件下,代價為小幅產量損失;通過本研究獲得的低鎘基因敲除品系在鎘污染地區具有較好利用潛力。
秈稻;鎘;基因編輯;育種
水稻是一種易于富集鎘的作物[1],而耕地酸化或污染導致土壤中活性鎘含量增加,又加劇了水稻中鎘的積累[2],導致稻米鎘超標事件頻發。過量攝入鎘嚴重危害身體健康[3],在我國南方部分地區,稻米鎘超標已經成為引起社會廣泛關注的食品安全問題。耕作方式的變革以及污染土壤的修復是解決稻米鎘污染問題的根本途徑,而選擇種植鎘低積累水稻品種可起到立竿見影的效果,是目前低成本快速解決稻米鎘污染問題的最佳選擇。研究者從現有水稻品種資源中篩選獲得了一些鎘低積累水稻品種[4-7],這可以解決部分稻米鎘污染的問題,然而受水稻品種的生態適應性限制,獲得的低鎘品種資源不適合在所有鎘污染區推廣,或者獲得的資源降鎘幅度有限,不適合在鎘污染程度較重的地區種植,因此生產中迫切需要適應不同地區生產需求的低鎘水稻新品種。
水稻中鎘吸收轉運相關蛋白及調控因子的鑒定為育種家通過分子手段快速培育低鎘水稻新品種奠定了基礎[2, 8-9]。研究顯示一些關鍵的鎘吸收轉運蛋白編碼基因可作為分子操作對象以創制低鎘水稻品種。OsHMA2[10-12]是一種鋅的轉運蛋白,參與鎘從根部向莖部的轉移,同時在莖節中通過韌皮部向發育器官轉運鎘,其功能缺失可使鎘和鋅同時減少,而且嚴重影響生長和產量,但過表達可增加籽粒中鋅含量并一定程度降低鎘積累;OsHMA3[13]是一種鎘的轉運蛋白,位于液泡膜上向液泡內轉運鎘,其功能是限制鎘從根向莖葉轉移從而將其截留在根內液泡中,此基因的超量表達可大幅降低籽粒鎘含量,而功能缺失將使地上部分鎘猛增;OsLCT1[14]亦是一種鎘的細胞膜離子外排轉運蛋白,同時可轉運鈣、鎂、鉀和錳,其參與鎘從莖葉向籽粒轉移過程,通過RNA干涉抑制其功能可使籽粒中鎘含量減少約50%,同時鐵含量增加,而且不影響植物生長及產量,亦不影響其他有益礦物元素的積累;OsLCD[15]的具體功能未知,其功能缺失可降低籽粒中約50%的鎘含量,不影響生長和產量;OsNramp5[16-18]是一種錳和鐵的轉運蛋白,是根中鎘吸收的主要蛋白,其功能缺失可使籽粒中鎘含量大幅降低(含量不到對照的3%),同時錳含量也降低一半以上,因品種或種植條件不同,OsNramp5功能缺失突變體可表現出大幅減產或產量不受影響兩種截然不同的結果。
CRISPR/Cas9等基因編輯技術的快速發展使得植物基因功能缺失突變體的創制變得十分便利[19],因此,可以通過基因編輯技術創制鎘吸收蛋白編碼基因的突變體以快速培育低鎘水稻品種。由前所述,、和基因功能缺失可使水稻籽粒鎘積累減少,而且在一定條件下對其他有益元素的積累和產量無影響或影響較小,因而可以作為基因敲除靶標以創制低鎘水稻品種。本研究旨在解決南方稻米嚴重鎘超標(如鎘含量1 mg/kg以上)地區的水稻生產問題,故選擇缺失可大幅降鎘的基因作為靶標,通過CRISPR/Cas9方法對其進行基因敲除,檢測基因失活對鎘和其他元素積累的影響,并考查突變體株高、產量和品質性狀的變化,評價其利用價值。
1 材料與方法
1.1 水稻品種
Kasalath由南京農業大學水稻研究所提供;華占和五豐B分別源于中國水稻研究所和廣東農業科學院水稻研究所;五山絲苗和中早35購自種子公司。
1.2 載體構建
CRISPR/Cas9載體為自行構建,載體以雙元載體pCUbi1390(由pCAMBIA1390骨架上添加玉米Ubiquitin 3啟動子構建而成)[20]為基礎,在I和HⅠ位點處插入水稻密碼子優化過的Cas9基因,繼續在Ubiquitin 3啟動子前的d Ⅲ位點插入方向與基因同向的水稻U6或U3啟動子和sgRNA骨架構件(構件中啟動子和sgRNA框架之間插入一段兩端為Ⅰ位點的R-基因表達盒以便于克隆sgRNA向導序列片段;構件中包含U6或U3啟動子的轉錄起始位點堿基G或A),最終載體命名為pCUbi1390Cas9-U6和pCUbi1390Cas9-U3(圖1),載體在大腸桿菌ccdB Survival? 2 T1R菌株(Invitrogen公司)中保存。其中,含有水稻密碼子優化過的Cas9基因原始載體由瞿禮嘉實驗室惠贈[21];水稻U6和U3啟動子擴增自日本晴基因組DNA,分別參考Feng等[22]和Miao等[21]的數據;sgRNA骨架來自張峰實驗室[23];R-基因表達盒來自從Invitrogen公司購置的gateway載體pENTR 1A。
利用在線工具CRISPR-P(http://cbi.hzau.edu.cn/ crispr/)[24, 25]設計用于敲除基因(RAP ID: Os07g0257200)的sgRNA向導序列。選擇兩個靶位點,均位于基因第10外顯子的正義鏈上[基因組ORF位置:3673?3692(位點1)和3685?3704(位點2),mRNA CDS位置:1006?1025(位點1)和1018?1037(位點2)]。構建載體時,先根據載體構建要求合成針對兩個靶位點的向導序列DNA接頭寡核酸,針對位點1為cttgGGCAGAGCTCC ACTATTAC(5′-3′;正義鏈)和aaacGTAATAGTGGA GCTCTGCC(5′-3′;反義鏈);針對位點2為ctagACTATTACCGGCACATACGCTGG(5′-3′;正義鏈)和tcgaCCAGCGTATGTGCCGGTAATAGT(5′-3′;反義鏈)。將兩條寡核酸退火后即為接頭,將接頭連接到用Ⅰ酶(Thermo scientific?;貨號ER1581)線性化的CRISPR/Cas9載體(第一個位點使用pCUbi1390Cas9-U6,第二個位點使用pCUbi1390Cas9-U3)后轉化大腸桿菌,挑單克隆測序,測序引物為OsU6-F(TTGAGCGATTACAGGC GAAAGTG;用于pCUbi1390Cas9-U6載體,檢測向導序列的正確性),OsU3-F(GGCATGCATG GATCTTGGAGGAAT;用于pCUbi1390Cas9-U3載體,檢測向導序列正確性),35S-F(TGACGCA CAATCCCACTATCCTTC;檢測載體完整性),Cas9-R-1(TCGAGCCTGCGGGACTTAGAG;Cas9 5′端引物,檢測載體完整性);C126(TCGTGA AGAAGACCGAGGTT;Cas9 3′端引物,檢測載體完整性,此引物由Miao等設計[21]),選取正確克隆擴大繁殖并提取質粒備用。
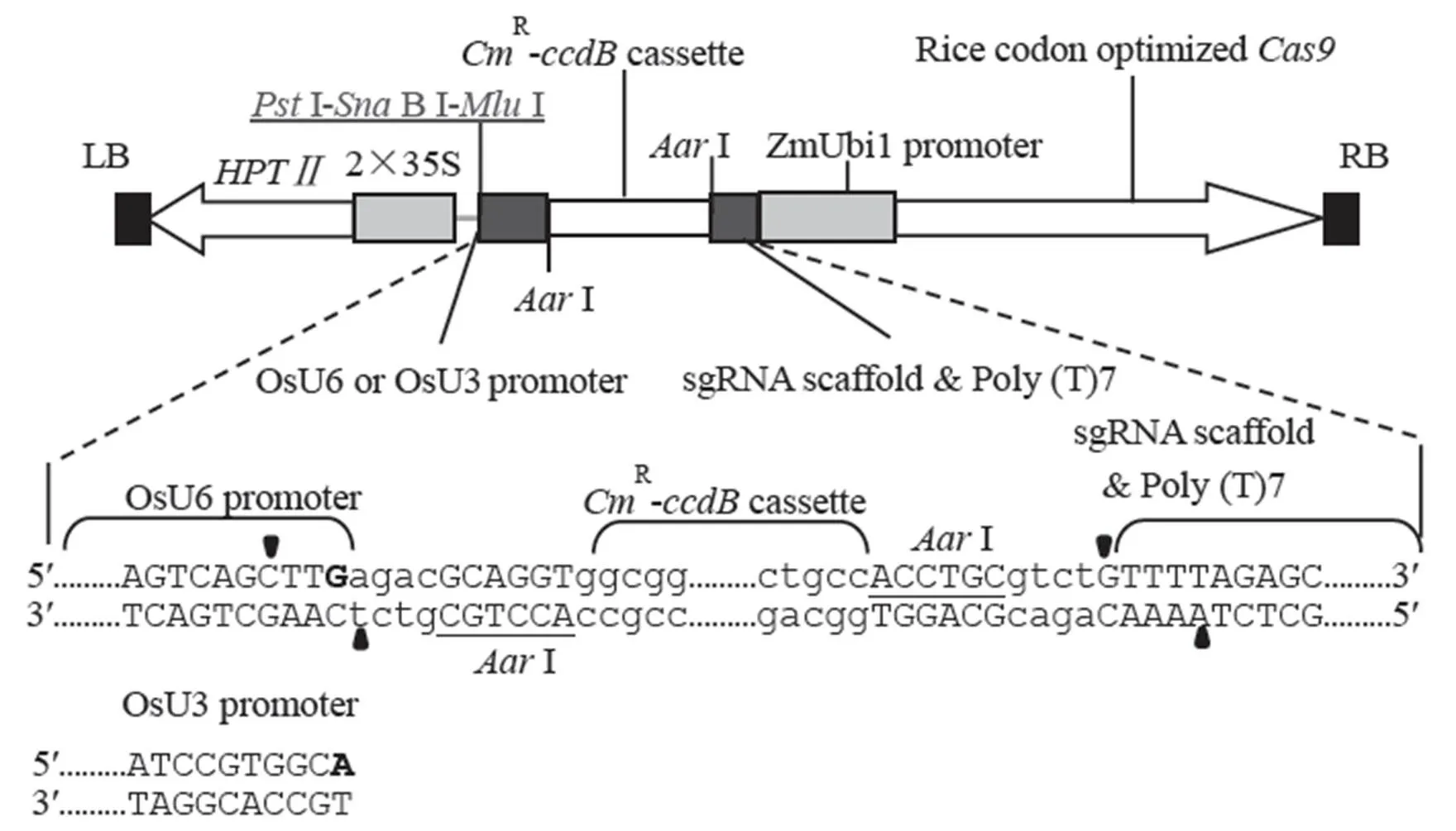
LB、RB分別為T-DNA的左、右邊界;HPTⅡ?潮霉素抗性標記;2×35S?串聯的35S啟動子;CmR-ccdBcassette?氯霉素抗性基因及自殺基因表達盒;sgRNA scaffold & Poly (T)7?sgRNA骨架及7個T堿基組成的終止子;ZmUbi1 promoter?玉米Ubi1基因啟動子;Rice codon optimized Cas9?水稻密碼子優化的Cas9基因;AarⅠ?供向導序列克隆的TypeⅡ限制性酶識別位點(黑色三角示剪切點);PstⅠ-Sna BⅠ-MluⅠ?三個單一限制性內切酶識別位點;圖中省略了HPT和Cas9基因各自表達所需的終止子CaMV 3′ UTR和Nos polyA信號序列;pCUbi1390Cas9-U6和pCUbi1390Cas9-U3載體的區別僅在于sgRNA啟動子及轉錄起始堿基不同。
Fig. 1. Structure of the T-DNA region of the CRISPR/Cas9 Ti binary vectors pCUbi1390Cas9-U6/U3.
1.3 遺傳轉化
將構建好的Ti雙元質粒載體轉化至EHA105農桿菌中,農桿菌感受態細胞制備方法采用CaCl2法,轉化采用反復凍融法[26]。對于品種Kasalath,遺傳轉化采用Toki等的方法進行[27];華占、五豐B、五山絲苗和中早35的轉化委托武漢伯遠生物科技有限公司完成。
1.4 水稻DNA提取
DNA提取用CTAB法[28],省略部分純化步驟。
1.5 轉基因陽性株篩選
用葉片切段潮霉素水溶液浸泡法[29]檢測T-DNA是否插入T0再生苗中,接著檢測篩除T1代轉基因陽性植株。
1.6 CRISPR/Cas9靶位點突變檢測
通過PCR產物直接測序的方法對轉基因陽性植株中的基因敲除靶位點進行初步檢測,若測序結果峰圖中有套峰則認為存在突變,繼續將片段克隆至質粒載體中進行測序,每個樣品至少5個陽性克隆,從中分析出兩個等位基因各自的基因型,若測序結果無套峰,則與對照進行序列比對,獲悉基因型。PCR引物為TTCGTGGCGCTGCTGATAAAC(正向)和AGAGCGGAGAAATATGGACGAAAGT(反向)。PCR體系包括5 μL 10×PCR緩沖液(含Mg2+;TaKaRa),4 μL dNTP混合液(各2.5 mmol/L),5 μL引物(正反向混合,各5 pmol/μL),0.5 μL TaKaRa(5 U/μL),2 μL二甲基亞砜(濃度不低于99.5%),2 μL DNA,31.5 μL H2O,總體積50 μL。PCR程序:95℃下2 min;94℃下30 s,58℃下30 s,72℃下60 s,35個循環;72℃下2 min。PCR產物直接測序所用引物為(擴增片段內,位于反義鏈)GTGCACCCCTACAATTCGTCAGT。測序委托杭州擎科生物技術有限公司完成。
1.7 元素含量分析
鎘、錳、鐵、銅、鉛、鉻、汞、砷、硒、鋅和鈣元素含量測定委托具有資質的專業第三方檢測機構青島科創質量檢測有限公司完成。田間按株系及姊妹系各自混收稻谷,脫殼成糙米(曬干,水分12%左右)后檢測,每份樣品不少于20 g,將樣品進行粉碎(過100目篩)后送檢。Cd測定樣品中部分抽取2個重復樣檢測,最后取其均值作測定值,其余樣品只測定一份樣品。元素檢測方法采用ICP-MS(電感耦合等離子體質譜)法。
1.8 材料繁殖及農藝性狀測定
T0代植株溫室內盆缽種植;T1分離群體在有防護措施的網室內種植,開花前篩選后拔除轉基因植株;已去除轉基因成分的T2或T3植株在大田種植,并用塑料薄膜墻圍住隔離;供鎘測定材料在晚季種植(2017年6?11月),供農藝性狀考查的中早35和五豐B品系作早稻(2018年4?7月)種植,華占和五山絲苗作中稻種植(2018年5?10月)。所有世代種植行距20 cm、株距17 cm,每行8株。元素含量測定所用材料種植時野生型對照設3次重復,在田間均勻排布,基因敲除株系按株系順序種植,同一轉化事件后代攜相同變異姊妹系視為重復;農藝性狀調查所用材料種植時不設重復。所有材料均按大田常規方法進行肥水管理。株高測定取莖稈基部至最長穗頂部的長度,有效分蘗測定取穗子實粒不少于5粒的分蘗,株高和有效分蘗數同時測定,連續測定一定株數(一般大于40株),邊株不測定;每穗總粒數和實粒數測定取樣10株,取樣時連續選10行,每行取一株,逐行錯位選株(即第1,2,3,4,5,6,7,8,9和10行分取第2,3,4,5,6,7,6,5,4和3株),邊株不取,穗子混合,每穗分別統計;千粒重測定取曬干稻谷去除空癟粒和雜質后用自動數粒儀數1000粒,重復2次;米質測定采用株系內所有植株的混合稻谷,經研磨成精米后測定,外觀品質指標通過萬深SC-E大米外觀品質檢測分析儀進行測定,其中,堊白度指堊白米粒中堊白面積與整粒米面積的比值,水分、蛋白含量、直鏈淀粉含量和食味分用近紅外分析儀檢測,儀器為InfratecTM1241(Foss),定量模型為廠商提供并經過用戶矯正,數值仍為估計值。
1.9 HRM分析
高分辨率溶解曲線分析利用Lightscanner 96系統(Idaho科技公司)進行,采用小片段擴增法。PCR引物及擴增子為CTGGGCAAGTCGAGTGCGAT(正向)和ATTACCTGCATGATGTACTGTCC(反向),擴增產物104 bp。PCR體系包括1 μL 10×PCR 緩沖液(含Mg2+;TaKaRa),0.8 μL dNTP混合液(各2.5 mmol/L),1 μL引物(正反向各5 pmol/μL),0.1 μL TaKaRa(5 U/μL),0.4 μL 二甲基亞砜(濃度不低于99.5%),1 μL DNA,1 μL LC-Green熒光染料,4.7 μL H2O,總體積10 μL,上加20 μL礦物油。PCR程序:94℃下2 min;95℃下5 s,60℃下20 s,72℃下20 s,共50個循環;72℃下1 min;95℃下3 min;16℃下2 min。將PCR體系連同石蠟油全部轉入專用PCR板(Bio-rad,貨號hsp9665)中進行HRM分析,分析溫度范圍為65℃~95℃。用系統自帶軟件分析數據。
1.10 數據分析
數據分析利用Microsoft Excel 2010及其共享Real Statistics統計插件(http://www.real-statistics. com/)進行。統計分析時,除特殊注明外,若比較分析中只含2組樣本數據,直接進行測驗檢測方差同質性后利用Excel自帶函數進行檢測(根據測驗結果選等方差或異方差模式);若包含3組以上樣本數據,則進行多重比較,利用Real Statistics統計插件行中位數版Levene檢驗檢測方差同質性,若同質后行方差分析(ANOVA)檢測組間差異,若結果顯著,采用Dunnett檢驗檢測突變體與對照差異;若方差不同質,采用Welch ANOVA檢測組間差異,若顯著,則采用Games Howell test檢測突變體與對照的差異顯著性。
不同品種突變體與對照相比的不同指標增減率數據為品種內各株系的平均值與各自野生型對照的平均值比較而得,4個品種總的增減率為4個品種各自平均增減率的均值。
2 結果與分析
2.1 OsNramp5基因編輯水稻品系的獲得
先以品種Kasalath為材料試驗CRISPR/Cas9系統對基因兩個靶位點的編輯效果。結果顯示在兩個靶位點中均成功創制突變,選擇位點1(CRISPR/Cas9系統中使用U6啟動子)作為進一步試驗的基因編輯靶位點,擴大試驗對象至純系品種五山絲苗和中早35以及雜種親本華占和五豐B。在針對靶位點1的試驗中,5個品種一共獲得97個獨立轉基因株系,對T0代(轉基因當代定義為T0代)植株的測序顯示,不同品種基因突變效率不同,突變率(突變轉化子數/總轉化子數×100%)為39%~90%,平均為58%(表1)。對這5個品種一共56個獨立基因編輯株系中的106個突變事件進行了分析,結果顯示一共出現了32種突變基因型(圖2-A),理論上其中25種突變類型為移碼突變,導致蛋白功能失活,7種突變類型(即當缺失或插入的堿基造成的總的堿基變化數為3的整倍數時,包括#05,#07,#12,#13,#17,#22和#24)導致部分氨基酸殘基發生改變,蛋白功能是否受影響未知。每個基因編輯株系所攜突變基因型見表2。針對靶位點2的基因編輯結果也在表1、表2和圖2-B中顯示。
進一步對靶位點1所有突變基因型的發生頻率進行了統計(圖2-C),發現主要發生的突變類型為缺失1~3個堿基和插入1個堿基的突變,占所有突變的66%,而且在這3種缺失突變中缺失的堿基位置并不具隨機性,而是缺失特定位置的堿基,而在插入1個堿基的突變類型中又以插入堿基A和T最為常見,未發現插入堿基C的突變類型;缺失5個堿基及以上的缺失突變以及插入缺失型的突變中每一種突變類型一般只發生1次,極少數發生2次。
在轉基因T1代通過葉片潮霉素抗性篩選獲得去除轉基因成分的植株,對這些植株中的靶位點進行測序檢測,篩選獲得純合突變株,利用這些純合株的后代(T2或T3)進行后續試驗。目前為止除品種Kasalath外,所有品種的多數突變基因型都獲得了純合后代,詳細信息見表2。
2.2 OsNramp5基因敲除降低水稻籽粒中鎘積累
在獲得去除轉基因成分的基因突變株系后,將品種華占、五豐B、五山絲苗和中早35的部分基因突變株系(T2代,除華占品系L01-#05/#05外余均為移碼突變)及其野生型對照分別在江西省A、B兩地種植(A地為鎘中等污染土壤,而B地為非污染土壤)。分別對從兩地種植獲得的種子(糙米)進行鎘含量測定,測試結果見表3。由表中數據可知,A、B兩地種植的基因編輯株系種子鎘含量較對照均大幅下降。在A地種植時,在對照品種鎘含量嚴重超標——約5~10倍于國標限定值(0.2 mg/kg;GB2762?2017)的情況下,不同品種敲除株系的鎘含量不及限定值的1/2,比對照降低91.7%~96.9%(4個品種的均值為94.8%);在B地種植時,對照和基因敲除株系鎘含量均低于國標限定值,其中不同品種基因敲除株系比對照降低76.2%~91.1%(均值85.5%),不到國標限定值的1/10。值得一提的是,華占品系L01-#05/#05鎘含量的降低與在其他品系相同,因此,雖然此株系內氨基酸序列只發生幾個氨基酸殘基的改變,同樣造成了基因功能的失活。總而言之,對于不同類型的水稻品種,敲除基因均可有效降低籽粒鎘積累。
表3數據亦顯示,無論在何地種植,不同品種野生型本身鎘積累存在一定差異,尤其是中早35,其鎘含量明顯比其他3個品種低,而敲除基因后,不同品種的鎘積累量變得相對一致。這一結果暗示了基因的功能差異可能是引起這些品種鎘積累差異的關鍵原因。
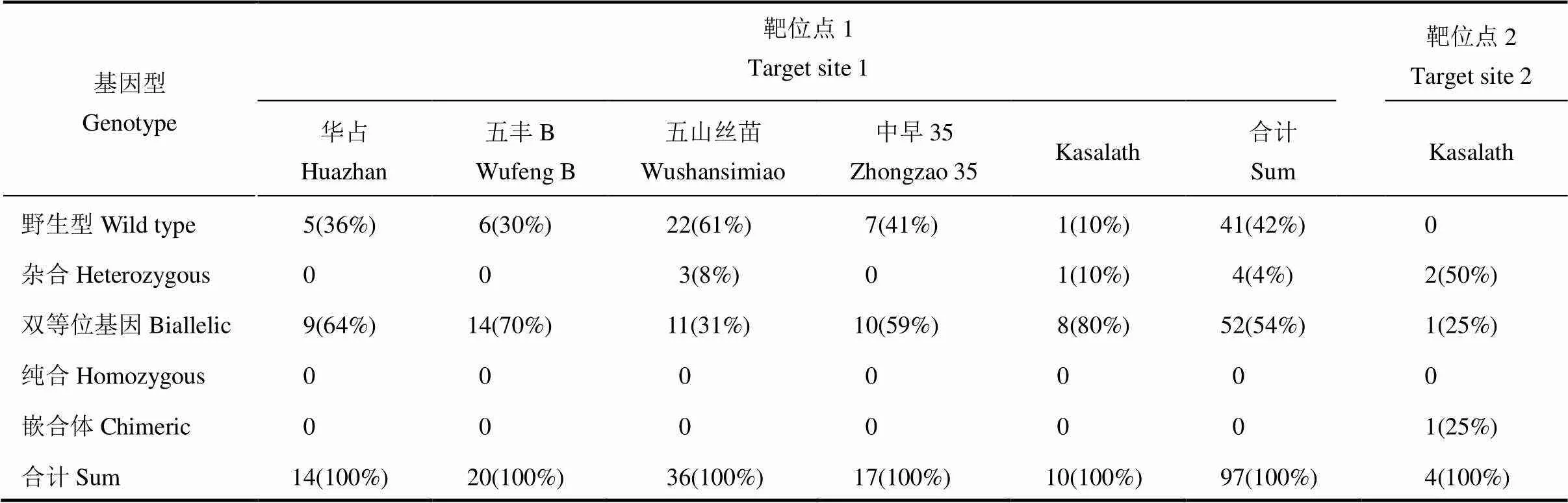
表1 不同品種陽性轉化株基因突變情況統計
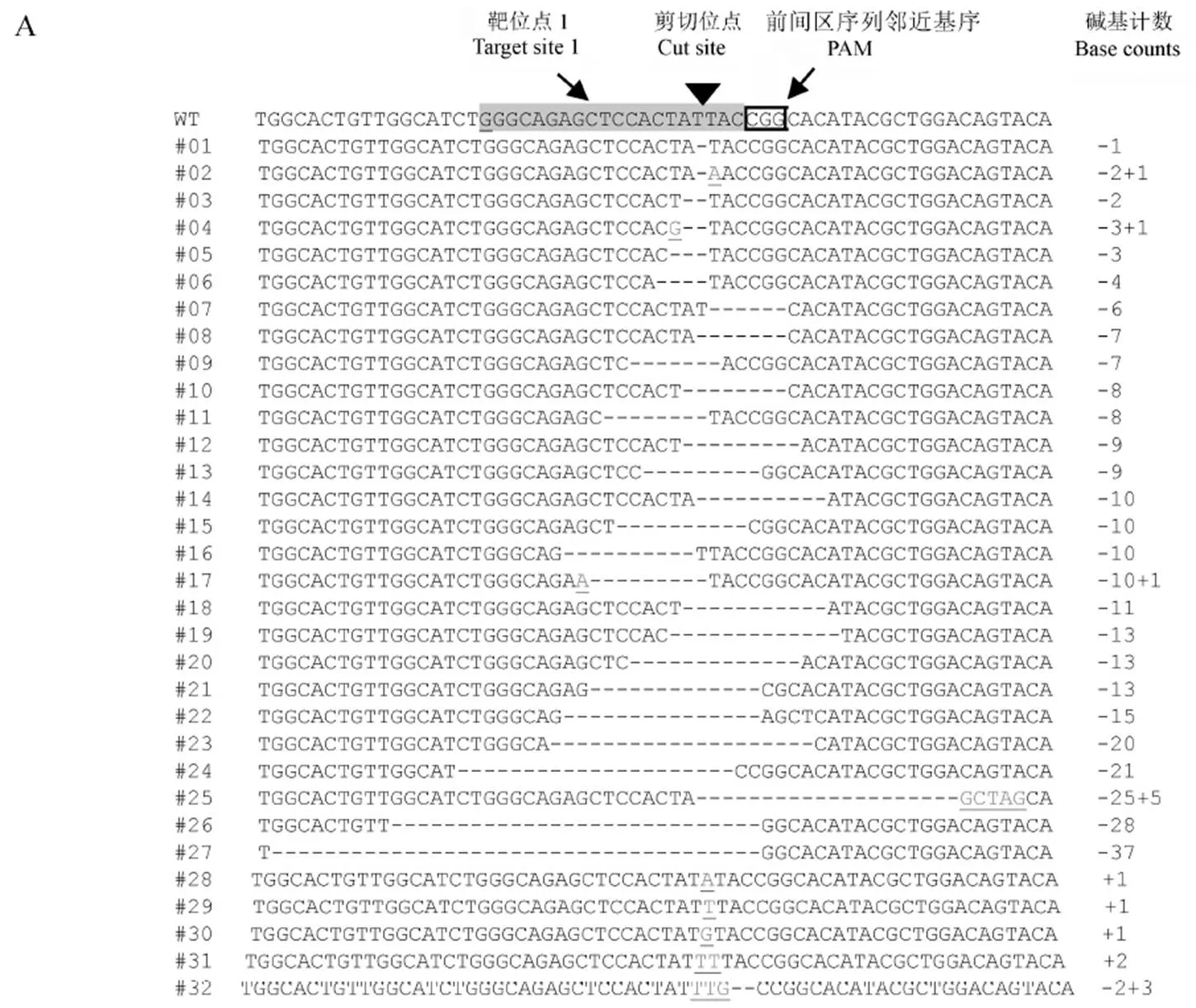
A和B,靶位點1和2的序列突變情況。WT?野生型;#**或##**,突變類型編號;野生型序列中Cas9靶位點、Cas9系統中U6和U3啟動子A圖和B圖分別為靶位點1和靶位點2的突變情況。起始轉錄堿基以及PAM位點分別用灰色陰影、加下劃線及加框標示,黑色倒三角指示Cas9切割位點,所有序列方向均為5′-3′;突變體序列中缺失堿基用短線代替,插入堿基加下劃線顯示,其余為與野生型相同者;圖右邊數字表示缺失(用“?”表示)或插入(用“+”表示)的堿基數。C?靶位點1不同突變類型發生頻率統計。突變體類型分3類,即包含缺失1 bp、2 bp、3 bp、4 bp和≥6 bp六種類型的缺失突變,包含插入1個堿基A、T或G及插入2個堿基TT四種類型的插入突變,以及同時含有缺失和插入即InDel;一共統計56個獨立轉化株系,106個突變等位基因。
A and B, The sequence changes in Target sites 1 and 2 of thegene, respectively. WT, Wild type; #** or ##**, Mutation type numbers; In the WT sequence, the target site, the cut site and the PAM site are indicated with the bases highlighted in grey (The underlined base “G” or “A” indicates the transcription start site of the rice U6 or U3 promoter in the CRISPR/Cas9 system), reverse black triangle and the bases in underlined bold font, respectively; In the mutation sequences, the deleted bases are indicated by dashed lines, the inserted ones are underlined, and those identical with the wild type are shown normally; The numbers on the right indicate the counts of the deleted or inserted bases. C, The occurrence frequency of various mutation types in Target site 1. The mutations were summarized as three types, which are deletions, insertions and InDels. The deletion type includes five subtypes, which are those with 1 bp, 2 bp, 3 bp, 4 bp and ≥6 bp deletion respectively. The insertion type includes four subtypes, which are those with one A, one T, one G and double T insertion, respectively. The data are derived from the analysis of 106 mutation alleles from 56 independent transgenic lines.
圖2 基因編輯株系中基因靶位點突變情況
Fig. 2. Target site mutations ofin the genetically edited lines.
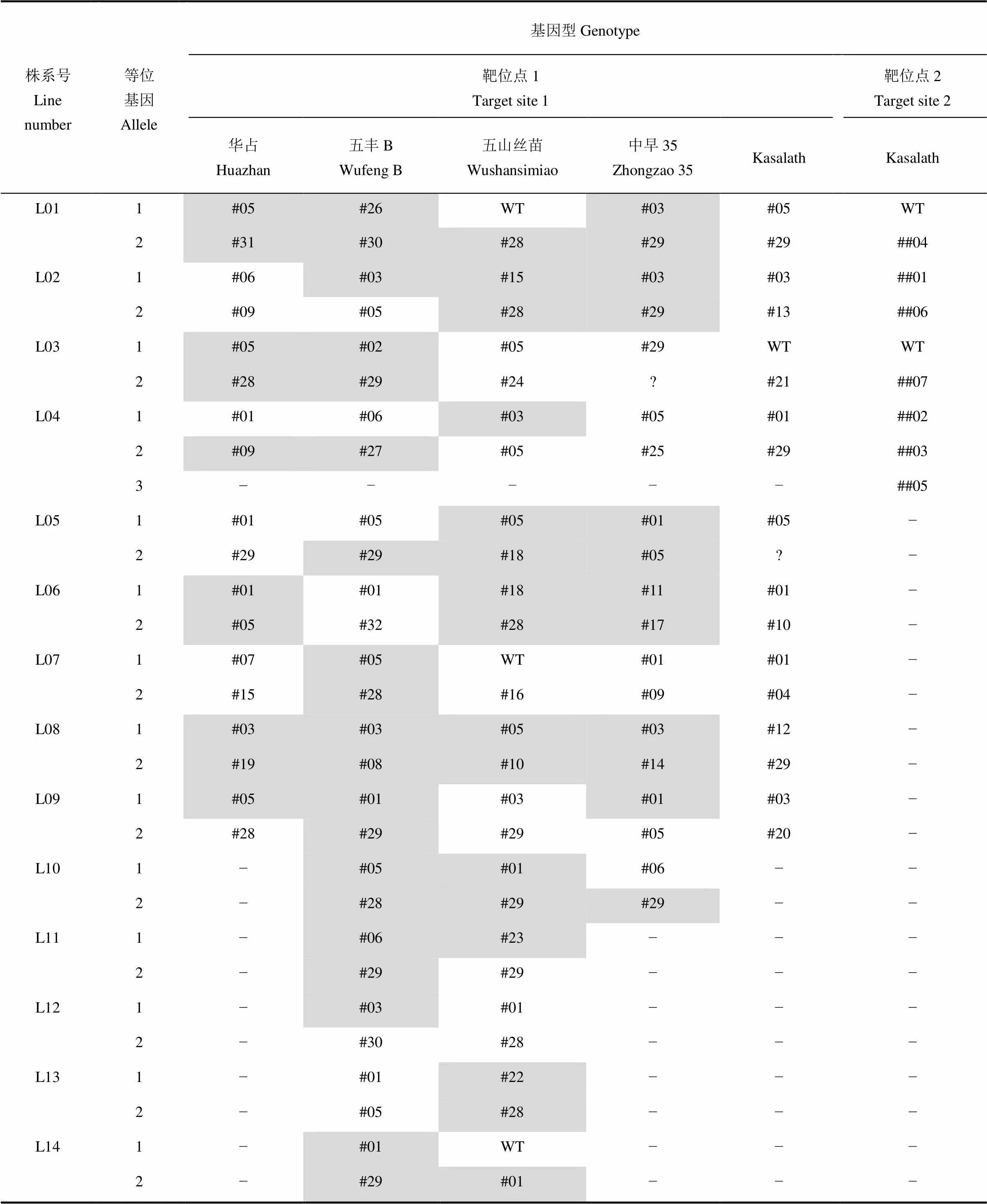
表2 五個品種基因編輯株系中OsNramp5靶位點的基因型
灰色底紋標識的單元格表示該基因型已經分離到去除標記且純合的植株;其他基因型正在分離純合植株;?,未確定;?,不適用。
The grey shading contents show that marker-free homozygous plants are available for the genotypes. For the other genotypes, the work to seek such plants was in progress; ?, Not resolved; ?, Not applicable.
2.3 OsNramp5基因敲除對籽粒中其他元素積累的影響
對鎘污染土壤(A地)種植的基因敲除株系及對照進行籽粒鎘含量測定的同時,也對錳、鐵、銅、鉛和鉻元素含量進行了測定。由表3可知,基因敲除后,所有品種錳含量均顯著降低,減幅34.2%~63.1%(均值52.7%);與此相反,鐵和鉻含量一定程度增加,不同品種鐵含量增幅11.4%~34.5%(均值21.4%),而鉻含量增幅更大,達到45.6%~75.5%(均值59.5%),但是,差異顯著性測驗結果顯示,無論是鐵還是鉻,這種增加只在部分品種中顯著;鉛元素含量在華占中顯著增加,增幅達79.1%,由于其值已略微超出國家限定值(0.2 mg/kg;GB2762?2017),這種變化值得關注,而其他品種鉛含量并未因基因敲除而顯著改變;銅元素含量變化較小(僅在中早35基因敲除株系中減少了13.3%)。此外,對華占基因敲除株系及野生型的汞、砷、硒、鋅和鈣元素含量進行檢測(數據未顯示),其中汞元素含量在所有樣品中均低于所用方法的檢測下限(30 μg/kg),因而基因敲除對汞積累的影響未知,而砷、硒、鋅和鈣的含量也無顯著變化。
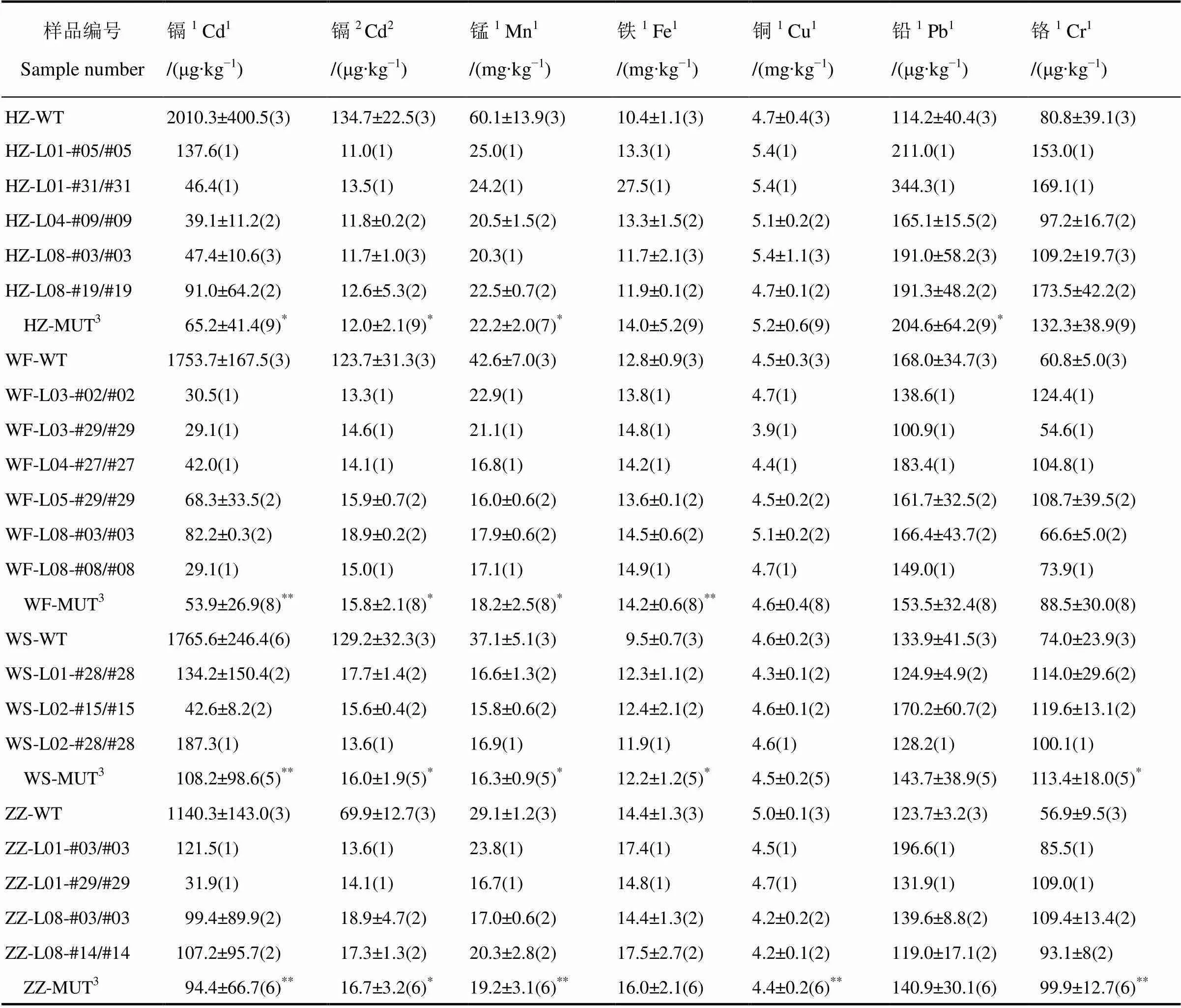
表3 OsNramp5基因敲除株系籽粒的礦物元素含量變化
1在鎘污染土壤種植;2在正常土壤種植;3數據為相同品種不同突變基因型觀測值的均值。樣品編號格式為“品種名縮寫-株系號-基因型”,具體信息見表2和圖2。HZ?華占;WF?五豐B;WS?五山絲苗;ZZ?中早35;WT?野生型;MUT?OsNramp5基因敲除株系。
數據格式為“均值±標準差(n)”或“均值(n)”;*,P<0.05;**,P<0.01;差異顯著性通過t測驗(雙樣本等方差或雙樣本異方差)進行檢測(先進行F測驗檢測方差齊性),統計分析時假設相同品種不同株系相互之間等同。
1Grown in a Cd-contaminated field;2Grown in a non-Cd-contaminated field;3Means of all the observed values of the genetic editing lines. The sample name is “cultivar name abbreviation-line number-genotype”, and one may refer to Table 2 and Fig. 2 for the information of the line numbers and genotypes. HZ, Huazhan; WF, Wufeng B; WS, Wushansimiao; ZZ, Zhongzao 35; WT, Wild type; MUT, Mutants(All the OsNramp5 mutation lines).
Values are “Mean±SD(n)” or “Mean(n)”;*, P<0.05; **, P<0.01; The significance was determined by Student’s t test (types: equal variances or unequal variances) after F test for variance homogeneity. In the analysis of statistical significance, all the genetic editing lines within one cultivar were considered to be identical.
被敲除后,水稻籽粒中鎘含量的大幅下降,錳含量亦減少一半左右,而鉻含量增加一半以上,鐵、鋅、鈣、硒、砷和銅積累受影響較小或不受影響,鉛可能在個別品種中有一定程度增加。
2.4 OsNramp5基因敲除對農藝性狀的影響
為了檢測敲除對農藝性狀的影響,對大田(非鎘污染土壤)種植的部分低鎘基因敲除株系(T3代)及野生型對照的株高和產量性狀進行了測定(表4)。4個品種基因敲除株系(同一品種內所有株系的指標均值)與對照相比發生了如下變化:株高稍降低,降幅3.5%~6.8%(4個品種的降幅均值為4.9%),所有株系與對照相比均差異顯著;有效分蘗呈增加趨勢,增幅1.9%~9.0%(均值5.5%),但由于分蘗性狀誤差較大,可能差異為誤差所掩蓋,差異并不顯著,但由于調查的樣本較大,我們認為這種增加是真實存在的;每穗總粒數變化無明顯規律;每穗實粒數、結實率和千粒重均呈下降趨勢,降幅分別為6.5%~10.3%(均值7.9%)、3.2%~9.0%(中早35基因敲除株系除外;均值5.3%)和1.4%~5.5%(均值3.4%),不過這種下降并非所有株系都差異顯著。通過計算理論產量,發現4個品種減產5.4%~9.4%(均值6.9%)。概而言之,敲除可引起株高、實粒數、結實率和千粒重輕度下降,而有效分蘗小幅增加,最終導致不容忽視的小幅減產。
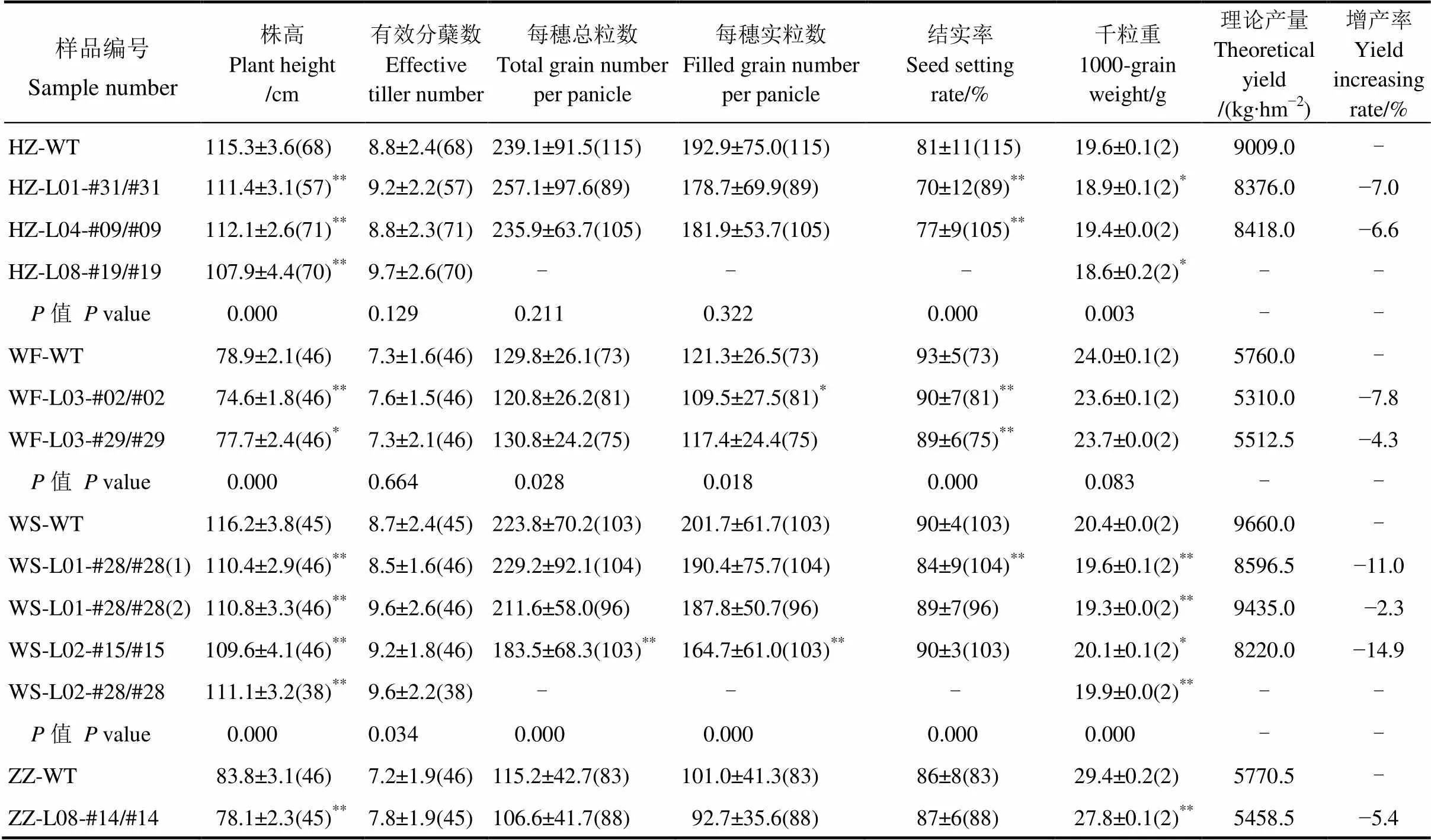
表4 OsNramp5基因敲除對株高、產量性狀的影響
數據格式為均值±標準差();*,<0.05;**,<0.01;?,未檢測或不適用;突變體與野生型之間顯著性測驗視適用情況采用測驗(含兩組數據時用;行測驗比較方差同質性后進行)或方差分析加多重比較(含三組以上數據時用;等方差時用ANOVA+Dunnett檢驗,異方差時用Welch ANOVA +Games-Howell test,組間方差齊性用中位數版Levene檢驗進行檢測,千粒重數據因<3未檢測方差齊性,視為等方差);結實率數據經平方根反正弦轉換后進行方差分析;產量依據“實粒數×有效分蘗數×千粒重/1000×270 000株/1000”估算;樣品編號格式為“品種名縮寫-株系號-基因型”,基因型WS-L01-#28/#28測試兩個姊妹系。品種名縮寫:HZ?華占;WF?五豐B;WS?五山絲苗;ZZ?中早35;WT?野生型。株系號見表2,基因型編號見圖2。
Values are Mean± SD ();*,<0.05;**,<0.01; ?,Not applicable or not determined; the significances between mutants and wild types were determined bytests aftertests for variance homogeneity (for comparisons with two groups of samples and for comparisons of the 1000-grain weight trait) or by ANOVA with post hoc multiple comparison analysis (for comparisons with three or more groups of samples; ANOVA + Dunnett’stests for groups with equal variances; Welch’s ANOVA + Games-Howell tests for groups with unequal variance; Median-edition Levene’s test was used to determine the homogeneity of group variances expect those of 1000-grain weight because of<3; groups of 1000-grain weight data were viewed as having equal variances); arcsine square root transformation for the data of seed setting rate was performed before ANOVA; the theoretical yield was estimated by “filled grain number per panicle × effective tiller number × 1000-grain weight / 1000 × 270 000 / 1000”. The sample name is “cultivar name abbreviation-line number-genotype”, and the information of the line numbers and genotypes is available in Table 2 and Fig. 2. Two sister lines were analyzed for the genotype WS-L01-#28/#28. Abbreviations of cultivar names: HZ, Huazhan; WF, Wufeng B; WS, Wushansimiao; ZZ, Zhongzao 35; WT, Wild type. The sample name is “cultivar name abbreviation-line number-genotype”. One may refer to Table 2 and Fig. 2 for the information of the line numbers and genotypes.
為了檢測敲除對大米品質的影響,我們亦對敲除突變體及對照米樣的外觀品質進行了測定,同時利用近紅外分析對蛋白含量、直鏈淀粉含量及食味進行了估計(表5)。4個品種敲除株系與對照相比,粒長輕微增加(增幅0.4%~2.7%,均值1.5%),粒寬略微變窄(減幅0.9%~3.9%,均值2.5%),最終所有株系均表現出長寬比輕微增加(增幅1.8%~5.0%,均值4.0%),而這種長寬比的增加引起堊白粒率和堊白度的小幅下降(降幅分別為6.7%~22.6%和0.6%~17.6%,均值分別為13.3%和10.0%)。食味指標測定結果顯示,與對照相比,4個品種敲除株系中蛋白質含量輕微增加(增幅1.8%~4.3%,均值3.2%),直鏈淀粉含量除品種五山絲苗外,其余品種均不同程度下降(降幅3.5%~12.6%,均值9.5%),而食味分幾乎不受敲除的影響(只有五山絲苗品種數據)。總之,敲除能引起籽粒長寬比小幅增加并一定程度降低堊白發生,同時引起蛋白含量輕微增加,而且在一些品種中導致直鏈淀粉含量小幅下降,對食味分幾乎無影。從應用角度來看,敲除對米質的影響是有利的。
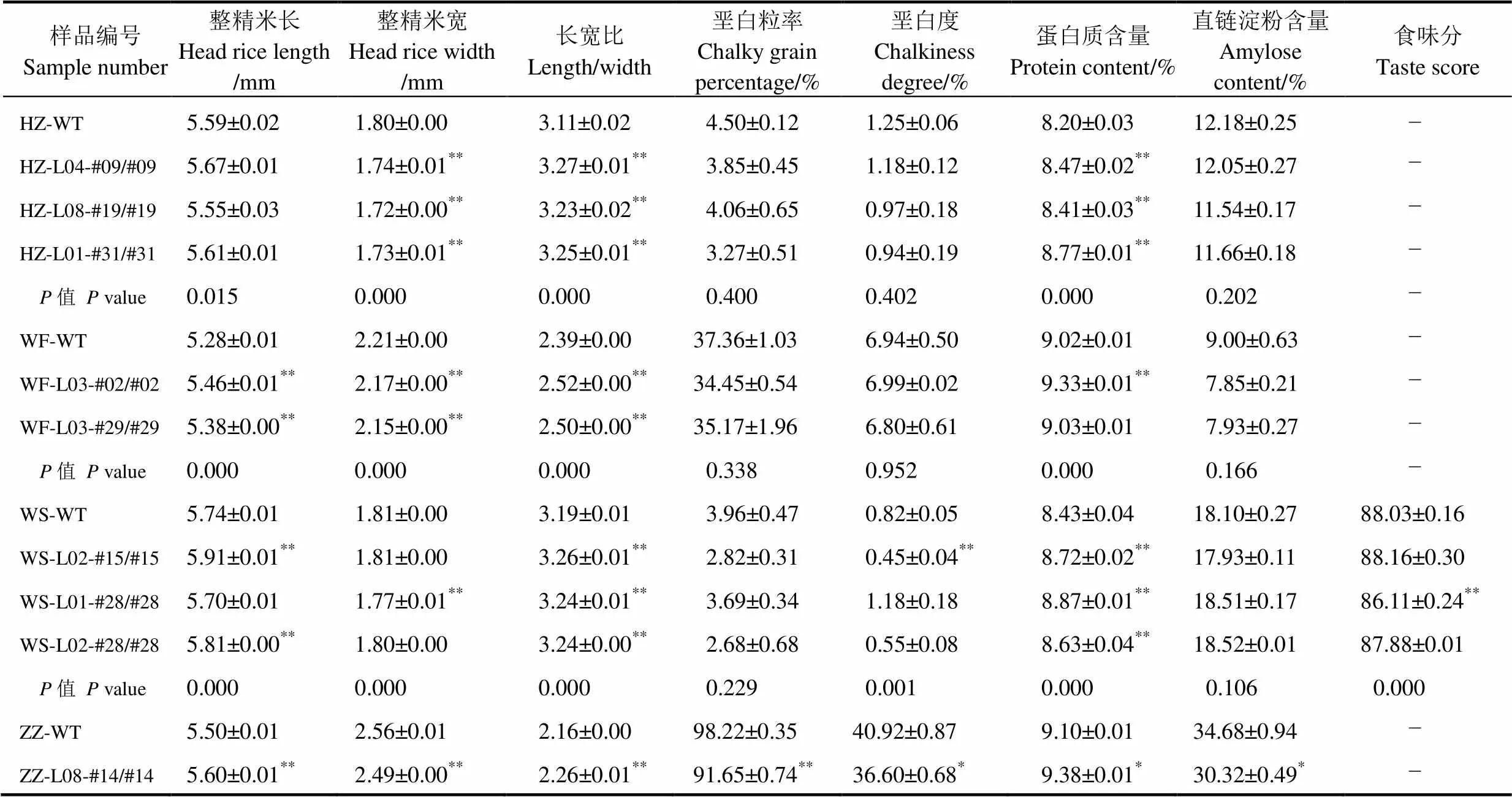
表5 OsNramp5基因敲除株系的米質變化
數據格式為均值±標準誤,重復3~4次,外觀品質每次分析整精米500粒以上,食味品質每次測定取5個次級樣品的平均值;?,含量超出模型檢測范圍;*,<0.05;**,<0.01。數據分析及樣品編號格式同表4,其中堊白粒率數據經平方根反正弦轉換后進行方差分析,同一品種樣品水分含量基本相同。品種名縮寫:HZ?華占;WF?五豐B;WS?五山絲苗;ZZ?中早35;WT?野生型。株系號見表2,基因型編號見圖2。
Values are Mean±SE,=3 or 4, for appearance quality more than 500 head rice grains were analyzed in each determination, and for eating quality five subsamples were included in each analysis; ?,Data out of the model range;*,<0.05;**,<0.01. Methods for data analysis are the same as those described in Table 4 and arcsine square root transformation for the data of seed setting rate was performed before ANOVA; The samples within the same cultivar had nearly identical water content. Abbreviations: HZ, Huazhan; WF, Wufeng B; WS, Wushansimiao; ZZ, Zhongzao 35; WT, Wild type. One may refer to Table 2 and Fig. 2 for the information of the line numbers and genotypes.
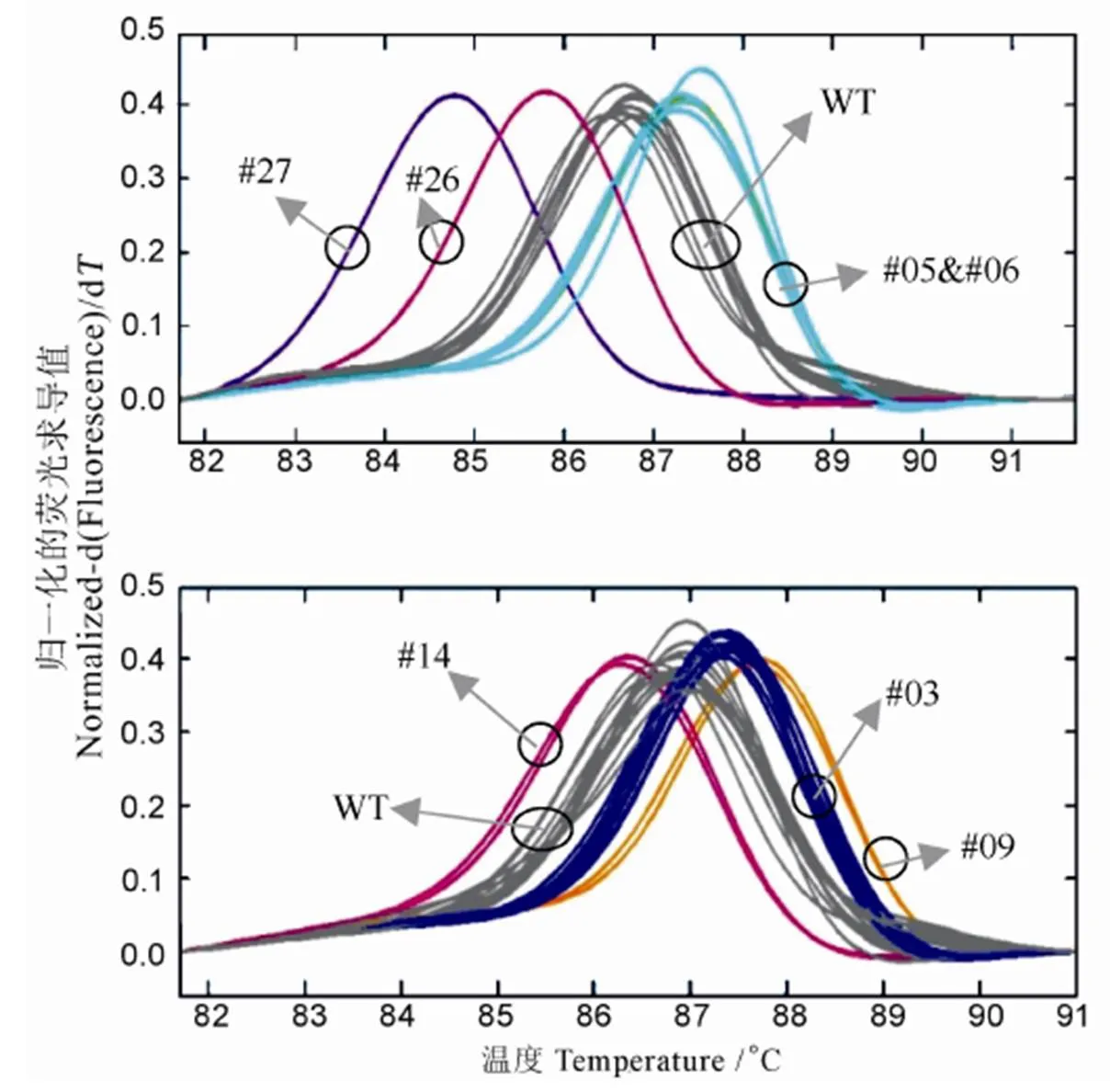
其中基因型WT(上圖)、WT(下圖)、#03、#05、#06、#09、#14、#26和#27的樣品數(n)分別為9、13、11、2、4、2、2、1和1。
Fig. 3. Melting curves of various mutation alleles and the wild type in HRM analysis.
2.5 通過HRM法對OsNramp5基因突變體和野生型進行基因分型
高分辨率溶解曲線(HRM)分析是對SNP和短片段InDel變異進行基因分型的高通量低成本方法。本研究獲得的基因突變體可用作低鎘基因供體以進行其他低鎘新品種的培育。為了在進一步的低鎘性狀轉育工作中采用HRM方法進行基因分型以提高分子標記輔助選擇的效率,本研究進行試驗,以篩選出易于分型的基因突變類型。結果顯示,在粗放檢測條件下(DNA粗放提取、PCR模板濃度不調為一致以及HRM分析時不加溫度內標),編號為#3, #05, #06, #09, #14, #26和#27(圖3)的突變基因型溶解曲線仍然能與野生型較好區分,解鏈溫度(m值)差異為0.5℃~1℃。因此在將來的低鎘性狀轉育工作中若使用HRM進行基因分型,可選擇含有前述幾種基因型的株系作為供體親本。
3 討論
在低鎘水稻創制中,理想的情形是,獲得的低鎘水稻籽粒鎘含量降低的同時其他有害元素含量不增加,有益元素含量不減少,同時農藝性狀較少或不受負面影響,而功能失活突變體似乎符合此種要求。
是水稻根部Mn和Cd吸收的關鍵離子轉運蛋白,亦在Fe吸收中發揮作用,這一發現分別由日本3個獨立研究團隊[16-18]所揭示。其中Ishikawa等[16]的研究顯示突變體在低鎘品種培育中具有重要應用價值,作者利用從粳稻品種越光離子束誘變突變體庫中鑒定獲得的基因功能失活突變體進行研究,發現突變能大幅度降低籽粒及稻草鎘積累(含量<0.1 mg/kg)的同時不改變Fe、Cu和Zn含量,亦不影響包含株高、產量和食味等在內的農藝性狀。而Sasaki等[18]利用粳稻品種中花11 T-DNA插入突變體進行的研究顯示,雖然功能失活使得籽粒及稻草鎘含量大幅度降低,但對農藝性狀的影響與Ishikawa等[16]報道的截然不同,其植株生長受到嚴重抑制并大幅減產(最高減產89%)。突變導致農藝性狀嚴重劣變的現象也為Ishimaru等[17]所報道。作者利用品種Tsukinohikari的RNA干涉株系進行研究,發現Mn缺乏時RNA干涉植株與野生型對照相比生長受到抑制,但Mn正常時無差異,揭示Mn的缺乏是導致農藝性狀劣變的原因。同樣,Sasaki等[18]通過補充Mn部分逆轉苗期生長抑制表型,進一步確認了Mn的缺乏導致了農藝性狀劣變。Ishikawa等[16]和Sasaki等[18]的研究都是在非Cd污染土壤大田條件下評價基因敲除對農藝性狀的影響,顯然兩個團隊的研究結果相互矛盾,這一點雖然Ishikawa等在其論文中已經提及,但并沒有給出解釋。我們認為,這種不一致可能是不同品種對Mn的需求不同所致,提示了當利用功能缺失突變基因對不同品種進行低鎘化改造時應該逐例評價其對農藝性狀的影響。
Tang等[30]通過CRISPR/Cas9技術創制了雜交稻品種親本華占及638S的基因敲除株系,并利用華占基因敲除品系及其與638S基因敲除品系配制獲得的雜種評價了突變對Cd、Mn、Fe、Zn和Cu元素積累以及株高、產量等農藝性狀的影響。在本研究中,我們亦在不同類型的秈稻(即早秈中早35、晚秈五山絲苗、三系恢復系華占和三系保持系五豐B;其中華占品種與Tang等研究中使用的相同)中展示了敲除后水稻籽粒中的Cd含量大幅下降,同時較大幅度下降的還有Mn含量,而Fe、Zn和Cu含量因品種不同而略微改變或不受影響,這與前述日本研究者在粳稻中的研究結果以及Tang等[30]在秈稻中的結果基本一致。與前人的研究不同,我們還測定了基因敲除株系中元素Pb、Cr、As、Se和Ca的含量變化,發現Pb在個別品種(華占)中含量呈增加趨勢,Cr在4個品種中亦不同程度增加,As、Se和Ca含量在檢測的品種華占中并無變化,由于Pb和Cr的轉運機制研究較少,尚不清楚其含量增加的具體原因。本研究顯示個別品種基因敲除株系中Pb含量的增加已經略微超過國標限定值(0.2 mg/kg;GB2762?2017),因而將來利用功能突變基因進行品種低鎘化改造時應該關注Pb含量的變化。Cr是人體必需的元素,由于本研究中籽粒本底Cr水平較低,我們認為基因敲除引起的Cr含量增加并不足以對健康造成危害(中國營養學會發布的2013版《中國居民膳食營養素參考攝入量》中推薦我國成年人總鉻適宜攝入量為30 μg/d;GB2762?2017食品中污染物限量標準規定稻米鉻含量須低于1 mg/kg),但是在Cr污染地區,若本底Cr含量比較高,這種效應就不能忽視,可能導致稻米Cr含量超標。
本研究進一步展示了基因敲除對水稻農藝性狀的負面影響,發現不同品種中株高和產量都小幅度降低,這一結果與Ishikawa等[16]以及Tang等[30]的結果矛盾,而與Sasaki等[18]和Ishimaru等[17]的結果一定程度吻合,只是本研究中產量減少的程度比Sasaki等[18]報道的小得多。值得注意的是,我們和Tang等[30]的研究使用了相同的品種華占為試驗材料,只是敲除靶位點不同,顯然敲除靶位點的差異不大可能是產生這種差異的原因,我們認為可能是種植條件的不同導致了這種矛盾結果。在本研究中,檢測對農藝性狀影響的植株種植在非Cd污染土壤(與Sasaki等[18]和Ishikawa等[16]的研究相同),而Tang等[30]的試驗在嚴重Cd污染土壤上進行,在鎘污染土壤種植時,一方面,由于Cd超量積累對水稻生長不利[31],野生型植株農藝性狀可能受到負面影響,而突變體因鎘積累較低而得以避免,另一方面,突變體株系中存在基因敲除導致的生長抑制效應,而野生型不存在這種效應,因而突變體與野生型中各自遭受的生長抑制程度相似,導致兩者農藝性狀表現無差別,而在一般土壤種植時,由于沒有Cd毒害,突變體與野生型對照的差別就只剩下基因敲除株系因功能失活而引起的生長抑制,因此差異得以顯現。此外,其他種植條件的不同(如Mn的供應)也可能是產生差異結果的原因。事實上,我們將華占和五山絲苗的各自兩個基因敲除株系在地點C種植時(正文提及的地點A、B之外的另一地點;該地稻米鎘含量只是輕微超標),小區測產方產量測定結果顯示突變體與野生型對照幾乎無差別(數據未顯示)。當然,以上猜想需要進一步試驗驗證。總之,不同品種及不同種植條件下,功能突變株系可能較野生型減產或無變化,提示生產中推廣通過基因敲除創制低鎘水稻品種時須謹慎,需要按照新品種的標準重新通過多年多點試驗確定推廣地域。需要指出的是,對于Cd嚴重超標稻米產區,一定程度的減產以換取稻米食用安全也是值得的。
本研究還揭示了當基因敲除導致減產時,產量的降低主要由實粒數、結實率和千粒重降低所致,并為有效分蘗數的增加所部分補償,這是Sasaki等[18]和Ishikawa等[17]的研究中未曾提及的。吳平等[32]利用F2分離群體進行分析的結果顯示,水稻株高和分蘗呈負相關關系。本研究發現基因敲除材料中伴隨株高小幅度降低分蘗略微增多,與前人報道吻合。
Allen等[33]通過在人體細胞中對40 000多個靶位點切割產生的大于1×109個突變序列進行分析揭示了CRISPR/Cas9切割基因組DNA產生雙鏈斷裂后細胞修復產生突變的規律,發現突變結果與切割位點鄰近序列特征息息相關,并可依據靶位點序列信息準確預測突變結果。我們通過該報道作者提供的網絡工具FORECasT(https://partslab.sanger. ac. uk/FORECasT)對本研究采用的基因靶位點突變結果進行了預測,預測結果中概率最高的前三種突變類型與本研究的實際結果完全吻合(數據未顯示)。因此,本研究結果還驗證了FORECasT在植物基因敲除突變結果預測中的準確性。
總之,雖然水稻中功能缺失可能導致不同程度減產,但是到目前為止,若想通過改良品種的辦法使嚴重鎘超標(如>1 mg/kg)稻米產區的大米鎘含量降至較低水平(如<0.2 mg/kg),除非通過轉基因過表達[13]、或基因片段[34],否則利用功能缺失突變體仍然是唯一的選擇。而對于稻米Cd輕微污染(如含量低于0.5 mg/kg)產區低鎘品種的培育,更優的選擇可能是利用[14, 35]、[15]和[36]等基因的突變體,因為這些基因功能缺失后能使鎘含量減少約50%的同時對其他元素含量和產量影響甚微。
謝辭:感謝江西省超級水稻研究發展中心蔡耀輝研究員、江西省農業科學院土壤肥料與資源環境研究所徐昌旭研究員和謝杰博士在鎘污染耕地信息提供及材料種植中的幫助。
[1] Grant C A, Clarke J M, Duguid S, Chaney R L. Selection and breeding of plant cultivars to minimize cadmium accumulation., 2008, 390: 301-310.
[2] Sebastian A, Prasad M N V. Cadmium minimization in rice. A review., 2014, 34(1): 155-173.
[3] J?rup L, ?kesson A. Current status of cadmium as an environmental health problem., 2009, 238: 201-208.
[4] Arao T, Ae N. Genotypic variations in cadmium levels of rice grain., 2003, 49(4): 473-479
[5] Liu J, Zhu Q, Zhang Z, Xu J, Yang J, Wong M H. Variations in cadmium accumulation among rice cultivars and types and the selection of cultivars for reducing cadmium in the diet., 2005, 85: 147-153.
[6] 王林友, 竺朝娜, 王建軍, 張禮霞, 金慶生, 石春海. 水稻鎘、鉛、砷低含量基因型的篩選. 浙江農業學報, 2012, 24(1): 133-138.
Wang L Y, Zhu C N, Wang J J, Zhang L X, Jin Q S, Shi C H. Screening for rice (L.) genotyeps with lower Cd, Pb and As contents., 2012, 24(1): 133-138. (in Chinese with English abstract)
[7] 葉新新, 周艷麗, 孫波. 適于輕度Cd、As污染土壤種植的水稻品種篩選. 農業環境科學學報, 2012, 31(6): 1082-1088.
Ye X X, Zhou Y L, Sun B. Screening of suitable rice cultivars for the adaptation to lightly contaminated soil with Cd and As., 2012, 31(6): 1082-1088. (in Chinese with English abstract)
[8] Uraguchi S, Fujiwara T. Rice breaks ground for cadmium-free cereals., 2013, 16(3): 328-334.
[9] Uraguchi S, Fujiwara T. Cadmium transport and tolerance in rice: Perspectives for reducing grain cadmium accumulation., 2012, 5(1): 5.
[10] Satoh-Nagasawa N, Mori M, Nakazawa N, Kawamoto T, Nagato Y, Sakurai K, Takahashi H, Watanabe A, Akagi H. Mutations in rice () heavy metal ATPase 2 (OsHMA2) restrict the translocation of zinc and cadmium., 2012, 53(1): 213-224.
[11] Takahashi R, Ishimaru Y, Shimo H, Ogo Y, Senoura T, Nishizawa N K, Nakanishi H. The OsHMA2 transporter is involved in root-to-shoot translocation of Zn and Cd in rice., 2012, 35(11): 1948-1957.
[12] Yamaji N, Xia J, Mitani-Ueno N, Yokosho K, Feng M J. Preferential delivery of zinc to developing tissues in rice is mediated by P-type heavy metal ATPase OsHMA2., 2013, 162(2): 927-939.
[13] Ueno D, Yamaji N, Kono I, Huang C F, Ando T, Yano M, Ma J F. Gene limiting cadmium accumulation in rice., 2010, 107(38): 16 500-16 505.
[14] Uraguchi S, Kamiya T, Sakamoto T, Kasai K, Sato Y, Nagamura Y, Yoshida A, Kyozuka J, Ishikawa S, Fujiwara T. Low-affinity cation transporter (OsLCT1) regulates cadmium transport into rice grains., 2011, 108(52): 20 959-20 964.
[15] Shimo H, Ishimaru Y, An G, Yamakawa T, Nakanishi H, Nishizawa N K. Low cadmium (), a novel gene related to cadmium tolerance and accumulation in rice., 2011, 62(15): 5727-5734.
[16] Ishikawa S, Ishimaru Y, Igura M, Kuramata M, Abe T, Senoura T, Hase Y, Arao T, Nishizawa N K, Nakanishi H. Ion-beam irradiation, gene identification, and marker-assisted breeding in the development of low-cadmium rice., 2012, 109(47): 19 166-19 171.
[17] Ishimaru Y, Takahashi R, Bashir K, Shimo H, Senoura T, Sugimoto K, Ono K, Yano M, Ishikawa S, Arao T, Nakanishi H, Nishizawa N K. Characterizing the role of rice NRAMP5 in manganese, iron and cadmium transport., 2012, 2: 286.
[18] Sasaki A, Yamaji N, Yokosho K, Ma J F. Nramp5 is a major transporter responsible for manganese and cadmium uptake in rice., 2012, 24(5): 2155-2167.
[19] Belhaj K, Chaparro-Garcia A, Kamoun S, Nekrasov V. Plant genome editing made easy: Targeted mutagenesis in model and crop plants using the CRISPR/Cas system., 2013, 9(1): 39.
[20] Peng H, Zhang Q, Li Y, Lei C, Zhai Y, Sun X, Sun D, Sun Y, Lu T. A putative leucine-rich repeat receptor kinase, OsBRR1, is involved in rice blast resistance., 2009, 230(2): 377-385.
[21] Miao J, Guo D, Zhang J, Huang Q, Qin G, Zhang X, Wan J, Gu H, Qu L J. Targeted mutagenesis in rice using CRISPR-Cas system., 2013, 23(10): 1233-1236.
[22] Feng Z, Zhang B, Ding W, Liu X, Yang D L, Wei P, Cao F, Zhu S, Zhang F, Mao Y, Zhu J K. Efficient genome editing in plants using a CRISPR/Cas system., 2013, 23(10): 1229-1232.
[23] Cong L, Ran F A, Cox D, Lin S, Barretto R, Habib N, Hsu P D, Wu X, Jiang W, Marraffini L A, Zhang F. Multiplex genome engineering using CRISPR/Cas systems., 2013, 339(6121): 819-823.
[24] Liu H, Ding Y, Zhou Y, Jin W, Xie K, Chen L L. CRISPR-P 2.0: An improved CRISPR-Cas9 tool for genome editing in plants., 2017, 10(3): 530-532.
[25] Lei Y, Lu L, Liu H Y, Li S, Xing F, Chen L L. CRISPR-P: A web tool for synthetic single-guide RNA design of CRISPR-system in plants., 2014, 7(9): 1494-1496.
[26] Holsters M, de Waele D, Depicker A, Messens E, van Montagu M, Schell J. Transfection and transformation of., 1978, 163(2): 181-187.
[27] Toki S, Hara N, Ono K, Onodera H, Tagiri A, Oka S, Tanaka H. Early infection of scutellum tissue with Agrobacterium allows high-speed transformation of rice., 2006, 47(6): 969-976.
[28] Allen G C, Flores-Vergara M A, Krasynanski S, Kumar S, Thompson W F. A modified protocol for rapid DNA isolation from plant tissues using cetyltrimethy- lammonium bromide., 2006, 1(5): 2320-2325.
[29] 劉巧泉, 陳秀花, 王興穩, 彭凌濤, 顧銘洪. 一種快速檢測轉基因水稻中潮霉素抗性的簡易方法. 農業生物技術學報, 2001, 9(3): 264-268.
Liu Q Q, Chen X H, Wang X Y, Peng T L, Gu M H. A rapid simple method of assaying hygromysin resistance in transgenic rice., 2001, 9(3): 264-268. (in Chinese with English abstract)
[30] Tang L, Mao B, Li Y, Lv Q, Zhang L, Chen C, He H, Wang W, Zeng X, Shao Y, Pan Y, Hu Y, Peng Y, Fu X, Li H, Xia S, Zhao B. Knockout ofusing the CRISPR/Cas9 system produces low Cd-accumulating indica rice without compromising yield., 2017, 7(1): 14 438.
[31] Rizwan M, Ali S, Adrees M, Rizvi H, Zia-Ur-Rehman M, Hannan F, Qayyum M F, Hafeez F, Ok Y S. Cadmium stress in rice: Toxic effects, tolerance mechanisms, and management: A critical review., 2016, 23(18): 17 859-17 879.
[32] 吳平. 應用RFLP標記分析水稻株高與分蘗的遺傳相關性. 中國科學C輯: 生命科學, 1996, 26(3): 264-270.
Wu P. Genetic correlation analysis of rice plant height and tiller number using RFLP markers., 1996, 26(3): 264-270. (in Chinese; the title was translated into English by us)
[33] Allen F, Crepaldi L, Alsinet C, Strong A J, Kleshchevnikov V, De Angeli P, Páleníková P, Khodak A, Kiselev V, Kosicki M, Bassett A R, Harding H, Galanty Y, Mu?oz-Martínez F, Metzakopian E, Jackson S P, Parts L. Predicting the mutations generated by repair of Cas9-induced double-strand breaks., 2018, 37: 64-72.
[34] Wang C, Guo W, Ye S, Wei P, Ow D W. Reduction of Cd in rice through expression of-like gene fragments., 2016, 9(2): 301-304
[35] Uraguchi S, Kamiya T, Clemens S, Fujiwara T. Characterization of OsLCT1, a cadmium transporter from indica rice ()., 2014, 151(3): 339-347.
[36] Hao X, Zeng M, Wang J, Zeng Z, Dai J, Xie Z, Yang Y, Tian L, Chen L, Li D. A node-expressed transporter OsCCX2 is involved in grain cadmium accumulation of rice., 2018, 9: 476.
Creation of Low-Cd-accumulatingRice by Disruption ofGene via CRISPR/Cas9
LONG Qizhang, HUANG Yonglan, TANG Xiuying, WANG Huimin, LU Ming, YUAN Linfeng, WAN Jianlin*
[,,;Corresponding author,:]
【】 To accelerate the breeding process of low-Cd-accumulating rice,【】the widely appliedpure-line cultivars, Wushansimiao and Zhongzao 35, and the popular parents of hybrids, Huazhan and Wufeng B were used as materials to createknockout lines via CRISPR/Cas9. The marker-freemutants together with the wild types were grown either in a Cd-contaminated field (Field A) or in a non-Cd-contaminated field (Field B) for subsequent experiments. The changes of the grain Cd content were investigated for plants grown in both Field A and Field B. The changes of the contents of other related minerals were also examined but only for those plants grown in Field A. The agronomic changes of the mutants were further assessed by examining those plants grown in Field B. 【】 Rice lines with mutations atwere successfully generated for all the four cultivars. On one hand, the grain Cd level inmutants grown in Field B was reduced by 85.5% on average (<0.02 mg/kg); as a result ofmutation, the Cdlevel in plants grown in Field A, was reduced by 94.8% (<0.1 mg/kg), meanwhile, the Mn level was decreased by 52.7%; on the contrary, the Cr level was elevated by 59.5% on average for the four cultivars and an increase of 79.1% in the Pb level was also observed but only inmutants of Huazhan; the levels of other mineral elements including Fe, Cu , Zn, Ca, Se and As were slightly or hardly affected. On the other hand, a slight decrease in plant height and an average yield loss of 6.9% were observed due tomutation for all cultivars; the yield loss could be ascribed to a reduction in seed setting rate and seed weight and was compensated by a slight increase in effective tiller number. 【】 Low-Cd-accumulating rice plants can be generated bydisruption at the cost of an acceptable yield loss and the low-Cd-accumulating rice lines produced in the present study may serve as promising cultivars to eliminate rice Cd contamination in many areas.
rice; cadmium; genetic editing; breeding
Q755; S511.034
A
1001-7216(2019)05-0407-14
10.16819/j.1001-7216.2019.9026
2019-03-05;
2019-05-24。
國家重點研發計劃資助項目(2016YFD0102102);江西現代農業科研協同創新專項(JXXTCX2015001-003);江西省科技計劃項目農業領域重點項目(20141BBF60001)。

