毛竹 Phyllostachys edulis retrotransposon 7(PHRE7)轉座子的克隆與鑒定
蔣政勤,周明兵,2,鄭 浩,季 航,徐芷馨
(1.浙江農林大學 省部共建亞熱帶森林培育國家重點實驗室,浙江 杭州 311300;2.浙江農林大學 浙江省竹資源與高效利用協同中心,浙江 杭州311300)
毛竹Phyllostachys edulis為禾本科Gramineae剛竹屬Phyllostachys下的一個種,在中國竹產業中有十分重要的地位。轉座子是真核生物中一類重要的轉座元件,在毛竹基因組中有廣泛分布。轉座子又可稱跳躍因子,其實質是基因組染色體上能自主復制和位移的一段脫氧核糖核酸(DNA)序列,可以直接從基因組內的一個位點移到另一個位點[1],其轉座的發生會導致自身堿基位置的改變和(或)拷貝數的增加。轉座子根據轉座方式不同可分為DNA轉座子(DNA transposon)和核糖核酸(RNA)反轉錄轉座子(retrotransposon)。DNA轉座子是以DNA為中間媒介,通過 “剪切—粘貼”的方式移動,最早在玉米Zea mays中發現[2]。RNA反轉錄轉座子是以RNA為中間媒介,依賴于反轉錄酶,反轉錄成DNA后,通過 “復制—粘貼”的方式移動,于1984年在玉米中首先被發現[3]。RNA反轉錄轉座子又在酵母和動物基因組中被發現,后被證實在植物基因組中廣泛存在[4]。例如在小麥Triticum aestivum基因組中轉座子高達60%[5],玉米基因組中轉座子比例高達85%[6]。RNA反轉錄轉座子根據其結構又可以分為長末端重復序列(long terminal repeat,LTR)和非長末端重復序列(non-LTRs)[7]。 其中含有長末端重復序列的 LTR反轉錄轉座子是目前研究較多的一類反轉錄轉座子,幾乎在所有高等植物基因組中都有分布[8],對植物基因組的結構、功能和進化有重要的作用。一個結構完整的LTR反轉錄轉座子長度通常為2~18 kb,兩端各有1個長度為100~5000 bp序列同源的長末端重復序列LTRs。LTR末端為反向重復序列,結構通常為 5′-TG-3′和 5′-CA-3′, 在 5′和 3′末端兩側通常具有 4~6 bp 的末端靶位點重復序列(target site duplications,TSDs)[9]。LTR反轉錄轉座子除兩端的反向重復的長末端重復序列外,其結構組成還包括引物結合位點(primer binding site,PBS),多嘌呤序列(polypurinetract,PPT),還有與轉座機制有關的GAG(gag protein)開放閱讀框和POL(polymerase)開放閱讀框。目前,已在多種植物中發現具有轉錄活性的LTR反轉錄轉座子,根據對活性的LTR轉座子序列、進化關系和結構特征的分析,發現活性LTR反轉錄轉座子在結構上具有相似性[10]:均含有2個 LTR區域、gag基因區、pol基因區(pr,int,rt,rh);LTR區具有1個或多個順式調控元件;各個結構域均具有相應的關鍵保守氨基酸。根據轉座子的長度、LTR的長度和物種分布,植物中的Ty1/Copia類轉座子可分為4種類型,分別為Tork,Retrofit,Oryco和 Sire; Ty3/Gypsy類轉座子可分為 6 種類型, 分別為 Tat, Athila, CRM, Reina, Del和 Galadriel[11]。LTR反轉錄轉座子是宿主染色體結構變異和基因組進化的重要調控因子[12],與植物性狀穩定表達有關。在正常的生長環境條件下,多數LTR反轉錄轉座子不表達,呈靜止狀態存在,在受到逆境脅迫或DNA甲基化水平波動劇烈的情況下會被激活轉座,調節植物抗逆基因的表達,從而發揮遺傳可塑性來適應不同環境條件。例如,云南地方水稻Oryza sativa品種 ‘月亮谷’ ‘Yuelianggu’(傳統品種,Acuche)的幼苗在高鹽處理后,檢測3個反轉錄轉座子相關基因的表達水平,發現在高鹽脅迫下,3個基因都被誘導快速轉錄,表達量均明顯增加[9];陸地棉Gossypium hirsutum品系 ‘棕彩選1號’ ‘Zongcaixuan No.1’在低溫脅迫下,抵御低溫起關鍵作用的T10和T18轉座子在低溫脅迫后均呈上調表達[13],禾本科單子葉植物高羊茅Festuca arundinacea在干旱脅迫下,它們的甲基化程度增加,干旱誘導T10和T18轉座子甲基化。DNA甲基化會抑制轉座子的活性[14]。所以當植物受到非生物環境脅迫時,例如在高溫、低溫、高鹽、輻照等逆境條件下,LTR反轉錄轉座子會被激活并插入到目的基因中進行轉座,影響周邊基因的表達模式和表達量[15],幫助植物適應不同的逆境脅迫。毛竹全基因組序列分析表明:LTR反轉錄轉座子在毛竹基因組含量最豐富,占整個基因組的37.3%(Ty1-copia,12.3%;Ty3-gypsy,24.6%)[16]。為探究毛竹LTR反轉錄轉座子在毛竹生長受到逆境脅迫時的響應模式,本研究在毛竹基因組數據庫中,選取1條具有完整結構的LTR反轉錄轉座子,命名為PHRE7,對PHRE7轉座子進行克隆與鑒定,系統分析了PHRE7在脅迫下的表達模式。本研究結果將為系統闡明LTR反轉錄轉座子非生物脅迫下的響應機制及揭示毛竹對環境適應的分子機制奠定基礎。
1 材料與方法
1.1 實驗材料
植物材料:毛竹幼葉、竹筍、竹根取自浙江農林大學省部共建亞熱帶森林培育國家重點實驗室的翠竹園,樣竹長勢良好。毛竹實生苗均由同一株毛竹種子培育而來。毛竹種子均取自廣西省靈川同一株開花毛竹,為大小基本一致的飽滿種子。
實驗試劑:用于提取RNA的RNA Trizol試劑,克隆PHRE7反轉錄轉座子的pMD18-T克隆載體,LATaq酶等聚合酶鏈式反應(PCR)試劑及RNA的反轉錄所需的Prime ScriptTMRT Master Mix和SYBRPremix ExTaqⅡ(Tli RNaseH Plus)。
1.2 實驗材料處理
從同株毛竹上采集飽滿且大小基本一致的優質種子,分別進行輻照、甲基化抑制劑、高溫、低溫、高鹽等5種不同的處理,并留空白對照。
空白對照組種子處理:先用無菌水清洗種子,后用體積分數為70%乙醇浸泡消毒30 s,再用無菌水沖洗3次,然后在無菌水中浸泡24 h以恢復種子活力。取無菌培養皿,放入無菌濾紙后用無菌水潤濕,再用鑷子輕夾出毛竹種子,置于濾紙上靜待萌發。待長至3~4片葉后(2~3周)將其轉移至m(珍珠巖)∶m(蛭石)∶m(泥炭土)=1∶1∶1 的基質中生長。
輻照處理:挑選優質野生型毛竹種子450粒,150粒·組-1,分別接受 30,50和70 Gy等3個梯度的137Cs-γ射線輻照,輻照地點為浙江省農業科學院作物與核技術利用研究所輻照中心,做好不同梯度的標記,后續與不做任何處理的空白對照組在相同條件下培育到長出5片葉[17]。
甲基化抑制劑處理:毛竹種子分別用50.0,150.0和250.0 μmol·L-1的5-氮雜胞苷浸種24 h,種子數量同輻照處理一致,即每梯度150粒,但在5-氮雜胞苷處理前需用體積分數為70%乙醇消毒30 s,無菌水沖洗3次進行消毒處理。隨后與空白對照組在相同條件下培育。
高溫和低溫處理:前面階段與對照組的培育方法相同,待毛竹實生苗長出3~4片葉后,分別在培養箱進行高溫42℃處理4 h和低溫4℃處理16 h,每處理選取生長狀況良好毛竹幼苗10株,后續處理與空白對照組相同。
高鹽處理:前面階段與對照組的培育方法相同,待毛竹實生苗長出3~4片葉后,分別用0.1,0.2和0.3 mol·L-1氯化鈉溶液100 mL澆灌3 d[18-19],每處理選取生長狀況良好毛竹幼苗10株,后續處理與空白對照組相同。
采集空白對照組及5種脅迫處理下的毛竹實生苗的葉片,每處理重復3株,用液氮速凍,于-80℃保存備用。
1.3 方法
1.3.1PHRE7反轉錄轉座子克隆 采用十六烷基三甲基溴化銨(CTAB)法從毛竹嫩葉中提取毛竹基因組DNA[20],根據PHRE7的側翼序列設計1對克隆引物(PHRE7-F和PHRE7-R,具體序列見表1)。以毛竹提取DNA作為克隆模板,運用以上設計的克隆引物進行PCR擴增。具體擴增反應體系設計為:LATaq酶 0.5 μL,PHRE7-F 和PHRE7-R 各 0.8 μL,2×GC 緩沖液(buffer)25.0 μL , 三磷酸堿基脫氧核苷酸混合液(dNTP mix)4.0 μL,DNA 100 ng,加無菌水補齊50.0 μL。具體反應條件設計為:預變性95℃ 5 min;變性94℃30 s,退火44℃30 s,延伸72℃5 min,35個循環;終延伸72℃10 min,4℃保存。將PCR產物在質量分數為1%瓊脂糖電泳中分離,PCR產物連接到pMD20-T克隆載體上,膠回收目的片段并送生物公司測序。
1.3.2PHRE7反轉錄轉座子編碼域的相對表達量分析 分別提取毛竹未經處理葉片以及甲基化抑制劑、輻照、高溫、低溫、高鹽等5種脅迫處理后實生苗葉片的RNA,在反轉錄酶的催化作用下反轉錄成cDNA模板,具體方法參照Trizol法[21]。在RT,INT,RH序列3個可編碼蛋白結構域內分別設計特異性引物(表1),對PHRE7的表達水平進行熒光定量PCR(qRT-PCR)分析,內參基因為毛竹PheACT2[22]。熒光定量反應體系設計為 10.0 μL: 5.0 μL SYBRPremix ExTaqTMⅡ,0.4 μL cDNA,0.2 μL 底物-5(primer-5), 0.2 μL primer-3,4.2 μL無菌水[23]。反應條件如下:95℃ 7 min; 預擴增95℃ 10 s,58℃10 s,72℃15 s,共30個循環。每個樣品重復3次,按照2-ΔΔt的方法計算出不同脅迫處理條件下PHRE7的相對表達量。
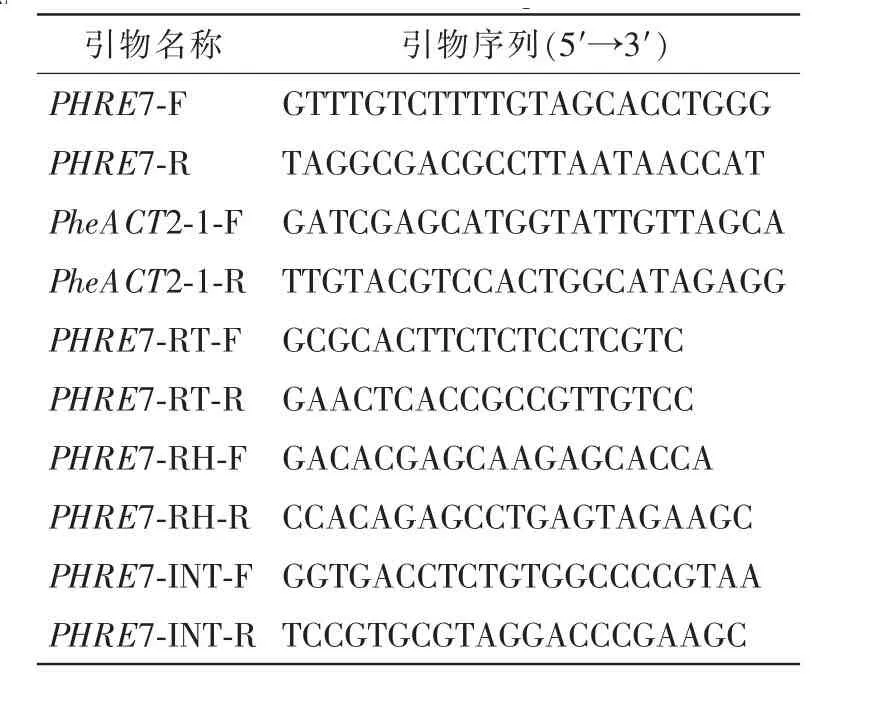
表1 引物序列Table 1 Primers sequence
1.3.3PHRE7反轉錄轉座子結構和拷貝數分析 下載毛竹基因組數據,通過LTRharvest軟件[24](http://www.zbh.uni-hamburg.de/forschung/genominformatik/software/ltrharvest.html)對LTR轉座子的長度、序列等進行分析,鑒定毛竹基因組LTR反轉錄轉座子,再利用LTRdigestion軟件[25](http://www.zbh.uni-hamburg.de/forschung/genominformatik/software/ltrdigest.html),解析毛竹LTR反轉錄轉座子結構并完成自動標注,通過cd-hit軟件[26]構建序列相似的非冗余基因集,分析各序列的相似程度,確定毛竹LTR反轉錄轉座子的拷貝數。根據毛竹LTR反轉錄轉座子拷貝以及轉座子結構,選取1個結構完整的LTR反轉錄轉座子作為研究對象,將其命名為PHRE7。PHRE7各個結構域由LTRdigestion軟件鑒定,由編碼區序列翻譯的氨基酸序列提交到非冗余蛋白質數據庫(NCBI),通過blastp進行比對確認,并由此建立PHRE7的結構圖。
1.3.4PHRE7反轉錄轉座子插入時間分析 將PHRE7的LTR序列輸入MEGA7軟件[27],通過Muscle方法比對PHRE7轉座子兩端LTR序列的同源性,計算得分化度k。根據公式t=k/2r[28](t為插入時間,r為LTR 序列的平均替換率,1.3×10-8bp·a-1[29]),計算得PHRE7 插入時間。
1.3.5PHRE7反轉錄轉座子LTR序列分析 通過克隆結果測序與該序列比對,確定序列完全一致性,明確開放性閱讀框中的各蛋白編碼區(PR,INT,RT,RH等)的位置。LTR反轉錄轉座子的5′端LTR的U3區域都有豐富的順式調控元件。為了分析鑒定轉座子LTR區域中順式調控元件的種類和分布情況,將LTR區域編碼序列放入PlantCARE在線軟件(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)[30]中,直接分析得出該區域內的各個順式作用元件并作出標記。
1.3.6PHRE7反轉錄轉座子進化地位分析 利用Gypsy Database 2.0(http://www.gydb.org/index.php/Main_Page)查找并下載Ty1-copia家族其他成員(Oryco,Sire,Retrofit,Tork)的代表性反轉錄轉座子RT氨基酸序列,與PHRE7轉座子的RT氨基酸序列比對,利用MEGA7軟件中的Muscle方法進行同源比對[31],找出最佳模型,并通過該軟件中的最大似然法(maximum likelihood,ML)構建進化樹[32],分析PHRE7所在的Ty1-copia家族亞家族位置。
2 結果與分析
2.1 PHRE7的克隆
以毛竹DNA作為模板,通過PCR擴增PHRE7轉座子,PCR擴增產物在質量分數為1%瓊脂糖凝膠電泳后,割膠回收目的片段,送生物工程公司測序。電泳檢測結果顯示PHRE7轉座子與預測的大小(6073 bp)基本一致(圖1)。
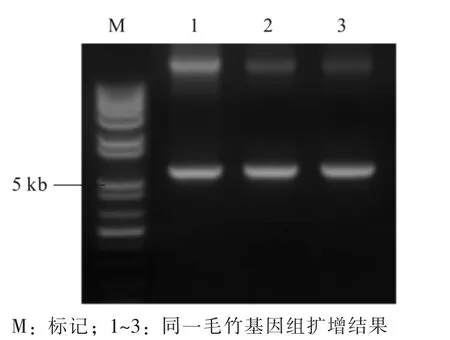
圖1 全長PHRE7轉座子PCR擴增電泳圖Figure 1 PCR result of full-length PHRE7
2.2 基因的序列分析
2.2.1PHRE7結構特征PHRE7轉座子全長為6073 bp,測序結果與預測一致。PHRE7轉座子組成為5′端和3′端的長末端重復序列和含5種酶的連續開放閱讀框(ORF),結構順序依次為5′-L-LTR-GAG-PRINT-RT-RH-LTR-R-3′(圖2)。各結構長度具體如下:左端LTR(L-LTR)長度為959 bp,右端LTR(LTR-R)為3′端LTR的反向重復序列,長度也為959 bp,相似性為96.7%。計算得插入時間約為126.92萬a前。開放閱讀框核苷酸編碼區總長度為1524 bp,共編碼氨基酸507個,GAG核心區位置在1030~1744 bp,主要與反轉錄轉座子RNA的成熟和包裝有關;PR核心區為是水解酶的編碼區,位置在1978~2224 bp,與反轉錄后多聚蛋白前體切割為功能性多肽有關;INT核心區是整合酶的編碼區,位置在2365~3004 bp,用于催化反轉錄轉座子插入宿主基因組的整個過程[33];RT核心區是反轉錄酶的編碼區,位置在3580~4312 bp,用于催化單鏈RNA或DNA合成DNA的過程,是轉座子轉座的必不可少的酶;RH核心區位置在4624~5020 bp,用于編碼核糖核酸酶H。核糖核酸酶H是水解酶的一種,負責原始RNA模板的水解。根據編碼區結構順序GAG-PR-INT-RT-RH,可確定PHRE7反轉錄轉座子是Ty1-gypsy家族成員。通過cd-hit軟件[26]對比毛竹基因組中的轉座子,鑒定出PHRE7相似的序列,即其拷貝。結果顯示:PHRE7存在相似的其他10個拷貝,根據拷貝位置不同,分別把10個拷貝命名(表2)為PHRE7-1,PHRE7-2,PHRE7-3,PHRE7-4,PHRE7-5,PHRE7-6,PHRE7-7,PHRE7-8,PHRE7-9和PHRE7-10,10個拷貝的結構也相對完整(圖2)。
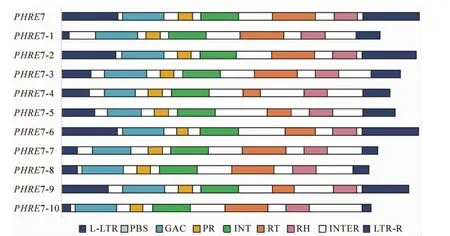
圖2 毛竹基因組中PHRE7及其10個拷貝的結構分析示意圖Figure 2 Structure of the PHRE7 and its copies in Phllostachys edulis genome
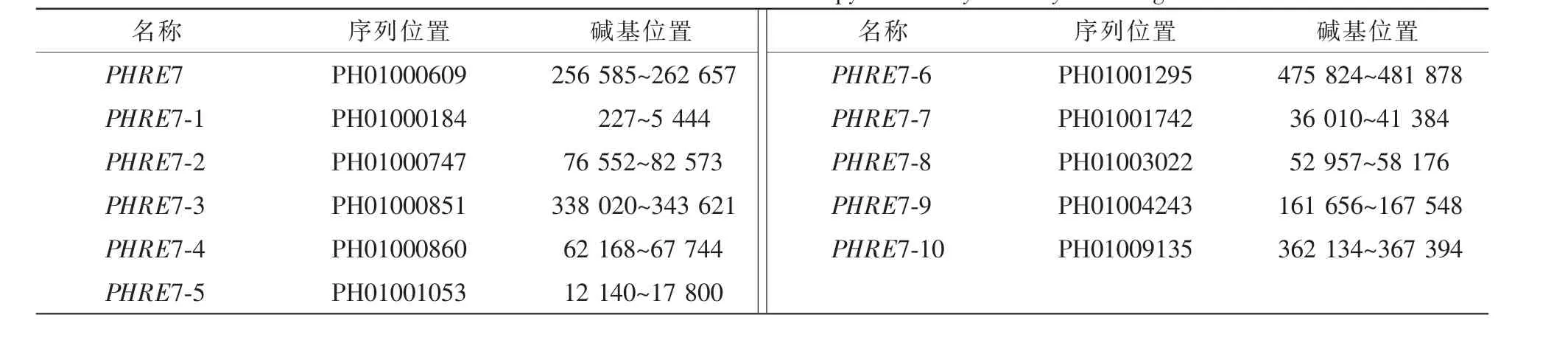
表2 PHRE7及其拷貝在毛竹基因組的命名及位置Table 2 Names and locations of PHRE7 and its copy in the Phyllostachys edulis genome
2.2.2PHRE7轉座子LTR序列分析 在LTR序列中,順式作用元件含量豐富。經分析得,核心啟動元件有TATA-box,CAAT-box,CCAAT-box,其中TATA-box有6個,CAAT-box有21個,CCAAT-box有2個,除啟動子元件外還有 2個光響應元件(4cl-CMA2b和chs-CMA2b),2個與脫落酸相關的調控元件(ABRE),3個壓力響應元件(SIRE),1個參與蛋白代謝調節的順式作用元件(O2-site)(圖3)。對毛竹基因組中LTR反轉錄轉座子PHRE7及其10個拷貝的插入時間進行分析,結果顯示:PHRE7的插入時間為126.92萬a前,PHRE7-1拷貝插入時間為223.08萬a前,PHRE7-2拷貝插入時間為150.00萬a前,PHRE7-3插入時間為142.31萬a前,PHRE7-4插入時間為261.54萬a前,PHRE7-5插入時間為126.92萬a前,PHRE7-6插入時間為403.85萬a前,PHRE7-7插入時間為442.31萬a前,PHRE7-8插入時間為238.46萬a前,PHRE7-9插入時間為219.23萬a前,PHRE7-10插入時間為307.69萬a前。經計算可知:在這11個序列中,PHRE7與PHRE7-5的插入時間相同,為126.92萬a前,在11個序列中插入時間最晚,說明PHRE7是其中結構完整且最為年輕的反轉錄轉座子(圖4)。
2.2.3PHRE7轉座子進化分析 為了探究PHRE7反轉錄轉座子和植物中其他LTR反轉錄轉座子的進化關系,利用保守性最高RT區域編碼序列構建了PHRE7和其他植物典型的反轉錄轉座子系統進化樹[34]。根據進化樹(圖5)可知:PHRE7屬于Ty1-copia家族中4個分支(Oryco,Sire,Retrofit,Tork)的Tork分支。
2.3 PHRE7在不同處理中的表達模式分析

圖3 PHRE7反轉錄轉座子LTR序列順式元件分析Figure 3 Analysis of the cis-regulatory motifs in PHRE7
為分析PHRE7的組織表達模式,在PHRE7轉座子6個結構域中選擇INT,RT,RH等3個結構域進行表達量分析。PHRE7反轉錄轉座子RT,RH,INT等3個結構域在毛竹竹根、竹葉、竹筍等3個不同部位中均有表達,且在葉中相對表達量最大,在竹筍和根相對表達豐度依次降低(圖6)。
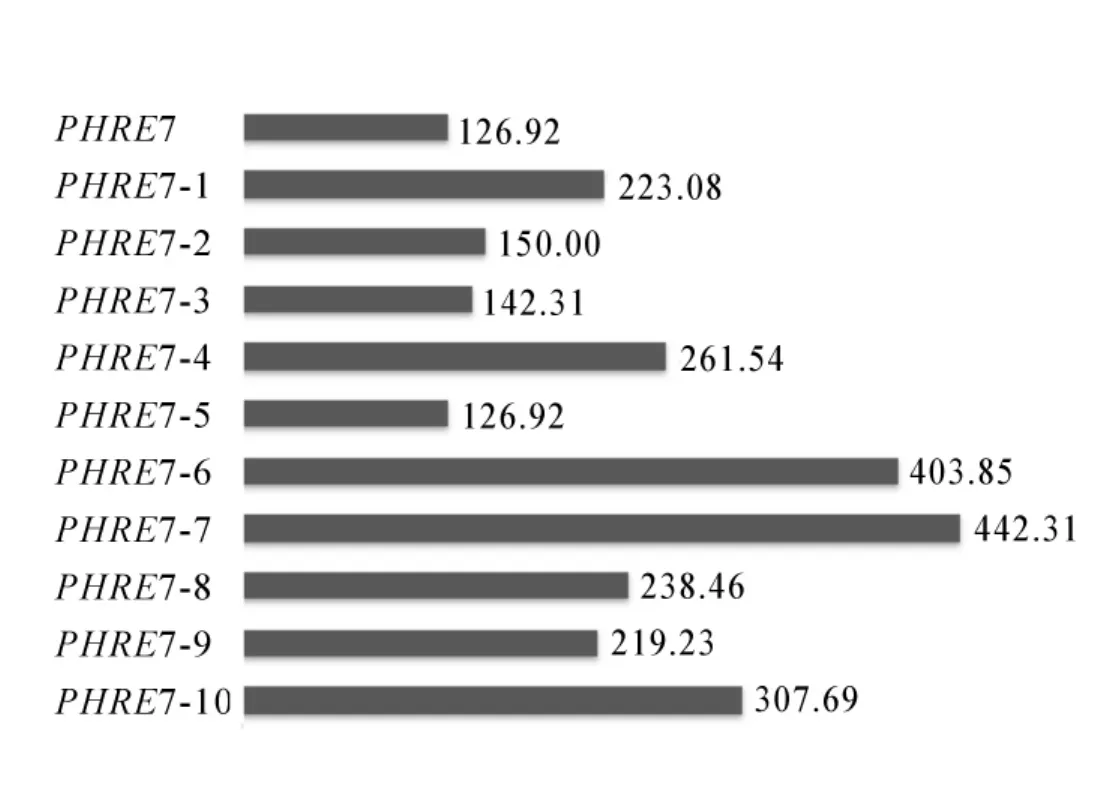
圖4 PHRE7反轉錄轉座子及各個拷貝的插入時間Figure 4 Insertion time of PHRE7 and its copies
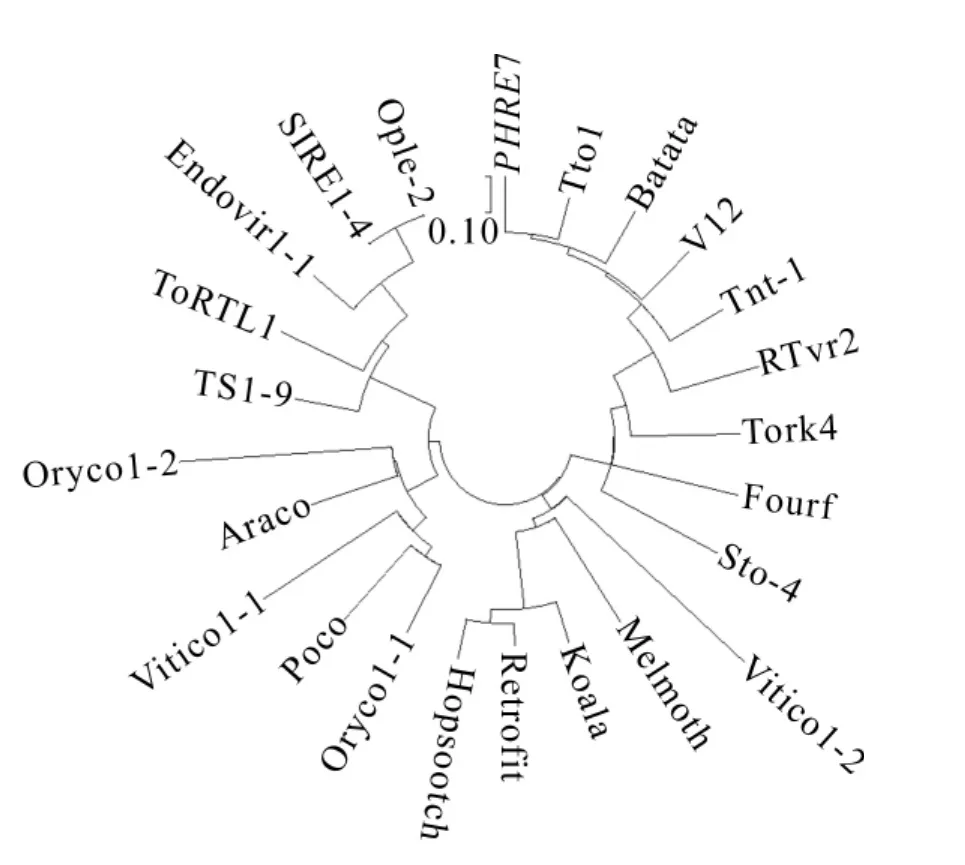
圖5 PHRE7反轉錄轉座子系統發育樹Figure 5 Phylogenetic tree of PHRE7 and other plant retrotransposons
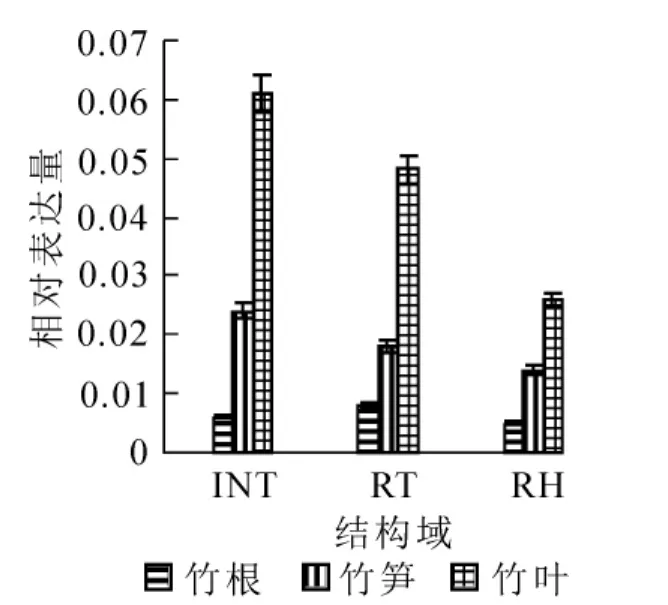
圖6 PHRE7反轉錄轉座子INT,RT, RH等3個結構域在毛竹竹筍、竹根、竹葉中的表達量Figure 6 Expression levels of INT,RT and RH domains of PHRE7 in root,shoot and leaf of Phyllostachys edulis
PHRE73個結構域(INT,RT,RH)在竹根、竹葉、竹筍不同部位的表達量上有差異,竹葉中的表達量均為最高。為了探討不同非生物環境脅迫條件對PHRE7反轉錄轉座子表達的影響,研究通過定量熒光PCR對經過不同處理(輻照、甲基化抑制劑、高/低溫、高鹽)的毛竹實生苗葉片中PHRE7轉座子的3個結構域(INT,RT,RH)表達進行了定量檢測和分析。
在0.1,0.2,0.3 mol·L-1高鹽處理后,高鹽脅迫下PHRE7的RT,RH和INT等3個結構域表達量呈現先上升再下降的趨勢,在0.1~0.2 mol·L-1時呈上升趨勢, 0.2 mol·L-1左右各結構域達到表達量最高值,0.2~0.3 mol·L-1表達量逐漸下降。3個結構域中INT結構域表達量變化幅度較大,RT和RH結構域表達量相對較接近,且變化幅度相對較小(圖7A)。
經 50.0, 100.0, 150.0 μmol·L-1濃度甲基化抑制劑處理, 各結構域的相對表達量隨甲基化抑制劑濃度的增大逐漸升高。50.0 μmol·L-1處理組相對空白對照組表達量顯著提升, 100.0 μmol·L-1與 150.0 μmol·L-1分別與前一甲基化抑制劑濃度處理組相比,表達量也均有上升。3個結構域中INT表達量最高,RT結構域表達量升高幅度比 INT小(圖7B)。
不同強度的輻照脅迫下,INT,RT,RH等3個結構域表達量均高于對照組。在30 Gy輻照強度下,各結構域表達量達最大值,后隨著輻照強度的增加,各結構域表達量呈下降趨勢。INT結構域中30 Gy輻照處理后的表達量與50 Gy輻照處理后的表達量比值接近于2,RH和RT結構域在50 Gy或70 Gy輻照強度處理下表達量均相近,INT結構域相對表達量與RH和RT結構域相比下降幅度最大(圖7C)。
4℃低溫和42℃高溫脅迫處理后,PHRE7反轉錄轉座子的INT,RT,RH等3個結構域的表達量均有提高,且高溫處理對表達量影響更大,上升更為顯著,表達量約為低溫處理的1.5倍。RT和INT結構域表達量上升幅度高于RH(圖7D)。
3 討論
PHRE7是1條結構完整的反轉錄轉座子,按照結構域排列順序鑒定屬于Ty1-copia分支,具備轉座所需的所有酶和相似度極高的長末端重復序列(96.7%)。PHRE7的LTR中含有豐富的順式作用元件,在外界的非生物環境的脅迫下可能會誘導轉座子激活進行轉座。PHRE7反轉錄轉座子的插入時間為126.92萬a前,在植物基因中,很多五六百萬年前插入的轉座子仍存在活性,例如水稻愈傷組織中的Tos17反轉錄轉座子插入時間約在700萬a前,現仍能夠進行轉座[35]。相比而言PHRE7較為年輕,故推測其是仍具有轉錄活性的反轉錄轉座子。
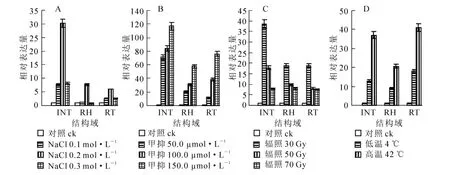
圖7 不同處理條件下PHRE7反轉錄轉座子INT,RT,RH等3個結構域的表達量Figurte 7 Expression levels of the INT,RT and RH domains of PHRE7 under different treatments
PHRE7反轉錄轉座子各結構域完整,該轉座子其他拷貝存在不同結構域間的缺失。轉座致使宿主基因組出現斷裂和重排的現象,還會影響宿主基因表達,導致宿主基因組處于不穩定的狀態。以上現象會激活宿主對LTR轉座子活性抑制機制,導致LTR轉座子在進化發展過程中,各結構域間出現基因片段丟失現象,控制該轉座子的自主轉座。所以,PHRE7反轉錄轉座子及其10條拷貝可能是由共同祖先演化變異而來,而PHRE7反轉錄轉座子在宿主基因調節機制下依然保持其完整的結構。
PHRE7在毛竹各部位的表達存在差異性。在毛竹竹筍、竹根和竹葉等3個不同部位表達量對比中,PHRE7在竹葉中的表達量相對較高。有研究證明:活性LTR反轉錄轉座子在不同部位間的表達具有選擇性和特異性,可能與LTR反轉錄轉座子長末端重復序列中的順式作用元件和其他元件的存在有關,各類元件與調控物質的作用不同,對LTR轉座子的表達量也存在一定影響。
毛竹實生苗在甲基化抑制劑、輻照、高鹽、高溫和低溫等5個不同處理后,3個結構域表達量均比對照高,表明PHRE7是1個具有轉錄活性的轉座子,轉錄活性受外界環境變化的影響。
LTR反轉錄轉座子的轉座活性與其甲基化程度有較大程度的關聯。通常情況下DNA甲基化可以作為基因沉默的一種表觀遺傳標記[36]。在毛竹基因組中,編碼區占比較小,非編碼DNA序列[37]大量存在。為保證基因表達的準確性,植物體要盡可能減少非編碼區基因對轉錄過程的干擾,因此在正常條件下,非編碼區DNA較少或基本不表達。而在甲基化抑制劑作用下,許多存在于非編碼區的轉座子會通過去甲基化作用解除限制使非編碼DNA得以激活表達[38],因此發生甲基化抑制劑作用下各結構域表達量顯著上調現象,且甲基化抑制劑的濃度越高,表達量越高,呈正相關。
逆境脅迫致使毛竹體內的激素水平、離子水平和甲基化水平等都發生一定程度的變化,從而影響轉座子的轉座活性[39],引起表達量的變化,這也是LTR反轉錄轉座子對逆境做出的一種適應[40]。輻射能引起植物變異。本研究結果顯示:輻照條件下,PHRE7轉座子表達量比空白對照組要高,但是隨輻照強度的增強表達量發生下調,其可能原因是高輻射對毛竹實生苗造成不可恢復的傷害,致使毛竹的自身調節功能不足以抵御外界輻照的傷害,各結構域表達量隨輻照強度的增強而下降。
在高鹽脅迫下,植物體內含水量會下降,水分過低會引起植物代謝和光合作用異常[41]。PHRE7的LTR序列中存在光響應元件(4cl-CMA2b和chs-CMA2b),表明PHRE7轉座子轉錄可能受光合作用調節。在高鹽作用下,PHRE7表達量隨鹽濃度的升高呈現先上升后下降趨勢。其可能原因是鹽濃度提升致使毛竹實生苗光合作用異常[42],轉座子得以激活表達,PHRE7結構域表達量升高。
PHRE7在4℃低溫和42℃高溫處理下,表達量較對照組均有上調,且42℃較4℃更為顯著。溫度脅迫是植物中常發生的脅迫類型,溫度對催化各類植物生理活動的酶活性有顯著影響,但極端溫度脅迫會造成基因組甲基化水平下降。在此效應下,PHRE7結構域的表達量呈上調變化。
本研究對毛竹反轉錄轉座子PHRE7的序列、結構以及在典型非生物環境脅迫下的表達量進行了系統的研究,發現PHRE7的表達量會在環境影響下有所變化,能對外界環境脅迫做出響應,是具有轉錄活性的LTR反轉錄轉座子,但具體的響應機制尚未清楚,仍需進一步的研究。

