松材線蟲Bx-ubc-3基因克隆及泛素通路鑒定
許佳瑤 陳俏麗 張瑞芝 李丹蕾
摘 要:泛素途徑可以調節許多關鍵的細胞功能。為探究松材線蟲中是否存在泛素途徑以及泛素途徑中的關鍵酶基因—泛素結合酶基因ubc3,構建松材線蟲cDNA文庫,并進行測序、ORF分析、同源性分析、系統發育樹構建、蛋白相互作用預測和分子動力學模擬等工作。克隆到長度為957bp的Bx-ubc-3基因片段。隨后的序列分析表明,其開放閱讀框長度為738 bp,編碼245個氨基酸;氨基6酸同源性分析結果表明,Bx-UBC-3與已知的泛素結合酶高度同源;蛋白互作和分子對接分析及分子動力學模擬結果表明,Bx-UBC-3的結合位點位于一個α螺旋、一個Loop1(L1)環與一個Loop2(L2)環;形成硫酯的Cys103位于L2上,構成催化裂隙;Bx-UBC-3和Bx-Rbx1之間以及Bx-UBC-3和Bx-泛素之間形成氫鍵。這些結果表明松材線蟲中存在一條理論泛素通路,其中的關鍵酶Bx-UBC-3是開發高效抗線蟲生物藥劑的潛在靶標。
關鍵詞:松材線蟲;泛素結合酶3;E2;泛素途徑
中圖分類號:S763.11 ? ?文獻標識碼:A ? 文章編號:1006-8023(2019)05-0009-07
Abstract:The ubiquitin pathway is able to regulate many critical cellular functions. In order to explore the existence of the ubiquitin pathway and a key enzyme gene in ubiquitin pathway, ubiquitin-conjugating enzyme named as ubc3 in B. xylophilus, a cDNA library of B. xylophilus was constructed and sequenced, ORF analysis, homology analysis, phylogenetic tree construction, protein interaction prediction, docking and molecular dynamics simulation were carried out. The full-length cDNA generated by the nematode spliced leader method was 957 bp. Analysis of the Bx-ubc-3 cDNA sequence indicated that its open reading frame (ORF) was 783 bp and 245 amino acids were encoded. Comparison of the identified amino acid sequence with the current databases revealed extensive homology of Bx-ubc-3 to all known ubiquitin-conjugating enzymes. The modeling and prediction of protein interaction, docking and molecular dynamics simulations showed that the binding sites in Bx-ubc-3 were located in α helix, Loop 1 (L1) ring and Loop 2 (L2) ring. The thioester-forming Cys103, which was located in L2, constituted a catalytic cleft. The interfaces of Bx-ubc-3 to Bx-Rbx1 and Bx-ubc-3 to Bx-Ubiquitin were also supported by several obvious hydrogen bond interactions. Results show that there is a theoretic ubiquitin pathway in B. xylophilus, and support the notion that Bx-ubc-3 is a potential target for developing potent antinematodal drugs against B. xylophilus.
Keywords:Bursaphelenchus xylophilus; ubiquitin-conjugating enzyme 3; E2; ubiquitin pathway
0 引言
泛素化是一種重要的轉錄后修飾方式[1],控制著真核生物整個蛋白質組。泛素途徑是由泛素激活酶(Ubiquitin-activating enzyme, E1)、泛素結合酶(Ubiquitin-conjugating enzyme, E2)和泛素連接酶(Ubiquitin-protein ligase, E3)介導的級聯反應[2],是細胞內蛋白質選擇性降解的重要途徑。E2是泛素途徑的關鍵酶,它與E1、E3通過蛋白間的相互作用使泛素與靶蛋白結合,形成一條多泛素鏈,從而使靶蛋白被26S蛋白酶體識別降解[3]。E2家族蛋白都有著相似的催化結構域,這些催化結構域都包含形成E2-泛素硫酯復合物的活性位點半胱氨酸(thioester-forming cysteine)。在對酵母的研究中,ubc-3(CDC34的同源基因)被證明是E2家族成員[4],編碼Ⅲ類E2,負責細胞周期G1期的蛋白降解[5]。盡管已有研究表明E2在酵母等模式生物的細胞中起著重要作用[6],但是目前國內外對于植物寄生線蟲中這類分子的研究仍然非常有限。
在模式生物秀麗線蟲(Caenorhabditis elegans)中,E2家族的UBC-18與UBC-3共同參與對同一底物的泛素化。UBC-18率先給底物標記一個泛素分子,隨后UBC-3負責在標記有一個泛素分子的底物上形成泛素鏈[1]。對秀麗線蟲相關基因的抑制或敲除均可引起秀麗線蟲不育或胚胎致死[7]。
松材線蟲(Bursaphelenchus xylophilus)是松材線蟲病的病原體[8],是一種對東亞松林危害極大的入侵生物,現入侵中國、日本和韓國等東亞國家及歐州國家葡萄牙與西班牙[9],對當地松林生態造成了嚴重破壞,對林業經濟造成了巨大損失。松材線蟲的胚胎發育和生命周期與秀麗線蟲非常相似[10],因此假設泛素途徑蛋白有可能是開發殺線藥物的潛在靶點。
2011年公布的松材線蟲基因組數據[11]使得對松材線蟲泛素途徑的研究更加容易。本研究克隆到松材線蟲Bx-ubc-3基因,分析了可能參與控制Bx-UBC-3與泛素途徑其他組分相互作用的區域并推測其功能,分析了泛素途徑中關鍵要素Bx-UBC-3、E3和泛素形成的復合物的結構。分析該途徑中的組分不僅有助于進一步理解其功能,建立一條理論的泛素通路,為后期的試驗打下理論基礎,還可以為今后的抗線蟲藥物的開發提供必要的思路。
1 材料與方法
1.1 線蟲培養、cDNA合成與轉錄組測序
試驗所用到的松材線蟲分離自中國廣東省患松材線蟲病的馬尾松(Pinus massoniana)。利用PDA培養基培養的灰葡萄孢菌進行線蟲人工擴繁培養[12]。用M9緩沖液[13]洗脫松材線蟲懸浮液100 μl(混合蟲齡),用TRIzol試劑提取松材線蟲總RNA,并利用DNA酶降解基因組DNA后構建cDNA文庫[14]。將上述cDNA文庫送華大基因生物公司測序。使用美國生物技術信息中心(NCBI)的在線BLASTN工具對測序結果進行鑒定。鑒定出ubc-3同源基因片段,命名為Bx-ubc-3。
1.2 基因克隆
用線蟲SL法(method of nematode spliced leader)[15]從松材線蟲的cDNA文庫中擴增了松材線蟲Bx-ubc-3基因序列的5端片段。在此基礎上,以SL1(GGT-TAT-ATC CAA GTT TGA G)和Ubc3-WH-2A(TCG AGA ACG TCC ACA TCA TC)為靶點,用oligo(dt)引物和以Ubc3-WH-1S為靶點的引物(GTT CGGA-ACA GTA GAGA GA)擴增3端片段。將純化的DNA片段克隆到pGEM-T載體(Promega,美國)中并測序。將5端和3端片段組裝成全長編碼序列,并提交給GenBank。
1.3 氨基酸序列及同源性分析
利用NCBI在線工具 ORF Finder進行ORF分析。用BLASTX/BLASTP進行核酸序列和氨基酸序列的同源性分析,E-value閾值為1×10-10。利用Clustal W進行多重序列比對,并用MEGA 5.05結合JTT+G模型構建系統發育樹(phylogenetic trees)[16]。使用上述ORF序列作為查詢序列在完整的松材線蟲基因組序列數據庫的TBLASTN搜索中發現了同源序列。
1.4 泛素途徑相關蛋白互作的建模與預測
提交FASTA格式的泛素途徑相關蛋白氨基酸序列至SWISS Model服務器進行蛋白質結構建模。輸出的模型通過定型模型能量分析(Qualitative Model Energy Analysis,QMEAN)賦值而著色,從而表示模型區域的準確度[17]。利用STRING 9.0預測泛素結合酶與其他蛋白之間可能的相互作用[18]。
1.5 泛素途徑相關蛋白對接與分子動力學模擬
為了定位分子對接位點,利用SWISS-PDB Viewer計算了蛋白質的靜電表面電位,并繪制到分子表面。利用已經公布的基因組數據[11]進一步確定Bx-UBC-3的配體。通過將Bx-UBC-3與Bx-泛素/Bx-Rbx1(Bx-ring-box1)對接,用HEX 6.3證實蛋白質相互作用。
使用NAMD 2.9進行分子動力學的模擬。用VMD、PyMOl、BioEdit和POV-Ray等程序對蛋白質進行了注釋說明。
2 結果與分析
2.1 松材線蟲Bx-ubc-3基因克隆
線蟲SL法產生的全長cDNA為957 bp。將這個cDNA序列命名為Bx-ubc-3,并提交給GenBank(GenBank ID:EU333281)。
基于松材線蟲基因組數據的TBLASTN同源性搜索發現CADV01004468.1與Bx-ubc-3所在contig(E-value:1×10-131)相關。
2.2 氨基酸序列及同源性分析
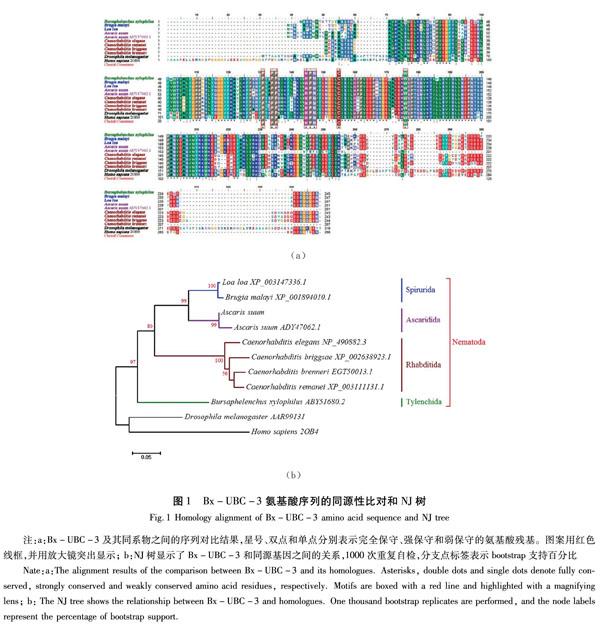
Bx-ubc-3的cDNA序列分析表明,其開放閱讀框(ORF)長度為783 bp,編碼245個氨基酸。將所鑒定的氨基酸序列與現有數據庫進行比較,發現與所有已知的泛素結合酶具有同源性,其中與秀麗線蟲ubc-3同源性為94%(GenBank ID:NP_490882;E-value:3×10-86),與馬來絲蟲(Brugia malayi)ubc-3同源性為90%(GenBank ID:XPY9001894010,E-vlaue:3×10-91)。
Bx-UBC-3與人(Homo sapiens)、黑腹果蠅(Drosophila melanogaster)和其他7種線蟲即非洲眼線蟲(Loa loa)、馬來絲蟲、豬蛔蟲(Ascaris suum)、秀麗線蟲、廣桿屬線蟲(C. briggsae)、C. brenneri和C. remanei的同源序列的多重比對表明,所有蛋白質的基序同源(圖1(a))。UBC酶超家族的一般特征基序是HPN(組氨酸-脯氨酸-天冬酰胺)三肽和E2活性位點,即形成硫酯的Cys103(半胱氨酸)[19]。在上述所有物種的序列中都有發現上述所有特征基序和氨基酸殘基(圖1(a)方框),包括Pro(脯氨酸)和Trp(色氨酸)殘基(圖1(a) Pro134、Pro135和Trp176)、HPN三肽基序(圖1(a) His145-Pro146-Asn147)和保守的Cys155殘基(圖1(a))。
線蟲UBC-3 NJ樹(圖1(b))顯示了5個簇[20]。因為沒有發現其他的墊刃目(Tylenchida)UBC-3,松材線蟲是系統發育樹中唯一的植物線蟲。松材線蟲與尾目(Spirurida)、蛔蟲目(Ascaridida)和小桿目(Rhabditida)在遺傳距離上遠大于人和果蠅。這一結果也得到了另一個基于Bx-UBC-3和100個同源基因而構建的NJ樹的證實。
2.3 Bx-UBC-3結構分析
用SWISMOST服務器在線預測Bx-UBC-3蛋白結構模型,進一步推測其功能。結果表明,Bx-UBC-3以β折疊為核心,兩側為4個α螺旋(H1-H4)、基本保守的310螺旋(h,殘基i和i+3之間有氫鍵)和兩個環(L1和L2)。L2是極性殘基Asp112(天冬氨酸)、Asp113、Gln115(谷氨酰胺)、 Gly117(甘氨酸)、Glu118和Glu122的酸性環。
人、果蠅和其他4種線蟲的同源序列同樣存在類似的結構變異。當Bx-UBC-3在同系物上疊加時,顯示出高度保守的N端序列。盡管總體結構相似,但這些蛋白在一些區域沒有顯著的構象特征。
2.4 泛素途徑相關蛋白間互作預測
以人同源細胞分裂周期34(CDC34,E-value:1×10-80)為模板預測了與Bx-UBC-3可能存在功能關系的基因相互作用蛋白的相互作用網絡。結果表明,Bx-UBC-3和SKP1-CUL1-F-box蛋白(SCF)E3之間存在蛋白質-蛋白質相互作用。7個蛋白與UBA-1的發生高度相關。這些蛋白中有3種是SCF相關蛋白:CUL1(cullin 1,the core component of multiple cullin-RING-based SCF)、RBX1(ring-box 1)和BTRC(β-transducin repeat, SCF的底物識別成分)。
2.5 泛素途徑相關蛋白Bx-UBC-3與SCF E3對接及結合位點分析
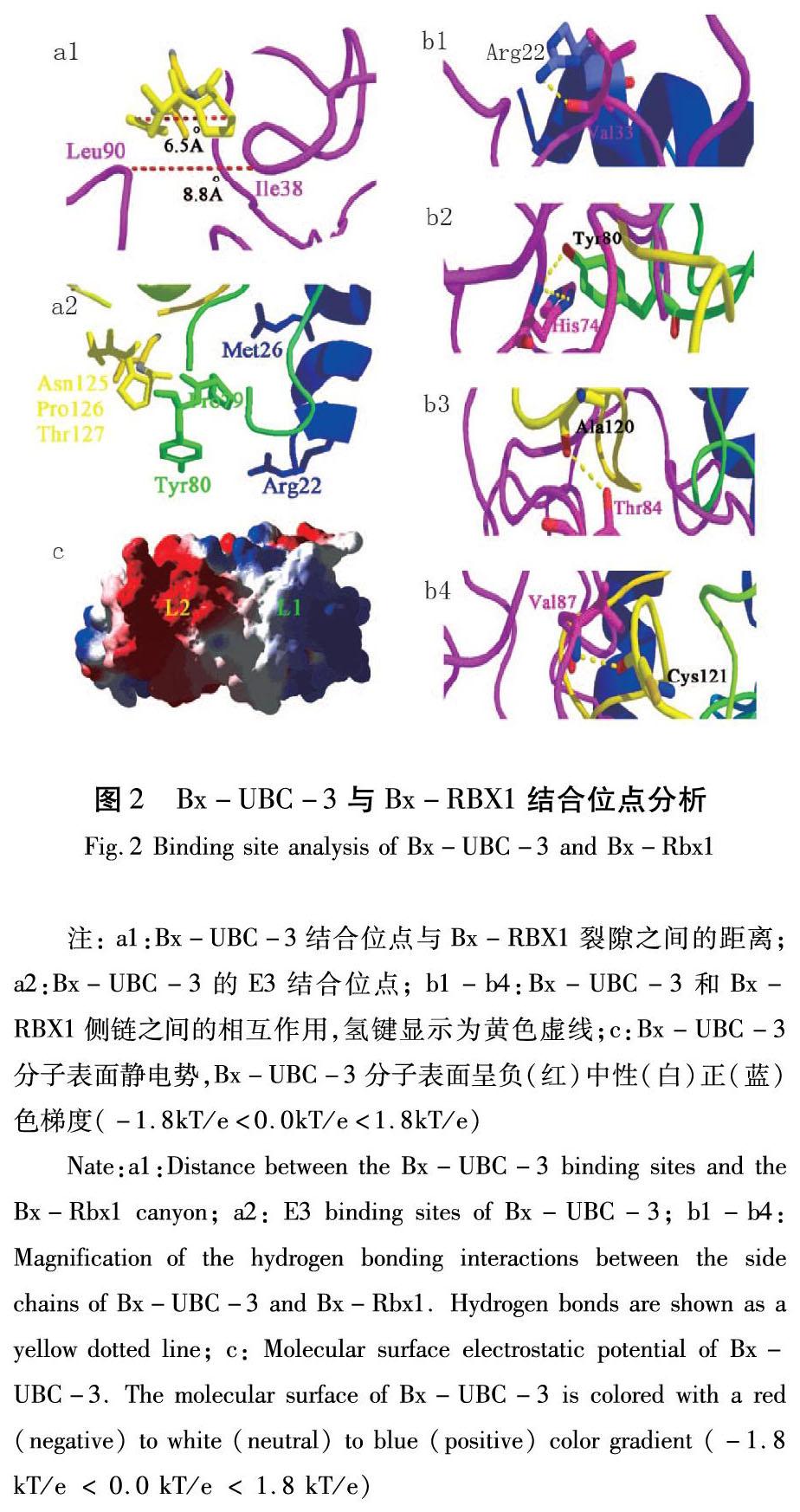
由于蛋白質間相互作用預測表明Bx-UBC-3與SCF相關蛋白相互作用,基于全基因組數據分析[11],發現了兩個與松材線蟲SCF相關的蛋白(Bx-Cul1和Bx-Rbx1)。基于已發表的X射線晶體結構數據,將Bx-UBC-3與Bx-Cul1/Bx-Rbx1復合物對接,建立了完整的Bx-UBC-3、Bx-Cul1和Bx-Rbx1三元復合物的模型。
為了識別候選的對接位點,計算了Bx-UBC-3和Bx-Rbx1的靜電表面電位,并將其繪制到分子表面。基于Bx-UBC-3、Bx-Cul1和Bx-Rbx1三元配合物和靜電表面,進一步預測Bx-UBC-3和Bx-Rbx1的相互作用。Bx-UBC-3位于Bx-Rbx1下方相對的靜電表面電勢裂隙上,在Bx-UBC-3上鑒定出帶正電荷的L2和帶負電荷的L1。
從Bx-UBC-3對接到Bx-Rbx1上,觀察到L1殘基Pro79和Tyr80(酪氨酸)可以與Bx-Rbx1結合(圖2(a2))。L2殘基Asn125和Pro126被埋入由Bx-Rbx1殘基Leu90(亮氨酸)和Ile38(異亮氨酸)形成的中心凹槽中(圖2(a1))。基于Bx-UBC-3和Bx-RBX1的界面特征,分析了4個氫鍵間的相互作用(圖2(b1-b4))。Bx-UBC-3中常見的E3結合位點是Arg22(精氨酸)(H1)、Pro79(L1)、Tyr80(L1)、Ala120(丙氨酸)(L2)和Cys121(L2)。
2.6 泛素途徑相關蛋白Bx-UBC-3與泛素對接及結合位點分析
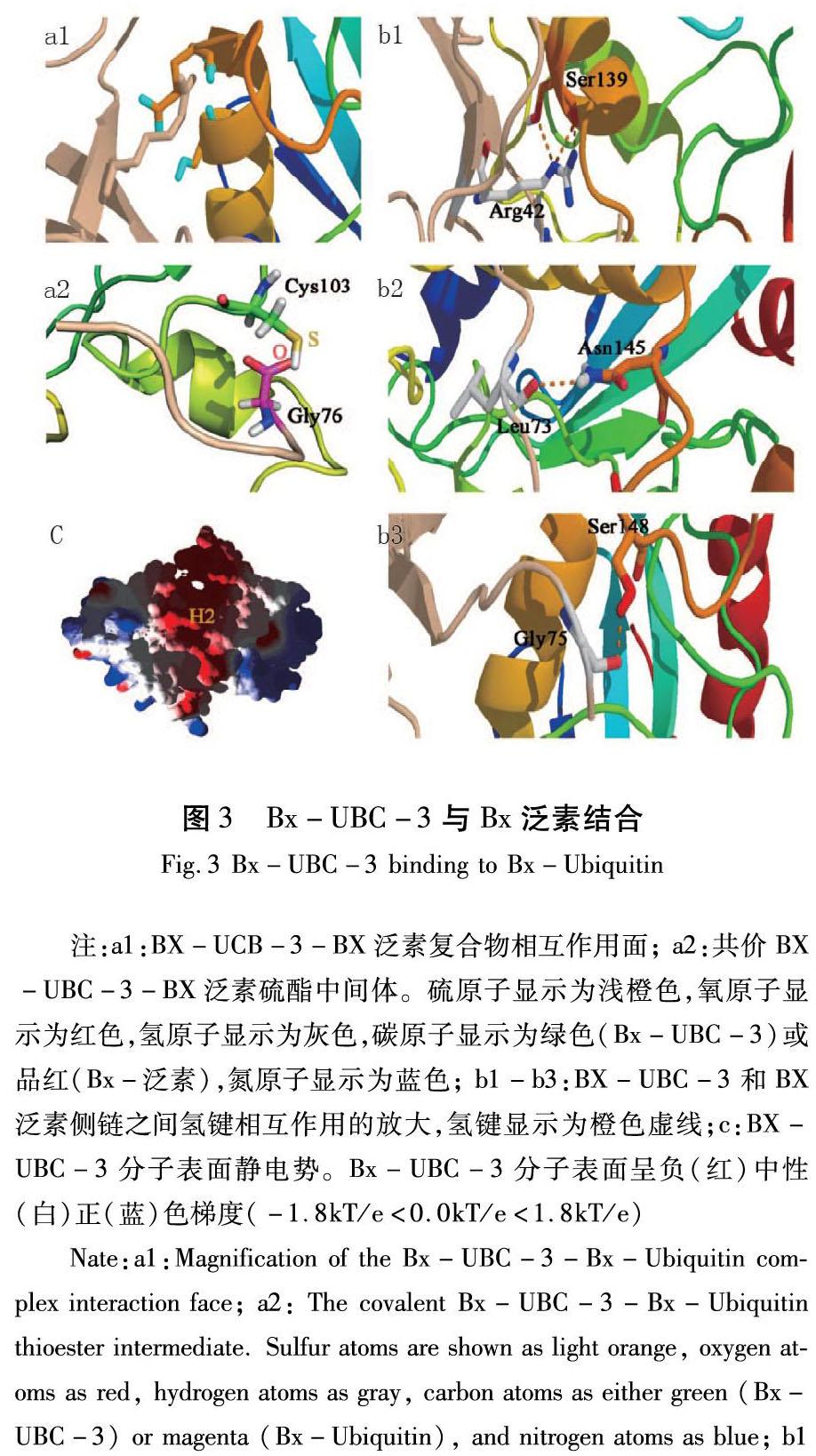
松材線蟲Bx-泛素基于全基因組數據鑒定。通過E2-泛素復合物結構比對,鑒定得到Bx-UBC-3與松材線蟲泛素復合物模型。在Bx-UCB-3與Bx-泛素復合體中結合位點中,Bx-UBC-的催化裂隙處中心是Bx-泛素的殘基Arg42和Gly76,Bx-UBC-3淺裂處的一側是殘基Ser139(絲氨酸)和Glu143,另一側是殘基Cys103。
模擬對接數據說明Bx-UBC-3和Bx-泛素通過硫酯模型相互作用。在Bx-UBC-3和Bx-泛素蛋白上發現了互補表面結構,Bx-泛素蛋白C端圍繞Bx-UBC-3蛋白,終止于Cys103(Bx-UBC-3)和Gly76(Bx-泛素)(圖3(a2))。形成硫酯的Cys103位于連接S4和H的相對非結構化區域(L2)上。
2.7 泛素途徑相關蛋白分子動力學模擬
通過分子動力學模擬研究了Bx-UBC-3的動態性能。同時計算了蛋白質結構的催化裂痕的柔度曲線和相關性(圖4)。繪制了Bx-UBC-3級聯軌跡的仿真幀(圖4(a)),并根據每個殘基的均方根偏差(root mea square deviation,RMSD)著色(圖4(b))。結合位點的RMSD值用紅星標記(圖4(d))。在酸性環L2中除了Ala120和Cys121外,大多數結合位點在平衡過程中表現出較少的運動。低RMSD值表明了模型的質量符合要求并可用于對接模擬。
分子動力學模擬證實,酸性環L2在Bx-UBC-3中具有最高的構象自由度。L2環與E2活性位點(形成硫酯的Cys103)緊密相連,其酸性插入特征是高構象變異(圖4(a)和圖4(c)),這可以用RMSD定量描述(圖4(d))。這些結果與其他E2 X射線晶體學的結果一致[21]。
蛋白質泛素化[22]過程中,E2的磷酸化是由蛋白激酶2介導的,酪蛋白激酶2依賴于保守Ser和酸性殘基之間的靜電排斥[23]。在動力學模擬中,L2中的酸性極性殘留物瞬間接近催化裂隙(從28.2到10.3 )(圖4(c))和Ser139所在的H2。這一結果表明,引入強負電荷基團的Ser139磷酸化可能導致與上述酸性殘基的靜電排斥,從而有利于L2開放構象。此外,L2在與Bx-RBX1對接(圖4(c))時呈現拓撲扭曲(32.7 )。這些結果表明,L2具有較高的構象自由度,證實了L2在這種相互作用中起著關鍵作用。
3 討論與結論
在過去的數十年中,不斷有新的泛素化酶及其參與的生理過程被發現[24]。現有的研究方法多數以蛋白降解過程為切入點,即目的蛋白通過泛素途徑被蛋白酶體降解的過程。誘導降解更直接的方法是將目的蛋白直接與蛋白質降解酶靶向連接。E2靶標的識別可通過E2生物酶工程還原,即在非細胞的體外條件下,將含有合適蛋白結合肽的E2融合到靶蛋白C末端[25]。這種蛋白質間的相互作用將降解酶與目標蛋白非共價結合的策略應用于蛋白酶體介導的降解過程。本試驗克隆了松材線蟲Bx-ubc-3基因,研究了該基因編碼的泛素結合酶Bx-UBC-3的晶體結構,模擬了相關蛋白的相互作用,基于這些研究,發現了一個理論泛素通路。本研究獲得的信息將有助于今后Bx-UBC-3的功能研究、工程化蛋白的開發利用,以及開發用于植物保護的Bx-UBC-3功能控制技術。對松材線蟲中泛素依賴途徑調控的蛋白質降解選擇性靶向性的進一步認識,也將為植物線蟲的有效防治提供新的思路。
本試驗克隆了松材線蟲的一種泛素結合酶(E2)基因Bx-ubc-3,并對其功能進行了研究。研究鑒定了Bx-UBC-3和Bx-泛素之間的相互作用。其結構研究表明了Bx-UBC-3與其配體的互作機理,說明了Bx-UBC-3是開發有效的松材線蟲藥物的一個潛在靶標,為Bx-UBC-3抑制劑的開發提供了理論基礎,同時也為松材線蟲的防治工作提供了新的思路。
【參 考 文 獻】
[1]DOVE K K, KEMP H A, BONA K R D, et al. Two functionally distinct E2/E3 pairs coordinate sequential ubiquitination of a common substrate in Caenorhabditis elegans development[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(32):E6576-E6584.
[2]BELLARE P, SMALL E C, HUANG X, et al. A role for ubiquitin in the spliceosome assembly pathway[J]. Nature Structural & Molecular Biology, 2008, 15(5):444-451.
[3]倪曉光,趙平.泛素-蛋白酶體途徑的組成和功能[J].生理科學進展,2006,37(3):255-258.
NI X G, ZHAO P. The components and functions of ubiquitin-proteasome pathway[J]. Progress in Physiological Sciences, 2006, 37(3):255-258.
[4]HAMILTON K S, ELLISON M J, BARBER K R, et al. Structure of a conjugating enzyme-ubiquitin thiolester intermediate reveals a novel role for the ubiquitin tail[J]. Structure, 2001, 9(10):897-904.
[5]SAHA A, LEWIS S M, KLEIGER G, et al. Essential role for ubiquitin-ubiquitin-conjugating enzyme interaction in ubiquitin discharge from cdc34 to substrate[J]. Molecular Cell, 2011, 42(1):75-83.
[6]LIU X, ZHANG Y, HU Z, et al. The catalytically inactive mutation of the ubiquitin-conjugating enzyme cdc34 affects its stability and cell proliferation[J]. Protein Journal, 2018, 37(2):132-143.
[7]AVGOUSTI D C, PALANI S, SHERMAN Y, et al. CSR-1 RNAi pathway positively regulates histone expression in C. elegans[J]. Embo Journal, 2012, 31(19):3821-3832.
[8]劉立宏,王峰,馬玲,等.松材線蟲乙酰膽堿酯酶1基因沉默及蛋白抑制劑[J].林業科學,2015,51(1):66-73.
LIU L H, WANG F, MA L, et al. BxACE1 gene RNAi and protein inhibition of Acetylcholinesterase in pine wood nematode Bursaphelenchus xylophilus[J]. Scientia Silvae Sinicae, 2015, 51(1):66-73.
[9]何龍喜,吉靜,邱秀文,等.世界松材線蟲病發生概況及防治措施[J].林業工程學報,2014,28(3):8-13.
HE X L, JI J, QIU X W, et al. General situation and control measures of pine wood nematode disease in the world[J]. Journal of Forestry Engineering, 2014, 28(3):8-13.
[10]HASEGAWA K, FUTAI K, MIWA S, et al. Early embryogenesis of the pinewood nematode Bursaphelenchus xylophilus[J]. Development Growth & Differentiation, 2004, 46(2):153-161.
[11]KIKUCHI T, COTTON J, DALZELL J J, et al. Genomic insights into the origin of parasitism in the emerging plant pathogen Bursaphelenchus xylophilus[J]. Plos Pathogens, 2011, 7(9):e1002219.
[12]王峰,馬玲,陳俏麗,等.松材線蟲熱激轉錄因子Bx-HSF-1基因的克隆及表達分析[J].林業科學,2016,52(12):92-98.
WANG F, MA L, CHEN Q L, et al. Cloning and expression analysis of the heat shock transcription factor Bx-HSF-1 gene in Bursaphelenchus xylophilus (Aphelenchida: Aphelenchoididae)[J]. Scientia Silvae Sinicae, 2016, 52(12): 92-98.
[13]陳俏麗,王峰,李丹蕾,等.‘大白谷CC-NBS-LRR蛋白編碼基因抗干尖線蟲侵染時空表達研究[J].中國農學通報,2015,31(21):279-283.
CHEN Q L, WANG F, LI D L, et al. Spatial-temporal expression of CC-NBS-LRR protein-coding gene resisting to infection of Aphelenchoides besseyi in Oryza sativa L. ssp. Indica[J]. Chinese Agriculture Science Bulletin, 2015, 31(21):279-283.
[14]WANG F, WANG Z Y, LI D L, et al. Identification and characterization of a Bursaphelenchus xylophilus (Aphelenchida Aphelenchoididae) thermotolerance-related gene: Bx-HSP90[J]. International Journal of Molecular Sciences, 2012, 13(7):8819-8833.
[15]WANG F, LI D L, CHEN Q L, et al. Genome-wide survey and characterization of the small heat shock protein gene family in Bursaphelenchus xylophilus[J]. Gene, 2016, 579(2):153-161.
[16]WANG B W, MA L, WANG F, et al. Low temperature extends the lifespan of Bursaphelenchus xylophilus through the cGMP Pathway[J]. International Journal of Molecular Sciences, 2017, 18(11):2320.
[17]BIASINI M, BIENERT S, WATERHOUSE A, et al. SWISS-MODEL: modelling protein tertiary and quaternary structure using evolutionary information[J]. Nucleic Acids Research, 2014, 42(W1):252-258.
[18]SZKLARCZYK D, FRANCESCHINI A, KUHN M, et al. The STRING database in 2011: functional interaction networks of proteins, globally integrated and scored[J]. Nucleic Acids Research, 2011, 39:561-568.
[19]ZHENG N, WANG P, JEFFREY P D, et al. Structure of a c-Cbl-UbcH7 complex: RING domain function in ubiquitin-protein ligases[J]. Cell, 2000, 102(4):533-539.
[20]CHEN Q L, LI D L, FENG W, et al. Trehalose metabolism genes of Aphelenchoides besseyi (Nematoda: Aphelenchoididae) in hypertonic osmotic pressure survival[J]. Biology Open, 2017, 6(5):664-672.
[21]ARAI R, YOSHIKAWA S, MURAYAMA K, et al. Structure of human ubiquitin-conjugating enzyme E2 G2 (UBE2G2/UBC7)[J]. Acta Crystallographica Section F, 2006, 62(4):330-334.
[22]LIU X, ZHANG Y, HU Z H, et al. The catalytically inactive mutation of the ubiquitin-conjugating enzyme CDC34 affects its stability and cell proliferation[J]. Protein Journal, 2018, 37(2):132-143.
[23]PAPALEO E, RANZANI V, TTRIPODI F, et al. An acidic loop and cognate phosphorylation sites define a molecular switch that modulates ubiquitin charging activity in cdc34-like enzymes[J]. Plos Computational Biology, 2011, 7(5):1-11.
[24]METZGER M B, PRUNEDA J N, KLEVIT R E, et al. RING-type E3 ligases: master manipulators of E2 ubiquitin-conjugating enzymes and ubiquitination[J]. Biochimica Et Biophysica Acta, 2014, 1843(1):47-60.
[25]CALLIS J. The ubiquitination machinery of the ubiquitin system[J]. The Arabidopsis Book, 2014, 12(12):e0174.

