黑曲霉Aspergillus niger全基因組DNA提取方法的改良與比較
蔡程山,王雨,白飛榮,翟磊,張天賜,胡海蓉,姚粟
(中國食品發酵工業研究院有限公司, 中國工業微生物菌種保藏中心,北京,100015)
黑曲霉(Aspergillusniger)屬于子囊菌門Ascomycota,曲霉科Aspergillaceae,曲霉屬Aspergillus中的一個常見種,廣泛分布于世界各地的糧食、植物性產品和土壤中。美國食品與藥品管理局于1987年將黑曲霉列為安全菌種[1],已廣泛用于淀粉酶、酸性蛋白酶、纖維素酶、果膠酶、葡萄糖氧化酶、檸檬酸、葡糖酸和沒食子酸等的生產。近年,研究發現部分食品工業用黑曲霉菌株具有產伏馬菌素能力[2-3],該毒素具有急性毒性和潛在致癌性,影響人類和動物健康[4-8]。辨別產毒菌株和非產毒菌株對我國食品工業用黑曲霉菌株使用安全性十分重要[1]。利用全基因組測序(whole genome sequencing,WGS)的分型方法,能夠在全基因組堿基序列的基礎上進行生物信息學分析,提高黑曲霉產毒菌株分辨能力。目前,由于黑曲霉細胞壁結構復雜、代謝產生大量黑色色素、胞內多糖、生物酶等原因,存在全基因組DNA提取難度大、可參考的全基因組數據少、全基因組分析方法尚不成熟的問題[9-11]。獲得完整性好、純度高的基因組DNA[12-14],是后續全基因組分析的前提,對黑曲霉產毒菌株分型辨別具有重要意義。
目前關于絲狀真菌全基因組DNA提取方法主要有CTAB法、試劑盒法和氯化芐法[15],細胞壁破裂方法主要有石英砂研磨法和液氮研磨法。黑曲霉屬絲狀真菌,細胞壁結構復雜堅固,富含細胞壁多糖和幾丁質,各種成分相互結合,使其具有一定強度[16],在進行全基因組DNA提取時很難將細胞壁破裂,而且細胞中存在大量多糖,使得DNA極少能溶出[17-21],如果長時間研磨會導致溶出的DNA斷裂,破壞DNA完整結構[22]。黑曲霉中存在大量色素,使得全基因組DNA的提取更加困難[9-11],常規DNA提取方法得到的DNA樣品通常A260/A280值>2.0,A260/A230值<2.0,樣品中有色素、RNA、糖類、鹽類或有機試劑的污染,雖可成功進行常規PCR擴增,但無基因組主帶,不滿足全基因組測序要求[23-24]。本文根據目前已有方法,針對黑曲霉菌株特性,優化了黑曲霉全基因組DNA提取方法,為黑曲霉基因組的分析研究奠定了基礎。
1 材料與方法
1.1 供試材料
1.1.1 供試菌株
該研究所用黑曲霉(Aspergillusniger)菌株,來源于20個省市,從23種食品基質中分離而來,目前保藏于中國工業微生物菌種保藏中心,菌株編號為CICC 2286、CICC 2629、CICC 2693、CICC 40040、CICC 40043、CICC 40046、CICC 40068、CICC 40087、CICC 40128、CICC 40130、CICC 40132、CICC 40133、CICC 40137、CICC 40408、CICC 40615、CICC 40755、CICC 40845、CICC 40846、CICC 40847、CICC 40849、CICC 40863、CICC 41115、CICC 41116、CICC 41178、CICC 41194、CICC 41195、CICC 41215、CICC 41255、CICC 41256、CICC 41257、CICC 41258、CICC 41568、CICC 41576、CICC 41586、CICC 41594。
1.1.2 試劑及儀器
溴代十六烷基三甲胺(cetyl trimethyl ammonium bromide,CTAB),Sigma公司;20 g/L RNA酶、20 g/L蛋白酶K,天根生化科技(北京)有限公司;Tris-飽和酚,Solarbio Sigma;氯仿,北京市通廣精細化工公司;異戊醇,西隴化工股份有限公司;異丙醇,北京化工廠;乙酸鈉,廣州化學試劑廠;DNA純化試劑盒,OMEGA;真菌DNA提取試劑盒,OMEGA;馬鈴薯葡萄糖瓊脂培養基,北京陸橋技術股份有限公司。
IPP 260恒溫培養箱,美墨爾特(上海)貿易有限公司;TProfessional PCR儀,北京世貿遠東科學儀器有限公司;EC 250-90電泳儀,美國ThermoEC有限公司;Bead Ruptor 12生物樣品均質器,奧然科技(北京)有限公司;核酸蛋白分析儀BioDrop, 豪沃生物科技(上海)有限公司;GelDoc EZ全自動免染凝膠成像分析系統,美國伯樂Bio-ra有限公司;AC2-4S1生物安全柜,新加坡藝思高科技有限公司。
1.2 菌株的培養與收集
將PDA培養基傾倒在培養皿內,待培養基冷卻凝固,在培養皿內平整鋪放無菌玻璃紙,用無菌棉簽蘸取培養好的黑曲霉試管斜面中的孢子,均勻涂布整個培養皿,25 ℃倒置培養。培養結束后,用無菌接種環將玻璃紙上的菌絲體刮至新的培養皿內待用。
1.3 黑曲霉培養條件確定
將培養平板分別25 ℃倒置培養1、2和3 d,觀察黑曲霉菌落生長狀態、菌體生物量、產孢子情況,確定黑曲霉全基因組DNA提取的最佳培養時間。
1.4 菌絲體細胞壁處理方法
生物均質器菌體破碎法:稱取200 mg菌絲體至含0.4 g的0.1 mm石英砂,0.4 g的1.5 mm鋯株,0.3 g 的3.0 mm鋯珠的無菌研磨管內,加入200 μL 的CTAB裂解液,擰緊管蓋,放入生物樣品均質器卡槽內,破碎時長2 min,顯微觀察破碎后的菌體樣品。液氮研磨法:將菌絲體加入無菌研缽中,倒入液氮,迅速研磨,在液氮蒸發凈前,再次加入液氮,繼續迅速研磨菌絲體至粉末狀。顯微觀察細胞壁破裂后的菌絲體樣品與生物樣品均質器細胞壁破裂法的結果差異。
1.5 DNA提取方法
菌絲體液氮研磨后應用真菌DNA提取試劑盒法、CTAB法和CTAB法結合試劑盒純化法,分別提取4株黑曲霉CICC 40137、CICC 40130、CICC 40132和CICC 41115全基因組DNA,對比不同方法所提全基因組DNA差異,篩選出適用于全基因組測序的黑曲霉DNA提取方法。
1.5.1 菌絲體液氮研磨后應用真菌DNA提取試劑盒法(方法一)
將菌絲體加入無菌研缽中,倒入液氮,迅速研磨,在液氮蒸發凈前,再次加入液氮,繼續迅速研磨菌絲體至粉末狀,稱取液氮研磨好的菌絲體粉末200 mg裝入1.5 mL離心管中,后續步驟按照真菌DNA提取試劑盒說明書準確提取。
1.5.2 菌絲體液氮研磨后應用CTAB法(方法二)
菌絲體研磨方法同1.5.1,稱取200 mg液氮研磨好的菌絲體粉末至1.5 mL離心管中,加入600 μL的CTAB裂解液,65 ℃水浴45 min,13 000 r/min離心10 min;取上清于一新離心管中,加等體積的V(酚)∶V(氯仿)∶V(異戊醇)=25∶24∶1,12 000 r/min離心10 min;取上清,加等體積的V(氯仿)∶V(異戊醇)=24∶1,12 000 r/min離心10 min;取上清,加等體積的異丙醇試劑,-20 ℃沉淀30 min,12 000 r/min離心20 min;收集沉淀,75%酒精沖洗,超凈臺真空干燥;100 μL TE溶解DNA,-20 ℃保存備用[25-26]。
1.5.3 菌絲體液氮研磨后應用CTAB法提取結合試劑盒純化法(方法三)
菌絲體研磨方法同1.5.1,稱取200 mg液氮研磨好的菌絲體粉末至1.5 mL離心管中,加入600 μL的CTAB裂解液和少量的聚乙烯吡咯烷酮(polyvingl pyrroliddone,PVP),65 ℃水浴45 min,13 000 r/min離心10 min;取上清于一新離心管中,加等體積的V(酚)∶V(氯仿)∶V(異戊醇)=25∶24∶1,12 000 r/min離心10 min;取上清,加等體積的V(氯仿)∶V(異戊醇)=24∶1,12 000 r/min離心10 min;取上清,加 0.7倍體積的異丙醇,0.1倍體積的3 mol/L NaAc,-20 ℃沉淀30 min,12 000 r/min離心20 min;收集沉淀,75%酒精沖洗,超凈臺真空干燥;100 μL TE 溶解DNA,加入1 μL的RNA酶,37 ℃水浴 1 h;加入400 μLV(氯仿)∶V(異戊醇)=24∶1,12 000 r/min離心10 min,重復2次。采用OMEGA公司cycle-pure DNA純化試劑盒進行純化,得到高純度基因組DNA,-20 ℃保存備用。
1.6 DNA樣品濃度測定
使用BioDrop測定DNA樣本濃度,根據A260/A280和A260/A230值判斷DNA純度[27]。
1.7 DNA樣品凝膠成像檢測
吸取5 μL基因組DNA樣品與6×Loading Buffer混合后,點樣于質量分數為0.8%的瓊脂糖凝膠中,染色劑為GoldView,在電泳緩沖液為 1×TAE,電壓為 110 V 的條件下電泳25 min,之后在凝膠成像儀上觀察DNA片段的大小、完整性及電泳條帶的清晰度,進行拍照。
1.8 全基因組DNA提取方法應用
將31株黑曲霉菌株分別用無菌棉簽蘸取培養好的黑曲霉試管斜面中的孢子,接種在鋪有玻璃紙的PDA培養皿內,涂抹整個培養皿,25 ℃倒置培養。培養結束后,用無菌接種環將玻璃紙上的菌體刮至研缽內液氮研磨,應用CTAB法提取并結合試劑盒純化法提取全基因組,對該方法進行應用驗證。
2 結果與分析
2.1 黑曲霉培養條件確定
黑曲霉培養1 d菌落為灰白色,菌體量少(圖1-a);培養2 d菌落為白色,菌落覆蓋整個平板,產孢量少(圖1-b);黑曲霉培養至第3天,菌落產生大量黑色孢子,菌體老化,細胞壁堅固,收集的菌絲體含有孢子,DNA提取過程中會溶出大量黑色素,影響DNA樣品的提取(圖1-c),因此選用收集培養2 d的黑曲霉菌絲體作為全基因組DNA提取最佳時間。
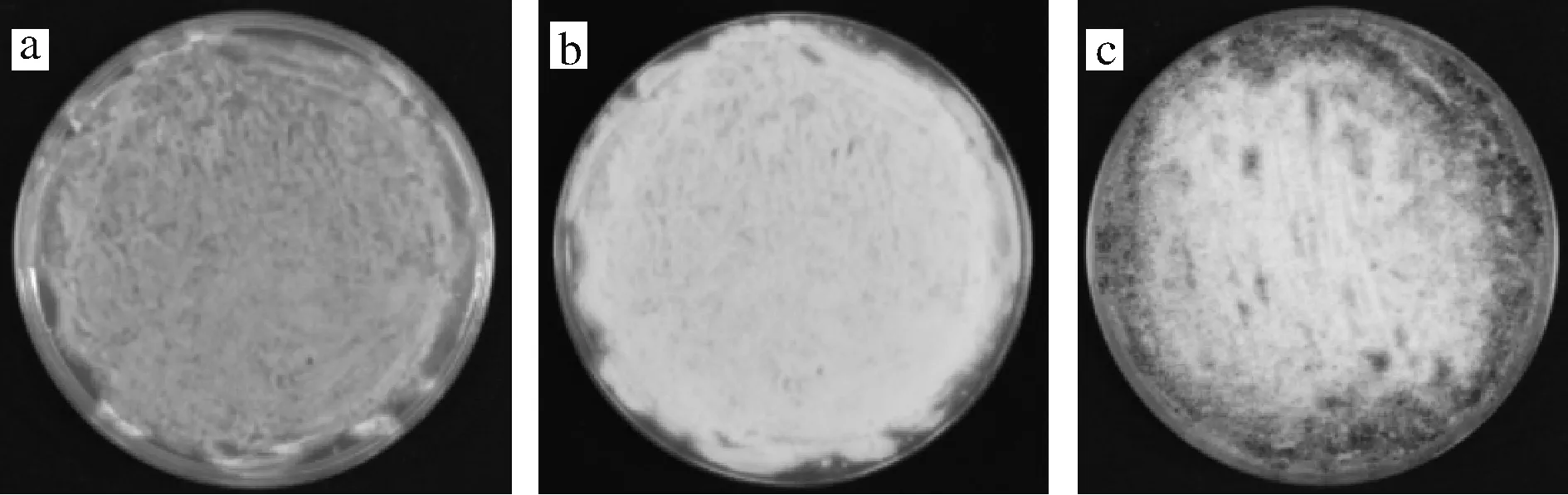
a-培養1 d;b-培養2 d;c-培養3 d
2.2 菌絲體細胞壁處理方法確定
將收集的菌體采用生物均質器研磨法和液氮研磨法進行顯微對比,兩種不同細胞破裂方法對菌體破碎情況明顯不同。圖2-a為研磨前菌體狀態,菌絲體結構完整;圖2-b為生物均質器研磨法結果顯微照片,大部分菌體細胞結構被破壞,但破碎結果不均勻,仍有部分菌體結構未破碎;圖2-c為液氮研磨結果顯微照片,菌絲體破碎充分、均勻,未觀察到完整菌體結構。與研磨前相比,兩種破壁方法雖然都有一定程度的破壞,但菌體破碎結果存在明顯差異,液氮研磨法研磨更充分均勻,因此選用為黑曲霉全基因組提取細胞壁處理方法。

a-研磨前菌體形態;b-生物均質器破壁結果;c-液氮研磨破壁結果
2.3 DNA樣品濃度檢測
采用BioDrop對3種方法的樣品基因組DNA進行測定,結果如表1所示。方法一為液氮研磨后應用真菌DNA提取試劑盒法測定結果,CICC 40137、CICC 40130、CICC 40132和CICC 41115四株菌DNA濃度分別為109.6、89.35、321.76和234.51 ng/μL,菌株CICC 40130、CICC 40132和CICC 41115的A260/A280值在1.8~2.0,A260/A230值均>2,證明DNA純度較高。但CICC 40137菌株A260/A280值為2.132>2.0,證明RNA含量較高;方法二為菌絲體液氮研磨后應用CTAB法測定結果,CICC 40137、CICC 40130、CICC 40132和CICC 41115四株菌DNA濃度分別為351.59、310.75、362.43和290.73 ng/μL,但4株菌A260/A280值均>2.0,證明DNA樣品中有RNA殘留,A260/A230值均<2.0,說明樣品DNA中含有較多的糖類、鹽類或有機試劑的污染;方法三為菌絲體液氮研磨后應用CTAB法提取結合試劑盒純化法測定結果,CICC 40137、CICC 40130、CICC 40132和CICC 41115四株菌DNA濃度分別為125.72、88.67、97.71和85.43 ng/μL,A260/A280值均在1.8~2.0,A260/A230值均>2.0,說明本方法提取到的基因組DNA樣品蛋白質和RNA含量很低,純度濃度均較好,吸光增色反應明顯,在260 nm處吸光度最高,證明基因組純度很高,優于方法一和方法二所提DNA樣品。
2.4 電泳檢測
分別對3種方法所得到的基因組DNA 進行電泳檢測,檢測結果如圖3所示。圖3-a為方法一提取的DNA樣品電泳結果,由圖可知未見到完整DNA主條帶,DNA長度均<5 000 bp。圖3-b是應用方法二提取的DNA樣品電泳結果,泳道上有黑色陰影,未顯示完整基因組DNA主帶,多為小分子片段,<5 000 bp;圖3-c為方法三所提DNA樣品電泳結果,CICC 40137、CICC 40130、CICC 40132和CICC 41115四株菌都有DNA主條帶,加樣孔附近滯留的物質也很少,基因組大小約為23 kb。方法一和方法二DNA鏈狀結構破壞嚴重,雖能滿足PCR擴增,但未見到全基因組DNA條帶。

表1 基因組DNA濃度檢測
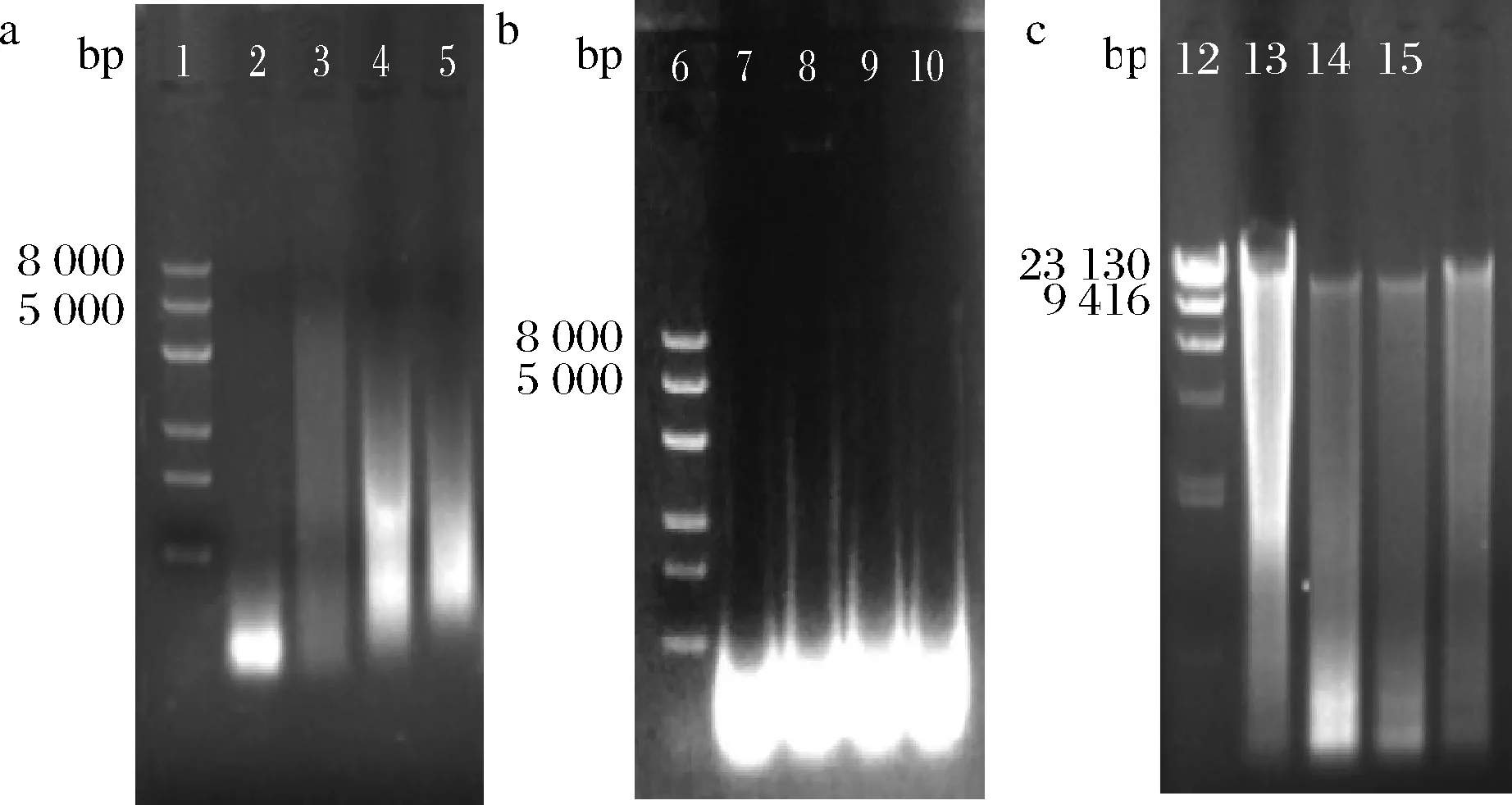
1、6-DL8000 Marker;2、7、11-CICC 40137;3、8、12-CICC 40130;4、9、13-CICC 40132;5、10和14-CICC 41115;15-λDNA/Hind Ⅲ Markera-方法一;b-方法二;c-方法三
2.5 全基因組DNA提取方法應用
如圖4所示,菌絲體液氮研磨后應用CTAB法提取,結合試劑盒純化法,對31株不同分離源黑曲霉菌株進行方法驗證結果,7泳道全基因組DNA條帶相對較弱,推測由于液氮研磨步驟研磨不充分,DNA溶出量少。8、11和13泳道的最前端出現明亮條帶,RNA酶與樣品中RNA反應不徹底,導致樣品中含有RNA。但31株黑曲霉DNA樣品均有23 kb大小的完整全基因組DNA主條帶。對該DNA樣品進行濃度測定,實驗結果如表2所示,濃度為52.24~212.37 ng/μL。全基因組測序要求DNA濃度>12.5 ng/ μL,DNA總量均在2 μg以上,31株黑曲霉DNA樣品滿足全基因組測序工作,證明該方法適用于黑曲霉全基因組DNA的提取。
3 結論
本研究通過對3種常見真菌基因組DNA提取方法比較分析,確定了一種高質量黑曲霉全因組提取方法,具體為菌絲體經液氮研磨后,采用CTAB法提取并結合試劑盒純化。該方法提取的基因組DNA條帶完整、純度高,可以滿足WGS要求,方法優點為:(1)確定了黑曲霉實驗菌株的培養條件和細胞壁破裂方法。因為黑曲霉主要成分為幾丁質,與細菌或者酵母相比其結構更加堅固。但是絲狀真菌培養過程中,細胞壁堅固程度是發生變化的,菌絲體在幼嫩時期細胞壁相對薄弱一些,老熟之后堅固,培養2 d的黑曲霉菌絲體提取全基因組DNA時間最佳[28]。液氮研磨相比生物樣品均質器破碎相比,細胞壁更加充分,確保DNA釋放。(2)CTAB裂解液中加入PVP、RNA酶和蛋白酶K。PVP可以去除多糖、酚類、醌類等物質[23,29-31],RNA酶去除RNA,蛋白酶K有效除去蛋白。(3)結合cycle-pure DNA純化試劑盒進行純化。黑曲霉菌絲體中富含色素、多糖、蛋白質[32],傳統CTAB 提取方法盡管對DNA產物進行洗滌除雜,但是色素物質殘留較多,在進行凝膠電泳檢測時,影響了DNA與染料的結合,因此看不到基因組完整條帶。應用試劑盒純化后雖然基因組DNA有一定損失,但是多糖、色素等雜質可充分除去,避免影響GoldView與基因組結合顯色,由電泳結果可知提升了DNA的純度,有清晰可見的陽性條帶。

1、16、34-λDNA/Hind Ⅲ Marker;2~15菌株編號分別為CICC 40087、CICC 40863、CICC 40046、CICC 40849、CICC 41586、CICC 40133、CICC 40845、CICC 40128、CICC 40040、CICC 40847、CICC 2286、CICC 41178、CICC 41594、CICC 40068;17~33菌株編號分別為CICC 2693、CICC 40043、CICC 40408、CICC 40615、CICC 40755、CICC 40846、CICC 41116、CICC 41194、CICC 41195、CICC 41215、CICC 41255、CICC 41256、CICC 41257、CICC 41258、CICC 41568、CICC 41576、CICC 2629
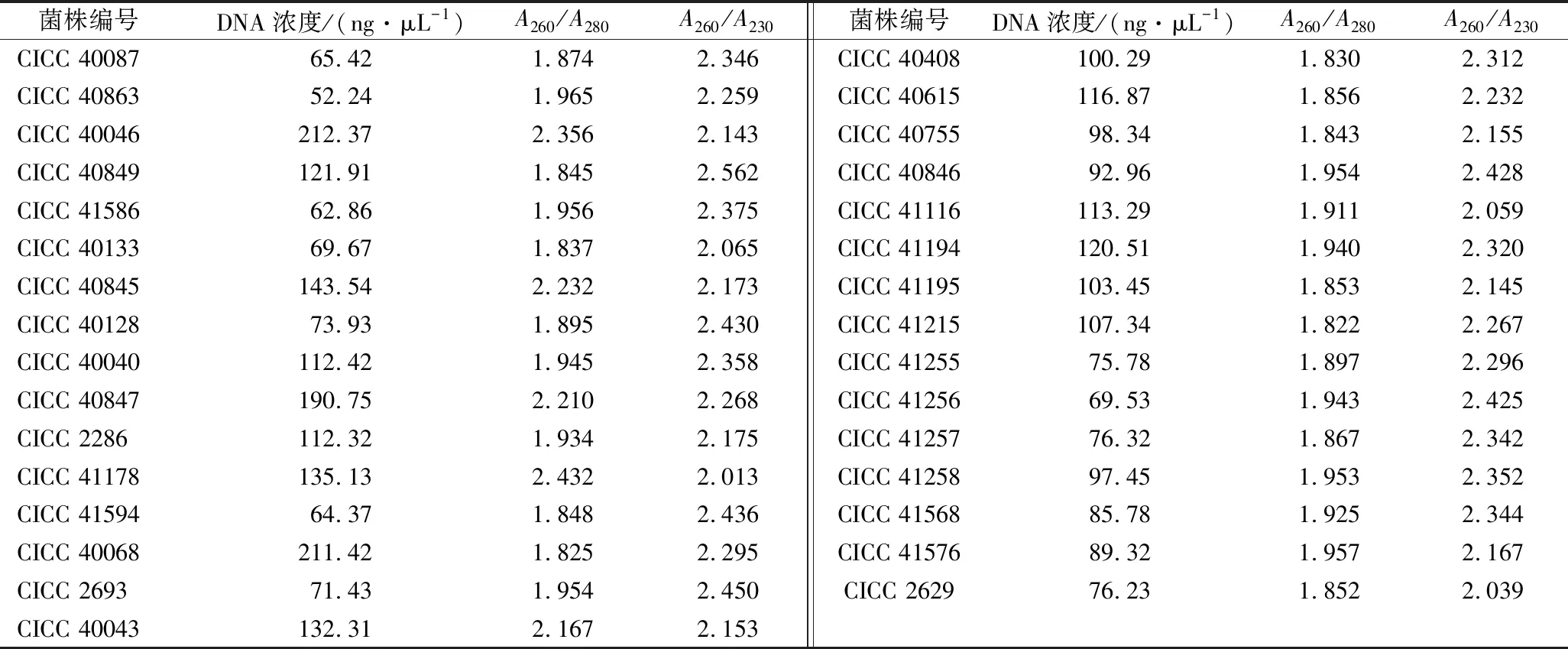
表2 不同分離源31株黑曲霉基因組DNA濃度
本研究確定黑曲霉全基因組DNA提取方法,可滿足WGS工作要求,為黑曲霉基因組數據庫的構建、菌株溯源、產毒基因分析奠定基礎,有助于產毒菌株和非產毒菌株快速準確辨別,對我國食品工業用黑曲霉菌株的使用安全性有重要意義。該法可作為實驗室提取黑曲霉高質量基因組DNA的方法推廣使用。

