基于異質網絡層次注意力機制的基因功能預測
萬美含,熊 贇,朱揚勇
(1.復旦大學 計算機科學技術學院,上海 200433; 2.上海市數據科學重點實驗室,上海 200433;3.上海先進通信與數據科學研究院,上海 200433)
0 概述
基因是具有功能性的DNA片段[1]。由于可通過功能產物的表達或基因表達調控來影響生物體性狀[2],因此確定基因的功能是生物學中的核心問題之一,其對了解疾病的生化過程、識別和驗證新藥物的靶點等都具有重要意義[3]。
基因組測序的快速發展使得生物數據庫中基因和基因組序列的數據規模爆炸式增長,但其中有大量的基因功能仍是未知的[4]。因此,通過已有的基因特性信息對基因的功能進行預測是目前的研究熱點。
目前,已有大量的基因功能預測方法被提出,總體可歸為兩類:一類是基于guilt-by-association原則的方法,即與相似的生物物質(如疾病)相連的基因應共享相同的功能[5],通過融合不同類型的生物數據,構建一個與基因功能相關的網絡來預測基因的功能[6];另一類是基于基因本體(Gene Ontology,GO)的方法,即基因本體通過結構化的術語以分子功能、生物過程和細胞成分3種屬性來描述基因,如文獻[7-9]利用基因本體計算不同基因之間的相似度,實現對基因功能的準確預測。本文結合上述兩類方法,將基因本體數據作為基因節點的屬性,使用多種數據源構建一個基因功能相關異質信息網絡。
近年來,注意力機制受到學者的關注[10],且在各個研究領域得到廣泛應用。在異質網絡表示學習方面,文獻[11]構建了HAN模型,通過引入層次注意力機制進行異質網絡節點表示學習,文獻[12]在其基礎上使用節點結構特征信息構建了HANE模型,但該模型僅適用于無節點屬性的異質網絡。本文將HANE模型擴展到屬性異質信息網絡(Attributed Heterogeneous Information Network,AHIN)中,構建一個具有節點屬性的基因功能相關異質信息網絡,并在此基礎上提出基于層次注意力機制的基因節點表示學習方法HAGE。
1 相關定義
本文通過結合多種類型的公開數據集,構建一個具有節點屬性的基因功能相關異質信息網絡,并在該網絡上應用基于層次注意力機制的網絡表示學習方法,為每一個基因節點生成一個節點嵌入向量,該向量可用于后續的基因功能預測任務。對上述過程中使用的相關概念進行形式化定義:
定義1異質信息網絡[13]是具有多種節點類型或(和)多種邊類型的網絡,表示為G=(V,E,T),其中,V是節點的集合,E是邊的集合。同時,φ:V→Tv是節點到節點類型的映射,φ:E→Te是邊到邊類型的映射,Tv和Te是預設的節點和邊的類型,并滿足|Tv|+|Te|>2,T=Tv∪Te。
由于本文使用的異質信息網絡是基于基因-疾病關系網絡、基因-miRNA關系網絡和miRNA-疾病關系網絡生成的,因此其中包含3種節點類型(基因、疾病和miRNA)和3種邊類型(基因-疾病關系、基因-miRNA關系和miRNA-疾病關系)[14]。
定義2網絡模式[15]是定義在節點類型和邊類型上的一個有向圖,表示為SG={Tv,Te}。
本文構建的基因功能相關異質信息網絡的網絡模式如圖1所示。

圖1 基因功能相關網絡模式

本文中使用的元路徑及其含義如表1所示。

表1 基因功能相關網絡中的元路徑及其含義



2 HAGE模型結構
在基因功能相關異質信息網絡的基礎上,本文提出基于層次注意力機制的基因節點表示學習方法HAGE,為每一個節點學習一個節點嵌入向量。HAGE模型主要包括3個部分,即節點特征抽取、節點層次的注意力機制和元路徑層次的注意力機制。
2.1 節點特征抽取
給定一個異質信息網絡G=(V,E,T)以及元路徑集合{P1,P2,…,Pm},對于V中的每一個節點vi,本文從2個方面考慮該節點的特征:節點的屬性信息ai以及該節點在網絡中的結構特征fi。
在構建的基因功能相關網絡中,基因節點屬性ai來自于基因本體數據,將每個基因對應的本體術語轉化為multi-hot編碼并作為基因節點的屬性。
對于節點網絡中的結構特征fi,本文使用基于元路徑的連接分布來描述。在異質網絡中,不同的元路徑具有不同的語義信息,因此,不同元路徑下相同節點間的連接分布也是不同的。對于同一對基因節點A和B,其通過元路徑基因-疾病-基因連接的路徑與通過元路徑基因-miRNA-基因連接的路徑完全不同,并且路徑的權重和數量也不同,因此,其連接分布也完全不同。

(1)

下面對連接強度矩陣IP進行歸一化,作為節點結構特征矩陣FP:
(2)
最后,將每個節點vi的節點屬性與其基于元路徑的結構特征進行拼接并作為節點的特征向量:
(3)
2.2 節點層次的注意力機制
首先在節點層次上使用注意力機制來學習基于元路徑鄰居節點的重要性,并通過聚合這些擁有不同權重的鄰居節點得到新的特征向量,即如果基因A具有功能f,其鄰居節點中功能與功能f相同或更近似的節點應具有更大的權重,通過聚合不同鄰居節點的嵌入向量及其權重來更新基因節點A的嵌入向量。

(4)
其中,anode是一個深度神經網絡,代表節點層次的注意力機制。對于給定的元路徑P,基于該路徑的所有鄰居節點共享anode。

(5)
其中,σ是激活函數,W是權重矩陣,aP是基于元路徑P節點層次的注意力向量。

(6)

(7)
給定一系列元路徑的集合{P1,P2,…,Pm},基于節點特征向量并利用節點層次的注意力機制可以得到m組新的節點特征向量{ZP1,ZP2,…,ZPm}。
2.3 元路徑層次的注意力機制
基于節點層次的注意力機制可以得到不同元路徑下新的節點特征向量,為得到最終的節點嵌入向量,需要對不同元路徑下的節點特征向量進行融合。
在異質網絡中,不同的元路徑代表不同的語義信息,因此,需要為不同的元路徑分配不同的權重。使用一個元路徑層次的注意力機制[11]來學習不同元路徑的重要程度βP。給定元路徑的集合{P1,P2,…,Pm}以及基于節點層次注意力機制得到的新的節點特征向量{ZP1,ZP2,…,ZPm},為每個元路徑Pi學習一個權重系數βPi,定義為:
βPi=ameta(ZPi)
(8)
其中,ameta是一個深度神經網絡,代表元路徑層次的注意力機制。不同的元路徑將學習到不同的權重,對基因功能預測任務更重要的元路徑將具有更大的權重。
為學習不同元路徑的重要程度,首先對基于節點層次的注意力機制得到的節點特征向量進行非線性變換,然后將變換后的特征向量與元路徑層次的注意力向量q的相似度作為元路徑的重要程度。因此,對于元路徑Pi,其重要程度wPi表示為:
(9)
其中,W是權重矩陣,b是偏置向量,q是元路徑層次的注意力向量。
得到每條元路徑的重要程度wi后,對它們進行歸一化處理,得到每條元路徑的權重系數βi:
(10)
對不同元路徑下的節點特征向量進行融合,得到最終的節點嵌入矩陣Z:
(11)
為提高模型的精度,本文增加一個全連接層用于分類,并利用部分有標簽的節點對模型進行優化,使用交叉熵作為損失函數:
(12)
其中,VL為擁有標簽的節點集合,Yl為節點的標簽,Zl為該節點的最終節點嵌入矩陣,C是分類器的參數。最后通過反向傳播對模型進行優化,學習節點的節點嵌入向量。
2.4 HAGE算法描述
注意力的計算可以在所有節點和元路徑下單獨計算,因此,HAGE模型支持并行運算。給定一個元路徑P,節點層次的注意力機制時間復雜度為O(VPF1F2K+EPF1K),其中,VP是節點的數量,EP是基于元路徑的節點對的數量,K是多頭注意力機制的數量,F1是節點特征的數量,F2是輸出的節點嵌入向量的維度。總體的時間復雜度與節點數量以及基于元路徑的節點對呈線性關系。
HAGE模型的算法描述如下:
算法1HAGE算法
輸入異質信息網絡G=(V,E,T),元路徑集合{P1,P2,…,Pm},節點屬性集合{ai,i∈V},多頭注意力機制數量K
輸出節點嵌入矩陣Z
for i∈V do
end
for Pi∈{P1,P2,…,Pm} do
for k=1,2,…,K do
for i∈V do
end
計算節點層次的特征向量
end
拼接得到節點層次的嵌入向量
end
計算元路徑層次的權重系數βPi;
end

反向傳播并更新HAGE模型的參數;
return節點嵌入矩陣Z
3 實驗結果與分析
3.1 實驗數據集
本文構建的具有節點屬性的基因功能相關異質信息網絡使用以下數據集:
1)使用DisGeNET[19]數據集構建基因-疾病關系網絡。每條邊的權重根據可靠性設為0~1,選取數據集中權重在0.3以上的3 833條基因-疾病關系來構建網絡。
2)使用miRTarBase[20]數據集構建基因-miRNA關系網絡。miRTarBase是一個手工收集的經過實驗驗證的miRNA及其靶基因關系的數據集,選取其中7 150對經過蛋白質印跡法以及報告基因分析驗證的基因-miRNA關系,并將權重設為1。
3)使用2個數據集構建miRNA-疾病關系網絡。第1個數據集來自文獻[21]提供的242條miRNA-疾病關系;第2個數據集來自miRNet[22]數據集,選取其中疾病名稱可以對應到OMIM編號的666條miRNA-疾病關系。將2個數據集進行融合,去除重復數據后,共有267個miRNA和59個疾病組成的878條miRNA-疾病關系。由于可信度較高,因此將權重設為1。
4)使用基因本體GO數據庫[23-24]中得到所有基因節點的本體信息,將其作為基因節點的節點屬性,共得到4 402個基因節點的基因本體信息。
5)使用MSigDB[25]基因集數據庫中的基因家族作為節點的標簽。MSigDB將數據庫中的基因集按照PubMed中文獻的定義進行分類,同一家族的基因具有相似的功能性,它們具有同源性或者生物化學活性。結果總共有1 185個基因節點獲得了所屬的基因家族標簽。
實驗數據集具體描述如表2所示。

表2 實驗數據集描述
3.2 對比算法
為評估本文方法的性能,選取以下算法作為對比方法:
1)GraphSAGE[26]。GraphSAGE通過聚集局部鄰居節點的特征來學習節點的節點嵌入向量。本文使用平均聚合器版本的GraphSAGE來證明為不同鄰居節點以及元路徑分配不同注意力的重要性。
2)GAT[27]。GAT是一個基于注意力機制的同質網絡表示學習方法,其注意力系數通過單層前饋神經網絡學習。本文在不同元路徑上使用GAT,選擇表現最好的作為最終結果。
3)HAGE w/o struc。HAGE w/o struc是HAGE的變種,其僅使用節點屬性作為節點初始特征向量,不考慮節點在網絡中的結構特征。
4)HAGE w/o node。HAGE w/o node是HAGE的變種,其不使用節點層次的注意力機制,僅為不同的基于元路徑的鄰居節點分配相同的權重系數。
5)HAGE w/o meta。HAGE w/o meta是HAGE的變種,其不使用元路徑層次的注意力機制,僅為不同的元路徑分配相同的權重系數。
3.3 實驗設置
隨機初始化模型參數,并且使用Adam[28]作為模型的優化器。其中,學習率設置為0.001,正則化參數設置為0.005,多頭注意力機制數量K設置為8,元路徑層次的注意力向量q的維度為128,最終的節點嵌入向量維度為128。實驗運行環境為64位Linux系統,GPU為NVIDIA GTX 1080 Ti。
3.4 節點分類
本文使用Micro-F1、Macro-F1、Average Precision 和AUC作為模型評價指標,實驗結果如表3所示。
由表3可以看出,在Micro-F1、Macro-F1、Average Precision和AUC這4種不同的指標下,HAGE模型的分類效果均為最優。相比于GraphSAGE和GAT 2種同質網絡表示學習方法,HAGE由于考慮異質網絡的特點即不同元路徑具有不同的語義信息,為不同的元路徑分配不同的權重,因此能夠取得更好的分類性能。與HAGE w/o struc、HAGE w/o node和HAGE w/o meta相比,HAGE的分類效果均有所提升,由此表明同時考慮網絡結構特征、節點層次以及元路徑層次注意力機制的重要性。
3.5 模型性能分析
為分析本文模型的效率性能,構建不同規模的屬性異質信息網絡進行實驗,結果如表4所示。

表4 本文模型時間效率
3.6 參數敏感性分析
對實驗中使用的參數敏感性進行測試,研究不同參數對模型結果的影響。
1)多頭注意力機制數量
為測試多頭注意力機制的效果,設置不同K值進行測試,當K=1時退化為單頭注意力機制,實驗結果如圖2所示。可以看出,隨著K值的增加,AUC的值也得到提升,當K=8時模型的分類性能最好。

圖2 多頭注意力機制數量對AUC的影響
2)元路徑層次的注意力向量維度
元路徑層次的注意力機制的分類效果受元路徑層次的注意力向量q的影響,因此,在不同維度的注意力向量q下進行測試,實驗結果如圖3所示。可以看出,當注意力向量q的維度為128時,模型的分類性能最好。
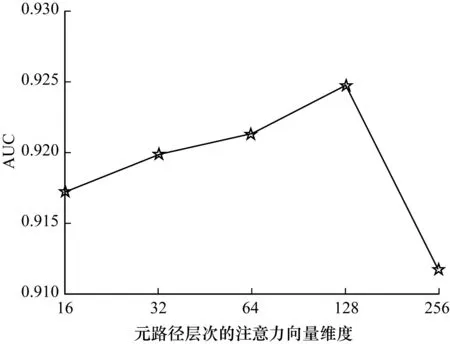
圖3 元路徑層次的注意力向量維度對AUC的影響
3)節點嵌入向量的維度
模型的分類效果受最終的節點嵌入向量Z維度的影響,因此對不同維度的節點嵌入向量Z進行測試,實驗結果如圖4所示。可以看出,模型的分類性能在維度為128時效果最好,后續隨著維度的繼續增加,AUC略微降低。

圖4 節點嵌入向量維度對AUC的影響
3.7 注意力機制性能分析
在學習基因節點的嵌入向量時,本文考慮了不同元路徑下的鄰居節點以及元路徑的重要性,并為它們分配不同的權重系數。為更好地理解權重的意義,分別從節點層次注意力機制以及元路徑層次注意力機制方面進行分析。
1)節點層次注意力機制
本文以基因CHEK2為例,其基于元路徑基因-疾病-基因(GDG)的鄰居如圖5所示,注意力權重系數如圖6所示。其中,基因CHEK2、BRCA2、RB1、BRCA1和TP53同屬于家族tumor suppressors(抑癌基因),RNASEL屬于家族protein kinases(蛋白激酶),HOXB13屬于家族homeodomain proteins(同源域蛋白),PIK3CA屬于家族oncogenes(致癌基因)。

圖5 基因CHEK2在元路徑GDG下的鄰居

圖6 基因CHEK2鄰居的權重系數分布
從圖6可以看出,具有相同或相似功能的同家族的鄰居基因節點的權重系數較大,其他家族的鄰居基因節點權重系數較小。其中BRCA1的權重最高,文獻[29]指出CHEK2和BRCA1參與的DNA修復有關,與乳腺癌發生有較密切的關系,因此,它們之間的功能關聯更密切。由此可見,本文模型可以較好地學習到基因節點層次的重要性。
2)元路徑層次注意力機制
為分析模型學習到的不同元路徑的權重系數是否反映了該元路徑對基因功能預測任務的重要性,對比僅使用該元路徑進行基因功能預測的結果以及該元路徑的注意力權重系數,結果如圖7所示。

圖7 3種元路徑的AUC與注意力權重系數對比
由圖7可以看出,單個元路徑的預測結果與該元路徑的注意力權重系數是成正比的,即該元路徑單獨進行基因功能預測時得到的AUC越高,其注意力權重系數越大。由此可見,本文模型能夠較好地學習到不同元路徑對基因功能預測任務的重要性。
4 結束語
本文提出基于異質網絡層次注意力機制的基因節點表示學習方法HAGE。結合不同來源的數據集構建一個具有節點屬性的基因功能相關網絡,使用節點屬性以及節點在網絡中的結構特征作為節點初始向量,并通過層次注意力機制為每一個基因節點學習一個節點嵌入向量,將其用于后續的基因功能預測任務。實驗結果表明,與GraphSAGE、GAT等方法相比,本文方法能夠取得較好的預測效果。下一步將把本文方法拓展到不同的生物數據集中進行預測,如蛋白質交互網絡、miRNA基因共表達網絡和代謝網絡等。

