肺腺癌相關長鏈非編碼RNA生物標志物識別
于曉慶
(上海應用技術大學 理學院,上海 201418)
肺癌是全球因癌癥導致人類死亡的最大因素。到目前為止,肺癌的5年生存率僅為18%[1]。在肺癌的亞型中,非小細胞肺癌約占85%。而肺腺癌(lung adenocarcinoma,LUAD)是非小細胞肺癌中最常見的惡性病理類型。近些年,隨著靶向藥物的出現,肺腺癌的5年生存率不斷在提高,但由于缺乏有效的診斷和預后生物標志物,死亡率仍然很高[2-3]。 因此,為了降低肺腺癌患者的死亡率,迫切需要尋找有效的分子標志物,為患者的診斷和臨床治療提供一定的理論依據。
長非編碼RNA(long noncoding RNA,LncRNA)是長度超過200個核苷酸的非編碼基因。過去很長一段時間,這些非編碼基因曾被認為是基因組的“暗物質”,并不具有生物學功能。然而,隨著實驗技術和計算機技術的發展,越來越多的研究表明,LncRNA通過不同的機制參與了細胞的幾乎整個生命過程,并在細胞分化、代謝、細胞增殖和凋亡、轉錄、染色質水平的表觀遺傳學狀態調控等許多基本和關鍵的生物過程中都起到了非常重要的作用[4-5]。同時,很多研究發現LncRNA與包括肺腺癌在內的多種癌癥的發展和生存都有關聯性,強調了LncRNA在肺腺癌發生和發展中的分子機制和生物學特性,表明某些lncRNA可以作為肺腺癌患者預后的分子標志物[6-7]。
個體化醫療的一個關鍵問題就是闡明疾病個體的分子機制。復雜疾病的發生,通常并不是由于單個分子的功能障礙導致,而是由于隨著時間和條件的動態變化,相關系統或者網絡的功能紊亂而引起[8-9]。一個樣本個體在特定狀態下的分子網絡,稱其為單樣本網絡。單樣本網絡是能夠準確描述疾病個體在特定條件下疾病狀態的關鍵個性化特征。對樣本個體在特定條件下的單樣本網絡進行評估和分析,能夠從系統和網絡水平揭示復雜疾病發生發展的分子機制。本文基于TCGA數據庫中肺腺癌的RNA-seq數據和臨床數據,利用單樣本網絡方法構建肺腺癌患者的單樣本網絡模型,分析和識別出6個與肺腺癌相關的lncRNA分子標志物。功能富集分析表明,6個lncRNA可能參與了肺腺癌患者細胞的代謝過程。本文的研究可以為肺腺癌的診斷、臨床治療以及預后提供可能的分子標志物。
1 數據準備
從TCGA (The Cancer Genome Atlas) 數據庫下載了肺腺癌的 RNA-seq 數據和臨床試驗數據,包括535個肺腺癌的組織樣本(tumor samples)以及59個癌旁的組織樣本(normal samples)。
2 方法
2.1 差異表達lncRNA的識別
我們使用R軟件包“edgeR”對肺腺癌表達數據中所有的lncRNA進行基因的差異表達分析。該軟件包為多組實驗提供了精確的統計方法,它的一個特殊功能是經驗貝葉斯方法,該方法即使在生物復制水平最低的情況下也可以估算基因特異性的生物變異。在我們的研究中,FDR<0.05且 |FC|>2為篩選差異表達的lncRNAs的臨界條件。
2.2 單樣本網絡的構建
單樣本網絡是基于單個樣本對于給定的一組參考樣本的擾動分析而構建的,它可以精確地刻畫個體樣本的疾病狀態[10]。單樣本網絡的構建需要兩組數據,一組是參照樣本數據,另一組是測試樣本數據。通常,這兩組數據有著共同的特征屬性。在本研究中,59個肺腺癌的癌旁樣本作為參照樣本組,535個肺腺癌的腫瘤樣本作為測試樣本組。首先,利用皮爾森相關系數(pearson correlation coefficient,PCC)計算每對lncRNA基因表達譜之間的相關性從而得到一個相關性網絡,稱為參照網絡(reference network),記為Nr。然后,再將一個測試樣本s添加到該參考樣本組中,利用上述相關性的計算方法,可以得到一個新的相關性網絡,稱為擾動網絡(perturbed network),記為Np。最后,通過比較參照網絡和擾動網絡之間的差異獲得一個差異網絡,被稱為樣本s的單樣本網絡(single-sample network),記為Nssn(Nssn=|Nr-Np|)(見圖1)。
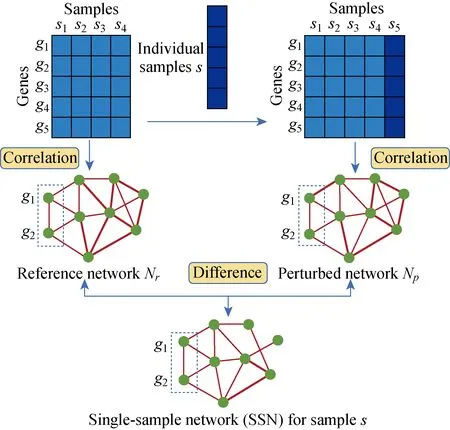
圖1 單樣本網絡構建流程圖Fig.1 Flowchart for constructing a single-sample network
按照上述方法,輪流將535個腫瘤樣本依次作為測試樣本添加到參照樣本組中,可以得到535個單樣本網絡。容易看出,如果測試樣本在基因表達模式上與參照樣本組的表達模式相似,即使將測試樣本添加到參照樣本組中,任何2個分子之間的PCC變化將非常小。相反,如果測試樣本和參照樣本組在表達模式上存在明顯差異,則將測試樣本添加到參照樣本組后將引起明顯的變化。可以看出,單樣本網絡是由樣本s決定的,因此它可以用來作為描述樣本s的一個特定特征。我們對所有的腫瘤樣本構建其對應的單樣本網絡并進行統計分析,篩選出可能與肺腺癌相關的lncRNA分子標志物。
2.3 生存分析
COX比例風險回歸模型,是由英國統計學家D.R.Cox 提出的一種半參數回歸模型。該模型以生存結局和生存時間為因變量,能夠分析各種影響因素對病人生存期的影響。COX回歸分析是醫學領域對于疾病預后分析最常用的統計方法之一。我們從TCGA數據庫下載了肺腺癌病人的臨床試驗數據,同時篩選掉沒有臨床數據的樣本,一共得到512個肺腺癌的樣本。利用單變量Cox回歸分析來評估生存率和每個lncRNA表達水平之間的關聯性。
2.4 功能分析
到目前為止,對與肺腺癌相關的lncRNA的功能機制的報道還很少。為了進一步驗證與肺腺癌相關的lncRNA的分子功能,利用相關性分析計算了并識別出lncRNA的共表達基因,通過對其共表達基因功能的探索,進而研究lncRNA的分子功能。這里,把計算結果中PCC>0.45,且P<0.01的編碼基因,認為是識別出的lncRNA的共表達基因。
Metascape(http://metascape.org/gp/index.html)是一個免費的基因注釋和分析資源,可以幫助研究者了解一個或多個基因的相關功能[11]。我們通過Metascape對共表達基因進行Go和KEGG功能富集分析。這里,P的臨界值設定為P<0.01。
3 結果與討論
3.1 差異表達分析結果
從下載的RNA-Seq數據中,我們提取了 14 823 個lncRNA的表達數據。 其中,腫瘤樣本未表達數量超過10% 的lncRNA數據被剔除,共得到了 4 311 個lncRNA。通過edgeR差異表達分析得到了 1 178 個差異表達的lncRNA,其中包括851個表達下調的lncRNA和327個表達上調的lncRNA(見圖2)。圖2中3個部分分別表示表達下調、無差異表達、表達上調的lncRNA。
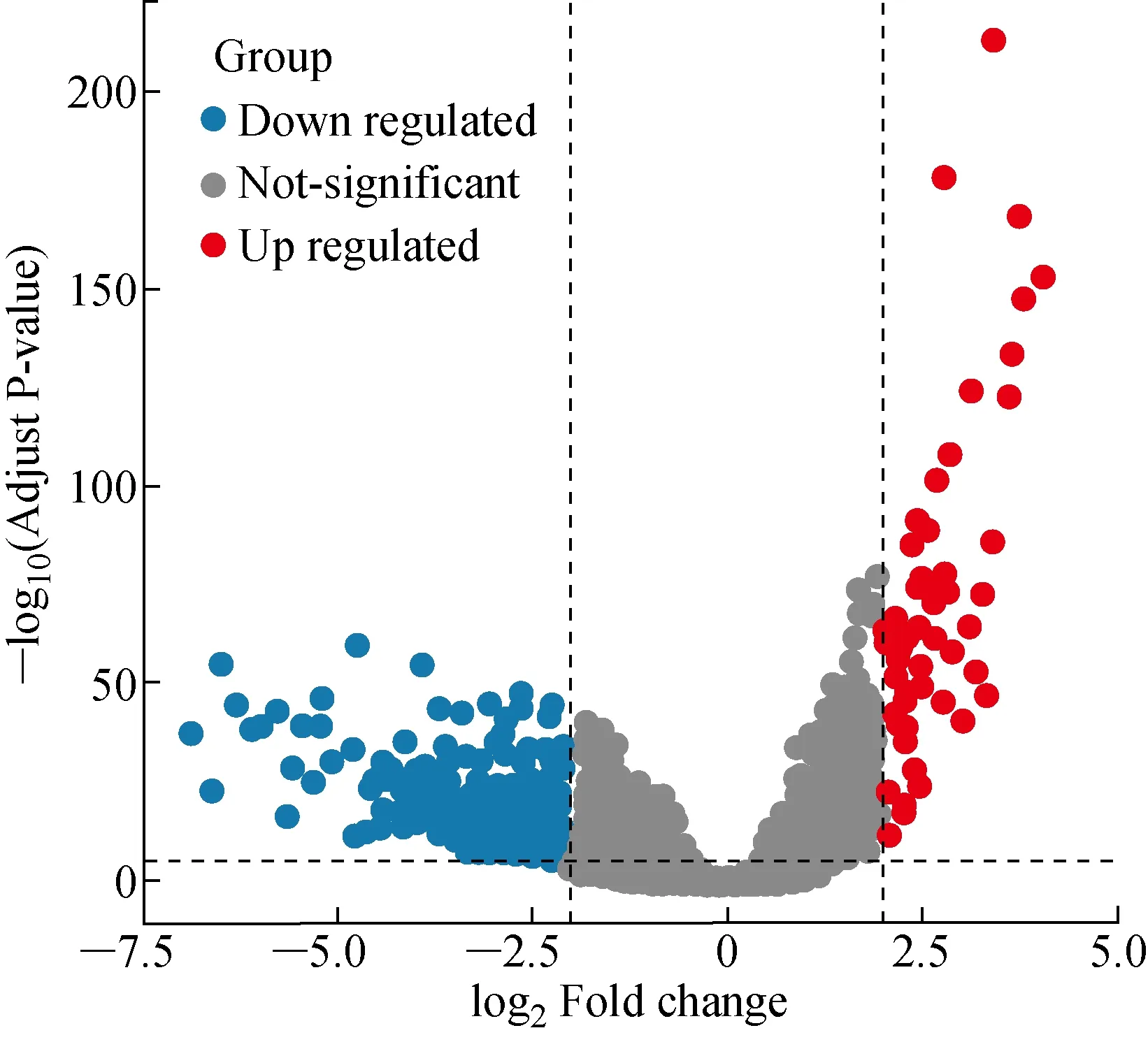
圖2 差異表達lncRNAs的火山圖Fig.2 The volcano plot of the differentially expressed lncRNAs
3.2 單樣本網絡分析結果
為了方便計算,我們把通過2.2得到的單樣本網絡轉化為一個鄰接矩陣 ΔD。容易知道,ΔDi,j表示單樣本網絡中第i個lncRNA和第j個lncRNA在參照網絡和擾動網絡中對應邊的PCC的差值,記為ΔPCC。基于假設:如果某個lncRNA可能是肺腺癌相關的分子標志物,則在差異網絡中,由該lncRNA連接的所有邊對應的ΔPCC的總和將比其他lncRNA對應的總和更高。這里,用向量SD來表示 1 178 個lncRNA在每個單樣本網絡中連接的所有邊所對應的ΔPCC的總和,記作
SD=(SD1,SD2,…,SDi,…,SD1 178)T
(1)
式中,SDi可通過以下方式計算:
(i=1,2,…,1 178,j=1,2,…,1 178)
(2)
為了找到與肺腺癌相關的lncRNA分子標志物,我們對每個單樣本網絡中所有lncRNA的SDi值進行排序。SDi的值越大,其成為肺腺癌的分子標志物的可能性也就越大。對于所有535個單樣本網絡,我們用一個矩陣M1 178×535來表示
M=
i=1,2,…,1 178,j=1,2,…,535
(3)
式中,SD1,j>SD2,j>…>SDi,j>SD1 178,j,i表示第i個lncRNA,j表示第j個腫瘤樣本。
我們取上述矩陣M的前10行(即在每個腫瘤樣本排序中的前10個lncRNA)進行統計分析。在535個腫瘤樣本中,出現頻率最高的30個lncRNA作為我們通過單樣本網絡方法識別出的可能與肺腺癌相關的候選lncRNA分子標志物。
3.3 生存分析結果
從肺腺癌患者的臨床數據中選擇整體生存期(overall survival,OS)進行單因素cox風險分析,結果顯示在30個lncRNA中有6個與患者的OS顯著相關(P<0.05)。6個lncRNA的詳細信息見表1。基于這6個lncRNA建立了一個風險得分評估模型,式為:OS-risk score=0.034×LINC00460+0.057×RP11-284F21.9+0.426×RP11-539E17.5+0.227×CTD-2377D24.6+0.053×RP1-27K12.4+0.007×RP11-783K16.5。式中的6個系數分別為6個lncRNA在生存分析回歸模型中對應的風險系數,基因名代表該lncRNA的表達數據。利用上述公式對512個肺腺癌患者進行風險得分計算,并根據所有患者風險得分的中位數將患者分為高低風險兩組。這512個肺腺癌患者在預后模型中的分布以及生存時間的分布見圖3(a)。ROC曲線被用來評估該風險得分評估模型的預測性能,結果顯示該模型在生存時間為1年、3年和5年時間的AUC值分別為0.68、0.7和0.71,這說明在5年的生存期內,隨著生存時間越來越長,該風險評估模型對于患者生存預后的準確性也越來越好,見圖3(b)。同時也采用KM生存曲線對這些患者的生存情況進行了分析,結果顯示低風險組患者的死亡率顯著低于高風險組(HR=2.72,95%CI=2.13~3.48,P<0.000 1),見圖3(c)。

圖3 基于6個lncRNA的風險得分對肺腺癌患者進行預測的結果(a)風險得分分布情況和肺腺癌患者的生存狀態;(b)整體生存情況預測的風險得分ROC曲線分析;(c)高風險組(n=256)和低風險組(n=256)患者整體生存情況的KM分析Fig.3 The result of predicting the overall survival of patients with LUAD based on the risk score of the 6 lncRNAs (a)The risk score distribution and patients’ survival status,(b)The receiver operating characteristic (ROC) analysis of the risk score for predicting the overall survival,(c)Kaplan-Meier analysis of patients’ overall survival in the high risk (n=256) and low-risk (n=256)
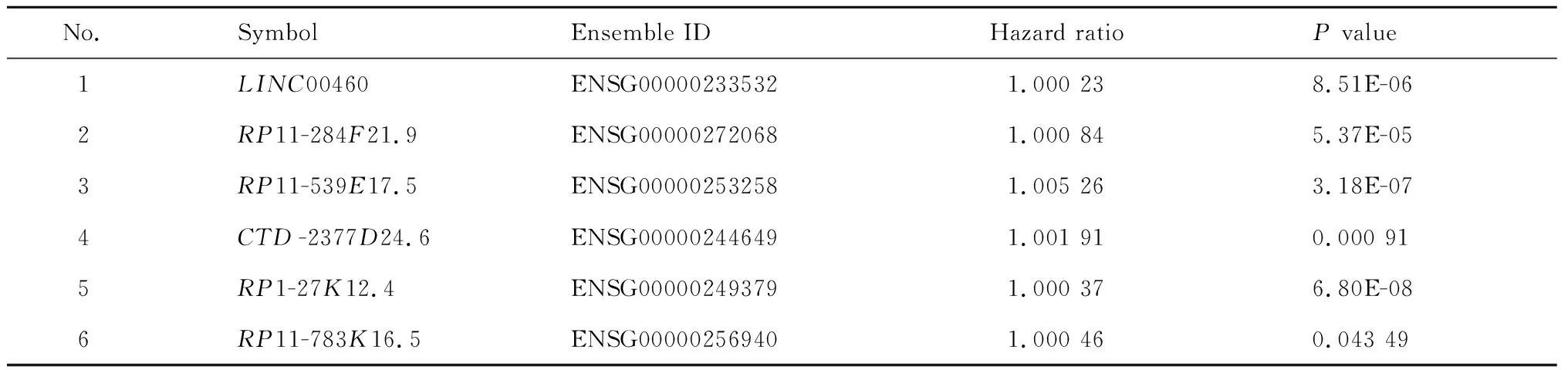
表1 與肺腺癌具有顯著預后關系的6個lncRNATab.1 The 6 lncRNAs significantly associated with prognosis of LUAD patients
3.4 功能富集分析結果
在共表達基因分析中,與6個lncRNA至少1個相關的蛋白質編碼基因就被認為是顯著相關的共表達基因。通過皮爾森相關分析計算,一共得到了52個共表達基因,這些基因主要與LINC00460、RP11-284F21.9、RP11-539E17.5、CTD-2377D24.6、RP11-783K16.5這5個lncRNA相關,見表2。采用Metascape對52個共表達基因進行Go和KEGG功能富集分析,見圖4。圖4中每個條形代表富集到的一簇功能,可以看到共表達基因主要與輔因子代謝過程、類固醇代謝過程以及細胞酮等代謝過程有關,而這些代謝過程是疾病產生和發展過程中的重要影響因素。

表2 5個lncRNA的共表達基因Tab.2 The co-express genes of the 5 LncRNAs

圖4 共表達基因功能富集分析(a)共表達基因功能富集熱圖;(b)共表達基因富集GO的詳細網絡結構Fig.4 Functional enrichment analysis of the co-express genes (a) Heatmap of the functional enrichment analysis of the co-expressed genes,(b)Detailed net structure of enriched GO term of the co-expressed genes
4 結 語
基于TCGA數據庫中肺腺癌的表達數據,利用基因差異分析,單樣本網絡方法以及生存分析識別出與肺腺癌相關的6個lncRNA生物標志物。利用單樣本網絡識別lncRNA 生物標志物,是對樣本個體特定條件下動態分子網絡進行分析研究,對于揭示復雜疾病在網絡水平的LncRNA分子機制有著積極的指導意義,也為從系統水平識別人類復雜疾病相關LncRNA生物標志物提供了一個新視角。

