環境DNA技術在水生態領域應用研究進展
趙彥偉,陳家琪,董麗,麻曉梅,白潔,田凱
(北京師范大學環境學院,水環境模擬國家重點實驗室,北京 100875)
對生物資源與生態系統進行全面系統的調查評估,是生態環境研究的重點,也是保護全球生物多樣性的重要舉措。基于形態學鑒定的傳統生物監測方法對樣本的完整性和鑒定人員的技術要求高,尤其對于隱蔽性強、生活習性難以掌握、形態變化大的物種,鑒定難度較大,這些均為生物監測和物種保護工作的開展帶來了困難。因此,需要一種快速高效、侵入性低且受物種自身影響小的技術來解決當前的生物監測問題。
環境DNA(Environmental DNA,eDNA)作為當前重要的技術手段之一,為環境生態的相關研究提供了新思路與新方法,其推動著物種鑒定工作由形態學識別向分子生物學過渡。任何物種都有自己特有的DNA 序列,皮膚、糞便、唾液、配子、分泌物等都會攜帶著自身的DNA 片段,其脫落到環境中后,即可通過一系列分子生物學操作流程檢測到環境中物種的存在信息[1],這即是eDNA 技術的基本原理。eDNA 技術直接從環境樣品中提取DNA,無需對任何物種進行分離,可將生物監測對環境帶來的影響降到最低[2?3]。涉及eDNA的研究最早開始于20世紀80年代,經過近年的發展,該技術的研究逐步深入,采集范圍逐步擴大,尤其在水生態領域得到了廣泛的應用。
相較于其他的生物鑒別技術,eDNA 是一項新技術領域,因此受到較多學者的關注。馬鴻娟等[4]、陳煉等[5]、張輝等[6]對eDNA 技術進行了較為全面的綜述,對該技術的發展歷程、通用操作流程及其在生物多樣性研究中的應用進行了系統的介紹。本文在這些綜述基礎上,吸收了近年eDNA 技術在水生態領域的最新研究成果,強調了水生態研究中采樣環境、目標物種變化情況下不同技術環節的特點與比較,重點總結了eDNA 技術在水生態領域的應用,分析了該技術應用的局限性,并根據水生態系統特點提出了相應改進措施,對eDNA技術未來發展趨勢進行了展望。
1 eDNA技術的操作方法
eDNA 技術主要包括樣品采集、DNA 提取、PCR擴增及測序、生物信息學分析。在不同的研究中,采樣環境、目標物種等重要因素會有差異,從而使不同的技術環節方法有所變化。
在水環境應用中,為減少外界環境因子對水體中遺留的生物DNA 的影響,樣品采集時不宜對水體造成過大的擾動,通常采集研究區表層水即可[7]。樣品采集過程中要格外注意外來污染或交叉污染,必須嚴格佩戴一次性手套并及時更換,以免給最后的物種識別過程帶來干擾。在采集平行樣本的同時設計空白樣本[8],如將一瓶加蓋的蒸餾水保存在冰上,以檢查現場工作期間的污染。樣品采集后要立刻冷藏防止水體中生物DNA 降解,并在最短的時間內進行初步處理。
樣品采集后,采用濾膜法和沉淀法均可以達到濃縮樣品提取DNA 的目的,且兩種方法各有優勢,沉淀法所需水樣明顯少于濾膜法,但濾膜法可獲得更高濃度的DNA,當前濾膜法中的抽濾方式應用較多。DNA 的提取方法包括物理法、化學法、生物法等,也有研究選擇相應的DNA 試劑盒提取水環境中的DNA。有研究表明,應根據目標物種選取合適的DNA 提取方法,以保證提取到的DNA 濃度最佳,如提取真核生物DNA 時采用抽濾法與DNeasy Blood &Tissue Kit試劑盒相結合的方法檢出率更高[9]。
PCR 擴增首先需要進行引物的設計。引物設計需依不同的研究目標而定,如果是對研究區進行全面的生物多樣性評價,則要選擇通用性引物,以保證此引物擴增到的物種序列最全;如果是對特定物種進行監測評估,則需根據目標物種特點設計特異性引物。之后進行PCR 擴增,擴增后產物經瓊脂糖凝膠電泳檢測,若檢測結果符合要求,即可進行上機測序。
測序得到的原始序列需經過低質量過濾、序列拼接、去除嵌合體等一系列質控過程,得到的高質量有效序列將按照一定的序列相似性進行OTU聚類(OTU相當于傳統生物監測中的“種”),在每個OTU 中選取代表序列(通常為最長序列)與相應的參考數據庫比對進行物種的注釋分析,不同數據庫的鑒定范圍通常不同(表1),需根據目標物種選擇合適的數據庫。以OTU聚類和物種注釋結果為基礎,可得到區域內的物種組成與分布、群落間結構差異等信息,還可進一步挖掘物種之間的進化關系,探究不同環境因子對物種群落結構的影響等。

表1 常見基因數據庫及鑒定范圍Table 1 Common genetic databases and appraisal scope
2 eDNA技術在水生態研究中的應用
2.1 生物多樣性監測
2.1.1 物種的識別與監測
生物多樣性是衡量某一區域物種豐富程度的重要指標,是環境生態學領域的重要基礎性工作,生物多樣性監測的難點在于快速準確地識別物種,eDNA技術因可以解決這一問題,而在浮游動物、藻類、魚類等生物類群的監測中得到了應用(表2)。其中,魚類是 eDNA 監測的重要類群之一,THOMSEN 等[18]發現eDNA 技術可監測到形態學監測方法識別不到的魚類;趙夢迪[19]、SIGSGAARD 等[20]、楊海樂等[21]將 eDNA技術與形態學監測方法對魚類的監測結果進行了比較,證明了eDNA 技術較高的覆蓋度和用于漁業生物多樣性監測的可行性。利用eDNA 技術,宋倫等[22]、LIU 等[23]、岳一鴻等[24]對藻類進行了監測,唐晟凱等[25]、胡愈炘等[26]對浮游生物進行了監測,這些研究證實了利用eDNA 技術對水生態系統中的物種進行識別與監測,全面評估區域物種資源狀況的潛力。
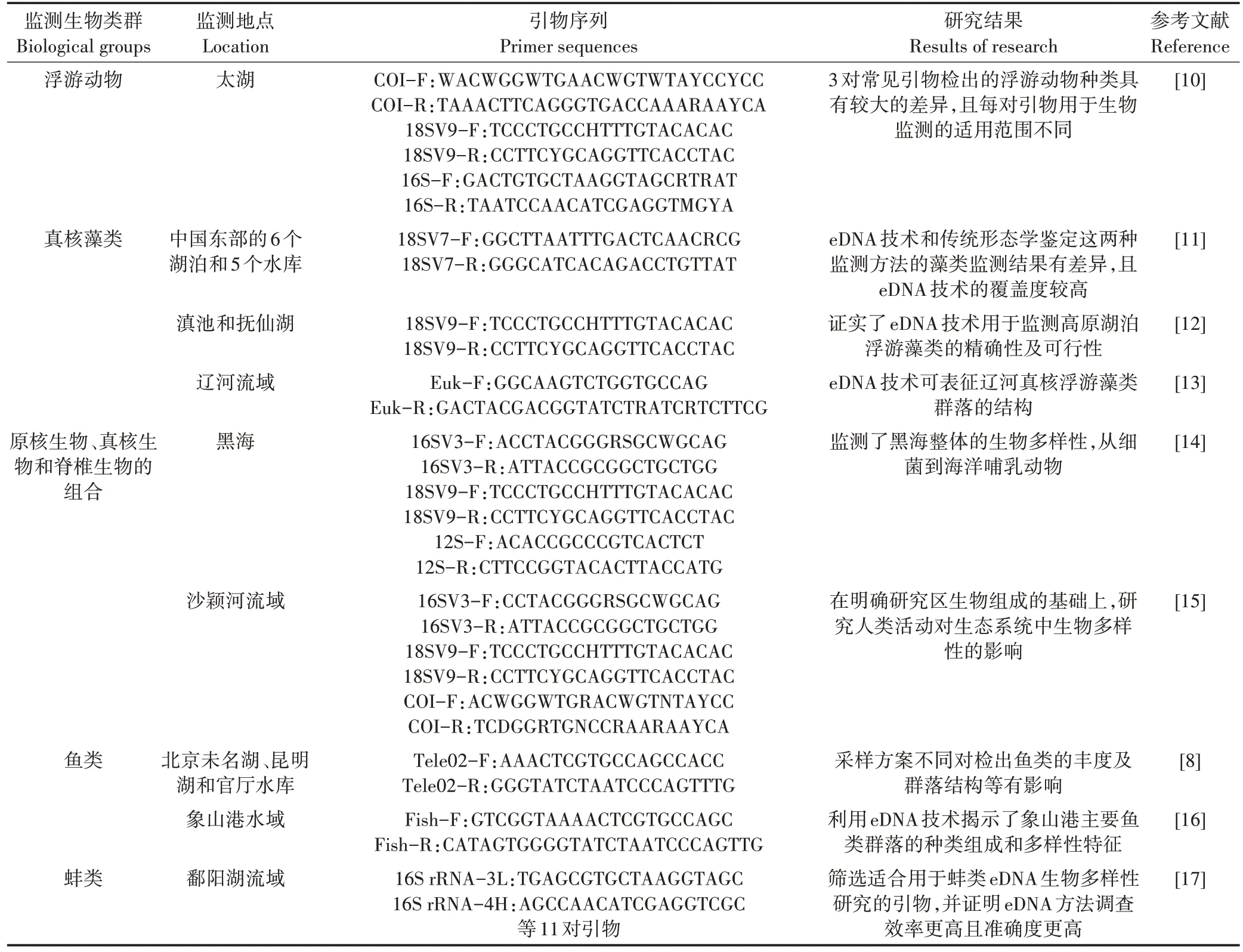
表2 eDNA技術用于水生態監測的應用實例Table 2 Application examples of eDNAinaquatic ecosystem monitoring
2.1.2 瀕危物種和入侵物種監測
瀕危物種保護和外來入侵物種防治是維持生態系統平衡、保護全球生物多樣性的重要舉措。一般情況下,入侵物種早期階段和瀕危物種的物種數量低,利用傳統的形態學監測方法難以及時發現,這增加了珍稀物種滅絕和外來物種大規模繁殖的風險,影響到生態系統結構的穩定性。盡早掌握瀕危物種和入侵物種早期階段的分布范圍、演變趨勢等基本信息是監測工作的關鍵[27?29],已有研究利用eDNA 技術完成了對典型入侵物種克氏原螯蝦的監測[30],還有研究利用eDNA 技術檢出了全球第四只斑鱉。利用eDNA 技術還實現了對美國牛蛙[31]、亞洲鯉魚[32]、斑馬貽貝[33]、藍腮太陽魚[34]等入侵物種,江豚[35]、歐白鮭[36]、野生娃娃魚[37]、澳洲麥氏鱸[38]、長鰭金槍魚[39]等瀕危物種的監測,這些證實了eDNA 技術在入侵物種的及時監測預警、珍稀物種的追蹤研究等方面的應用價值與潛力。
2.2 生物量評估
已有研究表明,某一物種釋放的eDNA 濃度和其生物量呈正相關關系[40],此結論為生物資源的綜合評估提供了一個新的思路。孫晶瑩等[41]選取太湖流域常見的5 種浮游動物為研究對象,利用eDNA 技術對其開展了生物量監測研究,發現在選擇合適引物的基礎上,eDNA 技術可實現對浮游動物的半定量檢測。李苗等[42?43]在建立優化中國對蝦(Fenneropenaeus chinensis)eDNA 操作流程的基礎上,進一步對eDNA在水體中的存留時間進行了研究,這些研究對中國對蝦進行定量的生物量評估至關重要。秦傳新等[44]分別綜述了實驗室可控環境和野外環境條件下利用eDNA 技術進行生物量評估的研究進展,并提出要更多地關注影響eDNA 釋放、遷移、降解的因素,進而為生物量評估提供重要的依據。盡管目前已有眾多針對魚類、兩棲動物等物種生物量估算的研究,但由于不同物種釋放eDNA 的濃度和速度不盡相同,想要實現跨類群的生物量評估還需要eDNA 技術更深入的發展。
2.3 生物食性分析
以往的食性分析多基于形態學鑒定、同位素追蹤等方法[45],對瀕危性、夜行性以及生活習性難以掌握的生物測定難度較大。近年來eDNA 技術被廣泛應用于動物食性的分析,其最大的優勢在于能進行快速精準的測定,有利于精確解析動物食物網的關系和生態系統的功能。周天成等[46]利用基因測序技術分析了一種海洋貝類——塔形馬蹄螺(Tectus pyramis)的食物組成并推測其屬于沉積物碎屑食性生物,測序結果共獲得41 個OTU,分屬11 個門類,其中有孔蟲、真菌、后生動物是其消化道中存在的重要類群。除此之外,eDNA 技術還被用于多種海洋生物的食性研究,如刺參、龍蝦、方星格蟲等[47]。
食性分析是生態學研究的重點,也是了解水生生物與環境關系及捕食者與被捕者關系的基礎,是對已有食物鏈研究的重要補充[48],對研究區內食物網結構的重建和生態調控具有重要意義。但世界上絕大多數生物的食性分析還處于較為粗略的定性描述階段,eDNA 技術將會成為人類更全面明確生物食性、研究食物鏈/網結構的有利工具。
2.4 生態風險評估
生態風險評估是生態毒理學研究中的重要且基礎內容,傳統生態毒理學方法強調對化學物質暴露過程的評估,忽視了對群落結構及生態系統基本功能的影響,評估結果具有一定的應用局限性。ZHANG[49]認為eDNA 技術開拓了生態毒理學研究的新時代,該技術既可解決當前生態風險評估存在的問題,又可擴展未來生態毒理學研究的領域和方法。YANG 等[50]、LI 等[51]利用eDNA 技術監測了水體中的原核生物、真核生物等生物群落結構,評估了其與pH、總氮、總磷等環境因子的關系;XIE 等[52]利用eDNA 技術評估了漏油對沿海沉積物中細菌、原生動物、后生動物群落的影響;YANG 等[53]探究了銅的不同濃度對濕地沉積物中原核生物和真核生物多樣性、群落結構以及生物間關系的影響。這些研究推動著eDNA 技術在生態系統監測、污染診斷和生態修復等方面更深入的應用。
2.5 生物流行病的預測與預防
疾病生態學是用生態學觀點研究環境、宿主和病原體之間關系及其發展規律的新學科,其能明確疾病進化和生態之間的相互作用,預測疾病爆發的方式和地點,指導相關部門的管理決策,有效預防生物流行病的傳播。當前,eDNA 技術已用于研究水體中的寄生蟲疾病[54]。CARRARO 等[55?56]、GOMES 等[57]通過研究證實了eDNA 技術用于預測預防水體寄生蟲爆發的潛力。其中CARRARO 等[55]以瑞士Wigger 分水嶺為研究區,以對本地鮭魚物種有極大影響的增生性疾病(Proliferative kidney disease,PKD)為研究對象,繪制了其病原體F.sultane的DNA 濃度分布圖,并建立了研究水傳播疾病的流行病學和疾病動力學模型;同時利用eDNA 技術對整個河流上游網絡中引發鮭魚疾病的固著無脊椎動物F.sultane及其寄生蟲T.bryo?salmonae的分布和豐度進行了估計,通過這兩種生物的生物量變化來預測PKD在研究區內的傳染狀況[56]。這項研究開啟了利用eDNA 技術估計目標物種在研究區內分布情況及有效預測預防流行病傳播的新模式,而傳統的調查方法迄今還無法做到這一點。
3 eDNA技術應用的局限性與改進
和傳統生物監測方法相比,eDNA 技術具有非入侵性、高靈敏度、不受環境條件限制等優勢,但也存在一系列有待完善的問題,在實際應用過程中存在局限,還不能完全取代傳統的調查方法,這些問題制約著eDNA技術的應用。
3.1 缺乏標準化的操作流程
選擇效果最佳、最經濟便捷的操作流程與方案一直是科研人員關注的焦點,隨著eDNA 技術的不斷發展及研究團隊的壯大,eDNA 技術實驗方案也在不斷增加。已有研究表明,不同的DNA 捕獲與提取方案[9,58]會對最終的監測結果產生影響。但因受采樣環境、采樣范圍、監測目標物種等多重因素的限制,當前缺乏一套通用的eDNA 技術標準化操作流程。研究者在研究過程中采取的操作方法不盡相同,導致研究結果的對比性差,存在一些爭議[7,28]。今后應加強eDNA 技術在水生態研究中應用的普適性研究,使eDNA 技術能更廣泛地應用于水生生態監測中。需要綜合考慮不同類型水生生物的生活特征,對eDNA 技術的整個操作流程進行優化,針對不同目標物種與研究目的,明確與細化采樣、提取、PCR擴增、分析等的操作指南與技術規范,從而建立一個規范統一的操作方案與流程,以推動eDNA技術更廣泛的應用與發展。
3.2 監測精度及準確性有待提高
eDNA 在水體中釋放的濃度及其分布受到水動力條件[59]、水體溫度及光照條件[60]、季節變化、生物活動等多種因素的影響,還與人類活動干擾強度密切相關,且eDNA 會隨著時間和各環境因子的改變而發生降解。當前研究大多未考慮外界環境條件變化對DNA 分布的影響[5],使得監測結果與實際情況存在一定的誤差。近年來國內外學者針對這一問題進行了有效探索,主要集中在季節變化對特定生物eDNA 的影響[35],水體溫度及營養條件與eDNA 降解之間的關系[61]等方面,但這些研究具有個案特征,并不能作為普遍的規律指導eDNA 的廣泛應用,eDNA 時空尺度研究的精確度還有待提升。為此,需要研究者們在模擬并建立外界干擾和物種分布之間對應關系的基礎上[7],積極探究影響eDNA 產生和降解速率的環境因素,進而更好地為生物量評估和生物流行病的預測預防等相關工作的開展提供理論基礎。
3.3 假陽性和假陰性錯誤無法避免
假陽性和假陰性錯誤是基于研究區目標物種的DNA 是否被檢出以及目標物種是否真實存在而產生的,主要受技術敏感性、引物偏倚性等因素的影響[35]。在實際操作中,假陽性和假陰性錯誤的出現可能會帶來較大的損失,尤其是在瀕危物種和入侵物種的監測中,物種的誤判對生態系統的影響是巨大甚至是毀滅性的。為了盡可能減少假陽性和假陰性錯誤的出現,需提高引物的通用性,嚴格控制實驗操作過程的準確性,設置陽性和陰性對照以保證實驗結果的可靠性,對實驗中的外來污染和交叉污染進行實時監控,還可以結合其他鑒定方法進行對比分析,校核實驗結果,從而提高eDNA技術用于生物監測的有效性和可靠性。
3.4 物種參考數據庫需要完善
基因測序序列和物種參考數據庫的比對是保障eDNA 技術應用準確性的關鍵環節。主要在于,物種參考數據庫的完整性和質量直接影響著特異性DNA條形碼的可用性,進而影響生物多樣性的監測結果,更決定著運用eDNA 技術進行物種鑒定的可靠性[62?63]。數據庫的不完整可能會導致大量的基因序列無法被注釋,造成海量遺傳信息的遺失。近年來,NCBI、SILVA、Greengenes、PR2 等一些常用的公共數據庫的數據在不斷更新和完善,也有研究者在積極建立研究區的本土生物基因庫,如:太湖浮游生物基因庫[64?65]、中國的底棲動物條形碼數據庫[66]等,但數據庫的容量依舊不足。為保證結果有效性和數據可用性,同時提高基因序列注釋到種水平的概率,可從兩個方面改進:一是建立本土基因庫,加強全球資源和成果共享;二是完善和優選現有數據庫,建立一個生物類群更多、基因序列更全的公共數據庫。
3.5 eDNA豐度和生物體數量的關系需要進一步明確
eDNA 技術只能從序列數量推斷OTU 的相對豐度,雖然可利用實時熒光定量PCR 等方法對特定DNA 進行絕對定量,但對于還原形態學方法中的物種絕對豐度或生物量的預期,仍然相差甚遠。目前多采用多引物法或僅對單一類群的特異基因測序結果進行分析,以彌補eDNA 監測技術還原度低的問題。近年來,物種的基因拷貝數、基因組大小、生物體積大小等因素逐漸被納入到矯正因子的參數選擇中[67],也有研究者在探索不依賴于形態學分類注釋的描述群落結構和多樣性的eDNA 監測和數據分析方法[68?69],這進一步豐富了eDNA 與生物體對應關系的研究體系。
4 總結與展望
在水生態監測研究中,傳統的形態學生物監測方法受耗時長、難度大、成本高等因素的限制,無法實現對水生生物的精準監測和識別。eDNA 技術目前雖然也存在著流程不規范統一、數據庫不完善等問題,但其具有的省時高效、靈敏度高、環境友好等優勢,正推動著該技術成為生物多樣性監測和評估的一個強有力工具。因此,eDNA 技術和傳統監測方法的互相補充才能為全球大規模、高效率的生物監測提供技術支撐。
eDNA 技術是分子生態學發展的重要產物,它將極大地提升人類從生態系統中獲取數據的能力,未來關于eDNA 的進一步發展將涉及技術和應用兩個層面。技術方面,在解決技術流程、監測精度、水生生物種數據庫等問題的同時,應加強新興測序技術及平臺的研發,將基因測序結果和監督式機器學習進行有效結合,實現水生態監測的智能化,不斷提高監測效率,使更大批量的測序成為可能,還應將eDNA 技術引入水生生物的食物網/鏈研究中,豐富eDNA研究的技術體系。應用方面,一是在穩步推進eDNA 技術應用于水域生物多樣性與水生態監測等常規研究的同時,還應拓展eDNA 技術在水生態系統的研究領域,加強該技術在水域食物鏈/網重構、流域營養鹽控制、流域生態調控與管理中的應用;二是要積極開展試點性研究,嘗試將eDNA 技術引入國家水環境與水生態監測體系,編制并完善與eDNA 技術應用及實施相關技術的指南、導則與方法,進而實現eDNA 技術在水生態環境監測領域更廣泛的應用。

