基于高通量測序的中國水仙根際土壤微生物群落組成
李媛媛 張瓊 陳小煌 張敬虎 陸鑾眉

摘要:【目的】明確中同水仙植株根際土壤微生物群落的組成特征。【方法】采用Illumina Miseq高通量測序技術對水仙根際土壤微生物樣品的保守基因區進行測序及生物信息學分析,闡明中同水仙根際土壤的細菌、真菌和^菌群落結構組成,并對水仙根際土壤微生物的優勢菌屬進行深入分析。【結果】共獲得優化序列175840條,基于97%序列相似度,聚類為2 680個OTUs。優勢細菌類群是綠彎菌門Chloroflexi( 30.86%)和變形菌門Proteobacteria(20.67%),真菌以子囊菌門Ascomycota(84.94%)為主,屬水平上球毛殼菌Chaetomium globosum( 28.15%)和散子囊菌Eurotiales( 25.01%)占較高比例。占菌類群主要為奇^菌門Thaumarchaeota( 51.40%)、深^菌門Bathyarchaeota(25.98%)和廣古菌門Euryarchaeota( 20.65%)。其中,來自奇古菌門的SCG類群(25.67%)和嗜酸性氨氧化古菌Candidatus Nitrosotalea( 12.93%)占較高比例。【結論】中國水仙根際土壤微域具有豐富多樣的微生物類群,這對于開發和利用水仙根際土壤微生物資源具有重要意義。
關鍵詞:中國水仙;根際土壤微生物;高通量測序;細菌;真菌;^菌
中圖分類號:S682
文獻標志碼:A
文章編號:1008-03 84(2021)08-0948-08
High-throughput Sequencing on Microbial Community in
Rhizosphere Soil of Chinese Narcissus
LI Yuanyuan 1, ZHANG Qiong 1, CHEN Xiaohuang 2, ZHANG Jinghu 1, LU Luanmei 1*
(I.Provincial Key Laboratory of Landscape Plants with Fujian and Taiwan Characteristics/College of the Bioscience and
Technology, Minnan Normal University, Zhangzhou, Fujian 363000. China; 2. College of the Chemistry, Chemical
Engineering and Environment. Minnan Nonral Universily. Zhangzhou, Fujian 363000. China )
Abstract: 【Objective】The microbial community in thizosphere soils ofChinese narcissuses was analyzed.【Method】TheIllumina Miseq high-throughput sequencing technology was used to obtain the relevant conserved gene regions of the microbesin soil specimens from Chinese narcissus planting lots. The community structures and distributions of dominant species ofbacteria, fungi, and archaea in the thizosphere soils were analyzed. 【Resultl】In total. 175 840 optimized sequences wereobtained and clustered from the specimens int0 2 680 representative OTUs with a 97% similarity. The dominant bacteria wereChloroflexi (30.86%) and Proteobacteria (20.67%). Among the fungi, Ascomycota (84.94%) significantly overshadowed theothers. with Chaetomiumm、 globosum (28.15%) and Asconycetes (25.01%) accounted for the greater proportions. On archaea,Thaumarchaeota (51 .40%), Bathyarchaeota (25.98%). and Euryarchaeota (20.65%) were the maj or phyla that had 25.67% SCGand 12.93% acidophilic ammonia oxidizing members. 【Conclusion】The thizosphere soils of Chinese narcissuses harboreddiverse and rich microbial species. The information obtained would aid the development and utilization of the natural resources.
Key words: Chinese narcissuses; thizosphere soil microorganisms; high-throughput sequencing; bacteria; fungi; archaea
0 引言
【研究意義】中國水仙(Narcissus tazetta L.var.chinensis Roem.)是石蒜科多年生草本植物,原產我國,已有一千多年栽培歷史,獨具天然麗質,芬芳清新,素潔幽雅,是我國十大觀賞名花之一,觀賞價值極高,在園林造景中也有廣泛應用。近年來,很多研究揭示了植物根際一土壤一微生物之間的相互作用機制,探明中國水仙根際土壤微生物群落分布特征對于解析中國水仙的生態生存機制具有重要意義[1-3]。但目前對中國水仙根際土壤微生物群落組成的認知仍然有限,尤其對水仙根際土壤真菌和古菌的群落水平鮮有報道,從而不能深入解析水仙根際土壤微生物與水仙生長發育的關系。【前人研究進展】水仙根際土壤微生物組成與栽培方式、地理位置及水仙的生長習性等密切相關[4]。體外噴施有效微生物群制劑,可顯著促進水仙花的生長發育[5]。已有研究證實,大量微生物類群具有促進植物生長的機制。Ryu等[6]從擬南芥根際土壤鑒定出2種菌株,它們釋放出的一些揮發性物質極大促進了擬南芥的生長。此外,從芽孢桿菌屬、假單胞菌屬和黃桿菌屬中,分離到多種可增強植物抗病性的促生菌菌株[7-9]。根際土壤微生物也包括可以抑制植物生長甚至導致植株死亡的根際“有害菌,”如根腐病病原菌[1O]、青枯病病原菌[11]等,它們普遍分布在根際土壤環境中,略微積累,就有可能影響根際土壤微生物群落的穩定,產生有害物質,抑制植物根系對土壤養分的吸收,嚴重可導致植物死亡。根際環境成為土壤一根系一微生物互作的關鍵區域,被稱作植物的第二基因組[12],根際土壤微生物影響著植物的生長發育,近年來一直是科學家們研究的熱點。【本研究切入點】福建省漳州市具有栽培水仙花的得天獨厚的地理環境,可提供研究水仙根際土壤微生物群落結構、功能及其與植物互作的理想土壤材料。然而水仙根際土壤菌群結構的組成特征及根際細菌、真菌和古菌的關鍵微生物類群組成亟待深入探討。【擬解決的關鍵問題】采集在漳州培育的漳州水仙根際土壤,利用高通量測序技術,對根際土壤中細菌、真菌和古菌群落的組成進行系統鑒定,分析水仙根際土壤環境中優勢的微生物類群,并對其生物功能進行探討,以期揭示中國水仙根際土壤微生物的群落分布特征,為解析水仙花的環境適應性機制以及建立科學合理的水仙花種植制度提供理論依據。
1材料與方法
1.1根際土壤微生物樣品采集
土壤樣品于2020年4月25日在福建省漳州市“水仙花海”園區進行漳州水仙金盞銀臺根際土壤的采集。用5點取樣法采集水仙根際土壤,去除土壤表層雜草和凋落物,在水仙植株的根部一側小心挖掘,待改側植株根系露出后,用毛刷刷取黏附在根表面的土壤。共采集5處樣地的水仙根際土壤,每處樣地選擇3株植株,每處土樣總采樣量約為50 g,各樣點土壤混勻后,封入滅菌的50 mL離心管,置于冰盒帶回實驗室。約20 g新鮮土樣用于根際土壤核酸樣品提取,剩余部分置于通風櫥中,靜置24h,磨細后過0.25 mm篩,用鋁箔紙收集,用于土壤理化性質測定。
1.2土壤理化性質測定
用梅特勒一托利多pH計測定土壤pH;土壤有機質含量采用重鉻酸鉀( K2Cr207)法測定[13];土壤的全氮含量采用凱氏定氮法測定[14];采用堿解擴散法[15]、NaHCO,浸提法[16]和醋酸銨浸提一火焰光度法[17]測定土壤堿解氮、有效磷和速效鉀含量。采用乙酸銨離心法測定土壤陽離子交換量( Cation ExchangeCapacity,CEC)[18];用梅特勒一托利多電導率儀測定土壤電導率;采用石墨爐原子吸收光譜法測定土壤中鉛( Pb)含量[19]。
1.3 DNA抽提和PCR擴增
根際土壤微生物DNA樣品依據PowerSoil?DNAIsolation kit( MoBio.U.S.)說明書的操作進行,DNA的質量、濃度和純度通過1%的瓊脂糖凝膠電泳和NanoDrop2000鑒定;使用338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')[20]對16S rRNA基因V3-V4可變區進行PCR擴增;使用524FlOextF(5'-TGYCAGCCGCCGCGGTAA-3')和Arch958RmodR(5'-YCCGGCGTTGAVTCCAATT-3,)[21]對古菌16S rRNA基因的特異區間進行PCR擴增;使用SSU0817F(5'-TTAGCATGGAATAATRRAATAGGA-3 ')和1196R(5'-TCTGGACCTGGTGAGTTTCC-3,)[22]對真菌18S rRNA基因的特異區間進行PCR擴增。擴增程序如下:95℃3 nun,35個循環(95℃30 s,58℃30 s,72℃30s),72℃15 min,最后4℃保存(PCR儀:ABI Verity? 96型)。PCR反應體系為:5×TransStart FastPfu 4 uL,2.5 mmol·L-l dNTPs 2 uL,上游引物(5umol·L-l) 0.8uL,下游引物(5 umol·L-I) 0.8 uL,TransStart FastPfuDNA聚合酶0.4 uL,模板DNA 10 ng,使用滅菌雙蒸水將體系補足至20 uL。每個樣本設置3個重復。
1.4 Illumina Miseq測序
使用2%瓊脂糖凝膠分離PCR產物,然后對相應膠條進行切割回收,利用MiniBEST Agarose GelDNA Extraction Kit( TaKaRa,Japan)進行回收產物純化,并用QuantusrM Fluorometer( Promega,USA)對回收產物進行檢測定量。建庫依據NEXTFLEXRapid DNA-Seq Kit進行,具體步驟如下:①在DNA片段兩端連上特定序列的接頭;②利用樣品本純化磁珠進行片段篩選;③通過PCR擴增實現原始模板的富集;④將PCR擴增后的文庫進行分選回收。采用Illumina Miseq測序技術(上海美吉生物醫藥科技有限公司)對文庫進行高通量測序分析。原始數據上傳至NCBI SRA數據庫(序列號:SAMA16825444,Genbank登錄號:PRJNA679159)。
1.5數據處理
利用Trlmmomatic軟件對接頭(Adapter)序列和低質量序列進行過濾[23],使用FLASH軟件實現測序數據的質控拼接[24],具體步驟如下:①去除尾部質量值的讀長( Reads)在20以下的堿基,設置50 bp的窗口,如果窗口內的平均質量值低于20,從窗口開始截去后端堿基,去除質控后50 bp以下的讀長,去除含N堿基的讀長;②根據讀長之間的重疊,將成對讀長重疊區域連接起來,重疊區長度不低于10 bp;③拼接序列的重疊區堿基錯配率不高于0.2;④根據序列首尾兩端的barcode和引物區分樣品,并調整序列方向,barcode允許的錯配數為0,最大引物錯配數為2。使用的UPARSE軟件[25],將序列相似性> 97%的定為一個OTU。用RDP classifierc2627]數據庫,選取OTU代表序列跟數據庫中的已知序列比對,獲得每個OTU的物種注釋信息,用Mothur[28]計算不同隨機抽樣下的Alpha多樣性指數,用R語言工具制作曲線圖。以OTU豐度為基礎,用ACE和Chao指數反映群落物種豐富度,用香農指數和辛普森指數反映群落多樣性,同時估測物種均勻度。
2結果與分析
2.1采樣地土壤理化特點
漳州位于福建省最南部,氣候溫暖,年平均氣溫21℃;雨量充沛,水源充足,土壤松軟,是水仙花的理想產地。對漳州圓山區“水仙花海”采樣地的理化特性進行分析,采樣地單位體積土壤有機質含量為29 g·kg-l,占比3%;土壤全氮含量為1.3 g·kg-l,占比0.13%,達到較高水平,其中速效氮含量豐富,在土壤中的含量為91 mg·kg-l。說明該生產區土壤有機質含量豐富,熟化程度高,水仙生長及采樣期間土壤的氮素供應能力較強[29]。此外,采樣區土壤質地屬黏質土,CEC含量較高,為13.4 cmol(+)·kg-l。土壤鉛含量在85 mg·kg-l,符合農業種植土壤標準。
2.2中國水仙根際土壤微生物多樣性
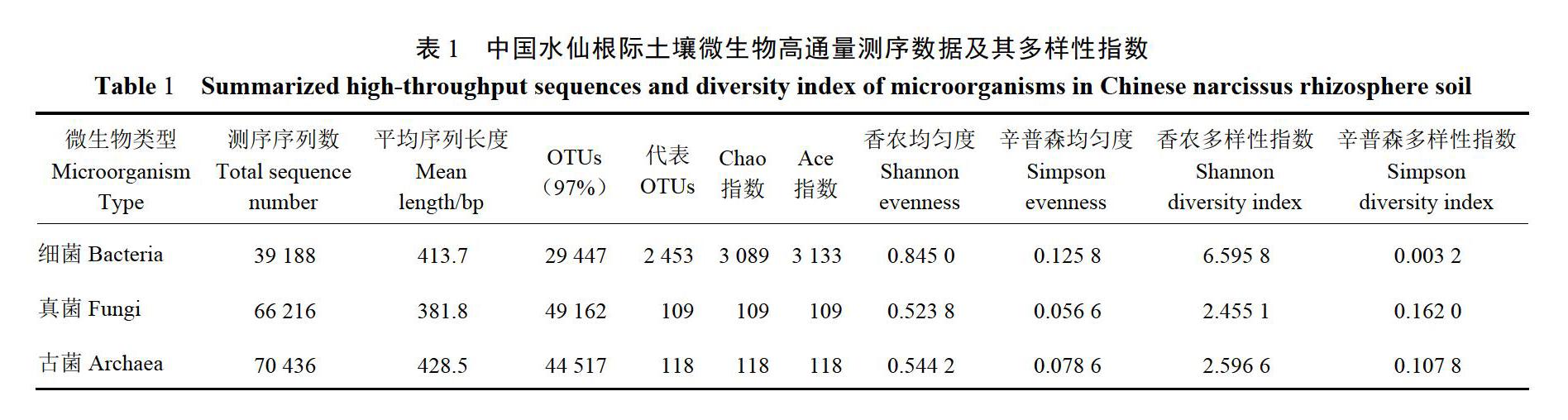
由表1可知,測序得到的細菌、真菌和古菌的有效序列分別為3.91萬、6.62萬和7.04萬條。按照序列相似性≥97%歸類為1個OTU分類單位的原則,分別得到細菌、真菌和古菌OTU分類單位2 453、109和118個。本次各樣品測得的序列均遠遠大于10 000,說明測序數據量足夠大,可以反映樣本中微生物多樣性的主要信息。從每個土壤樣品的OTUs中計算獲得微生物群落的a-多樣性指數,包括Chao、ACE、辛普森指數和香農指數,以此來反映微生物群落的豐富度和多樣性。結果表明,根際土壤環境中,細菌的多樣性明顯高于真菌和古菌,且細菌門下各類物種的數量接近程度也更高。真菌群落和古菌群落中鑒定到的物種數目、物種數量的相對密度均較為接近,同時明顯低于細菌群落。
2.3中國水仙根際土壤細菌群落的組成
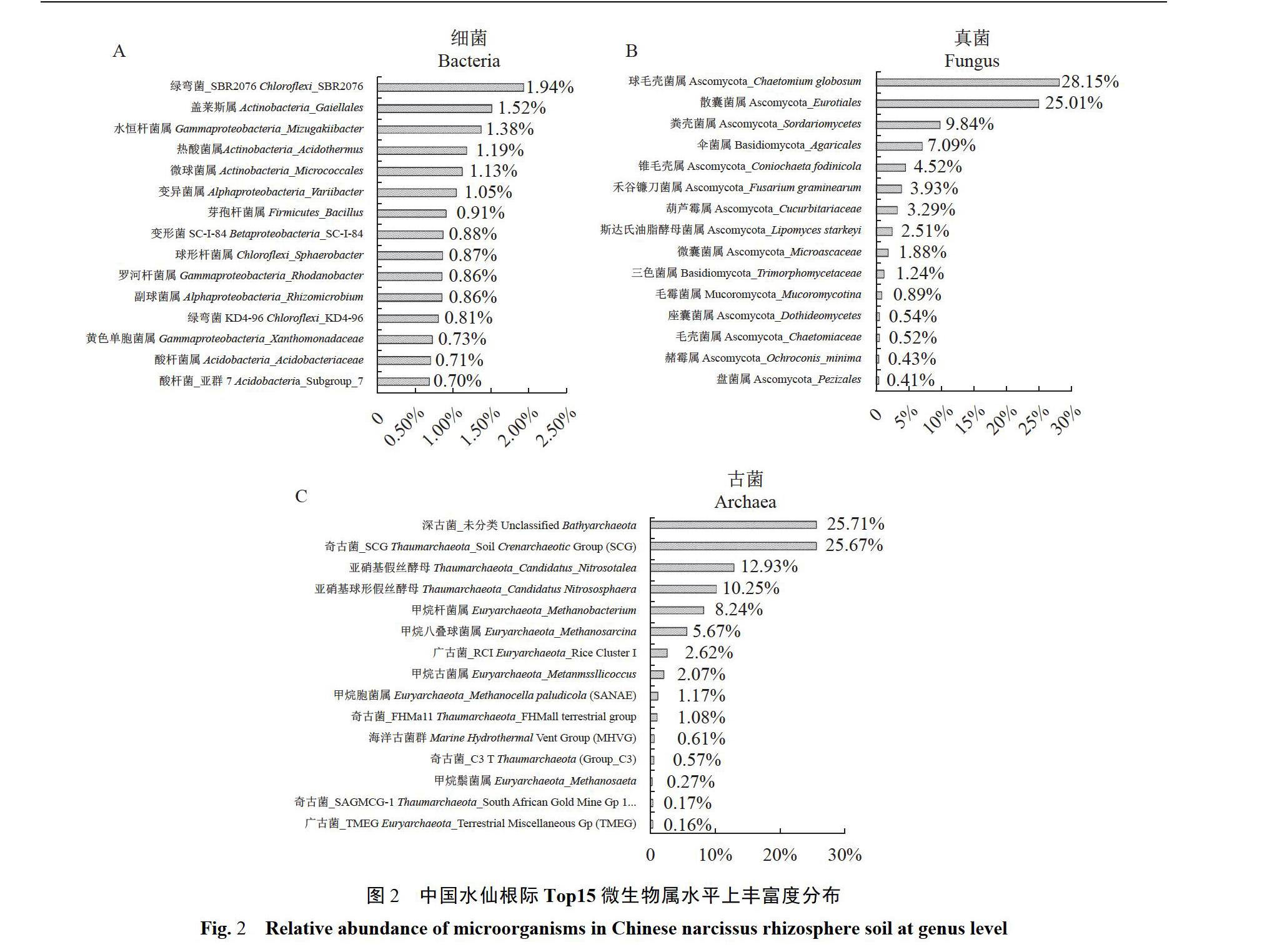
利用高通量測序的方法,根據OTU分類學注釋結果,按照門和屬2個水平提取序列,計算各類物種的相對豐富度。從圖1-A可知,在水仙根際土壤細菌群落中系統鑒定到超過40種不同門類的細菌,其中,13個門的細菌豐度總和高于總序列的97%。綠彎菌Chloroflexi( 30.86%)、變形菌Proteobacteria(20.67%)、放線菌Actinobacteria( 18.25%)和酸桿菌Acidobacteria( 13.6g%)是根際土壤細菌群落中的主要門類,總豐度高于總序列的80%。通過屬分類層次(圖2-A)分析,表明在細菌中占優勢的菌屬分別為SBR2076、蓋勒氏菌屬Gaiellales、水恒桿菌屬Mizugakiibacter、嗜酸柄熱菌屬Acidothermus、微球菌屬Micrococcales和Variibacter,這6個屬類各自的細菌豐度均高于總序列的1%,其他占比較少。屬水平上的統計分析表明,水仙根際土壤細菌群落中53.77%以上的細菌屬于未知。
2.4中國水仙根際土壤真菌群落的組成
在中國水仙根際土壤中檢測到6種不同門類的真菌,真菌門類群大比例集中于子囊菌門Ascomycota,占真菌總序列的84.94%,其他真菌門類所占比例相對較少,分別為擔子菌門Basidiomycota( 9.20%)、毛霉門3.38%、壺菌門Chytridiomycota( 0.3g%)、芽枝霉門Blastocladiomycota( 0.05%)和捕蟲霉門Zoopagomycota( 0.04%)(圖1-B)。通過屬分類層次(圖2-B)分析,豐度高于總序列1%的優勢真菌屬分別為球毛殼菌Chaetomium globosum( 28.15%)、散子囊菌Eurotiales( 25.01%)、糞殼菌Sordariomycetes(9.84%)、傘菌Agaricales( 7.Og%)、錐毛殼屬Coniochaeta fodinicola( 4.52%)、 禾谷鐮孢菌Fusarium graminearum( 3.93%)、葫蘆霉Cucurbitariaceae( 3.2g%)、油脂酵母菌Lipomycesstarkeyi、小囊菌Microascaceae( 1.88%)和三色霉菌Trimorphomycetaceae(1.24%),其他所占比例較少。
2.5中國水仙根際土壤古菌群落的組成
對水仙根際土壤中古菌群落多樣性的分析表明,古菌門類群主要包括奇古菌門Thaumarchaeota(51.40%)、深古菌門Bathyarchaeota( 25.98%)和廣古菌門Euryarchaeota( 20.65%),3個門的古菌豐度總和高于總序列的98%以上。其他門類,包括洛基古菌門Lokiarchaeota、 謎古菌門Aenigmarchaeota、Parvarchaeota等,所占比例相對較少(圖1-C)。通過屬分類層次(圖2-C)分析,表明在古菌中占優勢的菌屬主要為來自奇古菌門的SCG( Soil CrenarchaeoticGroup)類群和嗜酸性氨氧化古菌CandidatusNitrosotalea類群,在古菌總序列的占比分別為25.67%和12.93%,以及來自廣古菌門的甲烷桿菌屬Methanobacterium( 8.24%)、 甲烷八疊球菌屬Methanosarcina( 5.67%)、 Rice Cluster I(2.62%)、第七產甲烷古菌屬Methanomassiliicoccus( 1.17%)等。值得注意的是,屬水平上的統計分析發現,古菌群落中25.71%以上的序列來自深古菌門里的未知屬種。
3討論
植物根系是多細胞生物與土壤接觸的界面。植物根際土壤微生物組成及其分布,與植物的生產和健康密切相關。漳州地理位置特殊,北有高山阻隔寒流入侵,南有海洋調節,自然條件優越,氣候溫和,雨季集中[30],花田土壤松軟,是我國有名的水仙花主產區,有“天下水仙數漳州”的美譽。筆者以漳州市圓山麓腳下的水仙花種植園作為研究區域,以水仙花根際土壤微生物為研究對象,采用IlluminaMiseq高通量測序技術,實現了對以漳州水仙為代表的中國水仙根際土壤環境中不同微生物類群序列的初步研究。
從細菌群落結構及功能來看,研究發現,中國水仙根際最為優勢的細菌為綠彎菌、變形菌、放線菌和酸桿菌,其中,綠彎菌是中國水仙根際細菌群落中的優勢菌,占根際土壤細菌的比例約為30%。綠彎菌廣泛分布在土壤、海洋等各種環境中,是營養元素循環等生態學過程的重要參與者[31]。Fan等[32]研究表明,土壤綠彎菌與作物產量指示基因呈顯著正相關,提出綠彎菌是具有促生作用的土壤關鍵微生物菌群之一。變形菌也是水仙根際土壤占比較高的細菌群落之一,這與之前植物根際土壤細菌結構組成的研究結果基本一致,例如研究表明變形菌是大豆[33]、玉米[34]、柑橘[35]等常見農作物,以及梭魚草、美人蕉、紅花等觀賞植物根際土壤富集的主要菌群'3637]。在細菌屬水平的組成上看,可以發現根際土壤環境中占比較高的Mizugakiibacter、Acidothermus、Variibacter等大多數為促生菌,在已有研究中,作物枯萎病[38-39]、青枯病[40]等土傳病害發病較輕的土壤中這些類群的占比比較高,可作為水仙根際土壤促生細菌潛在功能菌株。
根際土壤真菌作為根際生態系統中的重要組成,是分解者中的先鋒物種,在維持根際生態系統穩定、代謝調節和抗病蟲害等方面有重要調節作用[41]。本研究發現,在中國水仙根際土壤真菌種群結構上,80%以上的真菌來自于子囊菌門,其次是擔子菌。在世界范圍內,土壤真菌群落中幾乎均以子囊菌為優勢型[42]。在屬水平上,本研究中的優勢真菌是來自盤菌亞門的球毛殼菌屬Chaetomium globosum和散子囊菌屬Eurotiales,與世界范圍內的土壤真菌調查結果一致[42]。根據Pietro等[43]、Park等[44]研究表明球毛殼菌是一種廣譜拮抗性真菌,從該菌發酵液中提取出的代謝活性物質對稻瘟病、小麥葉銹病、番茄晚疫病等多種病原菌有拮抗作用,在生物防治中具有巨大的開發應用潛力。
古菌是地球進化最早期的生命體之一,能適應嚴寒、酷暑等極端環境,同時驅動著土壤碳、氮、硫等營養元素的生物地球化學循環[45]。本研究中,古菌在土壤微生物群落及生物量亦占有不可忽視的比例。從古菌群落組成來看,古菌序列主要來自奇古菌門,其次來自廣古菌門,二者參與了土壤碳、氮和氫的生物地球化學循環。已知奇古菌具有氨氧化、反硝化等多樣的生理代謝特性和功能,易繁衍在有機質含量低、含水量較低,即氨態氮含量低的土壤[46]。本研究中,奇古菌SCG類群豐度最高,嗜酸性氨氧化古菌Candidatus Nitrosotalea豐度緊隨其后,與Shao等[47]對西北草原古菌群落所做的研究一致。本研究發現,廣古菌在水仙根際土壤古菌群落中亦占有相當大的比例,已有研究表明,廣古菌的相對豐度隨氨態氮的增加會顯著增加,而且還與土壤中的可利用硫正相關[48]。研究還發現,來自廣古菌門下的產甲烷古菌Methanobacteriu和甲烷八疊球古菌Methanosarcina是中國水仙根際土壤古菌群落豐度占比較高的類群,提示中國水仙種植地也是甲烷釋放量較多的土壤類型。其中,產甲烷古‘菌是迄今所知的在嚴格厭氧條件下生存,并以甲烷為特異代謝產物的古菌群,它們產生的甲烷氣體在全球碳循環中起到不可忽視的作用[49]。已知古菌作為最古老的生命體,對不同土壤環境變異的適應能力較強,本研究對水仙花根際土壤微生物中古菌關鍵種群的鑒定及其豐度的研究,可為土壤古菌的地理分布規律及功能意義提供重要參考。
4結論
綜上所述,本研究基于3個不同類型微生物樣品:細菌、真菌和古菌,初步揭示了以漳州水仙為代表的中國水仙根際土壤的細菌多樣性、真菌多樣性以及古菌多樣性,尤其是發現了之前未知的水仙根際土壤真菌類群和特有的古菌類群,這些結果將為開發和利用水仙根際土壤微生物資源奠定基礎。同時,研究結果對于今后了解水仙微生物群落環境適應性具有重要意義。
參考文獻:
[1] CHAPARRO J M. BADRI D v,VIVANCO J M Rhizospheremicrobiome assemblage is affected by plant development[J]. ISMEJournal. 2014.8 (4): 790803
[2]MENDES R, GARBEVA P, RAAIJMAKERS J M. The thizospheremicrobiome: Significance of plant beneficial. plant pathogenic. andhuman pathogenic microorganisms [J] FEMS Microbiology Reviews.2013. 37(5):634-663
[3] BARDGETT R D,CARUSO T Soil microbial community responsesto climate extremes: Resistance. resilience and transitions to altemativestates[J].Philosophical Transactions of the Royal Society of LondonSeries B Biological Sciences. 2020. 375( 1794):20190112
[4]王鑫鑫,楊珍珍,周婉柔,等水仙根圍細菌群落組成[J]微生物學通報、2017. 44(5):1081-1088
WANG X X.YANG Z Z ZHOU W R et al_Identification ofthizosphere bacterial communities of Narcissus tazetta[J]Microbiology China. 2017. 44(5):1081-1088( in Chinese)
[5]楊明俊,李娟,王永川,等水仙內生真菌的分離鑒定及聚類分析[J]中草藥,2014.6 (11):1625-1630
YANG M J,LI J, WANG Y G,et al Isolation and cluster analysis ofendophytic fungi in Narcissus taetra var chinensis [J]. ChineseHerbal Medicines. 2014.6(11):1625-1630.( in Chinese)
[6] RYU c M. FARAG. MOHAMED A, et al Bacterial volatiles promotegrowth in Arabidopsis EJl Proceedings of the National Academv ofSciences of the Unired States礦America. 2003. 100 C8J 4927-4932.
[7] MENDES R.KRUIJT M.DE BRUIJIN I.ct alDeciphicring theRhizosphere Microbiome for Disease-Suppressive Bacteria [J].Science,2011.332(6033):1097-1100.
[8]HU J WEI Z.FRIMAN V P et al Probimtic diversity enhancesrhizosphere microbiome function and plant discase supprssion [J].American Society for Microbiology.2016.7(6):e01790.16.
[9]黃秋斌,張穎,劉風英,等.蠟樣芽孢桿菌B3-7在大田小麥根部的定殖動態及其對小麥紋枯病的防治效果[J].生態學報.2014.34(10):2559-2566.
HUANG Q B ZHANG Y.LIU F Y et.alColonization dynamics ofBacillus cerens B3-7 onwhear roots and control efficiency agaonstsharp cycspot of where[J].Acta Ecologica sinica,2014.34(10):2559-2566(in Chinese)
[10]楊光柱,黃文靜,李云國,等,蘋果根腐病根際土壤真菌組成及多樣性研究[J],果樹學報,2020.37(6):875-881. YANG G Z,HUANG W J,LI Y G. et al Fungal community anddiversity in thizospheric soil with root rot in an apple orchard [J]Journal of Fruit Science. 2020. 37(6):875881(in Chinese)
[11]李得銘,翟子翔,鄧濤,等番茄青枯菌分離與二重PCR體系建立[J]分子植物育種,2020. 18 (11):209-215
LI D M. ZHAI Z X DENG T.et al_Isolation of ralstoniasolanacearum and establishment of triple PCR svstem [J] MolecularPlant Breeding. 2020. lS(ll): 209-215.(in Chinese)
[12] BACH E M. WILLIAMS R J,HARGREAVES s K,et al Greatestsoil inicrobial diversirv found in micro-habitats [J]. S0il Biology andBiochemistry. 2018.1 18: 217-226
[13]錢寶,劉凌,肖瀟上壤有機質測定方法對比分析[J]河海大學學報(自然科學版),2011. 39(1):34-38
QIAN B, LIU L, XIAO X Comparative tests on different methods forcontent of soil organic matter [J]. Journal of Hohai UiversityNatural Sciences Edition). 2011 39(1):34-38(in Chinese)
[14]馬丹凱氏定氮法測定食品中蛋白質含量[J]計量與測試技術,2008. 35 (6):57-58
MA DKjeldahl detennination of protein content [Jl. Measurementand Testing Technology, 2008. 35(6) 57-58.( in Chinese)
[15]葉祥盛,趙竹青流動注射法與堿解擴散法測定上壤有效氮的比較[J]安徽農業科學,2011. 20:12166-12167. 12178
YE X S,ZHAO Z Q Comparison of soil available nitrogenconcentration between flow injection method and alkali-diffusioninethod [J].Journal of Anhui Agricultural Sciences. 201l,20:1216612167. 12178. (in Chinese)
[16] BOWMAN R A. COLE c V. An exploratory method for fractionationof organic phosphorus from grassland soils EJl. Soil science. 1978.125(2):95-101
[17] SIMARD R R Ammomum acetate-extractable elements In Soilsampling and methods of analysis[M]. Lewis Publisher: Boca Raton.FL. USA. 1993(1): 39-42
[18] MEIER L P,KAHR G Determination of the cation exchange capacity(CEC) of clay minerals using the complexes of copper(ll) ion withtriethvlenetetrainme and tetraethylenepentamine [J].Clavs and ClayMinerals. 1999. 47(3):386388
[19]梁淑軒,孫漢文石墨爐原子吸收光譜法分析藥用植物中微量營養元素的含量[J]光譜學與光譜分析、2002. 22 (5):847-849
LIANG S X. SUN H W Detennmation of trace elements in inedicinalplants by Grapphite Furnace atomic absorption spectrometry [J]Spectroscopy and Spectral Analvsis. 2002. 22(5):847-849 (inChinese)
[20] CHEN c, ZHANG J N. LU M. et al Microbial communities of anarable soil treated for 8 vears with organic and inorganicfertilizers [J].Biology and Fertility of Soils. 2016. 52(4):455-467
[21] 11 B X. CHEN H L,LI N N. et al Spatio-temporal shifts in thearchaeal community of a constructed wetland treating river water [J]The Science of the Total Environment. 2017,6051606: 269-275
[22] ZHAO s c,QIU s J,XU x P,et al Change m straw decompositionrate and soil microbial community composition after straw addition indifferent long-term fertilization soils [J] Applied Soil Ecology, 2019.138: 123-133.
[23] BOLGER A M. LOHSE M. USADEL B. et al. Trimmomatic: aflexible trimmer for Illumina sequence data [J] . Bioinformotics. 2014.30 (15) : 2114-2120.
[24]MAGOC T. SALZBERG S L. FLASH: fast length adjustment of shortreads to inprove genome assemblies [J].Bioinformotics. 2011.27 (21) : 2957-2963
[25] EDGAR R C UPARSE: highly accurate OTU sequences frommicrobial amplicon reads [J]. Nature Methods. 2013. 10 (10) :996-998
[26] LAN Y M. WANG Q. COLE J R. et al. Using the RDP classifier topredict taxonomic novelty and reduce the search space for findingnovel organisms EJl . PLoS One. 2012. 7 (3) : e32491.
[27] QUAST C, PRUESSE E. YILMAZ P. et al. The SILVA ribosomalRNA gene database project: Improved data processing and web-basedtools [J]. Nucleic Acids Research. 2013. 41(Dl): D590-D596.
[28] SCHLOSS P D. WESTCOTT S L. RYABIN T, et al. Introducinginothur: open-source. platform-independent. community-supportedsoftware for describing and comparing microbial communities [J].Applied and Environmental Microbiology, 2009. 75 : 7537-7541 .
[29]伍海兵 .張青青.梁晶.城市綠地土L壤肥李質量綜合評價方法初探[J] .上壤通報. 2020. 51 (4): 795-800.
WU H B. ZHANG Q Q, LIANG J A comprehensive evaluationmethod of soil fertility quality in urban green space[J]. ChineseJournal ofSoil Science. 2020. 51 (4): 795-800 ( in Chinese )
[30]賴煥雄,鄭小琴 .漳州市近48年氣候特征分析[J].安徽農業利學,2010. 38 (15) : 8056-8060
LAI H X. ZHENG X Q. Characteristics of climate change inZhangzhou City in Recent 48 years [J]. Journal of Anhui AgriculturalSciences. 2010. 38 (15): 80568060. (in Chinese)
[31] DAVIS K E R. SANGWAN P. JANSSEN P H. Acidobacteria.Rubrobocteridae and Chloroflexi are abundant among very slow-growing and mini-colony-forming soil bacteria [J]. EnvironmentalMicrobiology. 2011, 13(3): 798805.
[32] FAN K K. DELGADO-BAQUERlZO M. GUO X S. et alBiodiversity of key-stone phylotypes detennines crop production in a4-decade fertilization experiment [J].The ISME Journal. 2021. 15:550-561
[33] HAN Q, MA Q, CHEN Y. et al. Variation in thizosphere microbialcommunities and its association with the svmbiotic efficiency ofthizobia in soybean [J]. The ISME Journal. 2020. 14 (8): 19151928.
[34] WALTERS W A. JIN Z. YOUNGBLUT N. et al. Large-scalereplicated field study of maize thizosphere identifies heritablemicrobes [J]. PNAS. 2018. 115 (28) : 73687373
[35]XU J. ZHANG Y. ZHANG P. et al. The structure and function of theglobal citrus thizosphere microbiome [J]. Notural Commication.2018. 9 (1) :4894
[36]雷旭.李冰曉.等 .復合直流人工濕地系統不同植物根際微生物群落結構[J] .生態學雜志. 2015. 34 (5): 1373-1381
LEI X. LI B. LI X. et al Rhizosphere microbial community of threeplants in vertical-flow constructed wetland [J]. Chinese Journal ofEcology. 2015. 34 (5): 1373-1381 (in Chinese )
[37]楊美玲.張霞.王紹明 .等.基于高通量測序的裕民紅花根際上壤細群落落特征分析[J] .物學通報、 2018. 45 (ll) : 2429-2438
YANG M L. ZHANG X. WANG S M. et al High throughputsequencing analysis of bacterial communities in Yumin safflower[J].Microbiology China, 2018. 45(ll): 2429-2438. ( in Chinese )
[38] CHEN S J. ZHU Y. SHAO T Y. et al. Relationship betweenthizosphere soil properties and disease severitv in highbush blueberry(Vaccinium corymbosum) [J]. Applied Soil Ecology. 2019, 127:187-194.
[39] GAO L. WANG R. GAO J M. et al Analvsis of the structure ofbacterial and fungal communities in disease suppressive and diseaseconducive tobacco-planting soils in China[J]. Soil Reseorch. 2019.58(l) : 35-40
[40] QI G F. CHEN S. KE L X. et al Cover crops restore declining soilproperties and suppress bacterial wilt by regulating thizospherebacterial communities and improving soil nutrient contents [J].Microbiologicol Research. 2020. 238: 126505
[41] TEDERSOO L. BAHRAM M. POLME S. et al Global diversity andgeography of soil fungi [J]. Science, 2014. 346(6213 ): 1078.
[42] EGIDI E. DELGADO-BAQUERIZO M. PLETT J M. et al A fewAscomvcota taxa dominate soil fungal communities worldwide EJl .Noture Communication. 2019. 10: 2369.
[43] PIETRO A D. RELLA M G. PACHLATKO J P. et al. Role ofantibiotics produced by Chaetomimn globosum in biocontrol ofPytbium ultimum. a causal agent of damping-off [J]. Physiology andBiochemistry. 1992. 8(2): 131-135
[44] PARK J H. CHOI G J. JANG K S. et al Antifungal activity againstplant pathogenic fumgi of chaetoviridins isolated from Choetomiumglobosum [J]. FEMS Microbiology Letters. 2005. 252(2) : 309313.
[45] ANGEL R. SOARES M I M. UNGAR E D. et al. Biogeography ofsoil Archaea and bacteria along a steep precipitation gradient [J].TheISME Journal. 2010. 4 (4): 553563
[46]HONG J K. CHO J C. Environmental variables shaping the ecologicalniche of thaumarchaeota in soil: Direct and indirect causal effects[J].PLoS One. 2015. 10 (S) : e0133763
[47] SHAO K Q. JIANG X Y. HU Y. et al Thauinarchaeota affiliated withSoil Crenarchaeotic Group are prevalent in the alkaline soil of analpine grassland in northwestem China [J]. Annals of Microbiology.2019. 69(8): 867-870
[48]JIAO S, XU Y Q. ZHANG J. et al. Environmental filtering drivesdistinct continental atlases of soil Archaea between dryland andwetland agricultural ecosysterns [J]. Microbiome. 2019. 7(1): l-13
[49]段昌海,張翠景,孫藝華,等,新型產甲烷古菌研究進展[J].微生物學報. 2019. 59(6) : 981-995.
DUAN C H. ZHANG C J. SUN Y H. et al. Recent advances on thenovel methanogens[J].Acta Microbiologica Sinica. 2019. 59 (6) :981-995 (in Chinese)
(責任編輯 :林海清 )
收稿日期:2021-0328初稿:2021-0521修改稿
作者簡介:李媛媛( 1989-),女,博士,講師,主要從事環境微生物學研究(E-mail: livuanyuan9215@163 com)
*通信作者:陸鑾眉( 1968-),女,教授,主要從事觀賞園藝學研究(E-mail: 358070295@qqcom)
基金項目:福建省科技計劃對外合作重點項目( 201810014):漳州市自然科學基金項日(222020JlO);漳州市科技重大專項(222019ZDOl);閩南
師范大學校長基金項目( 4206/L21918)

