面向臨床研究的基因測序項目的設計原則、管理流程與質量控制標準
劉陽,許喆,程絲,石延楓,林金嬉,孟霞,姜勇,李昊
基因檢測是對受試者的DNA進行檢測以尋找可能的遺傳變異的過程。高通量測序是一種基因檢測技術,能夠對數百萬個DNA片段進行平行序列測定,因此可以同時對大量遺傳變異進行鑒定和分析。在臨床研究中引入基因測序,可以將受試者的遺傳變異與臨床表型、生物標志物等數據相結合,為疾病機制的研究和藥物靶標的開發提供豐富的資源[1]。臨床研究的樣本收集時間跨度往往以年為單位,一些大型研究還涉及多中心管理,因此面向臨床研究的基因測序項目對管理流程、分析手段及質量控制的標準化程度要求很高,需要與之相適應的項目管理框架。本研究旨在確定面向臨床研究的基因測序項目的設計原則,搭建項目管理與質控標準化流程,并將該流程應用于一項全國性多中心大型臨床研究,從而確定質量控制標準。
1 方法
1.1 基因測序項目框架的確定
1.1.1 文獻復習 通過文獻復習學習生物醫學研究中的基因測序數據分析流程[2-5],調研國際、國內大規模測序隊列的建設現狀[6-9],制訂面向臨床研究的基因測序數據處理流程。
1.1.2 專家咨詢 遴選國內基因組學、生物信息學、統計學、生物樣本庫、臨床試驗、數據管理等領域的專家,調研面向臨床研究的基因測序項目的需求,完善基因測序項目設計原則并確定管理規范。
1.2 基因測序項目的設計原則 通過整理吸納文獻復習結果和專家咨詢意見,面向臨床研究的基因測序項目需要能滿足可擴展性、可重復性及可溯源性的需求。針對這3點需求,分別確定了如下設計原則:
1.2.1 可擴展性 隨著對疾病機制研究的深入,越來越多疾病相關的遺傳變異被發現。在進行臨床研究基因檢測的設計時,如果僅包含當時所了解的疾病相關遺傳變異,一方面無法涵蓋今后可能報道的新變異,另一方面也很難進行探索性的挖掘。全基因組測序(whole genome sequencing,WGS)是對受試者的所有DNA進行的高通量測序,可以覆蓋和識別整個基因組的變異,包括單核苷酸變異(single nucleotide variant,SNV)、短插入/缺失變異(insertion-deletions,INDEL)以及更長的結構變異,能夠滿足臨床基因研究的可擴展性需求。
1.2.2 可重復性 科學研究的統計功效需要有一定的樣本量,而高通量測序技術依賴復雜的試劑、硬件和訓練有素的人員[10],一個批次所能測定的樣本有限,因此臨床研究的基因測序項目通常涉及多個批次。批次效應(batch effect)是指樣本之間的變化不是來自真實的生物學差異,而是來自實驗或技術之間的差異[11-12]。批次效應會降低研究結果的可重復性,導致假陽性和假陰性關聯,甚至可能會產生誤導性生物學或臨床結論[13]。解決批次效應,依賴于全面的研究項目設計、可靠的質量控制方案、仔細的執行過程記錄,以及恰當的統計建模方法,這些在臨床研究基因測序項目的方案設計中都應該考慮到。
1.2.3 可溯源性 研究人員進行大規模臨床研究的課題分析時可能會發現感興趣的現象需要回溯到單獨的樣本,這就對基因測序數據的可溯源性提出了要求。臨床研究往往涉及多中心、長時間的樣本收集,對于基因測序項目,一方面是管理上的挑戰,另一方面也是機遇,因為遺傳數據本身能夠對樣本質量和來源形成反饋。在基因測序項目管理流程的搭建中,應當充分利用這一點。
1.3 基因測序項目管理流程的應用 數據處理流程搭建好后,需要在真實的臨床研究中確定合適的質控標準。中國國家卒中登記Ⅲ(China national stroke registry-Ⅲ,CNSR-Ⅲ)隊列是住院的急性缺血性腦血管事件患者的全國前瞻性登記研究,共有15 166例缺血性卒中患者或TIA患者,涉及201家醫院,是一個典型的多中心隊列[14]。CNSR-Ⅲ研究設計了遺傳亞組并進行了全基因組測序,是非常好的基因測序項目管理流程應用場景,因此本研究將在CNSR-Ⅲ隊列上實施并確定質控標準。
2 結果
2.1 樣本處理
2.1.1 樣本類型 遺傳研究的目標是檢測患者的生殖系基因變異,因此理論上來說,來源于患者的任何細胞都含有同樣的變異,都可以用于基因測序[4]。為了取樣方便,臨床研究中通常保留患者的外周血白細胞用于基因測序。采集血樣標本需要送到指定實驗室分離血清、血漿和白細胞,隨后進行中心化儲存和基因測序安排。在CNSR-Ⅲ研究中,預先確定了171家醫院納入遺傳亞組,在這些醫院入組的12 603例患者參與了基因測序項目,其中有1308例沒有提供足夠的白細胞。
2.1.2 DNA提取 常用的將基因組DNA從細胞中提取出來的方法有兩種,其一是采用酚氯仿法提取,其二是使用具有獨特分離作用的磁珠(如DP329磁性血液基因組DNA試劑盒)。提取既可以手工進行,也可以使用全自動儀器(如核酸蛋白提取系統)進行。
2.2 測序數據生成
2.2.1 DNA質量評估 提取出的基因組DNA需要經過質量評估,DNA總量、濃度和片段長度均合格的樣本才可用于基因測序。常見的對基因組DNA的濃度進行定量測量的儀器有Qubit 2.0熒光儀和Gemini XPS酶標儀;對DNA質量的評估一般在瓊脂糖凝膠上進行電泳,以確保基因組DNA沒有大量降解。合格樣本的標準見表1。在CNSR-Ⅲ項目中,經過DNA提取和質量評估,381例患者的DNA含量不足或質量不合格。
2.2.2 測序平臺選擇 大規模隊列研究通常采用的高通量基因測序儀器為Illumina?測序儀或Ion TorrentTM測序儀[15]。出于人類遺傳資源數據安全以及成本方面的考慮,國內的隊列研究也逐漸開始采用國產的華大智造DNBSEQ測序平臺,如ChinaMAP計劃[7]。不同測序儀所用的化學試劑、擴增方式、檢測方式、讀段長度等均有不同,各有利弊。CNSR-Ⅲ研究的測序項目由華大制造的BGISEQ-500型號的測序儀(以下簡稱BGISEQ)完成。除外沒有提供足夠白細胞的患者以及DNA含量不足或質量不合格的樣本,有10 914例患者的外周血白細胞樣本進行了基因測序文庫制備[16]。
2.2.3 文庫制備 “文庫”指的是帶有可用于測序的側翼接頭的DNA片段,不同的測序儀所需要的文庫大小及接頭序列不同,因此文庫制備方法與所選用的測序儀有關。BGISEQ的文庫制備步驟包括隨機打斷基因組DNA,選擇一定長度范圍內的DNA片段,末端修復,連接接頭,擴增連接產物,分離單鏈并與夾板寡核苷酸生成單鏈環化DNA,消化線性分子,最后對連接產物進行純化以得到最終文庫(質量控制標準見表1)。在CNSR-Ⅲ項目中,11例樣本經反復多次嘗試均無法成功制備文庫,159例樣本疑似有微生物污染,最終有10 744例樣本進行了基因測序。

表1 基因組DNA及測序文庫質控標準
2.2.4 測序 “測序”指的是應用特定技術對文庫DNA進行檢測并且使用與平臺相關的專用軟件進行初始分析及堿基檢出的過程。對合格的文庫進行滾環擴增以產生DNA納米球,然后將DNA納米球加載到規則陣列芯片上,并在BGISEQ平臺上進行測序。序列衍生原始圖像文件由BGISEQ基本堿基識別軟件在默認參數設置下處理,序列數據以FASTQ格式存儲,包含每一讀段的序列以及相應的堿基質量分數。圖1展示了基因測序實驗部分的流程圖。
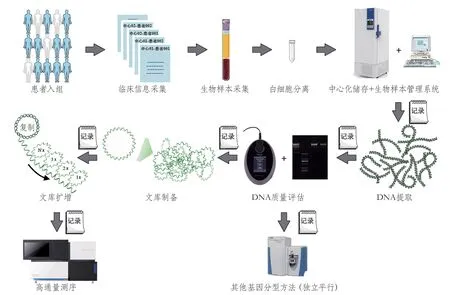
圖1 臨床研究中基因測序項目的實驗流程
2.3 測序數據生物信息學分析及質控
2.3.1 測序讀段清理 “讀段”指的是高通量基因測序儀檢測出來的DNA片段。測序儀的原始輸出文件中往往含有一些低質量的讀段,需要進行過濾。如果任一讀段含有測序接頭,或低質量堿基比例(堿基質量≤12)超過50%,或無法識別的堿基(“N”堿基)比例>10%,則移除該讀段對。之后對FASTQ文件進行質檢,使用Fastp軟件進一步過濾掉低質量的讀段和堿基[17]。
2.3.2 基因組比對 基因測序的讀段來自基因組DNA的隨機打斷,因此將質量合格的讀段比對回人類基因組,比對情況可以反映該數據來源樣本的DNA質量。利用在Sentieon軟件中實現的Burrows Wheeler校準工具將讀段比對到hg38人類參考基因組序列上[18],比對結果儲存在SAM或BAM文件中,該文件包括讀段序列、比對到染色體的位置以及比對質量等信息。完全相同的讀段通常是由于文庫制備時的PCR過程帶來的,因此需要去除。
2.3.3 雜合度 臨床研究的樣本可能會存在污染,如果污染來自微生物,會體現在文庫制備與測序數據質量上,在前述質控環節即應被鑒別與去除;而如果污染來自其他人源樣本,則需要基于測序數據基因組比對的結果進行深入分析。使用VerifyBamID軟件可以檢驗樣本之間的污染與混雜[19]。
2.3.4 測序數據質量評估 本研究通過對CNSR-Ⅲ項目10 744例樣本的數據質量進行整體評估,確定了9個單樣本質控項目,這些項目的內容與閾值見表2。質量評估結果為,15例樣本的10X覆蓋度<80%,1例樣本的錯配率>0.9%,267例樣本的人源污染率>0.03,這些樣本均被去除。圖2的上半部分展示了對每個樣本進行生物信息學分析及質控的過程。
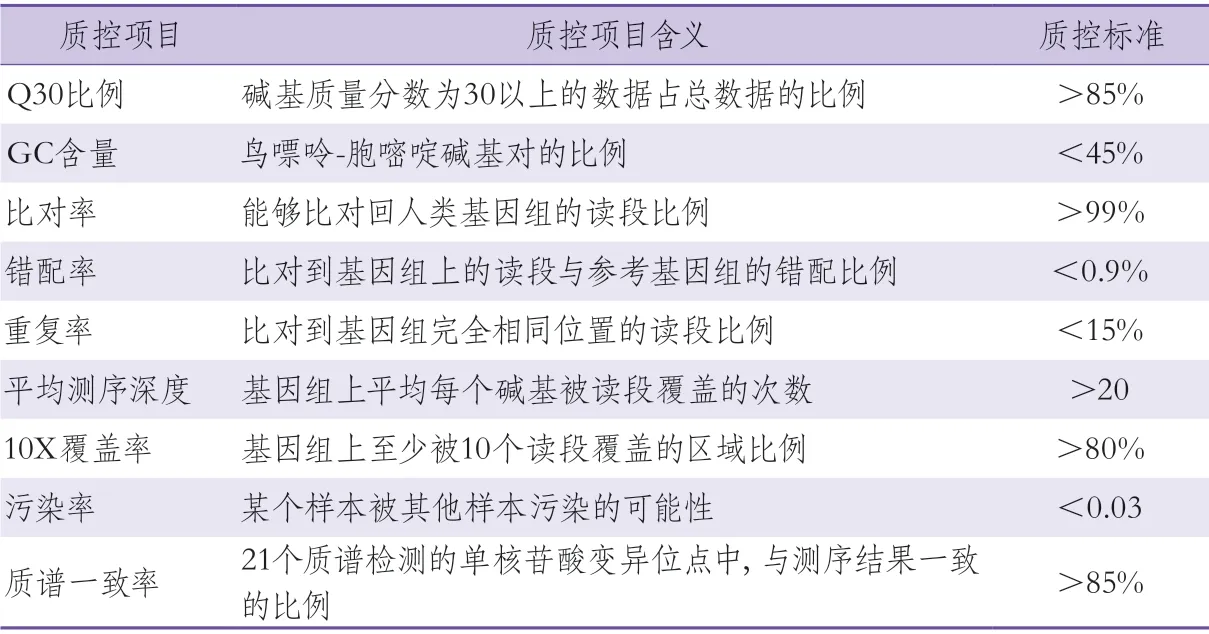
表2 中國國家卒中登記Ⅲ隊列9個單樣本質控項目的測序數據質控標準
2.4 遺傳變異鑒定
2.4.1 單樣本遺傳變異鑒定 GATK(genome analysis toolkit)最佳實踐指南是學術界最常用的高通量測序數據處理流程。根據該指南,首先,對樣本比對基因組的結果BAM文件進行堿基質量分數重新校準,目的是使用經驗誤差模型調整測序讀段的堿基質量分數;其次,使用Sentieon軟件實現的Haplotyper算法為每個樣本鑒定SNV和INDEL,最終生成VCF變異調用文件,該文件涵蓋每個變異在染色體上的位置、該位置原始的序列和變異序列,以及鑒定該變異的可信程度等信息。
2.4.2 變異聯合檢測 變異聯合檢測是指同時考慮所有樣本的變異檢測過程,該過程能利用一些樣本中的信息來推斷另一些樣本中最可能的基因型[2],從而提高低覆蓋區域中變異檢測的敏感性。利用GATK軟件可以將同一項目中所有樣本的變異調用文件整合為涵蓋全部樣本上全部變異的文件[20]。
2.4.3 變異質量分數重新校準 變異質量分數重新校準可以通過計算一個新的質量分數并以此為標準過濾遺傳變異,從而平衡鑒定變異的特異性和敏感性。使用GATK軟件對全部樣本進行硬過濾以去除高雜合度位點[20],接著將變異分為SNV和其他變異(包含INDEL和混合變異),并分別進行變異質量分數重新校準。在CNSR-Ⅲ項目中,兩類變異的過濾標準分別設定為敏感度99.0%和98.0%。圖2的下半部分展示了對全部樣本進行聯合生物信息分析及質控的過程。
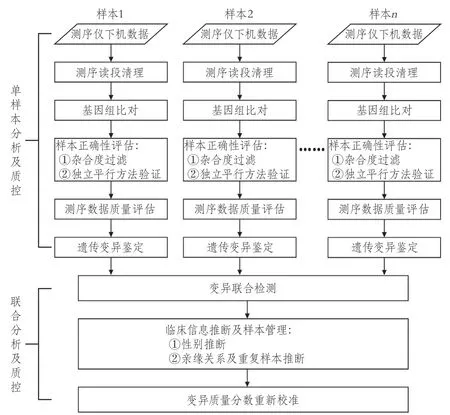
圖2 臨床研究中基因測序項目的生物信息分析流程
2.5 樣本臨床信息推斷
2.5.1 性別推斷 全基因組測序數據包含了個體的全部遺傳信息,因此可以推斷出樣本的性別。常見的推斷性別的原理有兩種:其一為根據性染色體深度,該方法同時可以判斷性染色體非整倍性;其二為根據X染色體雜合度,如可以使用Plink軟件判定X染色體雜合度異常的樣本[21]。
根據性染色體的深度進行性別推斷的方法:對于每個樣本,將X染色體和Y染色體的深度分別通過整個基因組的深度進行歸一化,并表示為二維圖上的一個點,兩個軸的坐標分別表示歸一化的X染色體深度和歸一化的Y染色體深度。根據臨床研究收集的基線信息對每個樣本按照性別進行標記后,在二維圖上自然地出現了邊界。在CNSR-Ⅲ項目中,僅憑歸一化的Y染色體深度0.075的簡單水平線即能夠將男性患者與女性患者分開,因而被選為性別推斷的閾值(圖3)。
同時,圖3中的離群散點代表了疑似性染色體非整倍性的樣本,由于納入這一步分析的樣本已經經過了測序質量的篩選,因此這些樣本更有可能是人群中自然的性染色體異常患者而非測序異常導致的。為了避免異常性染色體對遺傳相關機制研究產生影響,CNSR-Ⅲ項目中的這11例樣本被排除。同理,Plink軟件推斷出的13例異常X染色體雜合度樣本在后續分析中也被排除。
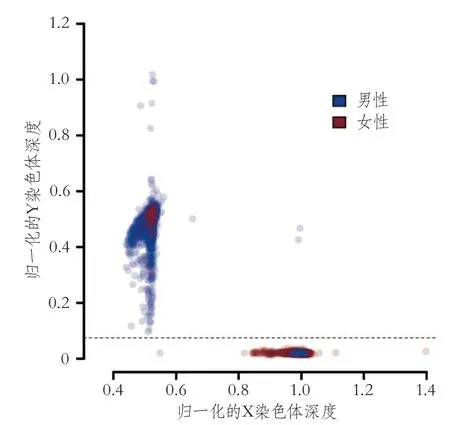
圖3 根據性染色體的歸一化深度對樣本進行性別推斷
2.5.2 親緣關系及重復樣本推斷 不同的臨床研究納入的患者不同,有的是針對家系的研究,有的則認為大部分受試者均無親緣關系,通過KING軟件可以推斷樣本之間的親緣關系以及群體中的重復樣本對[22]。CNSR-Ⅲ隊列作為一個前瞻性觀察性研究,沒有針對家系進行納入或排除,因此患者之間有可能存在親屬關系。使用KING軟件推斷樣本之間的潛在親緣關系,對于PI_HAT>0.125的個體對認為彼此之間是遺傳上相關聯的。
2.5.3 臨床樣本管理 基因測序項目不僅要保證數據正確,還要確保測序數據與生物樣本對應關系準確。因此,在納入排除樣本的流程中,需要考慮樣本推斷信息與臨床記錄不一致的情況(圖4)。在CNSR-Ⅲ項目中,將通過基因測序推斷的性別與臨床數據庫的性別進行對比,發現154例樣本的推斷性別與記錄性別不一致,且經過中心化項目管理部門與分中心研究者的校驗與核對,無法判斷是數據庫記錄錯誤還是樣本對應錯誤,因此刪除這些樣本。此外,經過臨床研究項目管理部門的工作,所有推斷出來的親緣關系均得到了分中心研究者確認,因此將38例患者排除在隊列之外。
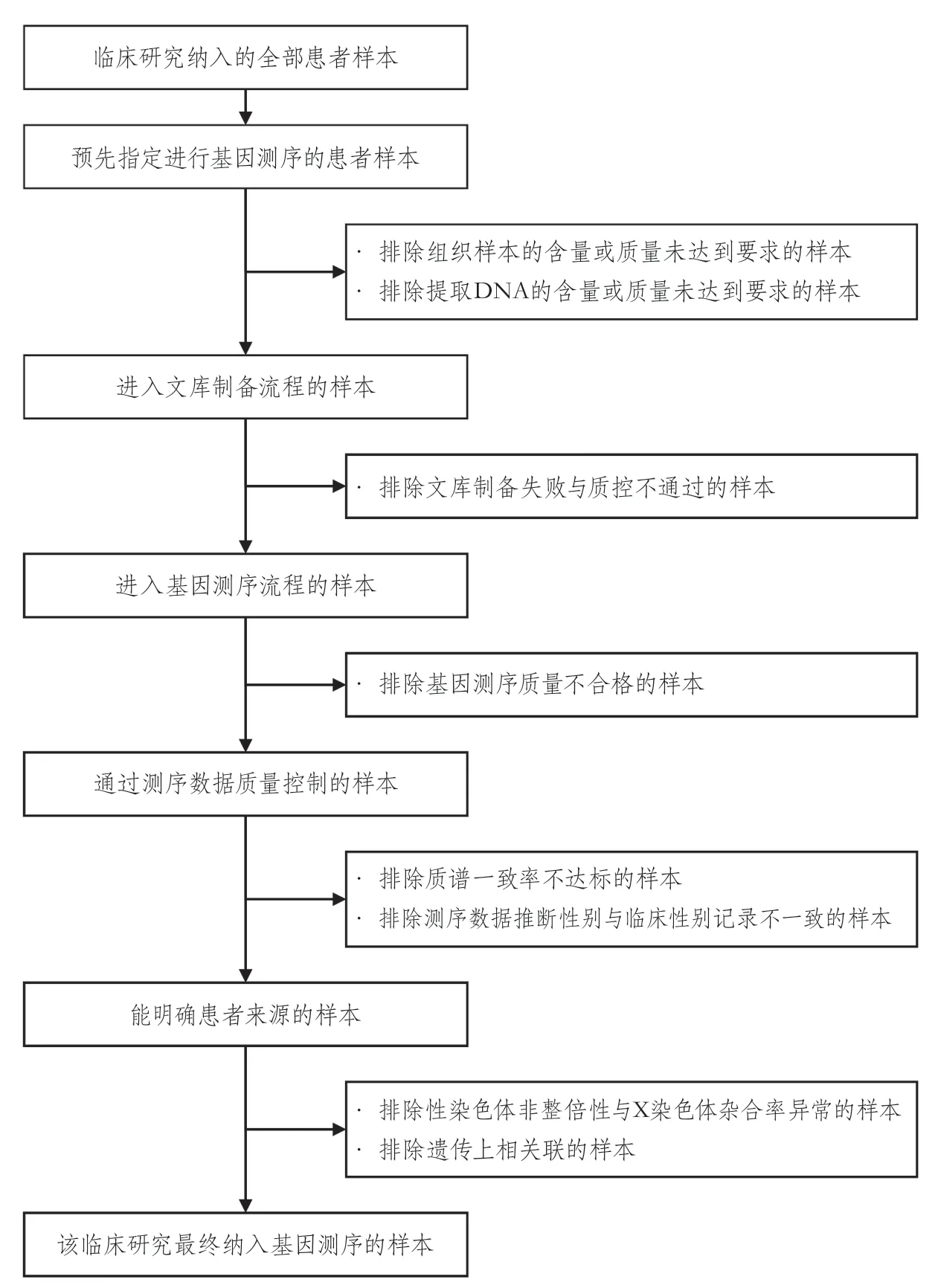
圖4 臨床研究中基因測序項目納入排除樣本的流程
2.6 基因測序項目的質量保證
2.6.1 基因檢測數據的相互驗證 對于大型臨床研究的基因測序項目,為了確保樣本在傳遞、提取、上機的過程中沒有被搞混,最好安排獨立平行的其他基因檢測方式,以驗證DNA樣本和測序數據來自同一個體。在CNSR-Ⅲ項目中,選取了21個雙等位基因指紋SNV,采用基于飛行質譜的基因分型方法進行項目質量管理[16],4例樣本因SNV分型一致率<85%而被排除。經過上述一系列樣本質控,有10 241例樣本最終可用。
2.6.2 樣本編號 涉及臨床研究的基因測序項目,為了避免患者隱私泄露以及人類遺傳資源數據安全遭到威脅,不僅需要對患者信息進行脫敏,而且建議不使用能追溯到其他信息(如患者基線、隨訪、生物標志物、影像等數據)的編號,可以采用一套新的唯一不重復編號并維護與其他信息編號的對應關系。在CNSR-Ⅲ項目中,以生物樣本送檢時的孔板編號為基礎編制了基因測序編號,在DNA提取和測序、SNV分型以及生物信息學分析的整個流程中均使用該基因測序編號,在后續涉及具體課題的分析中才對應回與其他信息能夠匹配的編號上。
2.6.3 樣本溯源 為了便于樣本溯源,應當記錄每個樣本在各個環節的信息。基因測序項目中需要保留的信息包括送樣批次及孔板排布信息、DNA提取批次及質控信息、引物和接頭名稱及序列、建庫批次及質控信息、測序批次及上下機時間、過程中的成功或失敗記錄及異常信息。如果樣本有重新送樣、提取、建庫情況,每次重復操作時的記錄也必須保留。測序過程可能會存在首次測序反應成功但數據量或質量不合格的情況,除了需記錄復測、加測時的生產記錄,還需記錄首次測序與復測、加測批次的對應情況,便于將多次測序的結果整合分析。上述所有記錄文件均須保留電子版和紙質版記錄備查。
2.6.4 批次效應控制 為了盡量降低測序反應中的批次效應,整個項目過程需采用預先指定的處理程序,測序儀和操作人員盡量保持一致,反應試劑盡量為同一生產批次。如果由于客觀原因無法保持一致,應盡量換用同品牌同貨號的產品并記錄每個樣本所使用的試劑批次、批號等,方便后續在數據處理階段去除批次效應。
2.6.5 實時反饋 臨床研究的基因測序項目往往樣本量大、運行時間長,如果待所有數據都接收后再進行質控,一旦發現問題,可能會給問題的解決或漏洞的彌補帶來麻煩。因此,在項目運行中應當采取實時反饋的機制,每接收一批數據應立即按照指定質控流程進行處理,并將不合格樣本或異常信息向檢測實驗室反饋,以便及時進行重測或加測,從而確保項目穩定和高效運轉。
2.7 數據安全與生物樣本安全
2.7.1 基因測序數據的安全 為了保障基因測序數據的安全,在生產過程中,測序儀要做到專機專用,并且項目開始前到項目結束后一定時期不可連接互聯網。測序數據生成后,不可使用網絡傳輸,可以采用硬盤等介質并進行加密傳輸,密碼需要采用與數據不同的途徑進行傳遞。此外,由于數據較大,為了避免傳遞過程中出錯,在將數據拷貝到傳遞介質之前需要生成MD5碼,將數據從傳遞介質拷貝到目的地后,再對MD5碼進行校驗,以確保數據完整和正確。基因測序成本高昂且難以再生,因此需要建立“兩地三中心”的容災備份解決方案。
2.7.2 生物樣本的安全保藏 用于基因測序的生物樣本及測序過程的中間產物含有人類遺傳物質,需要妥善保管。因此,在項目結束后,所用到的外周血白細胞、提取好的基因組DNA和測序文庫均必須返還中心化樣本庫,返還過程中需妥善包裝、冷鏈運輸。對于外周血白細胞,需要使用送樣時的原盒原孔原排布順序,已用完的白細胞也需要返還空管;對于基因組DNA和測序文庫,需要提交孔板排布表以及內容物的體積和濃度信息。為了避免運輸過程中樣本管順序被打亂,在包裝之前,需對每個樣本盒拍照,要求能看得清楚樣本盒的編號、板孔的狀態。
3 討論
高通量測序技術的實現改變了人類對健康和疾病的認識,如癌癥基因組圖譜(the cancer genome atlas,TCGA)、孟德爾基因組中心(centers for Mendelian genomics)和英國UK10K項目均采用高通量測序來進行疾病機制和健康狀況的研究[6,23-24]。臨床研究中基因測序的樣本量通常很大,檢測范圍更全面,因而對分析流程標準化、質控指標統一化以及項目管理精細化提出了更高的要求。本研究確定了面向臨床研究的基因測序項目的設計原則,搭建了項目管理與標準化數據質控流程,并在超過萬人的CNSR-Ⅲ隊列進行了驗證。
對質控標準的選擇是臨床研究基因測序項目的重點,特別是大規模多中心的臨床研究,由于不同分中心的醫院等級不同、設施設備不同、研究參與人員不同,因此樣本質量有所參差。對于質控標準的選擇,既不能太嚴格導致樣本量縮水增加檢測成本,又不能太寬松導致影響整體研究質量,需要在樣本量與樣本質量之間尋找到微妙平衡。本研究確定的基因組DNA質控標準適用于任何基因檢測項目,數據質控標準適用于大多數高通量測序項目(“平均測序深度”和“10X覆蓋率”兩個項目只適用于全基因組測序)。
通過生物信息學分析鑒別出的樣本性別有時與臨床信息中記錄的不同,親緣關系推斷出患者中隱藏的家庭和人口結構信息也不為研究者所掌握。此外,重復樣本可能暗示在樣本的傳遞、提取、上機的過程中有紕漏。上述不一致、不明確的情況均需反饋到中心化項目管理部門進行核驗與糾正,有時也需要回到分中心與研究者進行確認。這些現象在大型臨床研究中很難避免,也被其他研究所報道[7]。只要問題樣本所占的比例在一定范圍內,這其實不是壞事,通過對臨床研究的項目管理以及樣本質量形成閉環反饋,可以幫助找到并排除與臨床信息不對應的生物樣本,降低統計分析中的假陽性與假陰性。
本研究在基因測序項目的質量保證方面所做的工作(基因檢測數據的相互驗證、樣本重編號、全流程記錄、實時反饋等),盡最大可能降低了批次效應和系統偏差,保證了生物樣本安全與數據安全。然而,本研究還存在以下局限性:第一,樣本到達中心化生物樣本庫之前的步驟可控性差,院內采集患者生物樣本以及運輸過程中均存在弄混樣本的可能;第二,項目運行中人員、試劑、儀器等的更換難以控制;第三,盡管已經進行了非常詳盡的記錄,仍不可能窮盡批次效應的所有潛在來源。后續通過專門考慮批次效應的統計分析方案,可以進一步降低乃至消除批次效應[10],而這也依賴于本研究產生的可溯源性記錄。
總之,本研究所搭建的基因測序管理流程在CNSR-Ⅲ項目中應用成功,也為其他臨床研究中基因測序項目的管理與質量控制提供了參考與借鑒。
說明:本文涉及的部分生物信息學術語或數據庫名稱在國內尚無統一譯文,強行將這些術語或名稱翻譯成中文將影響讀者對原意的理解,因此本文對此類術語及名稱未進行翻譯。
【點睛】本研究搭建了一套基因測序項目框架,在中國國家卒中登記Ⅲ隊列上應用成功,可以為其他臨床研究中基因測序項目的管理與質量控制提供參考與借鑒。

