2 種帶魚科魚類線粒體COI 基因片段的比較分析
斯舒謹,褚夢潔,袁 帆,王忠明,楊天燕,朱文斌
(1.浙江海洋大學水產學院,浙江舟山 316022;2.浙江海洋大學海洋與漁業研究所,浙江省海洋水產研究所,浙江省海洋漁業資源可持續利用技術研究重點實驗室,農業農村部重點漁場漁業資源科學觀測實驗站,浙江舟山 316021)
帶魚Trichiurus lepturus 和沙帶魚Lepturacanthus savala 分別為隸屬于鱸形目Perciformes,帶魚科Trichiuridae,帶魚屬Trichiurus 和沙帶魚屬Lepturacanthus 是海洋中上層兇猛肉食性魚類[1-2]。帶魚在我國沿海均有分布,和大黃魚、小黃魚及烏賊并稱為中國的“四大海產”,也是東海漁場最重要的經濟魚類之一,歷史上漁獲量曾占全國總產量的80%以上[3-4]。沙帶魚產量相較帶魚少,分布于日本至澳大利亞、馬來半島及印度等近海水域,國內則常見于東海和南海[1-2],因背鰭金黃,福建沿海又有“黃鰭帶魚”或“黃金帶魚”的俗稱。20 世紀70 年代中期,隨著海洋捕撈強度的日益增加及環境的破壞,東海漁業資源面臨巨大壓力[5]。以帶魚為例,2008-2015 年間東海帶魚資源量發生劇烈波動,呈現持續下降趨勢。具體表現在群體組成低齡化和小型化、性成熟提前和初次性成熟最小肛長趨小以及單位漁獲量下降等方面[6-7]。近年來,隨著拖網和帆張網等主要帶魚捕撈作業方式減船轉產、伏季休漁延長、產卵場保護區設立、執法力度加大,帶魚資源狀況有好轉的趨勢。
線粒體DNA(mitochondrial DNA,mtDNA)嚴格遵守母系遺傳、具有分子結構簡單穩定、可溯源性強等特點,隨著DNA 測序技術和分子信息學的快速發展,mtDNA 在魚類系統學、種間雜交漸滲、群體遺傳學等方面得到廣泛應用,成為當今分子生物學研究中的一個熱門領域[8-10]。細胞色素c 氧化酶亞基I(cytochrome c oxidase subunit I,COI)基因是線粒體DNA 中13 個蛋白質編碼基因之一,因攜帶的遺傳信息量大、進化速率適中、解析能力強,主要應用于群體遺傳結構和系統發育分析、種類鑒定以及DNA 條形碼研究等領域[11-12]。
本研究分別選取我國舟山帶魚和北海沙帶魚為研究對象,采用線粒體COI 基因片段序列作為分子標記,探討2 種帶魚科魚類線粒體COI 基因片段序列的差異性,以期為帶魚和沙帶魚的種質資源保護研究和開發利用提供遺傳背景資料,并為帶魚科魚類分類和系統進化提供分子依據。
1 材料與方法
1.1 實驗材料
本研究實驗材料為2019 年7-9 月采集自舟山近海的帶魚和廣西沿海的沙帶魚,2 種魚類樣本共計52 尾(圖1)。所有樣品取背部新鮮肌肉組織存放于5 mL 離心管,加入無水乙醇置于-20 ℃保存備用。樣本數量、采集時間和地點信息如表1 所示。

圖1 2 種帶魚科魚類的采樣圖(括號數字代表樣本數量)Fig.1 The sampling locations of two Trichiuridae fishes (numbers in brackets represent the number of samples)
1.2 基因組DNA 提取及PCR 擴增
剪取適量背部肌肉組織,采用傳統的Tris 酚-氯仿法[13]提取帶魚和沙帶魚基因組DNA,將75%乙醇沉淀后經自然風干的DNA溶解于100 μL 滅菌水中,并放于4 ℃保存備用。用于擴增COI 基因序列的正反向引物分別為COI-F:5′-TCAACCAACCACAAAGACATTGGCAC-3′和COI-R:5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′。PCR 總反應體系為25 μL,各試劑劑量和引物劑量如表1 所示。

表1 PCR 反應體系Tab.1 The PCR reaction system
PCR 反應程序為:95 ℃預變性5 min;95 ℃變性0.5 min,54 ℃退火0.5 min,72 ℃延伸1.25 min 循環35 次;72 ℃延伸5 min。用濃度1.5%瓊脂糖凝膠電泳檢測PCR 擴增產物,凝膠成像系統拍照后,挑選條帶亮度較高的PCR 產物送至上海桑尼生物有限公司進行雙向測序。
1.3 數據分析
運用DNAstar Lasergene 7.1 軟件包[14]中的MegAlign 和Seqman 程序對測序結果進行比對和編輯;采用DnaSP 6.0 軟件[15]計算2 種魚類的單倍型數(H)、核苷酸多態位點數(S)、單倍型多樣度(Hd)和核苷酸多樣性指數(Pi);DAMBE 軟件[16]進行堿基替換飽和性分析;使用MEGA 6.0 軟件[17]分析序列堿基組成和變異位點情況,基于Kimura 2-Parameter(K2P)模型計算群體間遺傳距離并構建鄰接系統樹,系統樹各分支的置信度采用自展法重復1 000 次檢驗。
2 結果
2.1 COI 基因序列特征
經比對和校正去除序列兩端測序不準確部分,最終得到52 條長度為550 bp 的帶魚和沙帶魚COI基因同源序列。利用MEGA 軟件計算序列堿基組成如表2 所示。從堿基組成來看,沙帶魚的T 堿基含量最高,而帶魚C 含量最高,但2 種帶魚科魚類G 含量都最低,且A+T 含量大于C+G 含量,這與脊椎動物線粒體DNA 的特征相符[18]。堿基轉換明顯大于顛換,轉換/顛換(R)平均值為2.72,變化范圍在2.65~10.43 之間。4 種堿基在密碼子第1、2、3 位點的平均含量存在一定差別,其中第1 位點各堿基含量差別較小,第2 位點T 堿基含量偏高而A、G 堿基含量大致相當,G 含量偏低這一現象在密碼子第3 位點尤為突出(圖2)。在183 個三聯體密碼子中,第1 位點發生堿基突變的有37 個(27 個堿基轉換、10 個堿基顛換),第2 位點發生堿基突變的有3 個(3 個堿基轉換),第3 位點發生堿基突變的有1 個(1 個堿基顛換),由此可以看出密碼子第1 位點堿基突變數目明顯高于其它位點。

圖2 帶魚和沙帶魚COI 基因片段密碼子堿基組成Fig.2 Codon composition of COI gene fragment in T.lepturus and L.savala

表2 帶魚和沙帶魚COI 基因片段堿基組成Tab.2 Base composition of COI gene fragments in T.lepturus and L.savala
2.2 遺傳多樣性
2 種魚類COI 基因序列共檢測到25 種單倍型,其中24 尾帶魚樣品中檢測到13 種單倍型(DYhap1-DYhap13),28 尾沙帶魚樣品中存在12 種單倍型(SDYhap14-SDYhap25)。52 條同源序列存在變異位點總計105 個,約占序列總長度的19.1%,其中13 個帶魚COI 基因序列中有13 個變異位點、12 個沙帶魚COI 基因序列中有28 個變異位點(圖3)。沙帶魚平均單倍型多樣度(Hd)及核苷酸多樣性指數(Pi)分別為0.7169 和0.0050,保守位點數目為522,多態位點數目為28,占位點總數的5.09%,在28 個多態位點中,包括單一變異位點(singleton variable sites)21 個,簡約信息位點(parsimony informative sites)7 個;帶魚平均單倍型多樣度(Hd)及核苷酸多樣性指數(Pi)分別為0.884 1 和0.004 6,保守位點數目為537,多態位點數目為13,占位點總數的2.36%,在13 個多態位點中,包括單一變異位點4 個,簡約信息位點9 個(表3)。
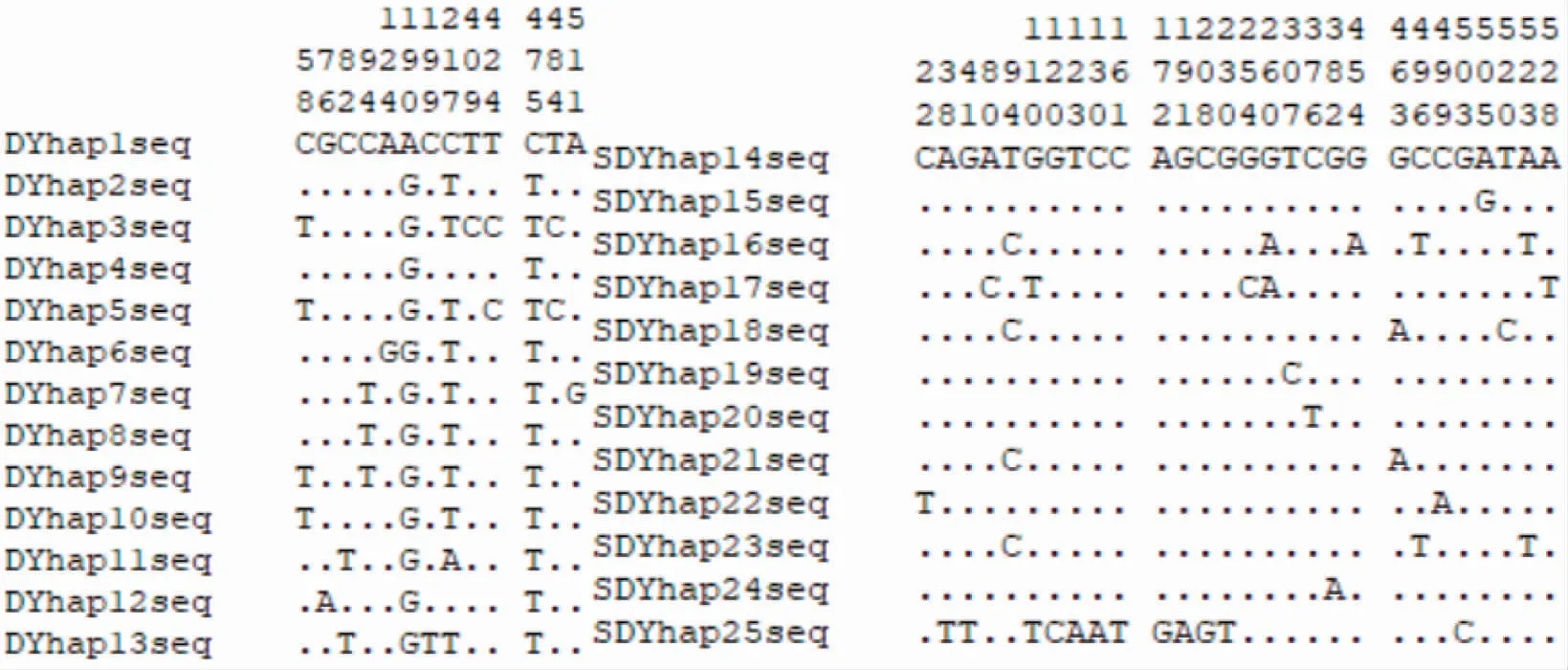
圖3 COI 基因序列片段單倍型變異位點分布(左圖為帶魚、右圖為沙帶魚)Fig.3 Variable sites of COI gene sequence from different haplotypes (T.lepturus is on the left and L.savala is on the right)

表3 基于線粒體COI 基因的帶魚和沙帶魚遺傳多樣性參數Tab.3 Genetic diversity indexesbetween T.lepturus and L.savala based on mtDNA COI gene
2.3 遺傳距離及系統發育
采用Kimura 2-Parameter 模型分別計算沙帶魚和帶魚種內個體間平均遺傳距離分別為0.004 9 和0.004 5,前者略大于后者。基于COI 基因序列得到的沙帶魚和帶魚種間的平均遺傳距離為0.165 5,種間的遺傳距離遠大于種內的遺傳距離。
使用DAMBE 軟件進行替換飽和性分析,并以堿基轉換數和顛換數為縱軸,以P-distance 模型校正的遺傳距離為橫軸做散點圖(圖4),顯示堿基轉換和堿基顛換均呈直線上升趨勢,COI 基因序列堿基替換數與遺傳距離呈顯著的線性關系且未出現平臺期,表明用于本研究的基因序列替換未飽和,適用于構建系統發育樹。圖5 為基于25 個單倍型構建的鄰接(neighbor-joining,NJ)關系樹,可以看出2 種帶魚科魚類分別聚類成獨立的兩支。從NCBI(National Center for Biotechnology Information)數據庫下載19 種帶魚科魚類COI 基因序列(表4),選擇本研究中2 種魚類單倍型數量最多的各1 條序列作為代表(DYhap2 和SDYhap14),以花鱸Lateolabrax japonicus(GenBank 登錄號:MF122427)作為外群,比對截取長度為504 bp 的同源序列,采用NJ 法構建系統發育關系樹如圖6 所示。
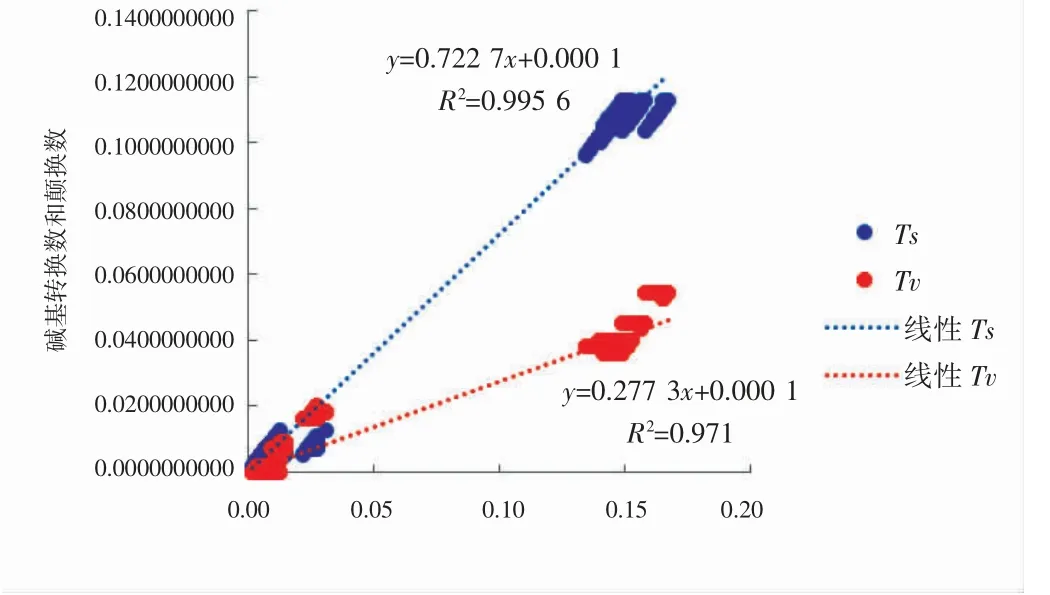
圖4 線粒體COI 基因堿基替代飽和度分析Fig.4 The substitution saturation analysis of mitochondrial COI gene

表4 用于構建系統進化樹的帶魚科不同物種序列信息Tab.4 Sequences of Trichiuridae fishes used for phylogenetic tree construction

圖5 基于單倍型構建的帶魚和沙帶魚NJ 系統樹Fig.5 NJ phylogenetic tree of T.lepturus and L.savala based on haplotypes

圖6 基于鄰接法構建的帶魚科魚類系統發育關系樹Fig.6 The phylogenetic tree of Trichiuridae fishes based on neighbor-joining method
3 討論
全球帶魚科魚類的主要捕撈海域包括西北太平洋、北印度洋和西非,其中西北太平洋是主要產區。我國近海5 種主要帶魚科經濟捕撈種類有沙帶魚、小帶魚Eupleurogrammus muticus 以及帶魚屬的帶魚、短帶魚T.brevis 和南海帶魚T.nanhaiensis。因前兩者產量較低且缺乏數據資料,在漁業統計時常被統計為帶魚。作為中國海洋魚類中捕撈產量最大的品種,20 世紀90 年代,我國帶魚科魚類捕撈量就已占全球總產量的60%[4,19],近年來平均年捕撈產量基本維持在100 萬t 左右[3]。
目前國內關于帶魚的研究主要集中在漁業生物學、種群動態、資源評估模型應用以及遺傳多樣性等方面。舟山帶魚口感鮮嫩、肉質細膩,是我國首批獲得海鮮類地理標志證明的產品,舟山帶魚有別于其他帶魚的優越品質是否與種質因素有關尚未得知。目前專門針對舟山近海帶魚種群遺傳學的研究不多,僅見李鵬飛等[20]曾對浙江近海帶魚的線粒體COI 基因進行比較分析,發現帶魚該基因變異程度較小;鄭文娟等[21]基于線粒體控制區對舟山海域3 個帶魚地理群體遺傳多樣性分析,發現群體間分化程度小、基因交流頻繁,遺傳多樣性處于中等偏下水平。本研究對采自舟山近海的24 尾帶魚線粒體COI 基因片段序列分析發現其平均單倍型多樣度(Hd=0.884 1)及核苷酸多樣性指數(Pi=0.004 6)均低于卞光明等[22]的研究結果(Hd=0.998 3,Pi=0.006 3)。該結果顯示近年來舟山近海帶魚遺傳多樣性水平呈現某種程度的下降趨勢。造成這一差異的原因一方面與采樣地點和取樣數量有關。另一方面,由于遺傳多樣性是生物多樣性的基礎,也是物種和種群長期續存的進化潛力,遺傳多樣性降低會引起生物對環境適應力的減弱并最終導致滅絕[23-24]。盡管我國實行的伏期休漁期制度對保護海洋資源起到了積極作用,但日益增長的市場需求對帶魚野生資源造成的捕撈壓力以及帶魚這種深海魚類難以突破人工繁殖的世界性難題,使得舟山漁場帶魚資源量恢復緩慢,種群數量的減少加之棲息地環境的改變使得舟山帶魚種質資源恢復面臨一定壓力。從2 種帶魚科魚類的遺傳差異來看,沙帶魚多態位點數目、平均核苷酸差異數和核苷酸多樣性均高于帶魚,表明較之帶魚而言,沙帶魚具有較豐富的遺傳資源和較好的開發潛力。
線粒體COI 基因作為DNA 條形碼(DNA barcoding),已廣泛應用于昆蟲、鳥類、甲殼類及魚類等物種的快速識別和鑒定,成為當今生態學研究的重要工具[25]。HEBERT,et al[26]分析比較了13 320 種生物的線粒體COI 基因序列,發現絕大多數生物的種內遺傳距離差異在0%~2%之間,大于2%非常少,種間遺傳距離則高達11.3%[26]。本研究發現沙帶魚和帶魚種間平均遺傳距離為0.165 5,約為種內平均遺傳距離(0.004 9和0.004 5)的33~36 倍,表明各物種的COI 序列間已經形成明顯“條形碼間隙”(Barcoding gap),即種間遺傳距離為種內遺傳距離的10 倍及以上[27]。基于K2P 遺傳距離構建的NJ 系統樹明顯分為2 支,帶魚和沙帶魚單倍型各自聚在一起,也表明兩種魚類可以得到很好地區分。結合NCBI 數據庫中下載的其他19 種帶魚科魚類COI 基因序列進行聚類分析,發現這些魚類按照尾鰭形態可分為鞭狀和叉狀2 大類群,前者包含了絕大多數常見的種類,其中帶魚屬和沙帶魚屬親緣關系最近,兩者聚類后再與小帶魚屬魚類相聚,小帶魚屬是最早分化出來的種類,其次為沙帶魚屬,而帶魚屬則是最新演化的種類;后者主要包含了叉尾帶魚屬和等鰭叉尾帶魚屬魚類,與蔡傳庚對10 種帶魚科魚類COI 基因聚類的結果相似[28]。有學者曾將帶魚科劃分為3 個亞科,其中等鰭叉尾帶魚亞科Aphanopodinae 包含了等鰭叉尾帶魚屬Aphanopus 和深海帶魚屬Benthodesmus[29]。但從本研究的聚類情況來看,兩者并未聚類到一起,彼此間的演化關系也不甚明確,還有待于今后增加樣品種類和數量并結合其他分子標記進行深入探討。

