達里湖高原鰍不同地理群體遺傳多樣性與遺傳分化
DOI:10.15928/j.1674-3075.202206130226
收稿日期:2022-06-13"""" 修回日期:2023-12-01
基金項目:山東省自然科學基金項目(ZR2021QC224);棗莊學院博士科研啟動基金項目(1020737)。
作者簡介:鞏政,1992年生,男,博士,講師,研究方向為魚類生態學。E-mail: zgongz@163.com
通信作者:潘保柱,1979年生,男,博士,教授,研究方向為淡水生態、生態水力、生態修復及河流生態水沙綜合管理。E-mail: zhuzipan@xaut.edu.cn
摘要:了解達里湖高原鰍(Triplophysa dalaica)不同群體遺傳變異現狀,為制定適宜的物種保護及水域生態系統管理策略提供基礎。選擇線粒體Cyt b基因作為分子標記,分析了2021年4月和7-8月采集于黃河流域的紅堿淖、大黑河和查干諾爾湖流域的輝騰河等3個地理群體,以及文獻記載海河流域的漳河群體的遺傳多樣性、遺傳分化及種群歷史動態。結果表明,70尾個體中共檢測到16個變異位點和13個單倍型;不同群體的單倍型多樣性范圍為0.233~0.791,核苷酸多樣性范圍為0.00020~0.00209。其中,輝騰河群體遺傳多樣性水平最高;紅堿淖與大黑河群體單倍型多樣性水平較高,核苷酸多樣性水平較低;漳河群體單倍型多樣性與核苷酸多樣性均較低。單倍型系統發育樹和進化網絡圖顯示,13個單倍型未聚類形成明顯的譜系結構,多數單倍型為單一群體獨有;除漳河群體外,其余群體的單倍型混雜連接,未遵循各自的地理分布格局。遺傳分化分析結果顯示,群體間遺傳分化指數為0.233~0.867,多為高度分化;分子方差分析表明遺傳變異主要來源于群體間(64.86%)。種群歷史動態分析未檢測到達里湖高原鰍各地理群體及總體在近期經歷過種群擴張。鑒于達里湖高原鰍已成為雅魯藏布江外來魚類,建議將達里湖高原鰍自然分布區內的各群體劃定為不同保護單元并跟蹤監測棲息地質量,同時加強對物種交易與放生活動的管控。
關鍵詞:達里湖高原鰍;遺傳多樣性;遺傳分化;種群歷史動態
中圖分類號:Q347""""""" 文獻標志碼:A""""""" 文章編號:1674-3075(2024)04-0092-07
高原鰍屬(Triplophysa)隸屬于鯉形目(Cypriniformes)條鰍科(Nemacheilidae),是一類高度適應高寒環境的中小型魚類,主要分布于青藏高原及周邊水系(朱松泉,1989;何德奎等,2006)。作為伴隨青藏高原隆升過程中地質運動和水系變遷而起源輻射的一個類群,高原鰍屬魚類物種分化活躍,隱存種繁多,近年來不斷有新種被發現;根據美國加州科學院Catalog of Fishes數據庫的收錄情況,目前已知有效種或亞種超過180個(Fricke et al,2022)。有關高原鰍屬魚類基礎生物學、生態學、基因組學及適應性進化等方面的研究被陸續報道,顯示出該類群重要的科研價值(Wang et al,2016;Feng et al,2017)。受物種繁多、演化歷史復雜及鑒別性狀難以辨識等因素限制,該屬魚類的分類厘定工作尚不完善。同時,相較高原鰍屬魚類豐富的物種多樣性,有關其種內遺傳多樣性的研究卻相對滯后,這對于評估物種保護地位及制定物種保護方案十分不利,亟需填補相關研究空白(Feng et al,2019)。
達里湖高原鰍(Triplophysa dalaica)是一種小型底棲高原鰍屬魚類,模式產地位于內蒙古東部內流湖泊達里湖,其主要鑒別特征為全身無鱗,腹鰭位置相對背鰭顯著靠后,鰾后室發達,長卵圓形,通過一細管與前室相連。達里湖高原鰍已知分布區集中在黃河蘭州段以下的干支流和內蒙古中東部的一些內流水體;近期該物種在海河流域也有新的分布記錄(周傳江等,2020),是分布界限向東部地區延伸最遠的高原鰍屬魚類。作為一種分布于干旱、半干旱地區的魚類,已有研究從生理學、轉錄組學和基因組學的角度報道了達里湖高原鰍對高堿性低溶氧水體的耐受能力(Wang et al,2015;武鵬飛,2017;Zhou et al,2021)。近年來,隨著內蒙古地區眾多內流河湖水量衰減或水源枯竭,魚類棲息地不斷萎縮和喪失,鹽堿脅迫程度不斷加劇;例如達里湖湖面面積逐年縮小,水位下降,漁業資源出現衰減的跡象(楊富億等,2021),以達里湖高原鰍為代表的淡水魚類的生存狀況受到嚴重威脅。然而,目前關于達里湖高原鰍種群生態學的研究資料仍較為匱乏,對不同地理群體的遺傳多樣性、譜系或群體分化等方面的現狀還知之甚少,物種保護策略制定及水域生態系統管理等相關工作缺乏科學依據。
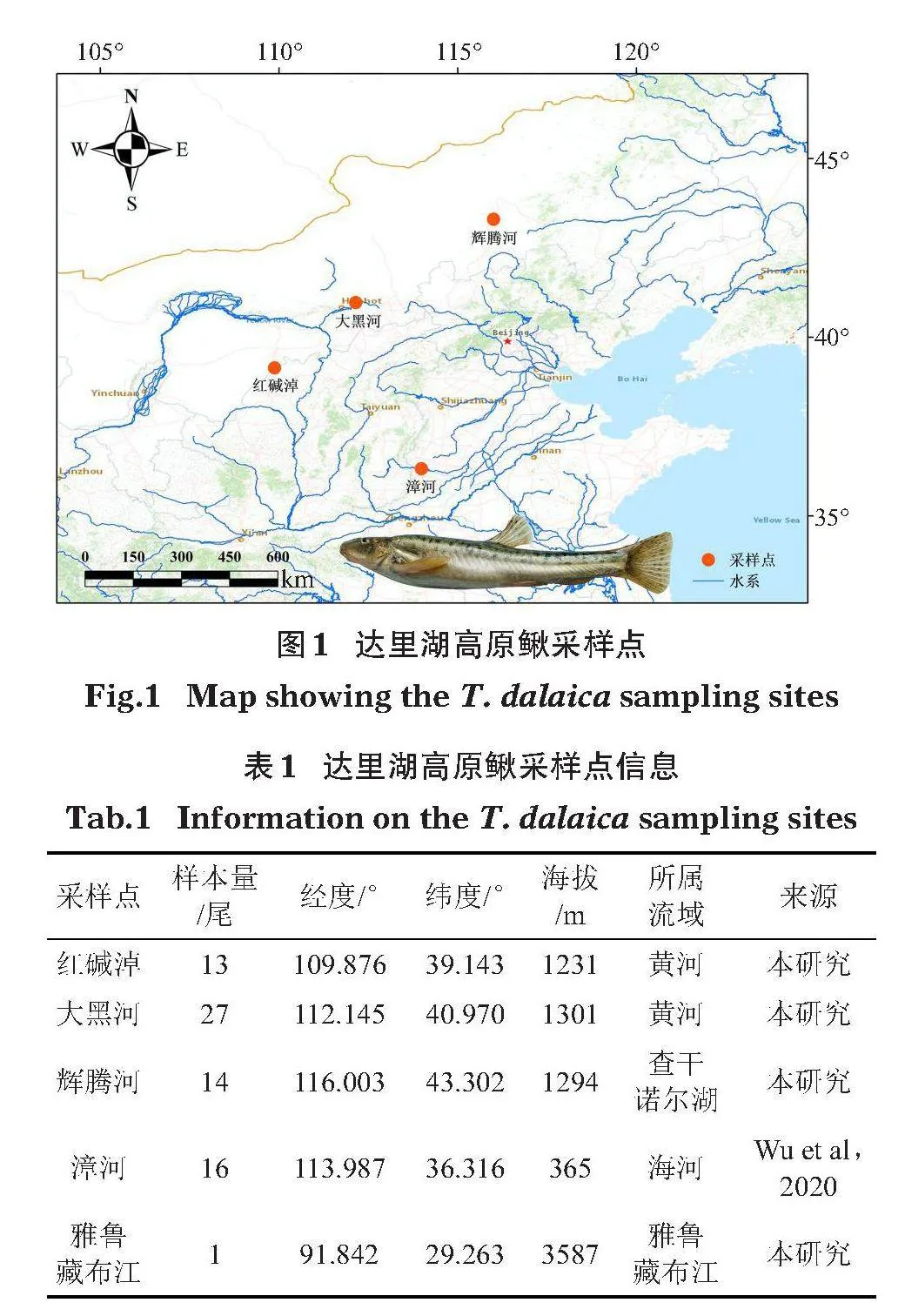
本研究在達里湖高原鰍分布區內選擇紅堿淖(陜西省榆林市)、大黑河(內蒙古自治區呼和浩特市)及輝騰河(內蒙古自治區錫林郭勒盟)3個樣點,采集達里湖高原鰍樣本,測定其線粒體Cyt b基因序列,結合已發表的海河支流漳河的樣本序列數據,分析達里湖高原鰍不同群體的遺傳多樣性、遺傳分化及種群歷史動態,以期為達里湖高原鰍的物種保護提供科學依據;結合在雅魯藏布江流域發現的1尾外來達里湖高原鰍個體,提出該物種的保護與管理對策。
1"" 材料與方法
1.1"" 樣本采集
2021年4月和7-8月在陜西省榆林市紅堿淖、內蒙古自治區呼和浩特市大黑河及內蒙古錫林郭勒盟輝騰河采集魚類樣本,漁具使用小型背攜式電魚機;采集到的達里湖高原鰍樣本參照《中國條鰍志》(朱松泉,1989)進行形態學鑒定和確認,隨后使用乙醚麻醉處死,保存于無水乙醇中,帶回實驗室冷藏于-20℃冰箱中備用;3個群體的樣本量在13~27尾。河南省安陽市漳河群體16尾樣本的序列數據引自Wu等(2020);4個樣點覆蓋了黃河、海河及內蒙古自流水體查干諾爾湖等達里湖高原鰍主要分布水系。雅魯藏布江的1尾達里湖高原鰍樣本于2020年5月采自西藏自治區山南市。采樣點及樣本的具體信息見圖1和表1。
1.2"" 基因組DNA提取、擴增及測序
采用略微改進的高鹽法從肌肉或鰭條組織中提取基因組DNA(Tang et al,2009),Cyt b基因擴增和測序引物選用L14724(5’-GACTTGAAAAACCACCGTTG-3’)和H15915(5’-CTCCGATCTCCGGATTACAAGAC-3’)(周傳江等,2020)。PCR反應體系為20 μL,具體組分為10×Buffer 2 μL,dNTPs 0.25 μL(10 mmol/L),引物各0.5 μL(10 μmol/L),TaqDNA聚合酶0.5 U,基因組DNA模板1~2 μL,加滅菌雙蒸水定容至20 μL。PCR反應條件:94℃預變性3 min;然后進入35次循環(94℃變性45 s,52~56℃退火45 s,72℃延伸1 min);最后72℃延伸8 min。采用0.8%濃度的瓊脂糖凝膠檢測PCR產物中是否含有目標片段,擴增成功后送至武漢天一輝遠生物科技有限公司進行純化和雙向測序。
1.3"" 序列整理與數據分析
獲得的基因序列由Dnastar軟件包(DNASTAR Inc.,Madison,USA)進行校對、編輯和拼接。拼接完成后的序列集使用MUSCLE 5.1軟件進行比對(Edgar,2004),利用MEGA 7.0軟件計算序列集的堿基組成、轉換/顛換值,并基于Kimura 2-Parameter(K2P)模型計算群體間的遺傳距離(Kumar et al,2016)。利用DnaSP 6統計序列集的變異位點、單一變異位點和簡約信息位點,計算單倍型數、單倍型多樣性與核苷酸多樣性等指數(Rozas et al,2017)。利用Arlequin 3.5軟件計算兩兩群體間基于1 000次重復抽樣的遺傳分化系數(FST),并進行分子方差分析(analysis of molecular variance,AMOVA)(Excoffier amp; Lischer,2010)。
選取武威高原鰍(Triplophysa wuweiensis)作為外類群,基于貝葉斯推斷(bayesian inference,BI)方法在MrBayes 3.1軟件中構建達里湖高原鰍單倍型的系統發育樹(Ronquist et al,2012)。建樹前首先利用PartitionFinder 2.3軟件基于修正的赤池信息量原則(corrected Akaike information criterion,AICc)在PhyloSuite平臺上篩選出最佳核苷酸替代模型(Zhang et al,2020)。貝葉斯法構建系統發育樹時,核苷酸替代模型根據篩選結果進行設定:nst=6,rates=invgamma;起始樹設置為隨機樹,基于馬爾科夫鏈蒙特卡洛方法設置4條鏈,運行200萬代,每1 000代進行1次抽樣,獨立運行2次,舍棄前50%的運行代數,剩余的樣本構建一致樹并計算后驗概率。單倍型之間的進化網絡圖在Popoart 1.7軟件中基于TCS法構建(Leigh amp; Bryant,2015)。
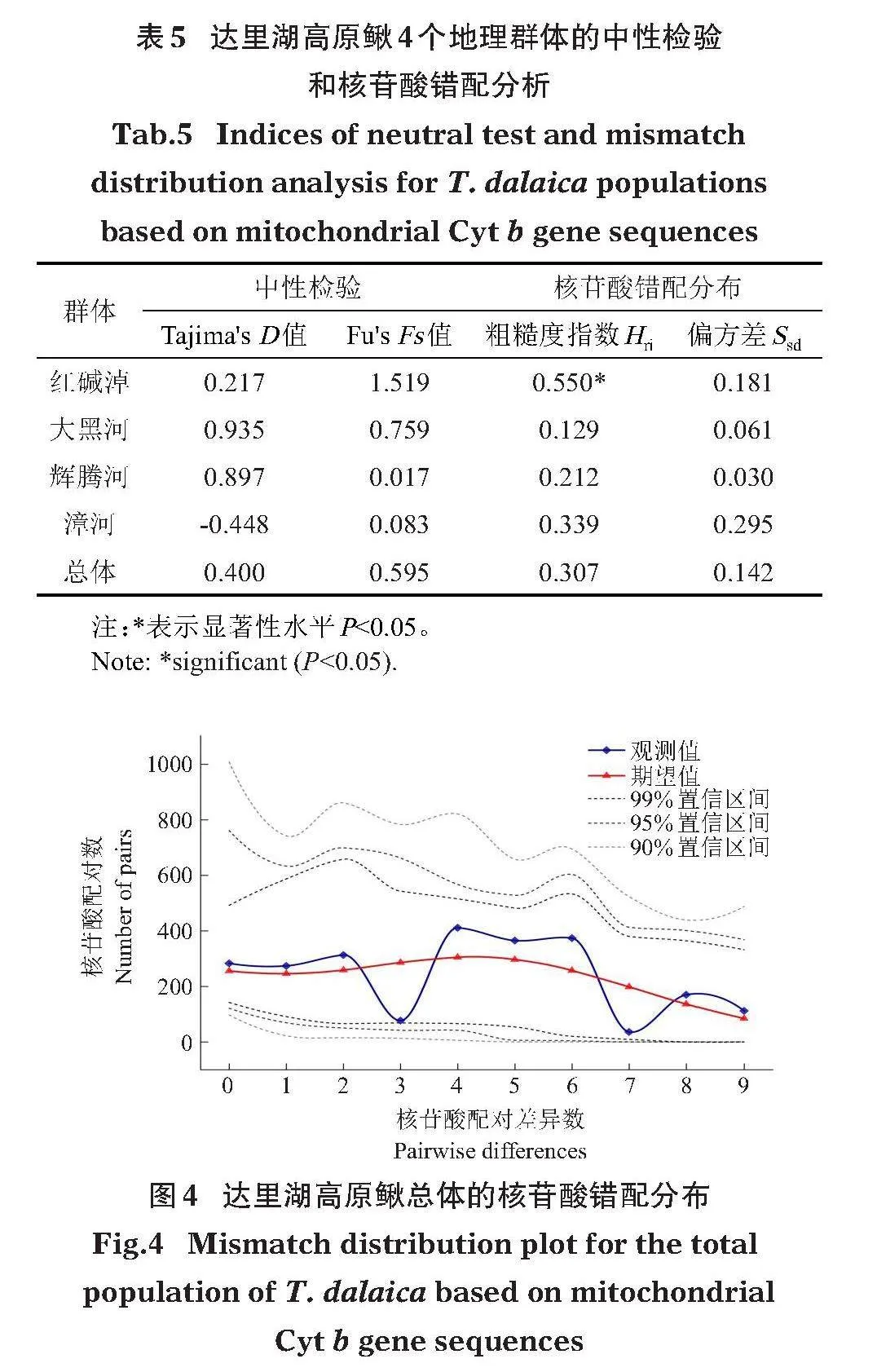
采用中性檢驗(neutral test)和錯配分布(mismatch distributions)追溯達里湖高原鰍的種群歷史動態。首先,在Arlequin 3.5軟件中計算中性檢驗指數Fu's Fs和Tajima’s D值;同時結合錯配分布粗糙度指數Hri和偏方差Ssd來初步檢驗種群是否經歷群體擴張;最后,基于核苷酸錯配分布曲線進一步確定種群是否經歷過瓶頸效應或群體擴張(Excoffier amp; Lischer,2010)。
2"" 結果與分析
2.1"" 序列信息
經整理獲得71尾達里湖高原鰍個體Cyt b基因序列集,長度為1 141 bp,基因的起始密碼子為ATG,終止密碼子為不完整的單堿基T;共檢測到16個變異位點,包括13個簡約信息位點和3個單一變異位點,無堿基的插入和缺失;變異位點的轉換(Ti)與顛換(Tv)比值為19.3。序列集中A、T、C、G的平均組成分別為26.1%、29.8%、27.6%和16.6%,A+T的含量(55.9%)高于G+C的含量(44.1%);堿基G含量最低,表現出明顯的反G偏倚。
2.2"" 單倍型及遺傳多樣性
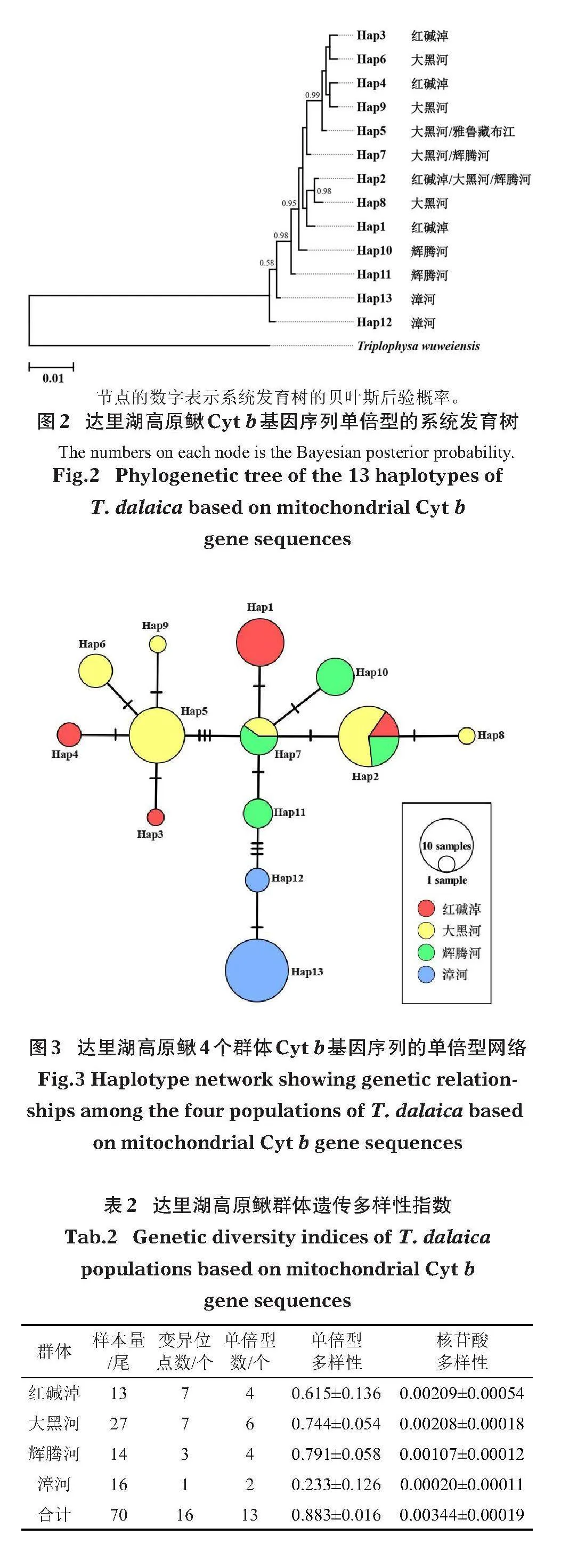
在4個群體70尾個體中共檢測到13個單倍型,包括3個共享單倍型和10個獨有單倍型,單倍型之間的平均K2P距離為0.4%。基于BI法構建的單倍型系統發育樹如圖2所示,13個單倍型聚成一個單系群,未形成明顯的譜系結構;除漳河群體的2個單倍型(Hap12和Hap13)位于系統發育樹的基部外,其他群體的單倍型混雜分布。采自雅魯藏布江的1尾個體單倍型定義為Hap5,與大黑河的部分個體同源。基于4個地理群體70尾個體構建的單倍型進化網絡圖(圖3)顯示,Hap7位于中心位置,其他單倍型均由其經歷1~5步突變生成;Hap2為紅堿淖、大黑河和輝騰河群體共享,Hap7為大黑河和輝騰河群體共享,其他單倍型僅為單一群體獨有。
對4個群體的遺傳多樣性水平進行分析,各指數的計算結果見表2。其中,漳河群體檢測到的變異位點最少(1個),紅堿淖群體和大黑河群體最多(7個);漳河群體檢測到的單倍型數目最少(2個),大黑河群體最多(6個)。漳河群體的單倍型多樣性與核苷酸多樣性水平均為最低,分別是0.233和0.00020;輝騰河群體的單倍型多樣性水平最高,為0.791;紅堿淖群體的核苷酸多樣性水平最高,為0.00209。總體的單倍型多樣性為0.883,核苷酸多樣性為0.00344。
2.3"" 群體間遺傳分化
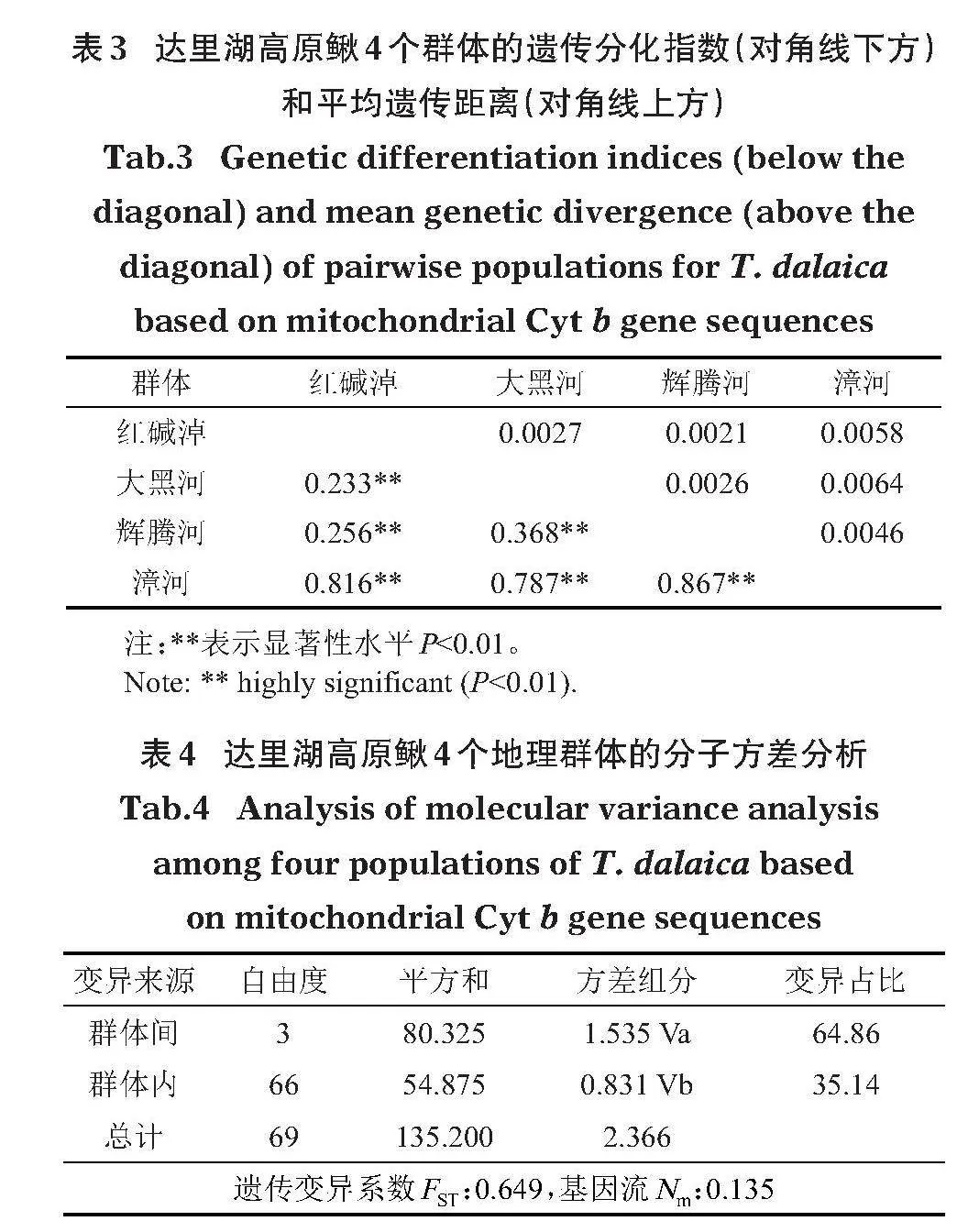
4個地理群體間的遺傳分化指數FST和平均遺傳距離見表3。不同地理群體間遺傳分化指數范圍為0.233~0.867,檢驗結果均達到極顯著水平(Plt;0.01)。其中,紅堿淖和大黑河群體的遺傳分化程度最小,輝騰河和漳河群體的遺傳分化程度最大。4個地理群體間的平均遺傳距離為0.0021~0.0064;其中,紅堿淖和輝騰河群體的遺傳距離最小,大黑河和漳河群體的遺傳距離最大。AMOVA分析(表4)顯示,4個群體的遺傳變異系數FST=0.649,基因流Nm=0.135;在總體遺傳變異中,來源于群體間的變異占比為64.86%,明顯高于群體內(35.14%)。
2.4"" 群體歷史動態
對4個群體及總體進行中性檢驗和錯配分布分析,中性檢驗結果顯示除漳河群體的Tajima's D值為負值外,其他群體及總體Tajima's D及Fu's Fs檢驗值均為正值,且檢驗結果均未達到顯著性水平;錯配分布檢驗結果表明除紅堿淖群體的Hri指數達到顯著性水平外,其他群體的Hri和Ssd指數的檢驗結果大都未達到顯著性水平(表5)。基于核苷酸錯配分布參數繪制總體的觀測值分布與期望值分布的擬合曲線,顯示觀測分布曲線為多峰形(圖4)。
3"" 討論
3.1"" 3個群體曾經或正在經歷遺傳瓶頸
遺傳多樣性是生物多樣性的核心組分之一,其高低通常能夠反映物種或種群對環境的適應能力:越高指示適應環境能力越強,反之亦然(Pinsky amp; Palumbi,2014)。Hd和Pi指數是評估物種或種群遺傳多樣性水平高低的2個重要指標。Grant和Bowen(1998)依據線粒體分子標記的Hd和Pi數值的高低將種群的遺傳多樣性分成4種情形:當二者均處于較低水平(Hdlt;0.5,Pilt;0.005)時,暗示種群近期經歷了遺傳瓶頸;當Hd較高(Hd≥0.5)而Pi較低(Pilt;0.005)時,推測種群可能在經歷衰退之后發生了快速擴張;當Hd較低(Hdlt;0.5)而Pi較高(Pi≥0.005)時,可能由于彼此隔離的種群發生了二次接觸,或大而穩定的群體正經歷強烈的遺傳瓶頸;當二者均處于較高水平(Hd≥0.5,Pi≥0.005)時,指示長期進化或二次接觸形成的大而穩定的種群。
本研究在4個達里湖高原鰍地理群體及總體中,海河流域的漳河群體屬于上述第1種情形,表明漳河群體近期經歷了遺傳瓶頸;黃河流域的紅堿淖和大黑河群體以及總體的遺傳多樣性模式屬于上述的第2種情形,推測種群可能正在經歷種群衰減之后的快速擴張;查干諾爾湖流域的輝騰河群體屬于第4種情形,種群遺傳結構穩定。本研究中涉及的黃河流域和海河流域的3個群體都曾經或正在經歷遺傳瓶頸,這可能是由河湖流量衰減、棲息地喪失等因素導致;其中黃河流域的種群可能由于群體間連通性較好,經歷遺傳瓶頸后不同群體間可以通過鄰近群體間的個體擴散進行基因交流和種群恢復。
3.2"" 群體間中高度遺傳分化
遺傳分化的強弱能夠指示遺傳變異異質性水平的高低,種群間的遺傳分化也被視為物種分化的早期階段。FST是一個反映種群遺傳分化水平的重要參數,常用來表示2個群體間基因流和遺傳漂變的程度。依據Freeland(2012)的劃分標準,除了同屬黃河流域的紅堿淖與大黑河群體之間的FST在0.05~0.25,屬于中度分化,其他配對群體間FST均大于0.25,屬于高度分化。這種群體間高度分化的現象推測與達里湖高原鰍作為定居型魚類、擴散能力弱、生長和繁殖的活動范圍較為局限等因素有關。4個地理群體間的平均遺傳距離均較小,說明基因序列差異不大,這與單倍型在系統發育關系上未分化出明顯的譜系結構相吻合,群體間的分化主要是由單倍型組成及比例的差異所導致。分子方差分析結果表明遺傳變異主要來自于群體間;4個群體間的基因流Nm為0.135,按照Slatkin(1987)的劃分標準,當Nmlt;1時,基因流不足以維持遺傳均質化,這暗示遺傳漂變等因素可能會推動達里湖高原鰍不同地理群體間發生進一步分化。單倍型系統發育樹和進化網絡圖表明,不同群體間的單倍型多為各自獨有,僅有少量單倍型混雜分布于不同群體中;除漳河群體的單倍型位于系統樹基部位置外,其他群體單倍型并未呈現出對應的地理分布格局。
3.3"" 進化歷史上種群規模相對穩定
利用中性檢驗和核苷酸錯配分析探討種群的歷史動態,負的Tajima's D值指示近期群體擴張而產生的豐富而稀有的等位基因;Fu's Fs檢驗是建立在溯祖理論基礎上的中性檢驗,對于經歷過擴張的種群,Fu's Fs也為顯著的負值(俞丹等,2019)。錯配分析中,Hri和Ssd是描述錯配分布曲線波峰的參數:處于平衡種群的指數一般較大,曲線呈多峰分布;當種群擴張時,指數較小,曲線呈單峰泊松分布(Barbosa et al,2013)。本研究中Fu's Fs和Tajima's D均未檢驗到顯著的負值,且錯配分布曲線為明顯的多峰形,不支持達里湖高原鰍群體在歷史上經歷過擴張事件。已有研究表明一些鰍超科魚類,如董氏須鰍(陳浩等,2019)和短尾高原鰍(鞏政等,2022)等,由于受到區域地質運動或近期氣候波動的影響,經歷過遺傳瓶頸和群體擴張。本研究結果顯示達里湖高原鰍在進化歷史上種群規模相對穩定,暗示該物種對冰期事件等氣候波動具有較強的適應能力。
3.4"" 物種保護與管理
遺傳結構和譜系地理格局的研究可為物種保護及管理方案的制定提供科學依據,對于空間上非連續分布的淡水魚類來說尤為重要(周偉等,2017)。作為一種小型定居型魚類,達里湖高原鰍喜歡棲息在湖泊沿岸帶和水流平緩的河段中,本研究發現多數群體和總體呈現出單倍型多樣性高、核苷酸多樣性低的現象,不同群體之間均出現了中高度遺傳分化。因此,在制定物種保護策略時,我們應將各地理群體作為不同保護單元進行管理,關注不同保護單元的棲息地質量,監測其種群規模與遺傳多樣性。
本研究報道的采自雅魯藏布江山南段的1尾達里湖高原鰍樣本,屬于雅魯藏布江外來魚類。通過走訪調查得知,達里湖高原鰍以其斑斕的體色、優良的抗逆性等特點而被開發為養殖品種和觀賞魚類,隨著人工繁殖和苗種培育技術的成功而被運輸擴散至全國各地。例如,Wang等(2016)研究的達里湖高原鰍樣本就來自成都市白家水產市場,其Cyt b基因序列所屬單倍型(GenBank登錄號:KT213590)與本研究中的Hap2同源。推測雅魯藏布江的達里湖高原鰍入侵個體是作為觀賞魚類售賣后經放生等人類活動而進入該江段。因此,建議在非原產地加強對達里湖高原鰍售賣、飼養和放生等環節的管控,防范其進入自然水體引發生態風險。
志謝:感謝西安理工大學朱慶威、李剛、楊子杰、郭善嵩和胡競翔等同仁在采樣過程中提供幫助。
參考文獻
陳浩, 楊銀盆, 張慧, 等, 2019. 三個地理種群的董氏須鰍遺傳多樣性及種群歷史動態[J]. 水生生物學報,43(5):931-938.
鞏政, 劉艷超, 馬聰敏, 等, 2022. 基于線粒體Cyt b基因的雅魯藏布江中游短尾高原鰍遺傳多樣性研究[J]. 淡水漁業,52(4):31-39.
何德奎, 陳詠霞, 陳毅峰, 2006. 高原鰍屬Triplophysa魚類的分子系統發育和生物地理學研究[J]. 自然科學進展,16(11):1395-1404.
武鵬飛, 2017. 達里湖高原鰍鹽堿適應性研究[D]. 上海:上海海洋大學.
楊富億, 文波龍, 李曉宇, 等, 2021. 達里諾爾濕地水環境和魚類多樣性調查Ⅴ. 達里湖的魚類多樣性及其持續健康發展[J]. 濕地科學,19(3):267-276.
俞丹, 張智, 張健, 等, 2019. 基于Cyt b基因的雅魯藏布江下游墨脫江段及察隅河墨脫裂腹魚的遺傳多樣性及種群歷史動態分析[J]. 水生生物學報,43(5):923-930.
周傳江, 胡博, 馬文文, 等, 2020. 河南省高原鰍屬新紀錄種:達里湖高原鰍[J]. 河南師范大學學報(自然科學版),48(4):113-124.
周偉, 高天翔, 王俊, 等, 2017. 烏鱧群體遺傳多樣性和遺傳結構分析[J]. 水產學報,41(10):1521-1532.
朱松泉, 1989. 中國條鰍志[M]. 南京:江蘇科學技術出版社.
Barbosa A M, Real R, Mu?oz A R, et al, 2013. New measures for assessing model equilibrium and prediction mismatch in species distribution models[J]. Diversity and Distributions,19(10):1333-1338.
Edgar R C, 2004. MUSCLE: multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Research,32(5):1792-1797.
Excoffier L, Lischer H E L, 2010. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources,10(3):564-567.
Feng C G, Wu Y J, Tian F, et al, 2017. Elevational diversity gradients of Tibetan loaches: the relative roles of ecological and evolutionary processes[J]. Ecology and Evolution,23(7):9970-9977.
Feng C G, Zhou W W, Tang Y T, et al, 2019. Molecular systematics of the Triplophysa robusta (Cobitoidea) complex: extensive gene flow in a depauperate lineage[J]. Molecular Phylogenetics and Evolution,132(2):275-283.
Freeland J R, 2012. Molecular ecology[M]. 2nd Edition. Chichester: John Wiley & Sons.
Fricke R, Eschmeyer W N, Van Der Laan R, 2022. Eschmeyer's Catalog Of Fishes: Genera, Species, References[DB/OL]. [2022-06-05]. http://researcharchive.calacademy.org/research/ichthyology/catalog/fishcatmain.asp.
Grant W, Bowen B W, 1998. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity,89(5):415-426.
Kumar S, Stecher G, Tamura K, 2016. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution,33(7):1870-1874.
Leigh J W, Bryant D, 2015. Popart: full-feature software for haplotype network construction[J]. Methods in Ecology and Evolution,6(9):1110-1116.
Pinsky M L, Palumbi S R, 2014. Meta-analysis reveals lower genetic diversity in overfished populations[J]. Molecular Ecology,23(1):29-39.
Ronquist F, Teslenko M, van der Mark P, et al, 2012. MrBayes 3.2: efficient Bayesian phylogenetic inference and model choice across a large model space[J]. Systematic Biology,61(3):539-542.
Rozas J, Ferrer-Mata A, Sánchez-DelBarrio J C, et al, 2017. DnaSP 6: DNA sequence polymorphism analysis of large data sets[J]. Molecular Biology and Evolution,34(12):3299-3302.
Slatkin M, 1987. Gene flow and the geographic structure of natural populations[J]. Science,236(4803):787-792.
Tang Q Y, Getahun A, Liu H Z, 2009. Multiple in-to-Africa dispersals of labeonin fishes (Teleostei: Cyprinidae) revealed by molecular phylogenetic analysis[J]. Hydrobiologia,632:261-271.
Wang Y, Shen Y J, Feng C G, et al, 2016. Mitogenomic perspectives on the origin of Tibetan loaches and their adaptation to high altitude[J]. Scientific Reports,6:29690.
Wang Y, Yang L, Wu B, et al, 2015. Transcriptome analysis of the plateau fish (Triplophysa dalaica): Implications for adaptation to hypoxia in fishes[J]. Gene,565(2):211-220.
Wu H H, Gu Q H, Zhou C J, et al, 2020. Molecular phylogeny and biogeography of Triplophysa stone loaches in the central Chinese mountains[J]. Biological Journal of the Linnean Society,130(3):563-577.
Zhang D, Gao F L, Jakovli? I, et al, 2020. PhyloSuite: an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies[J]. Molecular Ecology Resources,20(1):348-355.
Zhou C J, Hu B, Tang Y T, et al, 2021. The chromosome-level genome of Triplophysa dalaica (Cypriniformes: Cobitidae) provides insights into its survival in extremely alkaline environment[J]. Genome Biology and Evolution,13(8):evab153.
(責任編輯"" 熊美華)
Genetic Diversity and" Genetic Differentiation of Different Geographic
Populations of Triplophysa dalaica
GONG Zheng1,2, WANG Zi‐xuan1, ZHANG Yi‐chi3, ZHAO Geng‐nan3, PAN Bao‐zhu3
(1. College of Life Sciences, Zaozhuang University, Zaozhuang 277160, P. R. China;
2. Key Laboratory of Ecological Impacts of Hydraulic-Projects and Restoration of
Aquatic Ecosystem of Ministry of Water Resources, Institute of Hydroecology,
Ministry of Water Resources and Chinese Academy of Sciences, Wuhan 430079, P. R. China;
3. State Key Laboratory of Eco-hydraulics in Northwest Arid Region of China,
Xi’an University of Technology, Xi’an 710048, P. R. China)
Abstract:Triplophysa dalaica, a small loach in the Cobitidae family, primarily inhabits the mainstem and tributaries below the Lanzhou section of the Yellow River and some inland water bodies in central and eastern Inner Mongolia. To date, only a few studies have been carried out on the population genetics of T. dalaica. Thus, examination of the genetic diversity, genetic differences among geographically separated populations, and the population history of T. dalaica is needed for species conservation and management. In this study, we explored the genetic diversity and genetic differentiation of four geographic T. dalaica populations (Hongjiannao and Dahei Lakes in the Yellow River basin, Huiteng Lake in the Chagannuoer Lake basin and Zhanghe River in the Haihe Lake basin) by analyzing the mitochondrial Cyt b gene sequences of 70 specimens from the four locations. A total of 16 polymorphic loci and 13 haplotypes were identified in the four populations. The haplotype diversity index (Hd) and nucleotide diversity index (Pi) of the four populations ranged from 0.233 to 0.791 and from 0.00020 to 0.00209, respectively. The genetic diversity of the T. dalaica populations in Huiteng River was the highest. Although the haplotype diversity of the Hongjiannao and Dahei Lake populations was higher, the nucleotide diversity was lower. The haplotype diversity and nucleotide diversity of the Zhanghe population were both lower than those in the other two basins. The phylogenetic tree and evolutionary network of the 13 haplotypes did not cluster into a distinct clade structure, and most haplotypes were unique to a single population. Except for two basal haplotypes in the Zhanghe population, the haplotypes were interconnected in a mixed manner, rather than following their respective geographical distribution patterns. Genetic differentiation analysis shows that the range of genetic differentiation index (Fst) among populations was 0.233 to 0.867, indicating high degrees of differentiation. Analysis of molecular variance (AMOVA) demonstrates that a high proportion of the total genetic variance was attributable to variations between populations (64.86%). The population history shows no recent population expansion of T. dalaica within the various geographical groups or in the overall population. During an ichthyological survey, a" specimen of T. dalaica was collected in the Shannan reach of the Yarlung Tsangpo River and recorded as an alien species. In conclusion, the genetic diversity of T. dalaica varied in geographically separated populations, and the genetic differentiation among the populations were generally very high. Based on these findings, we recommend that the different populations of T. dalaica be treated as separate protection units, with monitoring of habitat quality, and that regulation of species trade and release should be strengthened.
Key words:Triplophysa dalaica; genetic diversity; population differentiation; demographic history dynamics

