我國圈養和野生東北虎種群線粒體基因組遺傳多樣性的比較研究


摘要 東北虎(Panthera tigris altaica)是體型最大的貓科(Felidae)動物之一,是極具代表性的珍稀野生動物。現有研究表明,我國野生東北虎種群遺傳多樣性較低、近交水平較高。盡管目前我國野生東北虎的數量在逐步增長,但通過人工干預來提高野生東北虎的遺傳多樣性會更利于其數量的恢復,通過野化放歸進行遺傳拯救是一種關鍵策略,但實施遺傳拯救之前,必須確定圈養個體與現存野生個體間的遺傳關系。采用糞便DNA的高通量測序數據組裝了51只橫道河子圈養東北虎和13只完達山、老爺嶺等地的野生東北虎的線粒體基因組,分析兩者之間的關系,評估線粒體基因組的遺傳多樣性。結果表明:圈養東北虎的遺傳多樣性高于野生種群,所有遺傳變異均為無害。部分圈養個體與野生種群同屬一個進化支,且具有野生種群所不包含的遺傳變異,可用于實施遺傳拯救。此外,圈養種群存在顯著的遺傳分化,一個與當前野生種群關系很遠的分支可能代表未知的地理種群,因此,建議對該遠緣分支開展野外來源的追溯,確定其譜系地理學地位和保護價值,使其成為恢復野外歷史遺傳多樣性的后備資源。
關鍵詞:東北虎;遺傳拯救;線粒體基因組;糞便DNA;遺傳多樣性
中圖分類號:Q953 文獻標識碼:A 文章編號:2310 - 1490(2024)- 02 - 0231 - 11
DOI:10.12375/ysdwxb.20240201
東北虎(Panthera tigris altaica)在維持東北亞森林生物系統的穩定性和完整性中扮演著重要角色。人口的增長和人類活動的日益增強、擴展,導致野生東北虎的棲息地遭到嚴重破壞[1],使之持續縮小和破碎化[2],嚴重影響了野生東北虎的生存。此外,氣候變化和環境污染等直接或間接造成的獵物數量下降,也給東北虎的生存帶來了很大挑戰[3]。因此,東北虎與其他虎亞種一起在國際自然保護聯盟紅色名錄(IUCN Red List)中被列為瀕危級(EN),同時被瀕危野生動植物種國際貿易公約(CITES)列為附錄Ⅰ物種。
目前我國境內野生東北虎包括4個相互隔離的小種群[4],各個小種群之間缺乏可用的生態廊道,無法進行自然的基因交流,發生了較為嚴重的近交,導致野生東北虎種群遺傳多樣性較低[5]。遺傳多樣性對于野生動物適應環境、抵御疾病等都具有極為重要的意義[6?7]。當小種群無法自我維持遺傳多樣性時,就需要通過人工輔助引入外源基因來提高遺傳多樣性,這被稱為遺傳拯救[8]。目前野生東北虎的處境需要開展人工輔助基因交流:一方面,通過建設生態廊道使不同種群之間得以自然交流[9];另一方面,將人工繁育的東北虎野化放歸,參與野生種群的繁殖,達到提升遺傳多樣性的目的[10]。野化放歸的策略首先要基于充分的遺傳學分析,從圈養種群中選擇與野生種群同屬一個進化分支且能提高野生種群遺傳多樣性的個體,進行野化訓練并放歸。在提高遺傳多樣性的同時應當權衡遺傳拯救的收益和風險,既要避免遠交衰退的產生還要避免有害基因的引入。本研究所考察的圈養種群來自國家東北虎種源基地橫道河子貓科動物飼養繁育中心,該基地先后從多國引進東北虎種源,目前已飼養超過1 000只東北虎,這一龐大的數量足以支撐野化放歸項目。本研究利用東北虎糞便提取DNA,并通過高通量測序獲取東北虎線粒體全基因組,用于圈養種群與野生種群遺傳多樣性評估和遺傳關系的比較,從母系遺傳層面為圈養東北虎野化放歸個體的遴選提供支持。
1 材料與方法
1. 1 糞便采集與DNA 提取
共采集64份東北虎糞便樣品,包括來自中國橫道河子貓科動物飼養繁育中心的51只圈養東北虎和13只完達山、老爺嶺等地的野生東北虎。13個野生東北虎的糞便樣本分別取自吉林省琿春市(8個)、黑龍江省鶴崗市蘿北縣(2 個)、吉林省敦化市(1 個)、吉林省舒蘭市(1 個)和黑龍江省東寧市(1個)[11]。野生東北虎的糞便樣本均通過擴增線粒體的一個271 bp的細胞色素b 基因片段進行物種確認;并選擇18個微衛星標記進行基因分析和個體鑒定[5]。采用本團隊創建的糞便DNA富集方法(periextractionenrichment using SDS,PEERS,專利號:CN113186185A)對糞便進行預處理,通過向糞便溶解液中加入終濃度為5% 的SDS,破壞東北虎的細胞,同時保持微生物的完整性,從而優先釋放東北虎的DNA。離心后采用Axygen Multisource GenomicDNA(Axygen,美國)試劑盒提取上清液DNA,即得到富集的東北虎總DNA。
1. 2 DNA 質檢
獲得的糞便DNA中只有宿主DNA占比足夠高才適合進行高通量測序。因此,分別以東北虎核基因GAPDH(nuDNA,NCBI序列號:NC056666. 1)和線粒體基因Cyt b(mtDNA,NCBI序列號:KF297576. 1)為模板,設計定量PCR引物,用于測定總DNA中二者的拷貝數;同時在細菌的16S rRNA 基因上設計1 對通用引物,用于測定細菌DNA(bDNA)的拷貝數。當mtDNA 與bDNA 的CT 值差值小于3、bDNA的CT值大于10、nuDNA的CT值小于25時用于高通量測序(表1)。
熒光定量PCR體系10. 0 μL:1. 0 μL DNA提取液,5. 0 μL TB Green Premix ExTaq Ⅱ酶(TaKaRa,大連),濃度為10 μmol/L 的上、下游引物各0. 4 μL和ddH2O 3. 2 μL。三對引物采用相同的反應程序:95 ℃預變性30 s;95 ℃變性5 s,58 ℃退火34 s,72 ℃延伸30 s,40個循環;95 ℃變性10 s+65 ℃退火5 s+95 ℃延伸15 s。利用9700 型PCR 擴增儀(Gene?Amp,美國)進行擴增反應。
1. 3 高通量測序及數據處理
合格的DNA在MGISEQ-2000測序平臺(華大智造,中國)進行高通量測序。下機數據使用FastQC進行可視化質量查驗;使用Trimmomatic去除低質量序列和接頭;用BWA(Burrow-Wheeler Aligner)完成線粒體數據的比對;用SAMtools 將測序數據中的read排序整齊,并用GATK去除重復序列,計算測序覆蓋度與測序深度。
1. 4 線粒體組裝與遺傳多樣性分析
使用NOVOPlasty 將整理后的序列組裝成完整的mtDNA基因組。組裝好的mtDNA用MAFFT進行序列比對,提取線粒體的D-loop區、2個編碼核糖體核酸的基因(12S rRNA和16S rRNA)和13個編碼蛋白質的基因,用于不同區域的分析。使用DnaSP6計算核苷酸多樣性(Pi )與單倍型多樣性(Hd),并使用POPART繪制單倍型網絡。用VCFtools計算遺傳分化系數(FST),基于最大似然法(ML)構建的進化樹和遺傳距離的計算由MEGA 11完成。
1. 5 注釋突變位點
選用東北虎線粒體全基因組(NCBI 序列號:KF297576. 1)作為參考序列,使用GATK完成SNP位點的檢測和篩選,檢測得到的SNP 位點使用軟件ANNOVAR 進行注釋和分析。在ANNOVAR 中的grantham. matrix 矩陣下,對所有突變進行Grantham評分,評估突變是否有害。評估主要基于氨基酸的物理/化學組成,一般認為,Grantham評分≥150的突變是有害的,評分lt;150的突變是良性的。
2 結果
2. 1 糞便DNA 質量與測序質量評估
所有糞便DNA 的質量濃度都分布在12. 5~47. 0 ng/μL,均滿足高通量測序要求。測序數據中質量達到Q20的堿基在95%以上,且堿基質量平均值波動較小,表明測序質量足夠高。堿基分離程度均勻,序列中GC含量的實際分布與理論分布接近。平均測序覆蓋度均為100%,表明數據質量可靠。
2. 2 組裝線粒體基因組
所有個體的mtDNA均被完整組裝,其中D-loop區中POLYC片段的長度在個體間變化較大,具體數目無法通過read比對來確定,屬于測量不精準區域,故取所有個體中最短的長度作為POLYC片段的長度,因而D-loop區的長度為1 180 bp,線粒體基因總長度為16 629 bp。其他各個基因區段均未見插入和缺失的堿基(表2)。
2. 3 核苷酸多樣性與單倍型多樣性
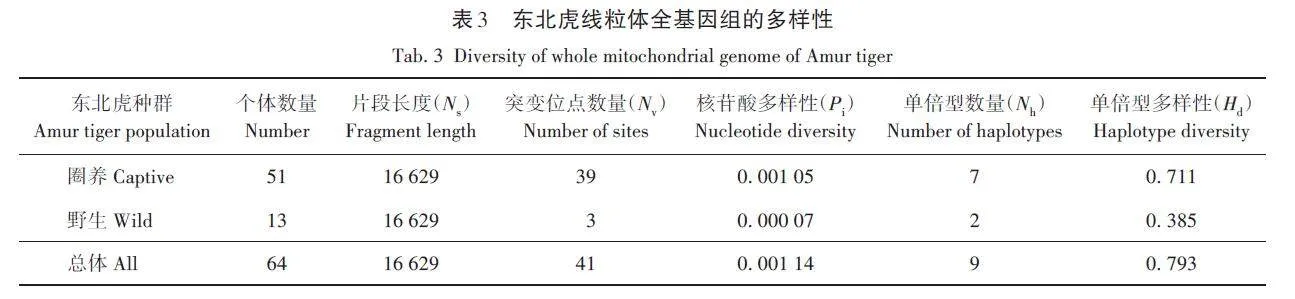
在64只東北虎的線粒體基因組中共鑒定到41個多態位點和9種單倍型。圈養東北虎的多態位點為39個,其中單次突變位點1個,簡約信息位點38個。野生東北虎的多態位點為3個,全部是簡約信息位點。圈養和野生東北虎單倍型數量分別為7、2種,二者之間不存在共享單倍型。圈養種群和野生種群的單倍型多樣性分別為0. 711和0. 385,核苷酸多樣性分別為0. 001 05和0. 000 07。對于所有樣本,單倍型多樣性為0. 793,核苷酸多樣性為0. 001 14。總體而言,圈養種群的核苷酸多樣性和單倍型多樣性要遠高于野生種群的多樣性(表3)。
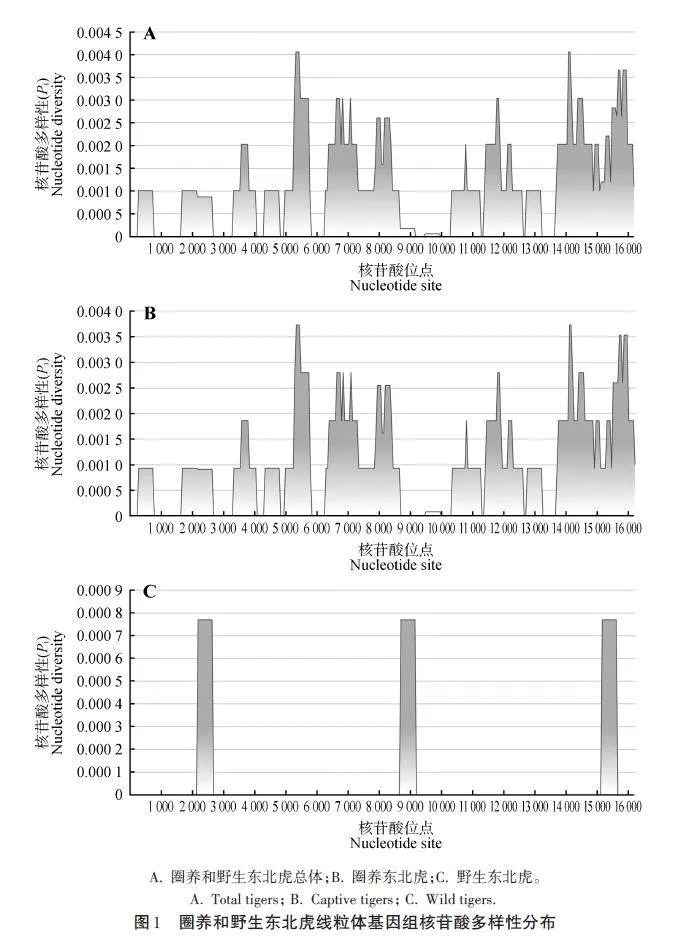
基于線粒體全基因序列,分別繪制圈養個體、野生個體和全部個體的核苷酸多樣性分布圖(圖1)。總體來看,線粒體基因組上核苷酸多樣性的分布以圈養種群為主導(圖1A);圈養種群的核苷酸多樣性(圖1B)要遠高于野生種群(圖1C),但在不同區域間,二者核苷酸多樣性差異較大,圈養虎在堿基序號4 900~8 600、10 300~13 200和13 600~16 000三個區域較高(圖1B),而野生虎主要表現在2 100~2 500、8 700~9 100和15 100~15 500三個區域(圖1C)。
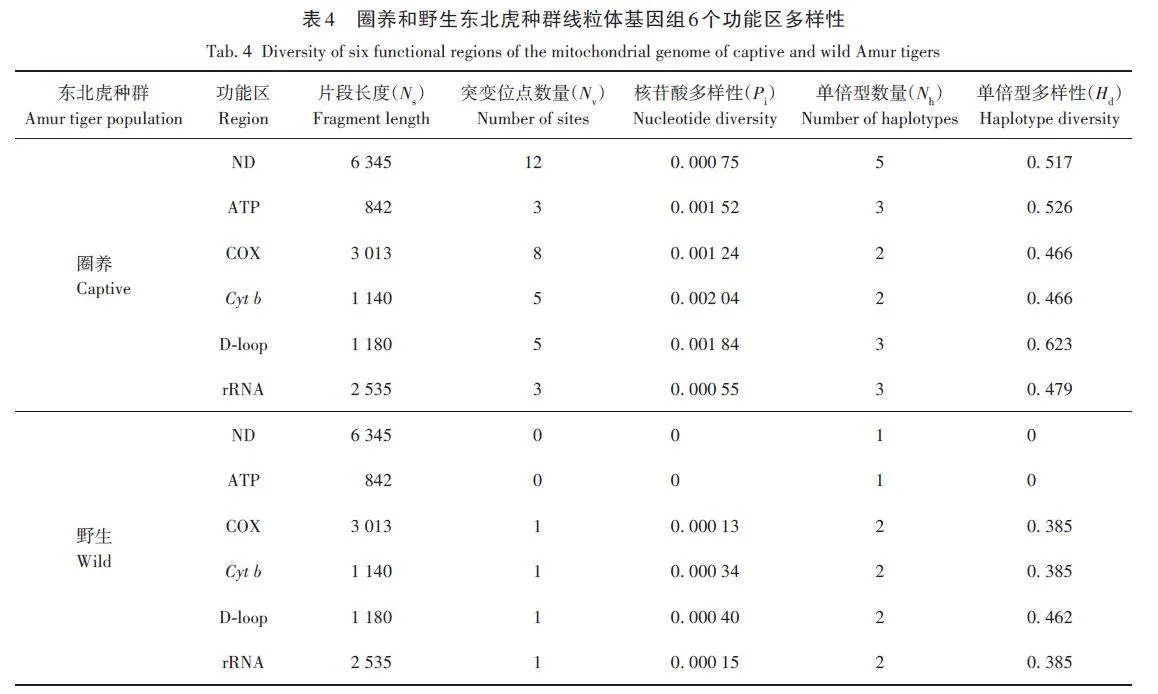
為了進一步分析線粒體基因組上不同功能區域的多樣性水平,將線粒體基因組分成6個功能區:ND編碼區、ATP編碼區、COX編碼區、Cyt b 編碼區、D-loop 區和rRNA 編碼區。各個區域的長度為其所包含的基因長度之和,其中ND4L 基因與ND4 基因存在7 個重疊堿基,ND5 基因與ND6 基因存在17個重疊堿基,ATP8 基因與ATP6 基因存在43個重疊堿基,故ND區與ATP區的長度分別為6 345 bp與842 bp。
圈養種群的Cyt b 基因具有最高的核苷酸多樣性(0. 002 04);rRNA 編碼區的核苷酸多樣性最低(0. 000 55);D-loop 區的單倍型多樣性最高,達到0. 623;編碼細胞色素C氧化酶的COX區和Cyt b 基因的單倍型多樣性最低,均為0. 466。野生種群中D-loop 控制區的核苷酸多樣性和單倍型多樣性最高,分別為0. 000 40 和0. 462;而編碼NADH 脫氫酶的ND區與編碼ATP合成酶的ATP區核苷酸多樣性和單倍型多樣性均為0,表現出極低的多樣性(表4)。
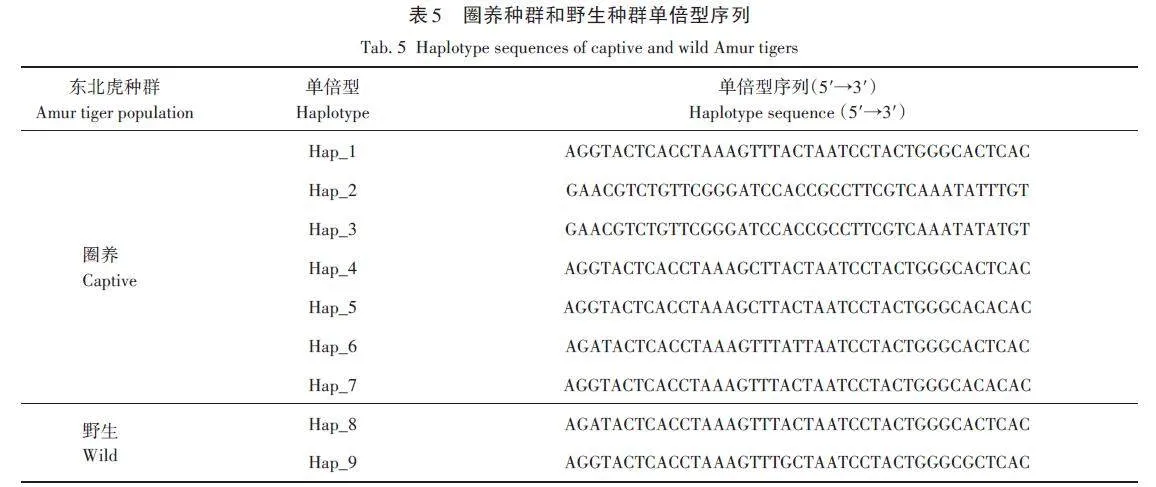
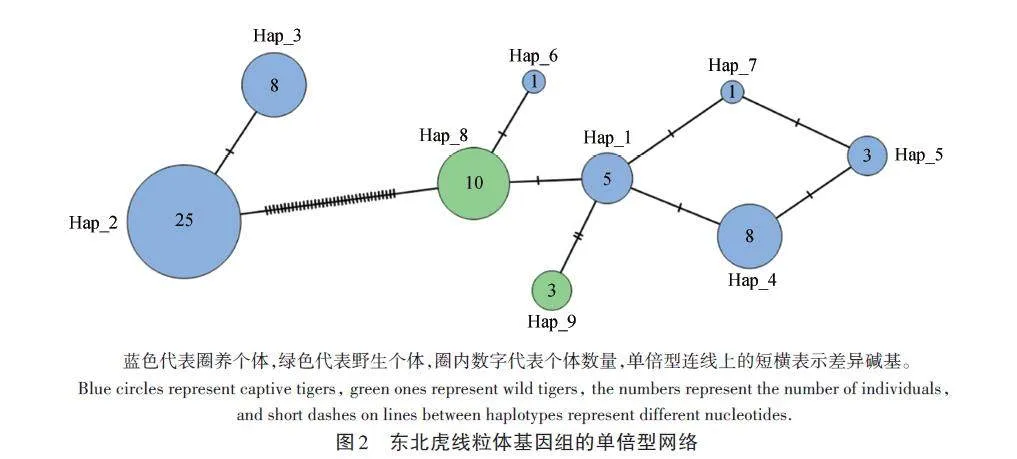
圈養種群和野生種群共檢測到9種單倍型(表5)。基于單倍型序列繪制的單倍型網絡顯示,東北虎的單倍型可以分為兩個大組,一組由Hap_2 和Hap_3組成,另一組由其他單倍型組成(圖2)。兩組之間的核苷酸差異遠大于組內,遺傳分化系數(FST)為0. 061。野生種群的單倍型全部包含在第2組中,且與圈養種群單倍型的差異只有1或2個核苷酸。單倍型的分化主要發生在圈養種群中。
2. 4 野生和圈養東北虎的母系遺傳關系
9種單倍型之間的Nei氏遺傳距離在0. 000 06~0. 002 35,平均為0. 000 76。其中,Hap_3 與Hap_9的遺傳距離最大,為0. 002 35;Hap_1 與Hap_4、Hap_7、Hap_8,Hap_2 與Hap_3,Hap_5 與Hap_4、Hap_7,Hap_6與Hap_8之間的遺傳距離最小,均為0. 000 06。圈養種群與野生種群單倍型Hap_8遺傳距離最近的為單倍型Hap_1和Hap_6,最遠的單倍型為Hap_3;與野生單倍型Hap_9遺傳距離最近的為Hap_1,最遠的為Hap_3(表6)。
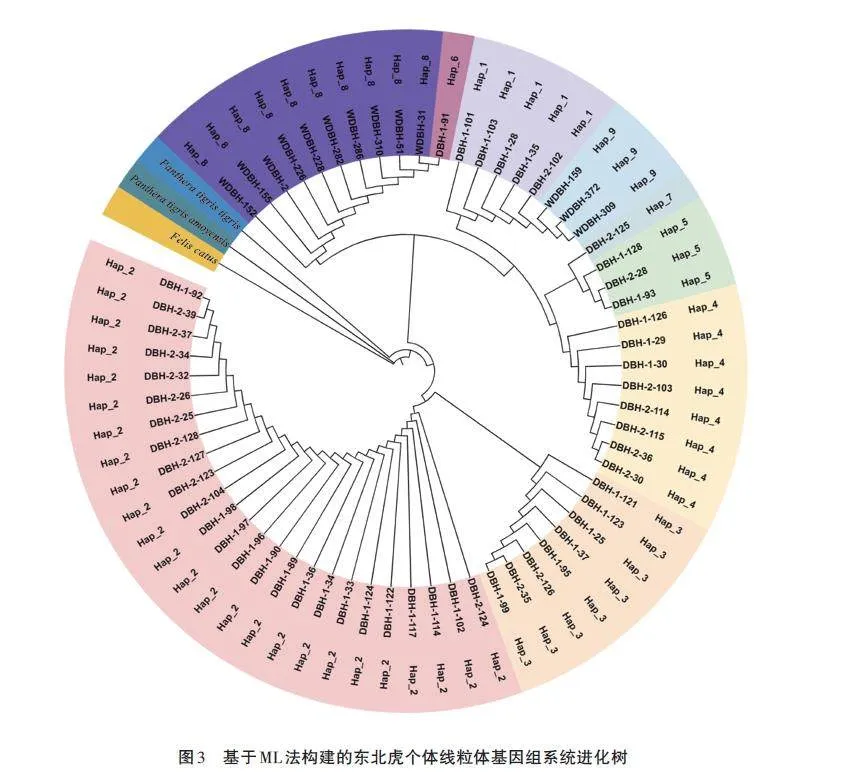
以家貓(Felis catus)、華南虎(Panthera tigrisamoyensis)、虎指名亞種(P. t. tigris)為外群,在東北虎個體的線粒體全基因構建的ML進化樹上,外群均位于東北虎的外側,說明該系統進化樹的結構正確。東北虎分為兩個大的分支,其中攜帶Hap_2與Hap_3的個體聚為第1個分支,其他個體聚為第2個分支。在第2個分支中,攜帶Hap_8的野生個體與攜帶Hap_6的圈養個體聚為一個小支,攜帶Hap_9的野生個體與攜帶Hap_1的圈養個體聚為一個小支(圖3)。這表明所研究的野生個體與圈養個體之間存在很近的進化關系,這一進化關系完全印證了單倍型網絡與遺傳距離計算的結果。
2. 5 線粒體基因組突變的有害性
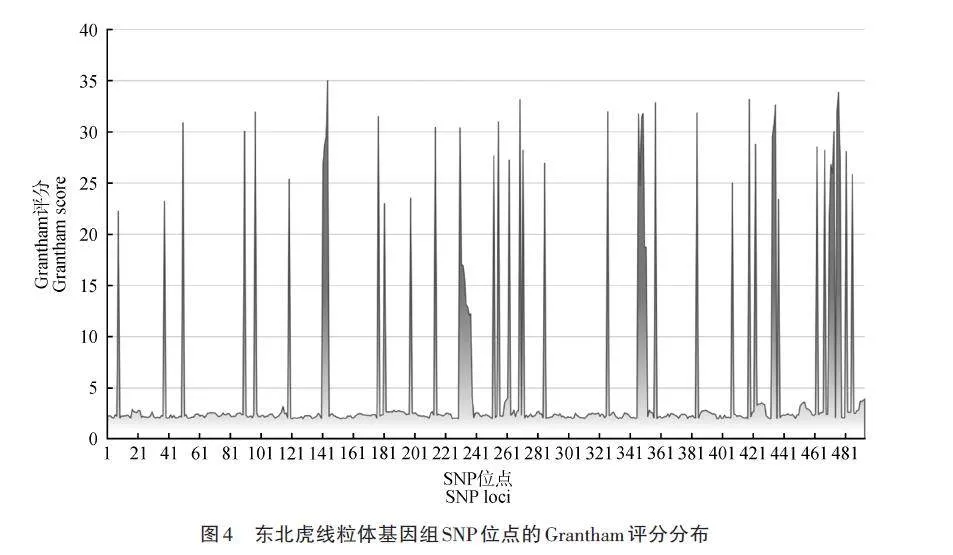
以東北虎線粒體基因組(NCBI序列號:KF297576. 1)為參照,64 只個體共篩選得到493 個SNP 位點。493 個SNP 位點的Grantham 評分最大值為34. 67,最小值為2. 00,意味著所有突變均為非有害突變( 圖4)。
3 討論
3. 1 糞便DNA 在線粒體基因組重測序中的可用性
線粒體DNA可以反映母系的遺傳變異[12],由于其具有高拷貝、高突變率的特點[13],被廣泛應用于系統發育和地理學[14]、群體遺傳學[15]及醫學研究[16]。糞便是較易獲得的非損傷性材料,可從中獲得DNA用于分子生態學和遺傳學分析[17?18]。但是,糞便中含有極其豐富的微生物和少量的宿主腸上皮細胞[19],其DNA的質量和可用性受到宿主消化能力、攝入食物、DNA 降解程度和糞便采集時機等影響[20?21],因此從糞便中獲取高質量的宿主DNA始終是個技術難點,好在糞便DNA中的mtDNA拷貝數高于核DNA,通過高通量測序數據更容易實現基因組的組裝[22]。本研究進行了有益的嘗試,從所有樣品的糞便DNA測序數據中成功組裝了完整的線粒體基因組,且平均覆蓋度均達到100%,達到了血液等高通量材料組裝的效果[23]。
日后可憑借本試驗的高通量測序數據以及組裝成功的完整線粒體基因組開展后續分析,這相較于傳統的局部片段或某一基因的分析更加全面、信息量更大[23?24]。本研究的DNA提取、質檢、測序和線粒體基因組的組裝方法等可為將來其他物種基于糞便的研究提供一定經驗。
3. 2 圈養和野生東北虎的線粒體基因組多樣性
我國動物園飼養東北虎的歷史可以追溯到新中國成立初期,如1952—1959年,哈爾濱動物園共收幼虎99只,平均每年12只[25]。彼時我國的東北虎數量較多,圈養種群的初始建群者來源廣泛,遺傳多樣性較高[25]。本研究所考察的橫道河子貓科動物飼養繁育中心的種群來源廣泛,先后從多個國家及國內動物園引進種源,因此推測這一種群攜帶的遺傳多樣性較高。Ning等[5]的研究表明,當前的東北虎野生種群已經處于中等近交水平,局部近交和遺傳漂變使其原有的遺傳多樣性在一定程度上呈現下降趨勢。
除此之外,還有研究表明,野生東北虎種群的遺傳多樣性較低。2003 年,Russello 等[26]在俄羅斯遠東地區對來自至少27只個體的82個野生東北虎糞便樣本進行分析,結果表明,野生東北虎線粒體控制區的單倍型多樣性水平極低,82個糞便樣本僅共享3種單倍型,其中最廣泛的一個單倍型占所有樣本的96. 4%,其余兩種單倍型僅占所有樣本的3. 6%。與之前Cracraft等[27]報道的14只圈養東北虎共享的4種mtDNA細胞色素b 基因單倍型相比,在一個突變率相對較低的基因區域中有著更高水平的單倍型多樣性,表明圈養個體可能比野生個體存在更大數量的遺傳變異。除東北虎之外,在其他物種的研究中也存在類似結果,如Zhu等[23]在對圈養與野生大熊貓(Ailuropoda melanoleuca)線粒體基因組編碼區的比較中發現,圈養大熊貓的遺傳多樣性高于野生大熊貓的遺傳多樣性。
本研究針對圈養與野生東北虎線粒體基因組的比較得到了類似結果,并驗證了本研究的推測,圈養東北虎的遺傳多樣性高于野生東北虎的遺傳多樣性,具有更高的遺傳變異水平。將線粒體基因組分區段考察,每個區段上野生種群的多樣性水平也都顯著低于圈養種群。這一結果雖然不能完全排除樣本量的影響(野生種群只有13只個體,而圈養種群有51只個體),但是與Ning等[5]的評估結果部分相符。也就是說,從線粒體全基因組看,圈養種群的遺傳多樣性要高于完達山與老爺嶺等地的野生東北虎的遺傳多樣性。相較于傳統的對線粒體基因組上某一基因或片段的研究,本研究放眼于整個東北虎線粒體基因組,并針對不同區域進行分段比較,從全局水平上對東北虎線粒體遺傳多樣性進行評估,使遺傳多樣性的評價更為全面、精準。
值得關注的是,在其他保護動物的研究中[28]也發現了相似結果。這可能是圈養種群的初始群體來自不同地理區域或不同親本個體。并且,圈養種群的繁殖受人工管理控制,以最大程度維持種群的遺傳多樣性水平。此外,圈養種群相對于野生種群來說,通常處于更加安全、穩定的環境中,圈養動物基本不會面臨野外的自然選擇壓力、環境威脅或食物競爭等不利生存的困境,從而使圈養種群有更多的個體生存下來并繁殖后代。這樣一來,圈養種群基因池中的各種變異就會有更大的機會傳遞給下一代。但需要注意的是,這些傳遞到下一代的遺傳變異可能不都是有利的,因此在提高種群遺傳多樣性的同時應該注意有害基因的引入。
線粒體基因組除了D-loop區以外,所有的編碼區都有其重要功能,有害突變往往影響其多方面的功能[29]。線粒體不僅被認為是能量的供應者,而且在與衰老相關的疾病發展中發揮著重要作用[30?31],當線粒體出現嚴重衰退時,會導致個體適合度降低而使個體甚至種群被自然界淘汰。研究顯示,虎在野生和圈養種群中都會受到有害突變的清除作用(purging)的影響,如虎指名亞種野生種群[32]和人工飼養的華南虎種群[33]。為了檢驗線粒體基因組的質量,本研究掃描了全基因組,在圈養和野生東北虎種群中均未發現明顯的有害突變,表明雖然線粒體基因組的遺傳多樣性豐富度不同,但都是安全的。
3. 3 圈養和野生東北虎的關系
本研究從圈養和野生東北虎種群的樣本中得到9種單倍型,其中圈養種群7種,野生種群2種,二者之間沒有共享單倍型。這說明圈養東北虎種群遺傳多樣性要高于野生東北虎種群,而從進化樹與單倍型網絡中可以看出,圈養種群的部分個體與野生東北虎屬于同一進化分支,具有較近的遺傳關系。因此,在利用該圈養種群部分個體實施遺傳拯救時,可以在增加遺傳多樣性的同時避免產生遠交衰退。
本研究的野生個體為隨機采集,而圈養個體主要是育齡虎,是該種源基地的核心群體。統計結果顯示,Hap_2和Hap_3分支對應的個體占全部圈養樣本的64. 7%,是圈養種群線粒體遺傳多樣性的主要組成部分。但是,它們與當前的野生種群距離甚遠,說明其可能來自當前分布區以外的某個地區的滅絕種群。但該分支具體代表哪個地理種群尚不明確,日后可將該樣本與中國、俄羅斯野外所有種群進行遺傳多樣性綜合分析,追溯其根源[34]。
4 結論與建議
在本研究現有樣本條件下,中國橫道河子貓科動物飼養繁育中心圈養種群線粒體基因組的遺傳多樣性較高,且所有的遺傳變異均無害。該圈養種群的部分個體與野生種群遺傳關系較近,遺傳多樣性高度互補,可用來實施野生種群的遺傳拯救,消除近交影響。應對圈養種群中的一些遠緣分支及時開展野外來源追溯,確定其譜系地理學地位和保護價值,使其成為恢復野外歷史遺傳多樣性的后備資源。
參考文獻:
[1] 馬建章, 張明海, 姜廣順, 等. 我國老虎及其棲息地保護面臨的挑戰與對策[J]. 野生動物學報, 2015, 36(2): 129-133.
MA J Z, ZHANG M H, JIANG G S, et al. Challenges and strate?gies for conservation of tigers and their habitats in China[J]. Chi?nese Journal of Wildlife, 2015, 36(2): 129-133.
[2] 龍澤旭. 東北虎棲息地變化、核心棲息地與廊道識別研究[D]. 哈爾濱:東北林業大學, 2021.
LONG Z X. Identification of habitat change, core habitats and cor?ridors for the Amur tiger[D]. Harbin:Northeast Forestry Univer?sity, 2021.
[3] 顧佳音. 中國東北虎種群現狀、冬季獵物選擇及運動特征研究[D]. 哈爾濱:東北林業大學, 2017.
GU J Y. The status of current population and distribution, winterprey selection and movement characteristics of Amur tigers (Pan?thera tigris altaica) in China[D]. Harbin:Northeast Forestry Uni?versity, 2017.
[4] QI J Z, GU J Y, NING Y, et al. Integrated assessments call forestablishing a sustainable meta-population of Amur tigers in north?east Asia[J]. Biological Conservation, 2021, 261: 109250.
[5] NING Y, ROBERTS N J, QI J, et al. Inbreeding status and impli?cations for Amur tigers[J]. Animal Conservation, 2022, 25(4):521-531.
[6] BIJLSMA R, BUNDGAARD J, BOEREMA A C. Does inbreed?ing affect the extinction risk of small populations?: predictionsfrom Drosophila[J]. Journal of Evolutionary Biology, 2000, 13(3): 502-514.
[7] XUE Y L, PRADO-MARTINEZ J, SUDMANT P H, et al. Moun?tain gorilla genomes reveal the impact of long-term population de?cline and inbreeding[J]. Science, 2015, 348(6231): 242-245.
[8] HEDRICK P W, ADAMS J R, VUCETICH J A. Reevaluatingand broadening the definition of genetic rescue[J]. ConservationBiology, 2011, 25(6): 1069-1070.
[9] 張明海,靳勇超,姜廣順. 中國東北虎自然保護地生態廊道空間規劃[J]. 自然保護地,2021,1(3):1-8.
ZHANG M H, JIN Y C, JIANG G S. Spatial planning of ecologi?cal corridors among the protected areas of Amur tiger in China[J]. Natural Protected Areas, 2021, 1(3): 1-8.
[10] BUCKLAND S, COLE N C, GROOMBRIDGE J J, et al. Highrisks of losing genetic diversity in an endemic Mauritian gecko:implications for conservation[J]. PLoS One, 2014, 9(6):e93387.
[11] LAN T M, LI H M, ZHANG L, et al. Population genomics revealsextensive inbreeding and purging of mutational load in wild Amurtigers[J]. bioRxiv, 2023. DOI: 10. 1101/2023. 05. 09. 539923.
[12] PERRY G H, MARIONI J C, MELSTED P, et al. Genomicscalecapture and sequencing of endogenous DNA from feces[J].Molecular Ecology, 2010, 19(24): 5332-5344.
[13] BROWN W M, GEORGE M, Jr, WILSON A C. Rapid evolu?tion of animal mitochondrial DNA[J]. Proceedings of the Na?tional Academy of Sciences of the United States of America,1979, 76(4): 1967-1971.
[14] MUELLER R L, MACEY J R, JAEKEL M, et al. Morphologi?cal homoplasy, life history evolution, and historical biogeogra?phy of plethodontid salamanders inferred from complete mito?chondrial genomes[J]. Proceedings of the National Academy ofSciences of the United States of America, 2004, 101(38):13820-13825.
[15] LIN L H, JI X, DIONG C H, et al. Phylogeography and popula?tion structure of the Reevese’s butterfly lizard (Leiolepis reevesii)inferred from mitochondrial DNA sequences[J]. Molecular Phy?logenetics and Evolution, 2010, 56(2): 601-607.
[16] INAK G, LORENZ C, LISOWSKI P, et al. Concise review: in?duced pluripotent stem cell-based drug discovery for mitochon?drial disease[J]. Stem Cells, 2017, 35(7): 1655-1662.
[17] DEDA O, CHATZIIOANNOU A C, FASOULA S, et al. Samplepreparation optimization in fecal metabolic profiling[J]. Journalof Chromatography B, 2017, 1047: 115-123.
[18] WANG B S, WANG Z L, TIAN J D, et al. Establishment of amicrosatellite set for noninvasive paternity testing in free-rangingMacaca mulatta tcheliensis in Mount Taihangshan area, Jiyuan,China[J]. Zoological Studies, 2015, 54: e8.
[19] VINCENT C, MEHROTRA S, LOO V G, et al. Excretion ofhost DNA in feces is associated with risk of Clostridium difficileinfection[J]. Journal of Immunology Research, 2015, 2015:246203.
[20] AGETSUMA-YANAGIHARA Y, INOUE E, AGETSUMA N. Ef?fects of time and environmental conditions on the quality of DNAextracted from fecal samples for genotyping of wild deer in awarm temperate broad-leaved forest[J]. Mammal Research,2017, 62(2): 201-207.
[21] KALIPPKE K, WERWITZKE S, VON HORNUNG M, et al.DNA analysis from stool samples: a fast and reliable methodavoiding invasive sampling methods in mouse models of bleedingdisorders[J]. Laboratory Animals, 2009, 43(4): 390-393.
[22] WANNER N, LARSEN P A, MCLAIN A, et al. The mitochon?drial genome and epigenome of the golden lion tamarin from fecalDNA using nanopore adaptive sequencing[J]. BMC Genomics,2021, 22(1): 726.
[23] ZHU Y X, DENG T, QIAO M J, et al. Comparison of geneticcharacteristics between captive and wild giant pandas based on13 mitochondrial coding genes[J]. Molecular Biology Reports,2022, 49(6): 4901-4908.
[24] SYNDERCOMBE C D. Mitochondrial DNA in forensic use[J].Emerging Topics in Life Sciences, 2021, 5(3): 415-426.
[25] 馬逸清, 解焱. 中國虎的起源和瀕危過程的研究[J]. 野生動物, 2010, 31(5): 262-269.
MA Y Q, XIE Y. On the origin of tigers in China and process ofendanger[J]. Chinese Journal of Wildlife, 2010, 31(5):262-269.
[26] RUSSELLO M A, GLADYSHEV E, MIQUELLE D, et al. Po?tential genetic consequences of a recent bottleneck in the Amurtiger of the Russian Far East[J]. Conservation Genetics, 2004,5(5): 707-713.
[27] CRACRAFT J, FEINSTEIN J, VAUGHN J, et al. Sorting out ti?gers (Panthera tigris): mitochondrial sequences, nuclear in?serts, systematics, and conservation genetics[J]. Animal Con?servation, 1998, 1(2): 139-150.
[28] 劉思霞,張馨元,晏鳴霄,等. 云南玉溪綠孔雀遺傳多樣性分析:與圈養種群的比較[J]. 野生動物學報,2022,43(4):943-954.
LIU S X, ZHANG X Y, YAN M X, et al. Genetic diversityanalysis of green peafowl in Yuxi, Yunnan, China: a comparisonwith captive population[J]. Chinese Journal of Wildlife, 2022,43(4): 943-954.
[29] SHPILKA T, HAYNES C M. The mitochondrial UPR: mecha?nisms, physiological functions and implications in ageing[J].Nature Reviews Molecular Cell Biology, 2018, 19: 109-120.
[30] LAGOUGE M, LARSSON N G. The role of mitochondrial DNAmutations and free radicals in disease and ageing[J]. Journal ofInternal Medicine, 2013, 273(6): 529-543.
[31] AMORIM J A, COPPOTELLI G, ROLO A P, et al. Mitochon?drial and metabolic dysfunction in ageing and age-related dis?eases[J]. Nature Reviews Endocrinology, 2022, 18: 243-258.
[32] KHAN A, PATEL K, SHUKLA H, et al. Genomic evidence forinbreeding depression and purging of deleterious genetic varia?tion in Indian tigers[J]. Proceedings of the National Academy ofSciences of the United States of America, 2021, 118(49):e2023018118.
[33] ZHANG L, LAN T M, LIN C Y, et al. Chromosome-scale ge?nomes reveal genomic consequences of inbreeding in the southChina tiger: a comparative study with the Amur tiger[J]. Mo?lecular Ecology Resources, 2023, 23(2): 330-347.
[34] STANGE M, BARRETT R D H, HENDRY A P. The impor?tance of genomic variation for biodiversity, ecosystems andpeople[J]. Nature Reviews Genetics, 2021, 22: 89-105.
基金項目:國家自然科學基金面上項目(32170517)

