圈養與野外近人環境獼猴腸道菌群的比較分析

摘 要 通過對來自北京動物園10只圈養獼猴(Macaca mulatta)和來自河南濟源五龍口風景區10只處于野外近人環境中的獼猴的糞便微生物進行鳥槍法測序,分析兩種不同環境下獼猴的腸道菌群組成和差異。結果表明:20份樣品共檢測出9個門、18個綱、22個目、31個科、46個屬和226個種。通過分析發現,圈養和野外近人環境群體共享了絕大部分細菌(200種),但是近人環境群體相比于圈養群體,獨有更多種類的微生物。圈養獼猴腸道微生物主要由厚壁菌門(Firmicutes)、擬桿菌門(Bacteroidetes)和綠彎菌門(Chloroflexi)組成。處于野外近人環境中的獼猴腸道微生物主要由厚壁菌門、變形菌門(Proteobacteria)和擬桿菌門組成。圈養獼猴和近人獼猴屬水平上的腸道微生物結構存在顯著差異(p=0. 001)。圈養獼猴和野外近人環境獼猴腸道菌群組成的多樣性與差異,可為獼猴的圈養以及景區野生動物管理提供指導和理論基礎。
關鍵詞:獼猴;腸道菌群;鳥槍法測序;宏基因組分析
中圖分類號:Q93 文獻標識碼:A 文章編號:2310 - 1490(2024)- 02 - 0269 - 07
DOI:10.12375/ysdwxb.20240205
腸道菌群作為機體的“第二套基因組”,不僅僅是一組隨機的微生物,而是一個在宿主生理和行為中扮演關鍵角色的復雜群落[1]。腸道菌群可以通過影響宿主代謝[2]、營養[3]、免疫[4]、行為[5]和發育[6]等,幫助宿主適應各種飲食變化、環境氣候變化以及病原體等[7]。因腸道菌群具有高度的可塑性,飲食結構、系統發育關系和周圍環境都會影響其結構和功能[8],從而幫助宿主對環境的變化做出快速反應[7]。圈養環境或人類干擾會導致非人靈長類動物失去原有的腸道菌群結構[9],并且向現代人類的腸道菌群結構靠攏[10]。
獼猴(Macaca mulatta)屬靈長目(Primates)猴科(Cercopithecidae)獼猴屬(Macaca),是國家二級重點保護野生動物,具有重要的科研價值[11]。同時獼猴是諸多旅游區、動物園的重要觀賞動物[12],能夠帶動旅游經濟。出于科研目的以及保護性目的所圈養的獼猴數量較多,對于其腸道菌群變化的探究有利于指導圈養種群的健康管理和科學喂養,但關于獼猴腸道健康的探究還較為缺乏,無論是在分類水平上還是在功能水平上,圈養和野外近人環境(野生無固定投食,但是和人類接觸密切)獼猴腸道菌群的差異都知之甚少。因此本研究擬開展對圈養和野外近人環境獼猴腸道菌群差異的探究,通過宏基因組測序,對比分析來自北京動物園的圈養獼猴與來自河南濟源五龍口風景區的野外近人環境獼猴腸道菌群的差異與結構,旨在為獼猴的圈養以及景區野生動物管理提供理論指導。
1 材料與方法
1. 1 樣品采集
圈養獼猴糞便樣品來源于北京動物園(10份),野外近人環境獼猴的糞便樣品來源于河南濟源五龍口風景區(10份)。采集的糞便樣本由RNALater液體于干冰中保存,并在一周內送至實驗室,-20 ℃保存備用。為了減少對動物棲息地的影響,在采集樣品過程中未攜帶對生物群系有不良影響的有毒物質。本研究嚴格遵守中國野生動物保護學會制定的議定書、美國靈長類動物學會(ASP)關于非人靈長類動物倫理待遇的原則和中國的法律要求進行。
1. 2 DNA 采集和鳥槍法測序
采用QIAamp DNA stool mini kit(Qiagen,Valen?cia,CA,USA)進行基因組DNA提取。按照試劑盒說明(每個糞便樣本0. 25 g)完成基因組DNA提取后,使用NanoDrop(ND-1000) spectrophotometer(Nano?Drop Technologies,Wilmington,DE,USA)測定DNA濃度,并通過1%的瓊脂糖凝膠電泳評估DNA降解程度。DNA 樣本于-20 ℃保存備用。通過IlluminaNovaSeq 6000進行每樣本10 Gb的鳥槍法測序。
1. 3 質量控制
使用Trimmomatic v0. 36剪切低質量測序片段:棄置小于70 bp的測序片段,并且當4 bp滑動窗口的平均質量小于20時,則從3'端刪除片段。使用bow?tie2 v2. 3. 5軟件,選擇“-very-sensitive”參數,比對參考NCBI 上的基因組過濾宿主或人類污染物(Ma?caca mulatta:assembly Mmul_10;Homo sapiens:as?sembly GRCh38. p13)。
1. 4 Alpha 多樣性與Beta 多樣性分析
使用R“ vegan”包計算Chao1指數和ACE指數,得出圈養以及野外近人環境獼猴間的Alpha 多樣性。基于R“ vegan” 包的Bray-Curtis dissimilarity在屬水平上進行主坐標分析(PCoA),并通過“stats”包進行非度量多維尺度分析(NMDS),得出兩個群體的Beta多樣性。
1. 5 宏基因組分類學分析
使用mOTUs v. 2. 0. 0生成分類學物種相對豐度表,并對物種豐度表進行過濾,舍棄最大相對豐度未超過1×10-3的微生物物種以及未映射的宏基因組測序片段。
1. 6 宏基因組功能分析
為了重建微生物基因組,了解每組的基因水平和功能水平特征,使用單樣本宏基因組組裝和功能注釋。簡單來說,使用MEGAHIT(v1. 2. 6)組裝,使用MetaGeneMark對長度超過300 bp的DNA長片段進行基因預測。將長度大于100 bp的氨基酸序列,使用Diamond v0. 9. 24 參考KEGG(v50)和CARD 數據庫進行功能注釋,參數為-d-q-e1e-5-k1,計算KEGG功能通路相對豐度和抗性基因相對豐度。
1. 7 差異分析
為了比較圈養和野外近人環境群體微生物和功能的差異,分別繪制物種水平和抗性基因的韋恩圖。此外,利用LEfSe軟件對物種、KEGG通路相對豐度進行差異分析,得到野外近人環境與圈養群體之間有差異的細菌和功能。
2 結果
2. 1 物種豐度
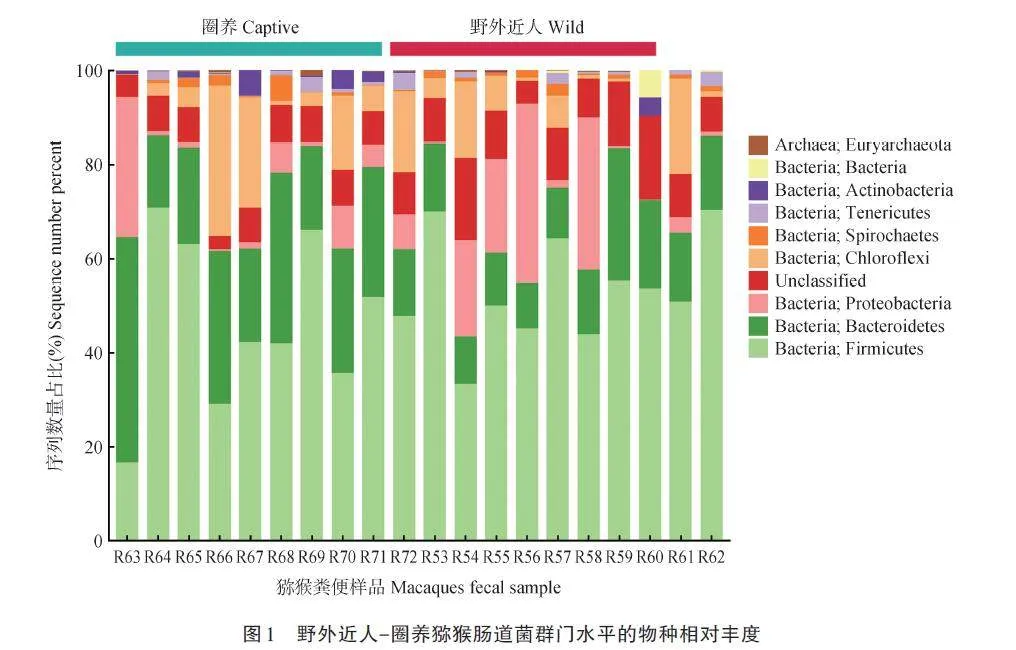
共計對20只獼猴樣本進行宏基因組測序和分析,發現9個門、18個綱、22個目、31個科、46個屬和226個種。根據物種注釋結果,選取各個樣本門水平上豐度排名前10的物種,繪制門水平物種相對豐度柱形圖(圖1)。在門水平上,圈養獼猴腸道微生物主要由厚壁菌門(Firmicutes,15. 18%~62. 15%)、擬桿菌門(Bacteroidetes,13. 48%~43. 47%)和綠彎菌門(Chloroflexi,0. 12%~30. 51%)組成,野外近人獼猴腸道微生物主要由厚壁菌門(23. 57%~66. 16%)、變形菌門(Proteobacteria,0. 07%~27. 42%)和擬桿菌門(6. 89%~25. 03%)組成。
2. 2 多樣性
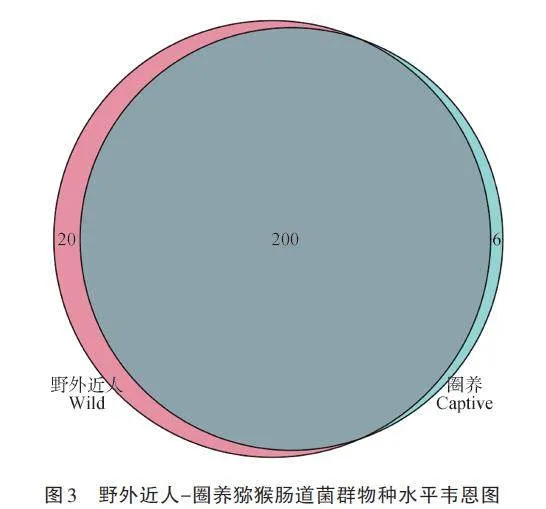
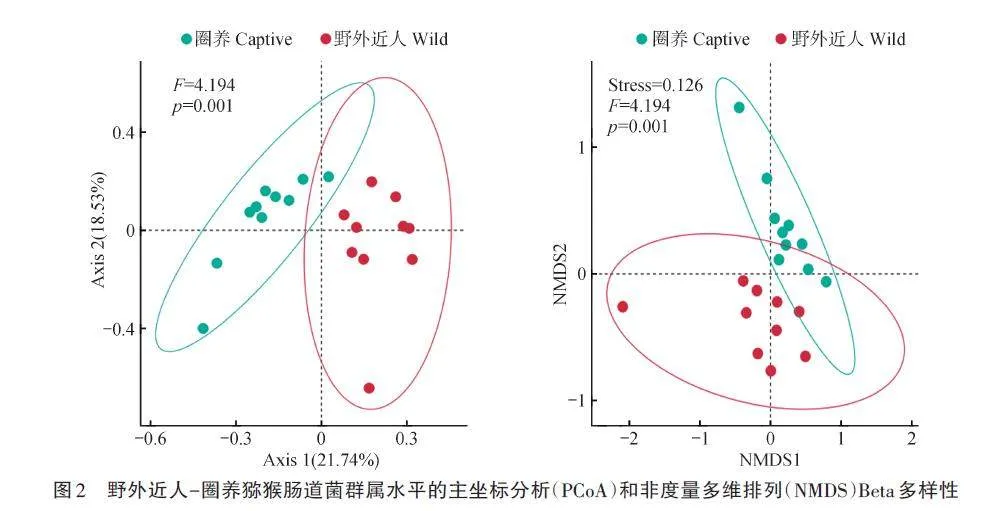
使用非度量多維尺度分析以及主坐標分析法分析屬水平上獼猴腸道微生物的Beta多樣性(圖2)。圈養獼猴和近人獼猴的屬水平上腸道微生物結構存在顯著差異(p=0. 001)。通過物種水平上的韋恩圖,比較圈養和近人獼猴群體腸道菌群的組成差異(圖3)。經過分析,發現圈養和近人群體共享了絕大部分細菌(200種),但是近人群體相比圈養群體,獨有更多種類的微生物。由圖3可知,近人群體特有20種細菌,而圈養群體特有6種細菌。
2. 3 基于屬水平上的物種差異
通過線性判別分析,在屬水平上判定野外近人組和圈養組間具有顯著差異的菌屬(圖4),結果表明,在野外近人組中具有顯著豐度差異的屬是不動桿菌屬(Acinetobacter)、霍爾德曼氏菌屬(Hold?emanella)、考拉桿菌屬(Phascolarctobacterium)、變形桿菌屬(Proteus)、月單胞菌屬(Selenomonas)、柯林斯菌屬(Collinsella)、庫特氏菌屬(Kurthia)、土壤芽孢桿菌屬(Solibacillus)、丁酸球菌屬(Butyricicoccus)、糞球菌屬(Coprococcus)和腸球菌屬(Enterococcus)。此外,在野外近人群體中存在更多的未知細菌。
在圈養組中具有顯著豐度差異的菌屬有普雷沃氏菌屬(Prevotella)、毛螺菌屬(Lachnospira)、螺桿菌屬(He?licobacter)、真細菌屬(Eubacterium)、雙歧桿菌屬(Bifido?bacterium)、瘤胃球菌屬(Ruminococcus)、琥珀酸弧菌屬(Succinivibrio)及Succinivibrionaceae unclassified。
2. 4 腸道菌群功能差異
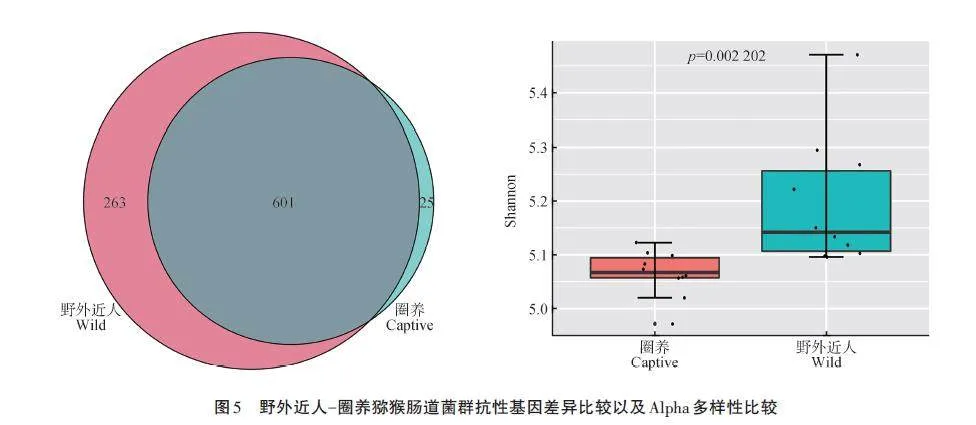
通過抗性基因韋恩圖(圖5)可知,野外近人獼猴所獨有的抗性基因要遠多于圈養獼猴所獨有的抗性基因。在進一步分析兩組間抗性基因的Alpha多樣性后,發現野外近人群體的抗性基因多樣性要高于圈養群體。這一發現與之前的部分野生圈養研究存在差異,如在Brisson等[13]對非洲圈養與自由放養的野生食草動物的研究中發現,相比于自由放養的野生食草動物,圈養動物糞便中的大腸埃希氏菌(Escherichia coli)更有可能攜帶抗性基因;Guo等[14]研究發現圈養大熊貓(Ailuropoda melanoleuca)的腸道微生物相比于野生大熊貓擁有更多的抗性基因。
3 討論
本研究對獼猴糞便進行了宏基因組測序,結果表明,圈養獼猴腸道微生物主要由厚壁菌門、擬桿菌門和綠彎菌門組成,野外近人獼猴腸道微生物主要由厚壁菌門、變形菌門和擬桿菌門組成。不論是圈養獼猴還是野外近人獼猴,其腸道微生物中的優勢菌群都包含厚壁菌門與擬桿菌門,這與人類健康腸道微生物群的主要結構相似,即主要由厚壁菌門和擬桿菌門組成[15]。
3. 1 圈養與野外近人環境獼猴腸道菌群存在顯著差異
本研究對獼猴的腸道菌群進行了Alpha多樣性和Beta多樣性分析,結果表明圈養獼猴和野外近人環境獼猴的腸道微生物在物種組成和功能上存在顯著差異。在其他物種的研究中,也存在這樣的差異,如在對綠尾虹雉(Lophophorus lhuysii)的研究中發現野生和家養的綠尾虹雉腸道菌群結構之間存在顯著差異[16];對鹿鼠(Peromyscus maniculatus)的研究中發現野生鹿鼠腸道菌群的Alpha 多樣性高于圈養鹿鼠[17]。這樣的差異可能是由食物種類不同導致。
3. 2 圈養與野外近人環境獼猴腸道菌群存在結構和功能差異
本研究進行了屬水平上的物種差異分析,發現野外近人組具有顯著豐度差異的屬一共有11個,其中變形桿菌代表著代謝紊亂和炎癥性腸病等疾病[18];霍爾德曼氏菌能夠改善葡萄糖耐量并調節GLP-1信號通路[19];不動桿菌能夠利用廣泛的有機化合物作為碳和能量的來源,降解多種有機污染物[20];考拉桿菌則可以利用擬桿菌屬(Bacteroides)和副擬桿菌屬(Parabacteroides)產生的琥珀酸鹽(succinate)生成短鏈脂肪酸,而高脂肪的飲食會使Bacteroides 和Para?bacteroides 的豐度增加[21]。因此,考拉桿菌的豐度會因為高脂肪的飲食而增加,意味著野外近人環境獼猴可能接觸到更多的有機污染物和高脂肪飲食。這可能是游客在風景區中旅游時,經常會向野生動物投喂自行攜帶的高熱量高脂肪食品。在圈養組中具有顯著豐度差異的屬一共有8個。其中毛螺菌是腸道菌群中重要的丁酸鹽生產者[22];螺桿菌引起腸炎和胃炎[23];真細菌產生丁酸鹽,在能量穩態、結腸運動、免疫調節和抑制腸道炎癥中起關鍵作用[24];雙歧桿菌能將芳香族氨基酸轉化為乳酸衍生物[25];普雷沃氏菌與高水平的碳水化合物以及水果和蔬菜的攝入呈正相關[26],代表圈養獼猴的飲食中所含碳水化合物的成分更多。在對黔金絲猴(Rhinopithecusbrelichi)的研究中也發現,相比于野生群體,圈養黔金絲猴具有更高相對豐度的普雷沃氏菌,并推測其原因是圈養群體主要飲食為水果及玉米粉[27]。
3. 3 野外近人環境獼猴具有更多特有的抗藥性腸道微生物
通過對獼猴腸道菌群的基因進行抗性基因檢測,發現相比于圈養獼猴,野外近人環境獼猴的腸道微生物中擁有更多的特有抗藥性基因。在對圈養、半圈養以及野生獼猴的研究[28]中,同樣發現半圈養(因為野外近人環境的獼猴糞便取樣地點位于風景區,獼猴雖身處野外,卻能夠經常與人類接觸,所以認為其與“半圈養”狀態類似)狀態下的獼猴擁有更高的抗性基因多樣性,推測是野外近人環境獼猴的糞便樣本來源于河南濟源五龍口風景區中的非圈養獼猴,在風景區中并未禁止游客同獼猴產生肢體接觸。因此野外近人環境獼猴擁有與更多、更復雜的人群接觸的機會,可能導致它們接觸到更多抗生素;而圈養獼猴生活在北京動物園,除了飼養員外極少與人類擁有親密接觸的機會,因此其腸道菌群擁有相對較少的抗性基因。
參考文獻:
[1] AMATO K R, YEOMAN C J, CERDA G, et al. Variable re?sponses of human and non-human primate gut microbiomes to aWestern diet[J]. Microbiome, 2015, 3: 53.
[2] VALDES A M, WALTER J, SEGAL E, et al. Role of the gut mi?crobiota in nutrition and health[J]. BMJ, 2018, 361: k2179.
[3] CHEVALIER C, STOJANOVI? O, COLIN D J, et al. Gut micro?biota orchestrates energy homeostasis during cold[J]. Cell,2015, 163(6): 1360-1374.
[4] LEE Y K, MAZMANIAN S K. Has the microbiota played a criti?cal role in the evolution of the adaptive immune system? [J].Science, 2010, 330(6012): 1768-1773.
[5] BRUCE-KELLER A J, SALBAUM J M, LUO M, et al. Obesetypegut microbiota induce neurobehavioral changes in the ab?sence of obesity[J]. Biological Psychiatry, 2015, 77(7):607-615.
[6] MUELLER N T, BAKACS E, COMBELLICK J, et al. The infantmicrobiome development: mom matters[J]. Trends in MolecularMedicine, 2015, 21(2): 109-117.
[7] SUZUKI T A, LEY R E. The role of the microbiota in human ge?netic adaptation[J]. Science, 2020, 370(6521): eaaz6827.
[8] BAJ A, MORO E, BISTOLETTI M, et al. Glutamatergic signal?ing along the microbiota-gut-brain axis[J]. International Journalof Molecular Sciences, 2019, 20(6): 1482.
[9] FRANKEL J S, MALLOTT E K, HOPPER L M, et al. The effectof captivity on the primate gut microbiome varies with host dietaryniche[J]. American Journal of Primatology, 2019, 81(12):e23061.
[10] CLAYTON J B, VANGAY P, HUANG H, et al. Captivity hu?manizes the primate microbiome[J]. Proceedings of the NationalAcademy of Sciences of the United States of America, 2016, 113(37): 10376-10381.
[11] 蔣學龍, 王應祥, 馬世來. 中國獼猴的分類及分布[J]. 動物學研究, 1991, 12(3): 241-247.
JIANG X L, WANG Y X, MA S L. Taxonomic revision and dis?tribution of subspecies of rhesus monkey (Macaca mulatta) inChina[ J]. Zoological Research, 1991, 12(3): 241-247.
[12] 王鈺煒, 路紀琪, 田軍東. 國內涉及非圈養獼猴的旅游區現狀調查[J]. 動物學雜志, 2022, 57(4): 514-520.。
WANG Y W, LU J Q, TIAN J D. Survey on the status of rhesusmacaque-involved tourism in China[J]. Chinese Journal of Zool?ogy, 2022, 57(4):514-520.
[13] BRISSON L, CARON A, MAZUY-CRUCHADET C, et al. Com?paring antibiotic resistance in free-ranging vs. captive Africanwild herbivores[J]. Journal of Wildlife Diseases, 2023, 59(2):224-233.
[14] GUO W, MISHRA S, WANG C D, et al. Comparative study ofgut microbiota in wild and captive giant pandas (Ailuropodamelanoleuca)[J]. Genes, 2019, 10(10): 827.
[15] JANDHYALA S M, TALUKDAR R, SUBRAMANYAM C, etal. Role of the normal gut microbiota[J]. World Journal of Gas?troenterology, 2015, 21(29): 8787-8803.
[16] JIANG D D, HE X, VALITUTTO M, et al. Gut microbiota com?position and metabolomic profiles of wild and captive Chinesemonals (Lophophorus lhuysii)[J]. Frontiers in Zoology, 2020,17(1): 36.
[17] SCHMIDT E, MYKYTCZUK N, SCHULTE-HOSTEDDE A I.Effects of the captive and wild environment on diversity of the gutmicrobiome of deer mice (Peromyscus maniculatus)[J]. TheISME Journal, 2019, 13: 1293-1305.
[18] RIZZATTI G, LOPETUSO L R, GIBIINO G, et al. Proteobacte?ria: a common factor in human diseases[J]. BioMed ResearchInternational, 2017, 2017: 9351507.
[19] ROMANí -PéREZ M, LóPEZ-ALMELA I, BULLICH-VILARRUBIASC, et al. Holdemanella biformis improves glucose toler?ance and regulates GLP-1 signaling in obese mice[J]. FASEBJournal, 2021, 35(7): e21734.
[20] KAMPFER P. Acinetobacter[M]//BATT C A, TORTORELLO ML. Encyclopedia of food microbiology. 2nd ed. London: Aca?demic Press, 2014: 11-17.
[21] WU F F, GUO X F, ZHANG J C, et al. Phascolarctobacteriumfaecium abundant colonization in human gastrointestinal tract[J]. Experimental and Therapeutic Medicine, 2017, 14(4):3122-3126.
[22] DAHIYA D K, RENUKA, DANGI A K, et al. New-generationprobiotics: perspectives and applications[M]//FAINTUCH J,FAINTUCH S. Microbiome and metabolome in diagnosis,therapy, and other strategic applications. London: AcademicPress, 2019: 417-424.
[23] WESLEY I V. Helicobacter[M]//ROBINSON R K, BATT C A,PATEL P D. Encyclopedia of food microbiology. San Diego:Academic Press, 2000: 1047-1052.
[24] MUKHERJEE A, LORDAN C, ROSS R P, et al. Gut microbesfrom the phylogenetically diverse genus Eubacterium and theirvarious contributions to gut health[J]. Gut Microbes, 2020, 12(1): 1802866.
[25] LAURSEN M F, SAKANAKA M, VON BURG N, et al. Bifido?bacterium species associated with breastfeeding produce aro?matic lactic acids in the infant gut[J]. Nature Microbiology,2021, 6: 1367-1382.
[26] WU G D, CHEN J, HOFFMANN C, et al. Linking long-term di?etary patterns with gut microbial enterotypes[J]. Science,2011, 334(6052):105-108.
[27] HALE V L, TAN C L, NIU K F, et al. Gut microbiota in wildand captive Guizhou snub-nosed monkeys, Rhinopithecus brelichi[J]. American Journal of Primatology, 2019, 81(10/11):e22989.
[28] JIA T, CHANG W S, MARCELINO V R, et al. Characteriza?tion of the gut microbiome and resistomes of wild and zoo-captivemacaques[J]. Frontiers in Veterinary Science, 2022, 8:778556.
基金項目:中國科學院戰略性先導科技專項(A類)(XDA23080201)

