瑤藥鴨腳風葉綠體基因組結構解析及密碼子偏好性分析





摘要:為探究老班瑤藥鴨腳風葉綠體基因組的結構特征、系統進化以及密碼子偏好性,以瑤藥鴨腳風為研究材料,對瑤藥鴨腳風葉綠體基因組進行測序和組裝。結果表明,鴨腳風的葉綠體基因組由大單拷貝區、小單拷貝區以及2個反向重復區組成,全長156 532 bp,包含123個基因,分別為8個核糖體rRNA基因、29個轉錄tRNA基因和86個蛋白質編碼基因。共檢測到84個SSR位點。密碼子偏好性分析顯示,鴨腳風的葉綠體基因最優密碼子偏好以A/U堿基結尾,自然選擇可能是其密碼子使用模式的主要影響因素。系統進化分析表明,鴨腳風與輻葉鵝掌柴親緣關系密切。以上研究結果為鴨腳風葉綠體基因組研究和應用提供了科學依據。
關鍵詞:鴨腳風;葉綠體基因組;密碼子偏好性;最優密碼子doi:10.13304/j.nykjdb.2023.0006
中圖分類號:Q949.95;Q732 文獻標志碼:A 文章編號:10080864(2024)12006314
鴨腳風[Schefflera octophylla (Lour.) Harms]為瑤族傳統中藥,別名鵝掌木、鴨掌木等,為五加科鵝掌柴屬,在中國主要分布在廣西、廣東、云南、海南、福建等地,多生長于山地、丘陵的溫暖、濕潤、半陽環境。鴨腳風在瑤族醫藥體系中具有重要地位,藥理學研究表明,其具有止痛、鎮靜、抗炎、抗真菌、解熱、利尿等功效[1]。除此之外,劉睿等[2]發現,鵝掌柴具有抗腫瘤的活性成分;龐素秋等[3]發現,鵝掌柴的可揮發油具有良好的抗炎鎮痛作用。
葉綠體為藥用植物提供大量合成具有生物活性的天然產物所需的能量和碳骨架,例如氨基酸和UDP葡萄糖的生物合成就發生在葉綠體中[4]。近年來,植物葉綠體基因組的研究已經成為葉綠體轉化技術和植物親緣關系鑒定的手段[5]。一般對于葉綠體基因組的研究有2種:一種是以全基因組測序為目標的結構基因組學(structuralgenomics);另一種是以基因功能鑒定為目標的功能基因組學(functional genomics)[6]。對于葉綠體基因組特性的研究首先需要對其葉綠體基因組進行測序、組裝和分析等[7]。葉綠體是與光合作用直接相關的細胞器,因此葉綠體基因組的功能跟光合作用具有較為緊密的關系[8]。植物基因的密碼子使用受到自然選擇、基因突變或者其他因素影響[9]。目前,許多藥用植物、特有植物等的葉綠體基因組信息得到了研究,但關于鴨腳風的葉綠體基因組結構尚未解析,基于葉綠體基因組的進化樹分析及其葉綠體基因組的密碼子偏好性等也有待于進一步研究。
因此,本研究在基因組水平上對鴨腳風葉綠體的基因組進行結構解析,分析其結構組成、功能、密碼子的使用偏好性及影響密碼子偏好性的因素,并篩選出鴨腳風葉綠體基因組的最優密碼子,為鴨腳風葉綠體基因組研究和應用提供科學依據,也為瑤藥鴨腳風遺傳背景、種質資源保護與開發利用及基因工程等方面的深入研究奠定理論基礎。
1 材料與方法
1.1 試驗材料
在廣西壯族自治區金秀瑤族自治縣大瑤山中采集瑤藥鴨腳風,經金秀瑤族自治縣瑤醫醫院羅秋香醫生鑒定為傳統瑤藥鴨腳風。
1.2 試驗方法及數據分析
1.2.1 鴨腳風葉綠體基因組提取與測序 取葉齡適中的健康葉片保存于液氮,使用天根生物科技有限公司植物基因組快速提取試劑盒提取瑤藥鴨腳風的葉綠體總DNA。利用1%瓊脂糖電泳檢測DNA質量和完整性,將達到測序要求的DNA樣品送往百邁克生物科技有限公司,使用IlluminaHiSeq 2500-PE150平臺進行測序。
1.2.2 鴨腳風葉綠體基因組組裝與注釋 序列采用Paired-End Clean Reads合并后用于葉綠體基因組組裝,采用Dierckxsens 的組裝方法[10],利用Novoplasty進行有參+無參的組合方案組裝,最終得到瑤藥鴨腳風葉綠體基因組數據。將篩選出的reads 用在線軟件GetOrganelle pipeline(https://github.com/Kinggerm/GetOrganelle)進行denovo 組裝。采用在線注釋軟件Annotation toolGeSeq(https://chlorobox.mpimp-golm.mpg.de/geseq. html)對瑤藥鴨腳風葉綠體基因組進行注釋,將注釋完整的鴨腳風葉綠體基因組序列使用CPGAVAS2 在線工具對注釋結果進行確認修正[11],并繪制鴨腳風葉綠體基因組物理圖譜。
1.2.3 鴨腳風葉綠體基因組特性分析 利用軟件MPI-MP的GeSeq(https://chlorobox.mpimp-golm.mpg.de/geseq.htm)分析瑤藥鴨腳風的葉綠體基因組的堿基組成情況,使用線上軟件MPI-MPCHIOROOBOX-OGDRAW對葉綠體基因組的功能基因進行分類,利用線上網站(https://webblast.ipk-gatersleben.de/misa)分析鴨腳風葉綠體基因組中核苷酸簡單重復序列(simple sequence repeat,SSR)的類型及數量。
1.2.4 鴨腳風葉綠體基因組密碼子偏好性分析 篩選出以ATG為起始密碼子、長度大于300 bp的編碼基因的序列(coding DNA sequence,CDS)作為分析樣本[12]。利用Codon W 1.4.2軟件對將篩選出來的CDS進行分析,計算瑤藥鴨腳風蛋白質編碼基因的相對同義密碼子使用度(relativesynonymous codon usage,RSCU)[13]。利用線上軟件Chip 分析CDS 的有效密碼子數(effectivenumber of codon,ENC)。使用在線軟件CUSP 計算各密碼子第1、第2和第3位堿基的GC含量,分別用GC1、GC2、GC3 表示,以及密碼子的平均GC量,用GCall表示。
利用數據處理軟件SPSS 19.0對各基因的密碼子數目(N)以及GC1、GC2、GC3、GCall和ENC值進行相關性分析;利用Excel 對GC3 與GC12(GC12=(GC1+GC2)/2)進行中性繪圖分析,計算出GC3 與GC12 的函數關系;利用R語言對GC3 和ENC值進行有效密碼子數繪圖分析(ENC-plot),根據標準曲線計算GC3 和ENC值的關系[14]。同時,利用標準方程求出ENC的期望值,計算ENC比值,公式如下。
利用Excel統計鴨腳風葉綠體基因組氨基酸第3位密碼子各堿基出現的頻率(分別用U3、C3、A3、G3 表示),并進行繪圖分析,以G3/(G3+C3)為橫坐標,A3/(A3+U3)為縱坐標制作四象分布圖,分析密碼子第3 位堿基是否存在偏好性及其影響因素。
有效密碼子數根據ΔRSCU 來確定,先將53條CDS根據ENC值按照從高到低來排序,從兩端各選擇10%的CDS,其中以5條高ENC值的CDS為低表達基因庫,以5條低ENC值為高表達基因庫。利用codon W1.2.4 軟件統計RSCU 值,將ΔRSCU≥0.08 的密碼子作為高表達優越密碼子。將RSCU值gt;1且ΔRSCU≥0.08的密碼子定義為鴨腳風葉綠體基因組的最優密碼子[15]。
1.2.5 聚類分析 從NCBI GenBank下載與瑤藥鴨腳風物種相似的完整基因組序列,包括鵝掌柴屬的Schefflera heptaphylla NC-029764 和Scheffleraactinophylla NC-049889,刺楸屬的Kalopanaxseptemlobus KC456167和Metapanax delavayi KC456165,人參屬的Panax notoginseng KJ566590 和 Araliaundulata NC-022810,五加屬的Eleutherococcuseleutheristylus NC-062720、Eleutherococcus gracilistylusNC-062721、Eleutherococcus trifoliatus MT754220、Eleutherococcus henryi NC-062719 和Fatsia japonicaNC-027685。使用MAFFT軟件對上述物種的基因序列進行比對,使用MEGA 7.0軟件采用最大似然法進行系統聚類分析,最后選擇最佳模型TVM+F+R2,1 000次重復,進行bootstrap檢驗分析[16]。
2 結果與分析
2.1 鴨腳風葉綠體基因組結構分析
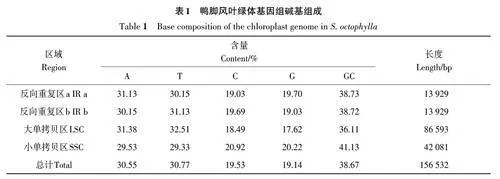
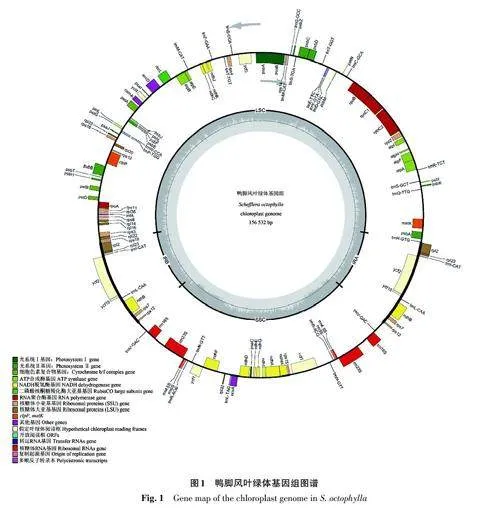
瑤藥鴨腳風葉綠體基因組的結構呈環狀雙鏈(圖1),堿基長度為156 532 bp,可分為4個區域:1 個長度為86 593 bp 的大單拷貝區(largesingle copy,LSC)、1 個長度為42 081 bp 的小單拷貝區(small single copy,SSC)、2 個長度為13 929 bp的反向重復區(inverted repeat,IR)。堿基A、T、C、G 的含量分別為30.55%、30.77%、19.53%、19.14%;平均G/C含量為38.67%;平均A/T含量為61.32%,具有明顯的A/T 偏向性(表1)。其中,小單拷貝區的GC含量最高,為41.13%;其次是反向重復區,GC 含量分別為38.73% 和38.72%;大單拷貝區的GC含量較低,為36.11%。
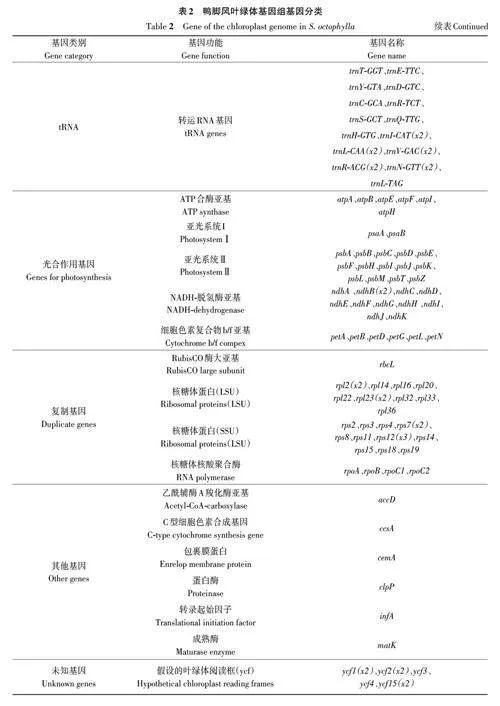
2.1.1 鴨腳風葉綠體基因組功能及分類 根據鴨腳風葉綠體基因組結構共得到123個基因(表2),其中包含86個蛋白質編碼基因、8個核糖體RNA基因(rRNA)和29個轉運RNA(tRNA);按照基因組功能劃分得到4大類別基因,主要包括光合作用基因、用于自我復制基因、其他基因和未知基因[17],比如乙酰輔酶A羧化酶亞基accD、包裹膜蛋白cemA、成熟酶matK。在這些基因中位于IR區的蛋白編碼基因有7個,ndhB、rpl2、rps7、rps12、rpl23、ycf2、ycf15;位于IR區的轉運RNA基因有2個,分別是trnI-CAT和trnL-CAA。位于SSC區域的基因有20個,其中包含12個蛋白質編碼基因,分別是ndhA、ndhD、ndhF、ndhE、ndhG、ndhI、ndhH、rps15、rpl32、psaC、ccsA、ycf;4個rRNA基因,分別是rrn16S(x2)、rrn23S(x2)、rrn4.5S(x2)、rrn5S(x2);以及4 個tRNA 基因,分別是trnVGAC、trnR-ACG、rnN-GTT和trnL-TAG。
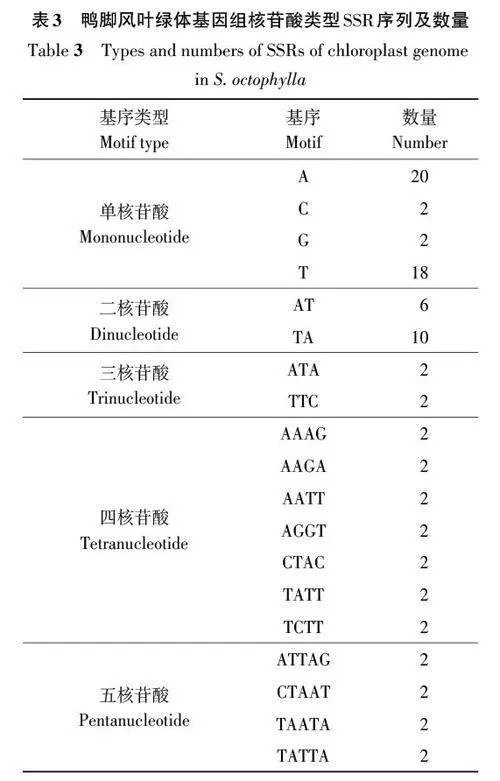
2.1.2 鴨腳風葉綠體基因組核苷酸類型及SSR分析 對瑤藥鴨腳風葉綠體基因組核苷酸進行SSR分析,結果(表3)表明,共檢測到84個SSRs,可分為5大類型:單核苷酸重復基序列(42個)、二核苷酸重復基序列(16 個)、三核苷酸重復基序列(4個)、四核苷酸重復基序列(14個)、五核苷酸重復基序列(8個)。其中,單核苷酸與二核苷酸數量較多,約占69%; 類型以A/T 為主,約占45%,AT/TA次之,約占19%。
2.2 鴨腳風葉綠體基因組中密碼子偏好性分析
由表4可知,CDS平均GC含量為38.71%,密碼子第1、第2和第3位的GC含量分別為47.22%、39.60%和29.78%,說明鴨腳風葉綠體基因的GC含量不高,且分布不均勻,其密碼子第3位堿基偏好以A/U結尾。
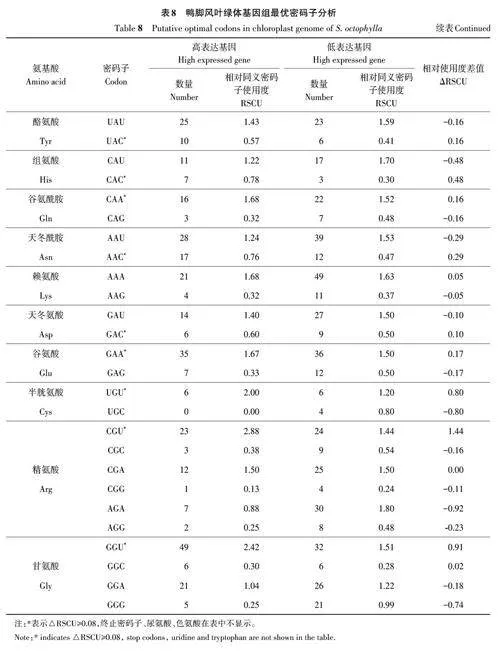
密碼子偏性的強弱可以根據ENC值來判定,當ENCgt;45 時其密碼子偏性較弱,反之較強[18]。鴨腳風葉綠體基因密碼子的ENC 值為40.35~56.14,平均48.28,表明其密碼子偏性較弱(表4)。相關性分析(表5)表明,GCall與GC1、GC2和GC3呈極顯著相關;GC1與GC2、GC3呈極顯著相關;ENC與GC3呈極顯著相關。由此表明,鴨腳風葉綠體基因密碼子前2 位堿基與第3 位堿基存在較大差異。
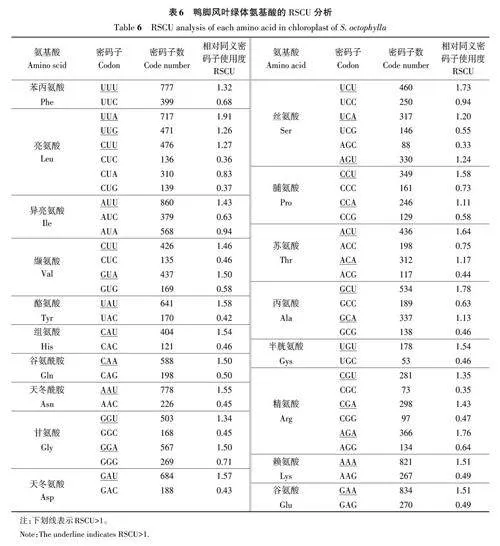
當RSCU=1 時,該密碼子不存在偏性[19];當RSCUgt;1時,該密碼子具有使用偏好性,且該密碼子為高頻密碼子[20]。分析鴨腳風各氨基酸的RSCU(表6)發現,RSCUgt;1的氨基酸有29個,其中有28個以A/U結尾,1個以G結尾,進一步說明鴨腳風葉綠體基因密碼子偏好以A/U結尾。RSCU分析及ENC分析結果均表明,鴨腳風葉綠體基因組密碼子的偏好性較低。
2.3 鴨腳風葉綠體基因組密碼子偏好性原因分析
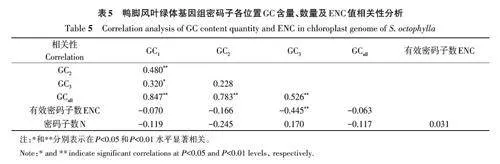
2.3.1 密碼子中性繪圖分析 密碼子中性繪圖可分析影響密碼子偏好性的因素[21],將鴨腳風葉綠體基因組密碼子第1和第2位堿基GC含量的平均值GC12 與GC3 進行中性繪圖分析,結果(圖2)表明,GC3 含量主要分布于0.2~0.4,而GC12 含量主要集中分布于0.3~0.5,二者間的相關系數為0.321 0(R2=0.103 1),相關性不顯著,且大部分基因主要分布于對角線的上方,由此說明瑤藥鴨腳風葉綠體密碼子的偏好性主要受自然選擇作用影響。
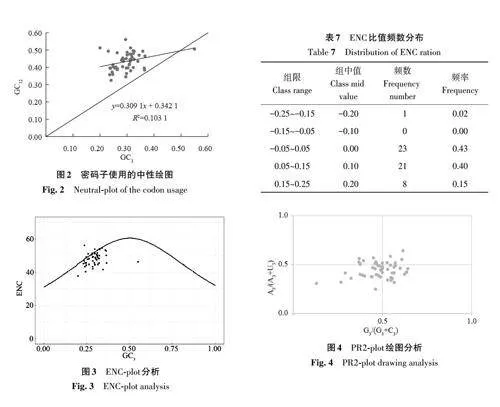
2.3.2 ENC-plot分析 ENC-plot可以直接反映基因密碼子的使用模式,分析突變和自然選擇對密碼子偏好性形成的影響[22]。由圖3可知,不同基因密碼子偏好性的差異較大。ENC 比值的分布頻率(表7)顯示,有23個基因分布集中在-0.05~0.05,占43%;有21 個基因分布在0.05~0.15,占40%;另外8個基因分布在0.15~0.25,占15%;1個基因在-0.25~-0.15。大部分基因都分布在距離曲線較遠位置,表明自然選擇作用對鴨腳風密碼子偏好性有較大影響。
2.3.3 PR2-plot分析 PR2-plot分析反映分析密碼子的使用是否受到自然或者其他因素的影響。由圖4可知,各CDS在4個區域內的分布不均勻,說明鴨腳風氨基酸密碼子的第3位堿基的使用可能受到自然選擇的影響。并且密碼子堿基U的使用頻率明顯高于A,C的使用頻率高于G,由此說明,鴨腳風葉綠體密碼子使用模式可能還受到其他因素的影響。
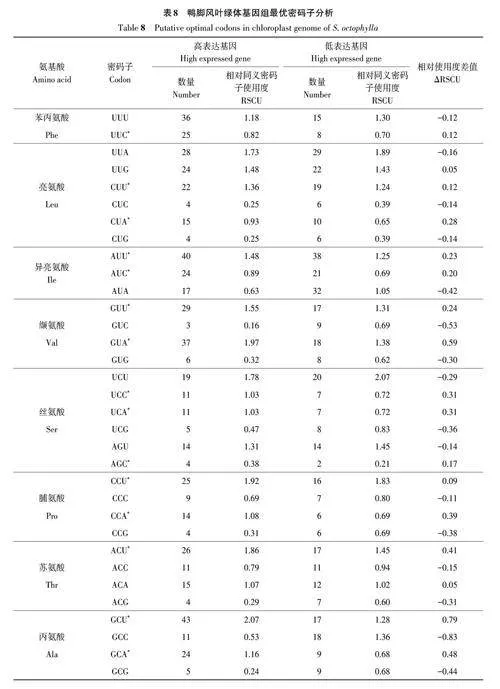
2.4 確定最優密碼子
由表8可知,ΔRSCU≥0.08的密碼子有24個,結合RSCUgt;1,共篩選出16 個最優密碼子,其中有9個以U結尾,有6個以A結尾,另1個以C結尾。最優密碼子分別為CUU、AUU、GUU、GUA、UCC、UCA、CCU、CCA、ACU、GCU、GCA、CAA、GAA、UGU、CGU、GGU。由此表明,鴨腳風葉綠體基因組密碼子偏好以A/U 結尾,具有A/T 偏向性。
2.5 系統進化分析
由圖5可知,從上到下4個分支分別是五加屬、刺楸屬、鵝掌柴屬、人參屬。瑤藥鴨腳風(Schefflera heptaphylla)與同屬的輻葉鵝掌柴(Schefflera actinophylla)親緣關系最近,其次是刺楸(Kalopanax septemlobus);輻葉鵝掌柴又與人參屬的三七(Panax notoginseng)關系較近;與五加屬相比,瑤藥鴨腳風與人參屬的關系更近。
3 討論
瑤藥鴨腳風是經典瑤藥之一,具有重要的開發和利用價值。本研究通過提取瑤藥鴨腳風葉綠體DNA 進行測序、組裝與注釋,得到長度為156 532 bp的葉綠體基因組序列,在分子水平上對瑤藥鴨腳風葉綠體的基因組進行結構解析,分析其組成、功能分類、密碼子使用偏好性及影響密碼子偏好性的成因。
植物葉綠體基因組一般為107~218 kb。本研究對鴨腳風葉綠體基因組測序得到長度為156 532 bp,與其他五加科植物(如三七、楤木、黃漆木等)的葉綠體基因組大小相似[23],可分為1個長度為86 593 bp的大單拷貝區(LSC)、1個長度為42 081 bp 的小單拷貝區(LSC)、2 個長度為13 929 bp 的反向重復區(IRa 與IRb,均為13 929 bp),G/C含量在4個區域的分布情況不同,平均G/C含量為38.67%,平均A/T含量為61.32%,說明密碼子使用偏向于A/T;總共編碼123 個基因,包括86個蛋白質編碼基因、29個tRNA基因和8個rRNA基因。鴨腳風葉綠體基因組與其他五加科植物(如人參等)的葉綠體基因組在基因數量上略有差異,其中蛋白質編碼基因總體上差異較小,而tRNA基因差異較大,大部分五加科物種的tRNA基因為37個[24]。
植物基因密碼子多存在偏好性現象,即某一或幾種特定密碼子的頻率超過其他同義密碼子,多為適應外界環境進化所致[25]。研究發現,影響物種密碼子偏好性的原因主要有堿基突變、基因表達水平、自然選擇等[26]。解析密碼子的偏好性,可以提高外源基因在寄主中的表達效率。本研究結果顯示,鴨腳風葉綠體基因密碼子第1、第2和第3位堿基的GC 含量分別為47.22%、39.60% 和29.78%,表明鴨腳風葉綠體基因不偏向使用GC,而偏向使用AT;其CDS 的ENC 值介于40.35~56.14,平均 48.28,說明偏性較弱;ENC與GC之間的相關性分析表明,其密碼子偏好以A/T(U)結尾,與五加科植物人參的研究結果一致[27];各氨基酸的RSCU分析來看,RSCUgt;1的CDS有29個,其中有28個以A/U結尾。由此表明,鴨腳風葉綠體基因組密碼子偏好使用A/U。中性繪圖及ENCplot分析表明,鴨腳風葉綠體基因組密碼子偏好與自然選擇有關,與小球藻[28]、杜梨[15]等一致。本研究共篩選出16 個最優密碼子,分別為CUU、AUU、GUU、GUA、UCC、UCA、CCU、CCA、ACU、GCU、GCA、CAA、GAA、UGU、CGU、GGU,其中有9個以U結尾,6個以A結尾,1個以C結尾。系統進化分析表明,瑤藥鴨腳風與輻葉鵝親緣關系最為密切,其次與刺楸的關系也較為密切,為進一步研究瑤藥鴨腳風遺傳背景、種質資源保護與開發利用及基因工程等方面的研究奠定了理論基礎。
參考文獻
[1] 陳蕾,朱霽虹,林瑞超. 鵝掌柴屬植物化學成分及生理活性研究概況[J].中藥材,2002,25(5):363-366.
[2] 劉睿,顧謙群,崔承彬,等. 密脈鵝掌柴的化學成分及其抗腫瘤活性[J].中草藥,2005,36(3):328-332.
LIU R, CU Q Q, CUI C B, et al . Chemical constituents of Schefflera venulosa and their antitumor activities [J]. Chin.Trad. Herbal Drugs, 2005,36(3):328-332.
[3] 龐素秋,金孝勤,孫愛靜,等. 鵝掌柴葉揮發油的成分分析及抗炎鎮痛活性[J]. 藥學實踐雜志,2016,34(1):56-58,78.
PANG S Q, JIN X Q, SUN A J, et al .. Constituent analysis, analgesic and anti-inflammatory activities of leaves essential oil of schefflera octophylla [J]. J. Pharm. Practice, 2016, 34(1):56-58, 78.
[4] 樊守金,郭秀秀.植物葉綠體基因組研究及應用進展[J].山東師范大學學報(自然科學版),2022,37(1):22-31.
FAN S J, GUO X X. Advances in research and application of plant chloroplast genome [J]. J. Shandong Norm. Univ. (Nat.Sci.), 2022, 37(1):22-31.
[5] 李曼芝,朱強,曾繼娟,等. 霸王葉綠體基因組測序及結構分析[J].天津農業科學,2021,27(10):13-16.
LI M Z, ZHU Q, ZENG J J, et al .. Sequencing and stuctural analysis of chloroplast genome in Zygophyllum xanthoxylum[J]. Tianjin Agric. Sci., 2021, 27(10):13-16.
[6] 李杰,蔡嘉慧,王慧中,等.藥用植物基因組測序及功能基因組學研究進展[J]. 杭州師范大學學報(自然科學版),2021,20(4):364-373,397.
LI J, CAI J H, WANG H Z, et al .. Advances in genome sequencing and functional genomics of medicinal plants [J]. J.Hangzhou Norm. Univ. (Nat. Sci.), 2021, 20(4):364-373,397.
[7] 路東曄,張磊,郝蕾,等. 臭柏葉綠體基因組結構與系統進化分析[J].西北植物學報,2018,38(8):1464-1475.
LU D Y, ZHANG L, HAO L, et al .. Analysis of chloroplast genome structure and phylogenetic evolution of Juniperus sabina [J]. Acta Bot. Bor-Occid. Sin., 2018, 38(8):1464-1475.
[8] 續晨,賁愛玲,蔡曉寧. 蝴蝶蘭葉綠體基因組密碼子使用的相關分析[J].分子植物育種,2010,8(5):945-950.
XU C, BEN A L, CAI X N. Analysis of synonymous codon usage in chloroplast genome of Phalaenopsis aphrodite psubsp.Formosana [J]. Mol. Plant Breed., 2010, 8(5):945-950
[9] 王宇飛,江媛,楊成金,等. 滇重樓葉綠體基因組特征及密碼子偏好性分析[J].分子植物育種,2021,19(22):7448-7458.
WANG Y F, JIANG Y, YANG C J, et al .. Analysis of chloroplast genome characteristic and codon usage bias in Paris polyphylla var. Yunnanensis [J] Mol. Plant Breed., 2021,19(22):7448-7458.
[10] DIERCKXSENS N, MARDULYN P, SMITS G. Unraveling heteroplasmy patterns with novoplasty [J/OL]. Nar. Genomics Bioinform., 2019, 2(1):11 [2023-02-01]. https://doi.org/10.1093/nargab/lqz011.
[11] GUO H J, LIU J S, LUO L, et al .. Complete chloroplast genome sequences of Schisandra chinensis: genome structure, comparative analysis,and phylogenetic relationship of basal angiosperms [J].Sci. China (Life Sci.), 2017, 60(11):1286-1290.
[12] 吳學俊,梁湘蘭,易子群,等. 湖北山楂葉綠體基因組密碼子偏好性分析[J].分子植物育種,2023,21(8):2508-2516.
WU X J, LIANG X L, YI Z Q, et al .. Analysis of codon bias in the chloroplast genome of Crataegus hupehensis sarg. (rosaceae) [J].Mol. Plant Breeding, 2023, 21(8):2508-2516.
[13] 郭松,梁湘蘭,黃青青,等. 紫九牛葉綠體基因組密碼子偏好性分析[J].廣西植物,2022,42(48):1426-1432
GUO S, LIANG X L, HUANG Q Q, et al .. Codon usage bias in the chloroplast genome of Ventilago leiocarpa [J]. Guihaia,2022, 42(8):1426-1432.
[14] 李曉娟,楊振艷,黃玉玲,等. 藥用植物華重樓(黑藥花科)葉綠體全基因組研究[J]. 熱帶亞熱帶植物學報,2015,23(6):601-513.
LI X J, YANG Z Y, HUANG Y L, et al .. Complete chloroplast genome of the medicinal plant Paris polyphylla var. Chinensis (melanthiaceae) [J]. J. Tropical Subtropical Bot., 2015, 23(6):601-513.
[15] 辛雅宣,董張宏,瞿紹宏,等. 杜梨葉綠體基因組密碼子偏好性分析[J].河北農業大學學報,2020,43(6):51-59.
XIN Y X, DONG Z H, QU S H, et al .. Analysis on codon usage bias of chloroplast genome in Pyrus betulifolia Beg [J]. J. Hebei Agric. Univ., 2020, 43(6):51-59.
[16] 嚴其偉,梁湘蘭,陶秋云,等. 老班瑤藥牛耳風葉綠體基因組特征及其密碼子使用偏好性分析[J]. 中國農業科技導報,2022,24(8):74-86
YAN Q W, LIANG X L, TAO Q Y, et al .. Analysis of chloroplast genome characteristics and codon usage bias of Fissisfigma polyanthum Merr [J]. J. Agric. Sci. Technol., 2022,24(8):74-86.
[17] LI Y, CHONG F, CAI M M, et al .. Complete chloroplast genome sequence of Amomum villosum and comparative analysis with other Zingiberaceae plants [J]. Chin. Herbal Med., 2020, 12(4):375-383.
[18] 朱沛煌,陳妤,朱靈芝,等.馬尾松轉錄組密碼子使用偏好性及其影響因素[J].林業科學,2020,56(4):74-81.
ZHU P H, CHEN Y, ZHU L Z, et al .. Codon sage bias and its influencing factors in Pinus massoniana transcriptome [J]. Sci.Silvae Sin., 2020, 56(4):74-81.
[19] 吳妙麗,陳世品,陳輝.竹亞科葉綠體基因組的密碼子使用偏性分析[J].森林與環境學報,2019,39(1):9-14.
WU M L, CHEN S P, CHEN H. Condon preference of chloroplast genome of Bambusoideae [J]. J. For. Environ.,2019, 39(1):9-14.
[20] 陸奇豐,駱文華,黃至歡.兩種梧桐葉綠體基因組密碼子使用偏性分析[J].廣西植物,2020,40(2):173-183.
LU Q F, LUO W H, HUANG Z H. Codon uasage bias of chloroplast genome from two species of Firmiana marsili [J].Guihaia, 2020, 40(2):173-183.
[21] 林濤,倪志華,沈明山,等.高頻密碼子分析法及其在煙草密碼子分析中的應用[J]. 廈門大學學報(自然科學版),2002,41(5):551-554.
LIN T, NI Z H, SHEN M S, et al .. High frequency codon analysis and its application in tobacco codon analysis [J]. J.Xiamen Univ. (Nat. Sci.), 2002,41(5):551-554.
[22] 沈宗芳,陸添權,張志榮,等.槲蕨屬葉綠體基因組密碼子偏好性分析[J].廣西植物,2021,41(2):266-273.
SHEN Z F, LU T Q, ZHANG Z R, et al .. Codon preference of chloroplast genome of Drynaria [J]. Guihaia, 2021, 41(2):266-273.
[23] 宋菊,龍月紅,林麗梅,等.五加科植物葉綠體基因組結構與進化分析[J].中草藥,2017,48(24):5070-5075.
SONG J, LONG Y H, LIN L M, et al .. Chloroplast genome structure and evolutionary analysis of Araliaceae [J]. Chin.Herbal Med., 2017. 48(24):5070-5075.
[24] 劉潮,李敏,任怡園,等.人參屬植物葉綠體基因組特征及其進化的研究[J].福建農業學報,2022,37(7):886-896.
LIU C, LI M, REN Y Y, et al .. Studies on the characteristics and evolution of chloroplast genomes in Panax [J]. Fujian J.Agric., 2022, 37(7):886-896.
[25] 王琳璇,王高烽,谷威,等.2種羌活藥用植物的葉綠體基因組密碼子使用偏性分析[J].安徽農業科學,2022,50(3):95-101.
WANG L X, WANG G F, GU W, et al .. Codon usage bias in chloroplast genomes of two medicinal species of Notopterygium H. de Boissieu [J]. J. Anhui Agric. Sci., 2022, 50(3):95-101.
[26] 楊祥燕,蔡元保,譚秦亮,等.菠蘿葉綠體基因組密碼子偏好性分析[J].熱帶作物學報,2022,43(3):439-446.
YANG X Y, CAI Y B, TAN Q L, et al .. Analysis of codon usage bias in the chloroplast genome of Ananas comosus [J]. Chin. J. Trop. Crops, 2022, 43(3):439-446.
[27] 石巖碩,趙永星.人參屬植物葉綠體基因組密碼子偏好性分析[J].分子植物育種,2022,20(19):6350-6361.
SHI Y S, ZHAO Y X. Analysis of codon preference in chloroplast genome of Panax ginseng [J]. Mol. Plant Breeding,2022, 20(19):6350-6361.
[28] 梁皓輝,符虹宇,黎釗坪,等.小球藻葉綠體基因組密碼子偏好性分析[J].分子植物育種,2020,18(17):5665-5673.
LIANG H H, FU H Y, LI Z P, et al .. Analysis on codon usage bias of chloroplast genome from Chlorella [J]. Mol. Plant Breeding, 2020, 18(17):5665-5673.

