基于遺傳算法的高維子空間聚類算法設計
黃白梅,章 政
(武漢科技大學 信息科學與工程學院,湖北 武漢 430081)
在現實世界中,高維數據占據著主導地位,例如文檔數據、WEB數據、基因微陣列數據、網絡通信數據等數據經常達到上千維甚至更高。在對高維數據進行聚類時,由于受到“維數災難效應”[1-2]的影響,同時高維數據空間中的一些不相關屬性維掩蓋了要尋找的目標簇,使得傳統的聚類算法在高維數據空間上進行聚類分析時失效。因此需要高維降維去掉不相關屬性維,通過對解空間中的全部屬性子集進行搜索,進而找到最密集的優良子集,再在低維空間中進行聚類分析。
傳統的搜索算法諸如貪婪算法等,在對其進行聚類分析時非常容易陷入局部最優解的困境而達不到理想的要求。遺傳算法是一種自適應全局優化的概率搜索算法,它在理論上可以克服局部最優解而搜索到全局最優解,因此被廣泛的應用于解決復雜的優化問題。
文中針對高維數據的特點,為了降低“維數災難效應”對聚類結果的影響,構建了一個基于遺傳算法進行高維數據聚類的框架,利用遺傳算法的全局搜索能力,挖掘高維數據空間中密集度高的數據子集,然后再在這個子集上進行聚類分析。
1 子空間聚類概述
目前對高維數據進行聚類的方法主要還是基于子空間聚類和全空間降維這兩個方面。
子空間聚類的方法(Sub-space Clustering)[2-5]是屬性子集選擇的一種擴展,在高維聚類方面顯示出了其獨有的優勢。它的基本思想是基于不同子空間可能包含不同的、有意義的類或簇,它在相同的數據集的不同子空間中搜索類或簇群,因此可以通過抽取出存在于子空間的類或簇來進行聚類分析。子空間聚類為每個類或簇搜索出其對應的子空間。根據搜索策略的不同,可以將子空間聚類方法劃分成為兩大類:自底向上的搜索方法(如 CLIQUE算法[3])和自頂向下的搜索方法(如PROCLUS算法[4])。也有一些算法結合自底向上的搜索方法和自頂向下的搜索方法(如 DOC算法[5])。子空間聚類方法對于處理不同類或簇存在于不同子空間里的高維數據結構模型比較有效,不過這類方法的計算復雜性非常高。
全空間降維則是通過縮減維數將高維數據空間歸約到較低維數據空間,然后再通過傳統的方法進行聚類分析,這類方法是在一個特征子空間里面尋找所有的類或簇,但是忽略了在高維數據空間里面不同的類或簇可能有不同的特征子空間。
這兩種聚類方法都有其優點與不足,目前尚沒有一種算法能夠適用于所有的情況,在實際應用中,我們應該根據具體問題的特點來選擇合適的聚類算法。同時由于在處理大規模高維數據時,容易陷入局部最優解的狀況。因此經常采用將各種全局優化搜索算法,如遺傳算法,粒子群算法,蟻群算法等,結合子空間聚類或者降維來處理的策略,達到最終尋找到最優解。遺傳算法(GA)[6],是通過模擬孟德爾.摩根的群體遺傳學說、達爾文的生物進化論的自然選擇和遺傳學機理的生物進化過程而形成的一種自適應全局優化概率搜索算法。它是一種高效的全局優化搜索算法,已被許多研究者應用到聚類分析中。
2 基于遺傳算法的子空間聚類算法
基于遺傳算法進行高維聚類的新算法利用遺傳算法的全局搜索能力對高維數據的特征空間進行搜索,因此其基本流程跟傳統的遺傳算法大致相同[7]。組成種群的個體由特征維和類中心點兩部分經過編碼后組成,每個個體對應著一個特征子空間;適應度值是遺傳算法對搜索進行評估的唯一依據,新算法中的適應度值表示個體所代表的特征子空間進行聚類的效果,適應度值越大,表明子空間數據對象的密集性越強,聚類越好。
2.1 編碼與初始化
常用的編碼方式有二進制編碼和實數編碼等,由于二進制編碼的染色體長度相對較長,且編碼的種群穩定性比實數編碼要差,文中選取實數編碼。個體的編碼空間[8]由(SUB,CEN)這兩部分組成,其中SUB代表特征子空間的實數編碼串,CEN代表類中心的實數編碼串。初始種群我們采用隨機生成的策略。隨機的選取(最大的特征維數目)個特征維和(最大的類數目)個數據對象進行編碼組成個體,然后迭代(預設的初始種群的規模)次,即完成初始種群的產生。
初始種群隨機生成的方案如下:隨機的在數據對象集中選取m個特征維的編號和n個數據對象的編號來進行編碼,構成初始染色體。例如某數據對象集有10維,由150個數據對象組成。取m=4,n=3,則一個染色體的基因即由4個特征維的編號和3個數據對象的編號組成。染色體的左部分基因表示由第5,8,3,2等4個特征維組成,染色體的右部分基因表示由該數據集的第32,12,50個數據組成,兩部分共同構成了一個染色體。通過編碼即完成了染色體的構造。
2.2 適應度函數的設計
適應度值是遺傳算法進行搜索的唯一依據,因此適應度函數設計的好壞直接影響著算法的搜索方向及收斂程度。本文基于類內距離,類間距離和信息熵提出了一種新的適應度評估函數。
在高維數據聚類中,目標簇通常只跟某些特征維有關,為了考察特征維在子空間聚類中所表現出來的性能,本文提出用特征維對子空間聚類的貢獻率來表征。
假設某子空間中含有 K個以{C1,C2,…,CK}為中心的類{A1,A2,…,AK},對每一個類 Ai(i=1,2,…,K),考慮 3 個函數:
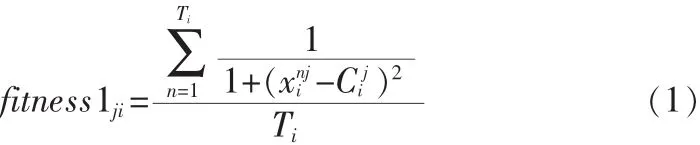
在這里,T表示數據集的數據對象個數,Ti表示數據集的第i個類的數據對象的個數,表示第i個類的中心點的第j維值,表示第n個類內第n個數據對象的第j維值。
fitness1ji體現了第j維對類 Ai的類內貢獻率-越小,表示第i類內某個數據對象的第j維值與中心點的第j維值距離越接近,fitness1ji就越大。則類Ai在特征維j上是稠密的,即稱維j對類i的貢獻大,反之,稱維j對類的貢獻小。
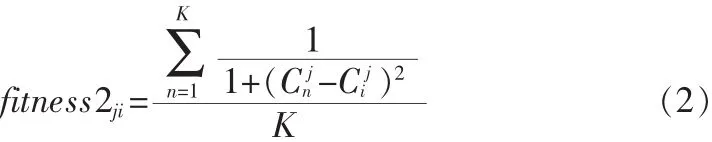
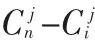

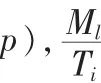


故維j對類i的貢獻率為:
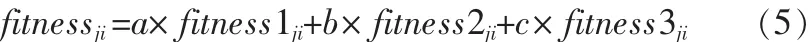
維j對此子空間聚類的貢獻率:

染色體(特征子空間)的適應度值:
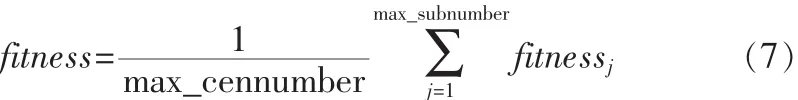
其中max_cennumber表示最大的特征維數目,max_cennumber表示最大的類數目a,b,c。為常數(在這里根據先驗知識取 a=1,b=-0.5,c=0.8)。
2.3 遺傳算子及控制參數
遺傳操作是遺傳算法的核心部分,遺傳操作有3個操作算子:選擇算子、交叉算子和變異算子,父代種群通過遺傳操作產生出子代種群來繁衍和進化[9]。
選擇算子:文中依據上述適應度函數作為選擇的依據,保留部分適應度函數值高的優良個體(根據先驗知識預先設定),進入到子代進行繁殖,然后采取輪盤賭選擇法,根據適應度函數值的大小選擇剩下的個體[8]。
交叉算子:交叉算子的主要參數是交叉概率pc,取pc?[0.4,0.9],根據pc對父代個體進行單點交叉操作[8],在基因位上通過互換基因,生成兩個新的子代個體。
變異算子:文中取基本位變異法[9],按照預設的變異概率pm進行變異操作,在基因位上對原基因值進行突變替換。pm取 0.01~0.2
迭代終止條件:采用世代數是否超過預設的參數值[11]的方法來作為遺傳算法終止的條件。
3 實驗對比與分析
為了驗證文中提出的基于遺傳算法進行高維聚類的新算法的聚類效果和性能,采用了一組真實數據集來進行實驗。在實驗中我們選取了經典的聚類算法k-means算法,子空間聚類算法PROCLUS[11]算法以及基于遺傳算法進行高維數據數據聚類的降維算法GA-HDclustering算法[8]與本文提出的算法進行了比較。通過比較錯誤率 (Error_degree),熵(Entropy)值,純度(Purity)值,Rand 統計量(RI)值這幾項指標的評判值來對其聚類結果進行評估和比較。
為了檢驗文中算法在實際高維數據中的有效性,選取了一組真實的數據集來進行實驗。數據集來自UCI機器學習的數據集(http://archive.ics.uci.edu/ml/),如表 1。

表1 真實數據集Tab.1 Real data set
該數據集最初是用來對乳腺癌病進行預測和診斷的。wdbc數據集記錄了569位女性的乳房腫塊的30個特征值,這些特征值是通過乳房腫塊的細針抽取數字圖像而計算出來的,它們體現了圖像中細胞核的特征。根據這30個特征值,可以將這569位女性分為兩類,一類患有乳腺癌者,共有212人;另一類未患乳腺癌者,共有357人。數據集客觀上分為兩類。
對于wdbc數據集,對其使用k-means算法、PROCLUS算法、GA-HDclustering算法以及文中提出的基于遺傳算法進行高維聚類的新算法分別進行實驗。運行GA-HDclustering算法和基于遺傳算法進行高維聚類的新算法時,為了便于比較,將需要設定的相關參數,此處取pc=0.8,pc=0.02,max_subnumber=25,max_cennunber=2,popsize=80,max_gen=350。
對這組數據集分別運行k-means算法、PROCLUS算法、GA-HDclustering算法以及基于遺傳算法進行高維聚類的新算法,計算各自的錯誤率并統計熵值、純度值以及Rand統計量值這3個有效性衡量指標,具體的實驗結果與分析如下。
數據集客觀上分為兩類,共有569個數據對象,每個數據對象有30維,各類分布如下:(各類數據對象數目)Class1:357,Class2:212。
各算法在對wdbc數據集聚類對應的特征子空間如表2所示。

表2 數據集的聚類特征子空間Tab.2 Clustering feature sub-space of
我們將每個算法的錯誤率標記為Error。k-means算法,PROCLUS算法,GA-HDclustering算法和基于遺傳算法進行高維聚類的新算法的錯誤率如表3所示。

表3 各個算法的錯誤率Tab.3 Error rate of each algorithm
通過上面4個算法對數據的聚類結果,看到基于遺傳算法進行高維聚類的新算法的錯誤率為0.143 2,比K-means算法的0.151 6和PROCLUS算法的0.153 4這兩個經典算法的總錯誤率要小得多,比GA-HDclustering算法的總錯誤率0.151 2也明顯偏小,聚類的精確性最高。
各算法在對該數據集進行聚類時,對應的熵值,純度,RAND統計量如表4所示。
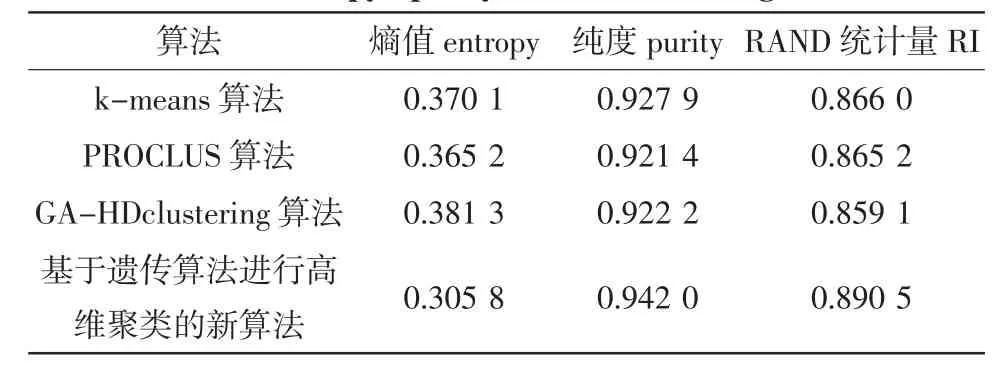
表4 各個算法對應的熵值、純度和RAND值Tab.4 Entropy,purity and RI of each algorithm
實驗結果說明,K-means算法和PROCLUS算法的熵值優于GA-HDclustering算法,K-means算法的純度和 RI值比PROCLUS算法和GA-HDclustering算法也略高。而基于遺傳算法進行高維聚類的新算法的熵值比這3個算法大幅度降低,純度和RI值也比這3個算法明顯提高。
4 結束語
文中提出的算法能夠高效地對高維數據進行降維,找到有效的特征子空間進行聚類;并且對數據進行聚類結果的錯誤率和評估指標Entropy值、purity值及RI值與其他算法相比,精確性和魯棒性更強;綜上所述,文中提出的算法能夠有效地進行高維數據聚類,降低“維數災難效應”的影響,是一種行之有效的高維數據聚類方法。但同時也存在一些問題值得進一步深入研究,文中提出的算法中的各個參數一般都是根據經驗及多次試驗進行設定的,下一步考慮研究各參數及其自動設置,另外該算法在對非特定的真實數據進行聚類時的魯棒性仍有待提高。
[1]Donoho D L.High-dimensional data analysis:the curses and blessings of dimensionality[C]//Aide-Memoire of a Lecture at AMS Conference on Math Challenges of 21st Century,2000.
[2]Parsons L,Haque E,Liu H.Subspace clustering for high dimensional data:a review[J].SIGKDD Explorations,2004,6(1):90-105.
[3]Agrawal R,Gehrke J,Gunopulos D.Automatic subspace clustering ofhigh dimensionaldata fordata mining applications[J].In Proc.ACM-SIGMOD Int.Conf.Management of Data (SIGMOD'98),(Seattle, WA),1998:94-105.
[4]Aggarwal C C,Procopiuc C.Fast algorithms for projected clustering[Z].In Proc.of ACM SIGMOD,1999.
[5]Procopiuc C M,Jones M,Agarwal P K,et al.A monte carlo algorithm for fast projective clustering[Z].Proc.ACM SIGMOD,2002.
[6]Galletly J E.An overview of genetic algorithms[J].Kybernetics,1992, 21(6):26-30.
[7]周明,孫樹棟.遺傳算法原理及應用[M].國防工業出版社,1999.
[8]孫浩軍,熊瑯環.一種高維數據聚類遺傳算法[J].計算機科學與工程,2010,32(8):94-98.
SUN Hao-jun,XIONG Lang-huan.A genetic algorithm for hign-domensional data clustering[J].Computer Science and Engineering,2010,32(8):94-98.
[9]潘正君,康立山,陳毓屏.演化計算[M].北京:清華大學出版社,1998.
[10]何宏,譚永紅.一種基于動態遺傳算法的聚類新方法[J].電子學報,2012,2(2):254-260.
HE Hong,TAN Yong-hong.A novel clustering method based ondynamicgeneticalgorithm[J].ChineseJournalofElectronics,2012,2(2):254-260.
[11]Aggarwal C C,Procopiuc C.Fast algorithms for projected clustering[Z].In Proc.of ACM SIGMOD,2004.

