一種基于信息容量的模體比較非比對度量算法
郭麗娟,張少強,花季偉
(天津師范大學計算機與信息工程學院,天津300387)
破譯基因組中復雜的基因調控網絡是一項極具挑戰性的課題[1-2],要實現這個目標,首先要在基因組中鑒別所有轉錄因子的結合位點[1,3-4].轉錄因子結合位點是轉錄因子結合的一組短基因序列,長度通常為6~25個堿基對(base pair,bp),具有觸發細胞轉錄調控的功能.屬于同一轉錄因子的結合位點通常具有特定的保守性和相同的長度,但它們也可以顯示出一定程度的變異,而這些結合位點位于一段非常長的非編碼序列中,這些都會導致它們的預測計算變得非常困難.同一轉錄因子的一組具有高保守性的結合位點通常被稱作模體,可以由實驗驗證,或者通過比較一組可能含有轉錄因子結合位點的方法預測.由于轉錄因子結合位點比它們周圍的DNA片段更保守,所以,許多從頭測序模體查找算法被開發出來用于識別轉錄因子結合位點.模體可以由位置賦權矩陣(position weight matrix,PWM) 和位置頻率矩陣(position frequency matrix,PFM)精確地表述出來[5-6].這2個矩陣是模體結合位點序列比對的變形,它們極大程度上反映了相應的轉錄因子的位置結合傾向性.因此,通過在這2個矩陣中掃描可能包含TFBS的序列即可以發現模體.
利用模體查找工具獲得一些假定的模體后,通過在一個模體數據庫中找到與假定模體匹配的模體,從而推斷出轉錄因子附屬于這些假定的模體[7];或聚類相同轉錄因子的相似子模體從而去除冗余模體、形成一個完整的模體.因此,在上述提到的應用中,需要一種有效的度量法用于捕獲不相關模體之間的細微差別,強調同種群間模體的相似度.目前,計算模體相似度的比對度量方法包括兩大類:一類是列相似度度量法,即從2個模體的位置頻率矩陣(或者位置賦權矩陣)中各取一列計算相似性,如SSD(sum of squared distances)[8-9]、pCS (p-value of Chi-square)[10]、ALLR (average log-likelihood ratio)[11]、AKL(average Kullback-Leibler,AKL)[12]和 PCC(pearson’scorrelation coefficient,PCC)[13]等;另一類是雙序列比對算法,利用列相似度度量法和一個空位罰分函數作為分數比對2個模體[14],在假定具有空位罰分函數的情況下,Needleman-Wunsch 算法[15]和 Smith-Waterman(SW)算法[16]都可用于查找最優比對.文獻[7]和文獻[17]對這些度量和比對算法進行評估后,建立了網絡服務器STAMP,用于集成這些帶有比對的度量法.除此之外,還有2個用于比較模體的非比對度量方法KFV(kmer frequency vector) 和 Mosta 包 的 AC(Asymptotic Covariance),它們分別由文獻[7]和文獻[15]提出.
上述度量方法僅用到了位置頻率矩陣,均沒有使用列信息容量(column information contents)和位置賦權矩陣.實際上,由于矩陣中所對應的列具有很高的相關性,2個總體信息容量低的模體也可能具有高的相似度分數.因此,如果2個模體某些列的信息容量很低,在應用這些度量方法比對前,低信息容量的列就要被刪除.上述帶有比對的度量方法在聚類相似模體方面具有較好的效果,但是它們基本不能從帶有低信息容量列的混雜模體中分離出真模體.此外,帶有比對的度量公式需要用到比對算法,而比對算法依賴的參數較多,運行所需時間也較多,因此基于非比對的度量公式可以更快速精確地進行模體比較.綜上所述,本研究提出一種帶有位置信息容量的相似度非比對度量法(information contents based similarity metric,ICBSM).算法中不僅包含位置頻率矩陣和位置賦權矩陣,還加入了每個位置的信息內容,并利用來自于 STAMP[17]、KFV[14]和 GLECLUBS[18-19]中的數據集對ICBSM算法進行評估,將該算法與國際上已經提出的算法進行比較分析.
1 ICBSM方法
1.1 ICBSM度量法的提出
設模體Motif由n個長度為L的序列組成,定義其位置頻率矩陣為

式(3)用于表示PWM1生成PFM2的可能性,其中 Alignment(1,2)是通過固定 PWM1、滑動 PFM2得到的矩陣列的比對.圖1為PFM在PWM上的滑動示意圖.

圖1 PFM在PWM上的滑動示意圖Fig.1 Representation of PFM sliding on PWM
由圖1可以看出,當用PFM在PWM上逐列由左向右滑動形成比對s時,在該比對s中,PWM的第i列與PFM 的第 s(i)列對應.
再用Motif2的PFM2和Motif1的PWM1進行比對,計算相似性:

1.2 算法驗證
為了驗證算法的性能,利用經過驗證的3個數據集對ICBSM進行測試和評估.數據集-1由Mahony等[3]從JASPAR庫中首次選出,該數據庫由96個真實的模體組成,這些模體屬于13個已知的不同結構的TF類.文獻[6]創建了數據集-2,用以測試KFV度量法對于識別冗余的位置頻率矩陣的顯著性能.該數據集由124個JASPAR的核心模體及每個核心模體的3個子模體組成,這些子模體通過隨機選取每個模體的2/3序列得到.數據集-3可由http://gleclubs.uncc.edu/pbs頁面下載,包含了大約105個假定的模體[18-19],這些模體來自大腸桿菌2000多組全基因組的同源基因間序列以及其他54個γ-變形菌門的參照基因組.關于3個數據集的詳細參數參見表1.

表1 用于測試與評估的3個數據集的參數Tab.1 Parameters of three datasets for testing and assessing
將ICBSM算法、STAMP工具包中的5個算法(ALLR,AKL,SSD,pCS,PCC)、KFV 法以及 Mosta算法中的AC應用到1個數據集上,針對聚類相關的真模體、過濾偽模體和找回模體等方面進行性能比較.利用STAMP平臺計算5個依靠比對的度量法得分(http://www.benoslab.pitt.edu/stamp/),Mosta包計算AC得分(http://mosta.molgen.mpg.de),KFV的網絡服務器計算KFV得分(http://bioinfo.uncc.edu/kfv/).
1.2.1 模體找回
帶有比對算法的列相似度度量法和非比對相似度度量法可以用于將待查模體與數據庫中的每一個模體進行比較,從而找回模體.如果在1個數據集中模體的相似度分數超過閾值,則表明這些模體被待查模體“命中”;如果有多個“命中”[6],則相似度分數最高的“命中”稱為“最佳命中”.通過使用“最佳命中法”把在數據庫中搜索模體的正確找回率定義為度量法的“性能精確度”.
與其他3個帶有最優比對[6]的列相似度度量法和非比對度量的AC法[14]相比,SSD、PCC和KFV度量法在查找模體時具有更高精確度,因此選出它們與ICBSM度量法進行對比,比較它們在數據集-1中找回同一個轉錄因子家族模體的能力.在數據集-1上,STAMP包的5個帶有比對設置的列相似度度量法中,結合SW的非空位比對算法PCC(PCC/SWU)和結合SW、空位延伸為0.5、空位開放為1的比對算法SSD/SW是最好的2個度量法和比對設置[7].根據文獻[6]的描述,當把4-mer和夾角余弦值用于向量構建和比較時,KFV會獲得最優結果.
本研究利用 ROC(receiver operating characteristic)曲線考察度量法在數據集-1和數據集-2中識別出相同轉錄因子的模體的性能.ROC曲線的繪制方法依據下述規則:給定1個由n個模體組成的數據集,其中這些模體的轉錄因子結構類已知,n個模體具有n(n+1)/2個組對,應用度量法分別計算出每一對的相似度分數.如果2個模體的相似度分數小于1個閾值或大于閾值但沒有“最佳命中”,則設定這2個模體為錯誤匹配,否則為正確匹配.如果由度量法計算出2個模體正確匹配,且這2個模體確實同屬于1個轉錄因子,則該正確匹配稱為“真陽性(true positive,TP)”,否則這個正確匹配為“假陽性(false positive,FP)”;如果2個模體由度量法計算出是錯誤匹配,且這2個模體確實屬于不同的轉錄因子,則該匹配稱為“真陰性(true negative,TN)”,否則這個錯誤匹配為“假陰性(false negative,FN)”.ROC曲線是在不同的模體相似度閾值下由真陽性率對比假陽性率的描述.
1.2.2 從混雜的模體中分離出真模體
一些基于遺傳系譜印技術的轉錄因子綁定位點的全基因組測序算法需要把任意轉錄因子的子模體和冗余模體合并成一個獨立的模體并剔除偽模體[8-9,13],即聚類相似模體,區分出不相關的模體.因此,研究人員需要一個不僅能精確計算出一對模體的精確度,而且還能有效區分出無關模體的度量法,這個算法可以為相同轉錄因子模體的2個子模體賦予足夠高的相似度值,為沒有任何進化關系的2個模體賦予足夠低的相似度值,從而在混雜的模體中分離出真模體.由GLECLUBS生成的數據集-3[8]由大量的混雜模體和一小部分的真模體構成,為從數據集-3中發現真模體,在Regulon數據庫中選出一組真模體用于在數據集-3上進行評估.該組真實模體是大腸桿菌的122個轉錄因子模體生成的大量的真的子模體.每個轉錄因子模體均是由n個結合位點構成(n≥3),度量法把每個轉錄因子模體隨機分成1個大小為k的子模體和1個大小為(n-k)的子模體,其中 k∈{1,2,…,[n/2]}.因此,每個大小為n的模體都可以生成[n/2]對的子模體.度量法對每個大小為k的子模體重復前面的分離過程,生成[k/2]對子模體的子模體.當每個子模體的大小為1時,過程停止.然后,利用這些度量法計算每對子模體間的相似度值[7,11],并在數據集-3上計算每對模體的相似度值.通過計算數據庫-3中每對模體相似度分數標準化后的分布和每對真的子模體的相似度分數標準化后的分布,查看2個分布的重疊區域.
2 ICBSM算法性能分析與結果
2.1 模體找回
對于從一個數據集中找回模體,本研究將模體比較的閾值設置為0.6,然后將ICBSM、KFV、PCC/SWU和SSD/SW算法在數據集-1上計算精確度,結果如表2所示.
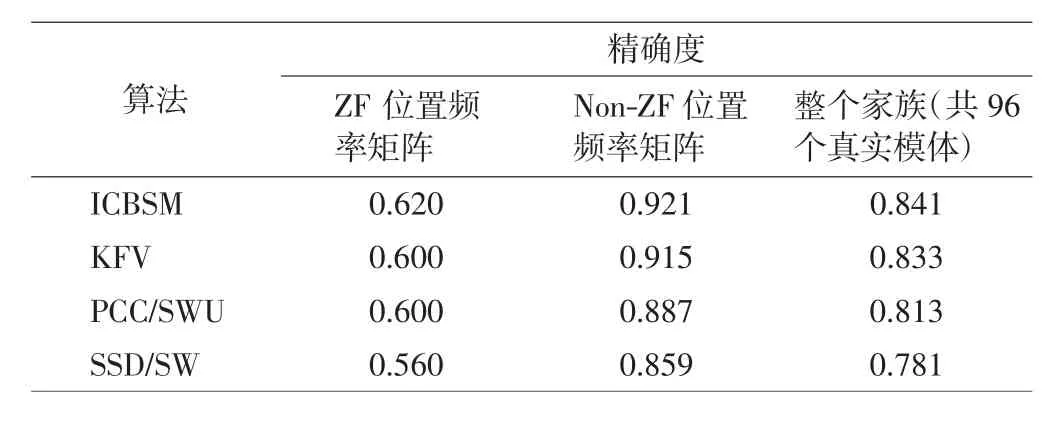
表2 在數據集-1上,ICBSM、KFV、PCC/SWU及SSD/SW模體找回的精確度Tab.2 Accuracy for searching motifs of ICBSM,KFV,PCC/SWU and SSD/SW on dataset-1
數據集-1可以分為包含25個真實模體的鋅指狀結構蛋白質家族(zinc-finger,ZF)和包含71個真實模體的非鋅指狀結構蛋白質家族(non-ZF).由表2中結果可知,對于ZF蛋白質家族、Non-ZF蛋白質家族以及整個蛋白質家族集合,ICBSM算法的模體找回精度最高,說明該算法在數據庫中能夠正確找回模體的能力最強,比其他3種度量法具有更卓越的策略.
為了將ICBSM與PCC/SWU、KFV(4-mer夾角余弦值)的最優策略做進一步比較,在模體比較閾值設置為0.6的情況下,在數據集-1和數據集-2上,對這3種策略的性能進行ROC分析,結果如圖2所示.由圖2可知,假陽性率相同的情況下,ICBSM度量法的真陽性率最高,即對于同1個數據集,ICBSM度量法能夠正確找回模體的能力比其他2種方法更強.
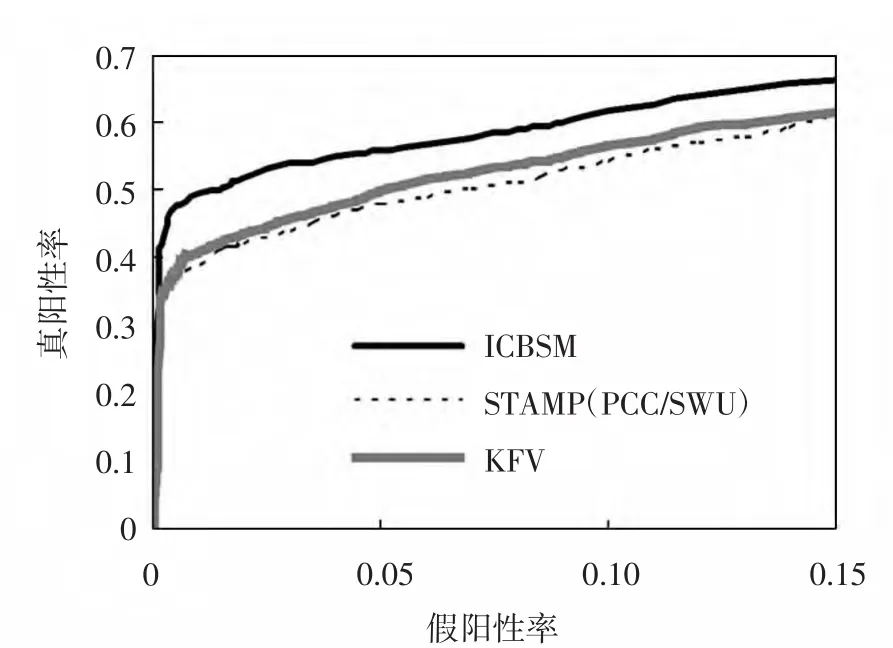
圖23 種度量法的ROC曲線圖Fig.2 ROC curves of three metrics
2.2 從混雜的模體中分離出真模體
用ICBSM度量法、STAMP工具包、AC度量法以及KFV度量法分別計算數據集-3的每對模體相似度分數以及每對真的子模體的相似度分數,并將這2個分數標準化形成曲線分布圖,以ICBSM度量法與AKL度量法曲線分布效果為例,結果如圖3所示.
圖3中“數據集-3模體”的曲線是在數據集-3中計算每對模體相似度分數標準化后的分布曲線,標有“真的子模體”曲線是每對真的子模體的標準化相似度分數的分布曲線.在數據集-3中,由于每對真的子模體具有相關性而大多數模體具有無關性,因此性能好的度量法應該可以把前一個相似度分布區域與后一個相似度分布區域分離出來,即圖3中2個曲線所圍成的2個區域的重疊部分越小,分離效果越好.ICBSM在計算模體的相似度分數時考慮了信息容量,因此可以從帶有低信息容量的混雜的模體中分離出真模體.
將ICBSM與其他度量法生成的相似度分布曲線的重疊區域比率進行比較,結果如圖4所示.在ICBSM的分布曲線下,2塊區域具有最小的重疊部分,這說明與其他度量法相比,ICBSM能夠更加精確地從混亂模體中分離出真模體.
3 結論
在生物信息處理過程中,由于很多應用都包含了模體比較的過程,因此提出一種基于列信息內容的用于模體比較的非比對度量法ICBSM,通過對比分析,結果表明:
(1)ICBSM度量法采用了帶有信息容量的非比對策略計算模體間的相似度分數,將信息容量添加到模體的位置賦權矩陣上,將一個模體的位置頻率矩陣在另一個模體的位置賦權矩陣上滑動,計算2個模體間的相似度.該算法依賴參數少,提升了計算效率.
(2)在模體比較的閾值設置為0.6的情況下,在數據集-1上,ICBSM度量法與KFV、PCC/SWU及SSD/SW相比較,其模體找回的精確度最高;同時,與KFV、PCC/SWU相比較,ICBSM的ROC曲線的真陽性率值也最高,這說明該方法在數據庫中找回模體的效果更好.
(3)由于ICBSM在計算模體相似度時考慮了模體的信息容量,因此它計算出的真的子模體的相似度分數標準化后的分布曲線與數據集中所有模體的相似度分數標準化后的分布曲線重疊率最低,說明該方法能夠精確地將真模體從混雜的模體中區分出來,為聚類相似模體、分組不相關模體提供了有效工具.
[1]CELNIKER S E,DILLON L A,GERSTEIN M B,et al.Unlocking the secrets of the genome[J].Nature,2009,459(7249):927-930.
[2] RISTER J,DESPLAN C.Deciphering the genome's regulatory code:the many languages of DNA[J].Bioessays,2010,32(5):381-384.
[3] REED J L,FAMILI I,THIELE I,et al.Towards multidimensional genome annotation[J].Nat Rev Genet,2006,7(2):130-141.
[4]ALEXANDER RP,FANG G,ROZOWSKY J,SNYDER M,et al.Annotating non-coding regions of the genome[J].Nat Rev Genet,2010,11(8):559-571.
[5] GUHATHAKURTA D.Computational identification of transcriptional regulatory elements in DNA sequence[J].Nucleic Acids Res,2006,34(12):3585-3598.
[6] STORMO G D.DNA binding sites:representation and discovery[J].Bioinformatics,2000,16(1):16-23.
[7]MAHONY S,AURON PE,BENOS P V.DNA familial binding profiles made easy:comparison of various motif alignment and clustering strategies[J].PLoS Comput Biol,2007,3(3):61.
[8]SANDELIN A,WASSERMAN W W.Constrained binding site diversity within families of transcription factors enhances pattern discovery bioinformatics[J].J Mol Biol,2004,338(2):207-215.
[9] WANG T,STORMO G D.Identifying the conserved network of cisregulatory sites of a eukaryotic genome[J].Proc Natl Acad Sci USA,2005,102(48):17400-17405.
[10]SCHONES D E,SUMAZIN P,ZHANG M Q.Similarity of position frequency matrices for transcription factor binding sites[J].Bioinformatics,2005,21(3):307-313.
[11]WANG T,STORMO G D.Combining phylogenetic data with coregulated genes to identify regulatory motifs[J].Bioinformatics,2003,19(18):2369-2380.
[12]KULLBACK S,LEIBLER R A.On information and sufficiency[J].Ann Math Statist,1951,22(1):79-86.
[13]PIETROKOVSKI S.Searching databases of conserved sequence regions by aligning protein multiple-alignments[J].Nucleic Acids Res,1996,24(19):3836-3845.
[14]XU M,SU Z.A novel alignment-free method for comparing transcription factor binding site motifs[J].PLoS One,2010,5(1):87-97.
[15]NEEDLEMAN S B,WUNSCH C D.A general method applicable to the search for similarities in the amino acid sequence of two proteins[J].J Mol Biol,1970,48(3):443-453.
[16]SMITH T F,WATERMAN M S.Identification of common molecular subsequences[J].J Mol Boil,1981,147(1):195-197.
[17]MAHONY S,BENOS P V.STAMP:a web tool for exploring DNA-binding motif similarities[J].Nucleic Acids Res,2007,35:253-258.
[18]ZHANG S,XU M,LI S,et al.Genome-wide de novo prediction of cisregulatory binding sites in prokaryotes[J].Nucleic Acids Res,2009,37(10):72.
[19]ZHANG S,LI S,PHAM P T,et al.Simultaneous prediction of transcri-ption factor binding sites in a group of prokaryotic genomes[J].BMC Bi-oinformatics,2010,11:397.

