重金屬脅迫下厚殼貽貝谷胱甘肽S-轉移酶基因表達分析*
劉慧慧 何建瑜 趙榮濤 薛超波
(1.浙江海洋學院 國家海洋設施養殖工程技術研究中心 舟山 316004;2.舟山市質量技術監督檢測研究院 舟山 316021)
厚殼貽貝(Mytilus coruscus)是一種棲息于海水中的常見雙殼經濟貝類,北至遼寧大連,南至福建東山均有分布,以浙江沿海資源量最大,其軟體部蛋白質含量較高,鮮味氨基酸和必需氨基酸組成豐富,微量元素構成合理,屬營養價值和經濟價值都較高的海產貝類(何建瑜等,2012),已發展為浙江沿海重要的經濟養殖品種。但隨著近年來沿海城市工業化進程的加速,厚殼貽貝繁殖的周邊海域水質、環境因子持續惡化,生境污染不斷加劇,作為一種典型的濾食性潮間帶生物,厚殼貽貝因具有個體較大,生活史較長,活動性差,容易采集,對環境污染物(如重金屬、病原微生物等)具有很強的耐受性等特征,常被作為環境指示生物來監測海區環境的變化。
谷胱甘肽 S-轉移酶(Glutathione S-transferase,GST)是多功能解毒酶超家族中的一員(雷安平等,2009;張妍,2012),主要在抗毒機制第II階段起作用,因此也稱為 II相解毒酶,在生物體內起到抵抗外源物質入侵和細胞毒性防御的作用(Saranya Revathyet al,2012;Umasuthanet al,2012),涉及細胞的去毒及毒素的解毒過程(Masellaet al,2005;Ohnumaet al,2011)。其作用機理是促進還原型谷胱甘肽(Glutathione,GSH)的活性基團—巰基與毒素結合形成親電子試劑(程煒軒等,2009),使親電的疏水化合物變成親水物質,易于從膽汁或尿液中排泄(Liaoet al,2006)。此外,GST通過催化過氧化氫的氧化還原反應,清除體內由代謝反應產生的氧自由基和過氧化物,參與體內免疫反應(羅凱婭等,2012),保護細胞免受外界脅迫造成的損傷(Blanchetteet al,2002;張永國等,2006)。另有研究證實,GST中pi型能參與細胞的增殖調控,并通過各種基因控制的酶在抗氧化反應中協同作用發揮功能(Salinaset al,1999,Fuet al,2012),可以作為生物受環境污染的指示標記物,其表達水平在一定程度上可以有效地反應生物體受環境毒物的損害程度(Saranya Revathyet al,2012),該特征在硬殼蛤(Mercenaria mercenaria)(Blanchetteet al,2002)及太平洋牡蠣(Crassostrea gigas)(Boutetet al,2004)中均有報道。
GST在生命活動過程中有解毒、抗氧化等保護細胞的作用,而且對污染物有敏感反應,常作為生物標志物。厚殼貽貝因對環境污染物具有耐受性,而常被用作污染指示生物來監測海區環境的變化。因此,為研究厚殼貽貝 GST基因在代謝活動中的作用,以及分析該基因在海洋環境污染監測中的指示作用,本文克隆了厚殼貽貝 GST基因,并采用實時熒光定量PCR技術,以其血液中GST為分子標記,監測鎘、銅等重金屬脅迫下 GST基因的相對表達情況,以期為厚殼貽貝的大規模安全養殖及正確運用其 GST分子作為環境污染預警提供指導。
1 材料與方法
1.1 材料
本次實驗成體厚殼貽貝于2012年6月購自舟山水產品市場,平均殼長9.3cm,平均殼高4.6cm,于25°C潔凈海水中暫養1周,每天換新鮮海水,定期投喂微綠球藻(Prochloroccus)和小球藻(Chlorella)。隨后將厚殼貽貝隨機分為2組,每組30只,并立即抽取各組的3只厚殼貽貝血液,提取總 RNA作為對照組,標記為0d。實驗采用 CuSO4·5H2O 和 CdCl2·5H2O 兩種重金屬進行脅迫處理,將兩組貽貝分別置于含銅(Cu2+濃度為20μg/L)和鎘(Cd2+濃度為200μg/L)的10L 海水中,每天換一半含有相應濃度重金屬的海水,定期投喂微綠球藻和小球藻,分別取處理后第5d、10d、15d、20d、30d的厚殼貽貝(每個時間點隨機取3只)血液提取總RNA。
1.2 總RNA提取和cDNA合成
用一次性針筒(5mL)快速從厚殼貽貝閉殼肌抽取血液,立即置于4°C 800r/min離心10min,沉淀用于總 RNA提取,方法按 TaKaRa公司的 Trizol Total RNA提取試劑盒推薦方法進行,獲得的總 RNA以1.5%非變性瓊脂糖電泳檢測,并置于紫外分光光度計(Bio-Rad,USA)下檢測其A260/A280值。以TaKaRa M-MLV RTase cDNA Synthesis Kit試劑盒(TaKaRa)對所提取的RNA進行反轉錄,獲得相應cDNA。
1.3 厚殼貽貝GST基因克隆
根據已知海水雙殼貝類紫貽貝(Mytilus galloprovincialis,AF527010)、地中海貽貝(Mytilus edulis,AY557404)、硬殼蛤(Mercenaria mercenaria,EU 024655)、河蜆(Corbicula fluminea,AY885667)、美洲簾蛤(Mercenaria mercenaria,EU024655)的GST基因通過Primer 5.0軟件在保守區設計兼并引物GST-F和GST-R(表1)。以厚殼貽貝血液 cDNA 為模板,克隆GST 基因。20μL 擴增反應體系:10×PCR Buffer 2μL,Mg2+(25mmol/L)2μL,dNTPs(2.5mmol/L)0.4μL,GST-F(10μmol/L)、GST-R(10μmol/L)各 0.8μL,ddH2O 13μL,Taq酶(5U/μL,TaKaRa)0.4μL,模板 cDNA 0.6μL。PCR擴增條件:95°C預變性4min,94°C變性1min,53°C退火 30s,72°C延伸 45s,循環 35次;最后 72°C 延伸10min。以DL 2000 Marker為標記,1.5 %瓊脂糖電泳檢測PCR產物,選取與預期大小一致的約500bp的條帶用瓊脂糖膠純化試劑盒(TIANGEN)純化后,送上海英濰捷基生物公司測序。
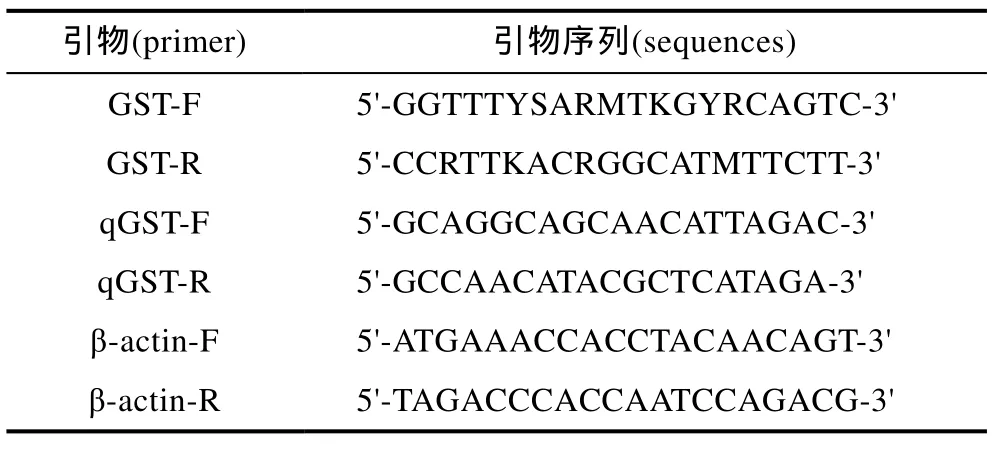
表1 厚殼貽貝GST基因克隆及熒光定量所用的引物序列Tab.1 Primer sequences for GST cloning and real- time quantitive PCR in Mytilus coruscus
1.4 厚殼貽貝GST基因克隆序列分析
將雙向測序獲得的 cDNA序列利用 DNAstar7.0軟件進行拼接,以 Clustal X1.8和 BLASTn(http://www.Ncbi.Nlm.Nih.gov/BLAST/)進行序列同源性比對,在MEGA4.1軟件中采取Neighbor-Joining算法構建系統發育樹,1000次重復計算Bootstrap值(Kumaret al,2004),進行系統發育分析。
1.5 厚殼貽貝GST基因表達實時熒光定量PCR檢測
根據GST基因測序結果設計熒光定量PCR引物qGST-F和qGST-R,采用RT-PCR法,以β-actin為內參,分析 Cu2+和Cd2+脅迫下厚殼貽貝血液中 GST的表達情況(引物 qGST-F、qGST-R、β-actin-F、β-actin-R見表1)。20μL PCR 反應體系:qGST-F(10μmol/L)與qGST-R(10μmol/L)各 0.8μL,2×SYBR?Premix Ex TaqTM Ⅱ(TaKaRa)10μL,cDNA 0.8μL,ddH2O 7.6μL,反應在ABI-7500型PCR儀上進行,采用兩步法進行擴增,即95°C預變性1min,95°C變性10s,59.6°C延伸45s,共40個循環,結束后,從55°C 緩慢升溫到95°C,制備熔解曲線。每次反應都設置陰性對照和無模板對照,每個反應3個重復孔。
1.6 數據處理
GST的相對表達量按用最小二乘法 2-ΔΔCT法計算(Pfaffl,2001),并利用SPSS 13.0進行單因子顯著性差異分析(ANOVA)和t檢驗,分別標記為顯著差異(P<0.05)和極顯著性差異(P<0.01)。
2 結果
2.1 基因擴增及進化發育分析
以反轉錄得到的血液cDNA為模板(總RNA濃度,A260/A280=1.91),用兼并性引物擴增得到一條約500bp的特異性條帶,與預期擴增產物片段大小接近(圖1)。經 3次測序確認后的序列提交 NCBI數據庫獲得GenBank序列號為 KC176684,通過BLAST比對發現,該序列與紫貽貝(AF527010)和地中海貽貝(AY557404)的 pi型 GST基因均有較高的相似性(93%—94%),初步判斷所獲得的GST分子為pi型。選取人、猴、鼠、巨蠣及部分雙殼貝類各種 GST亞型利用MEGA4.1構建厚殼貽貝GST的系統發育樹見圖2,結果發現,Alpha、Zeta、Kappa、Pi、Omega、Mu和Sigma等7種亞型的GST基因之間相互成簇,各聚成一支;且每一種亞型中脊椎動物和無脊椎動物的GST基因也相互分開,各聚成不同的亞支,說明各亞型差別明顯,具有相對獨立的保守結構域。厚殼貽貝GST與pi型GST雙殼貝類位于同一進化支,進一步說明本次實驗獲得的GST基因歸屬于pi型,這與 BLAST的比對結果相似,同時也說明厚殼貽貝GST與人、猴、鼠等在進化上具有較大的差別。
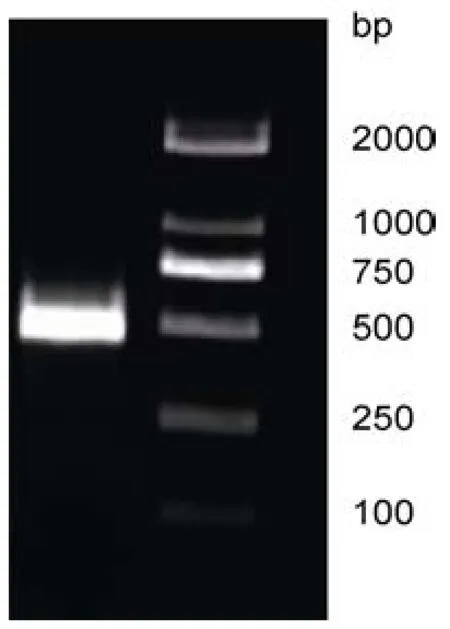
圖1 GST擴增產物電泳圖譜Fig.1 The gel electrophoresis pattern of GST PCR product
2.2 Cu2+及Cd2+脅迫下厚殼貽貝GST基因表達
GST和內參β-actin的擴增曲線均呈“S”形,復孔間擴增曲線重疊,指數區明顯、基線平整,陰性對照和無模板對照無擴增,熔解曲線均為單一峰,擴增效率達 100%±5%,說明定量 PCR反應體系良好,無非特異性擴增,確保相對定量結果的準確性(周向紅等,2011)。檢測發現,厚殼貽貝GST受銅脅迫前幾天一直處于低水平表達,至第10 d表達迅速達到最高,為對照組的6.95倍,接下來的10 d內一直保持相對較高的表達水平,約為對照組的2.5—3.5倍,之后開始下降,30d后GST表達量基本下降至對照組水平。Cd2+脅迫處理后,GST的表達值一直處于逐步上升狀態,15d時達到最高,約為對照組的6.11倍,之后表達逐漸降低,至30 d時仍略高于(1.49倍)對照組。上述結果說明,GST基因參與了重金屬的解毒過程,但是對于不同的重金屬解毒作用稍有不同。
3 討論
3.1 厚殼貽貝GST基因的序列分析
目前已發現的谷胱甘肽 S轉移酶亞型至少有 14種(Wanet al,2008;Umasuthanet al,2012),分別為alpha(α)、beta(β)、delta(δ)、epsilon(ε)、zeta(ζ)、theta(θ)、kappa(κ)、lambda(λ)、mu(μ)、pi(π)、sigma(σ)、tau(τ)、phi(φ)和omega(?)。這些亞型的分類基于對谷胱甘肽S-轉移酶基因N端結構域的氨基酸、結構、抗體反應以及對抑制劑的敏感性(Kimet al,2009)。本次實驗擴增得到厚殼貽貝GST序列經測序和Blastn比對初步判定為pi型,在系統進化樹中與雙殼貝類pi型GST位于同一進化支,與人、猴、鼠等進化上差別較大。此外,上述各種GST亞型之間相互成簇,各聚成一支,該結果與周向紅等(2011)對條斑紫菜 GST基因的分析類似。有學者(林群等,2009)提出,各種GST基因最終匯聚成同一樹根,可能是由于它們在起源上具有相同的祖先,由同一的GST基因發展變化而來。
pi型是眾多 GST亞型的典型代表,廣泛參與免疫細胞中毒素的代謝和親脂性化合物及其衍生物的產生過程(Kimet al,2009),因其具有抗氧化活性,并參與機體免疫防御,常被用作環境污染的指示標記物,其表達水平在一定程度上可以有效地反應生物體受環境毒物的損害程度,該特征在海洋雙殼貝類中表現明顯,已獲得的海洋貝類中的 GST基本屬于pi型(Yanget al,2004;Rheeet al,2007)。本研究已從厚殼貽貝中克隆出的該條基因核心序列,還有待于通過擴增基因全長確定其結構特征,并找到調控位點,為進一步利用該基因評估厚殼貽貝等海洋經濟貝類的養殖水體污染狀況奠定基礎。

圖2 厚殼貽貝GST基因與其他GST亞型之間的進化分析Fig.2 The relationship of M.coruscus GST-pi and other GST in phylogenetic tree
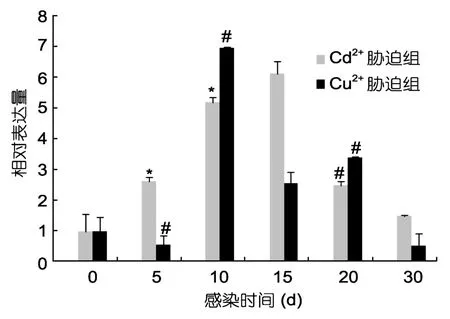
圖3 重金屬脅迫下厚殼貽貝pi型GST基因的表達分析Fig.3 Expression analysis of GST-pi from M.coruscus under stress of heavy metals
3.2 重金屬脅迫下厚殼貽貝GST基因的表達分析
通過相對定量表達分析發現,銅和鎘脅迫均能促使厚殼貽貝 GST基因表達升高,且表現為時間依賴型,該結果說明厚殼貽貝對重金屬刺激較為敏感,可以通過自身的代謝調控作用排出毒素,GST基因在該免疫過程中發揮了重要作用,參與重金屬的解毒過程,周向紅等(2011)用不同濃度重金屬(鉛)刺激條斑紫菜,其 GST表達出現類似的結果。有研究證實,GST參與重金屬解毒的過程主要通過兩種方式:一是 GST基因能夠催化還原型谷胱甘肽直接與重金屬離子共價結合,從而降低重金屬離子毒性并促進重金屬向液泡或質外體轉運(Adamiset al,2009);二是GST的過氧化物酶活性能利用還原型谷胱甘肽向氫過氧化物發動親核攻擊,使其還原為低毒的一元醇(monohydroxy alcohol),從而緩解重金屬脅迫產生的氧化脅迫(Edwardset al,2000)。
此外,銅和鎘脅迫后 GST基因的表達特征略有不同,其中Cu2+刺激10d時表達量達到最高,為對照組6.95倍,Cd2+刺激15d時表達量最高(6.11倍),隨后都開始下降,但 Cd2+刺激后的表達量下降較為緩慢,至30d時仍有較高的表達(約為對照組的1.49倍)。上述表達特征可能是由于 GST對不同重金屬的解毒能力有所不同。另外,海洋貝類中的 GST雖然主要為pi型,但仍有其他類型GST的存在,生物表現出的解毒作用亦是不同類型GST綜合作用的結果,Boutet等(2004)研究太平洋牡蠣受到碳氫化合物和農藥的污染后GST的表達水平,發現體內各種亞型的GST表達量都有明顯的變化,且與毒素類型以及濃度有關,與環境因子和個性特征無關。本研究還有待于進一步檢測不同濃度重金屬刺激作用下 GST的表達量,以確定生物的耐受程度,從而利用基因的表達情況指示水環境中重金屬的污染狀況。
3.3 厚殼貽貝GST分子對環境污染的指示功能
環境污染物是引發經濟貝類發生死亡的重要原因,至今已報道的環境污染物有重金屬、殺蟲劑、有機污染物、工業廢水和環境內分泌干擾物等,上述毒物進入細胞后,大量的等活性氧分子立即在細胞中產生,這些活性氧分子不但直接對細胞造成損傷,還將大量脂質分子氧化成為過氧化脂質,細胞膜的結構與功能會受到過氧化脂質的影響,機體正常的生命活動受到危害(梁旭方,2006)。GST分子具有細胞解毒和抗氧化雙重功效,當環境污染物對貝類產生刺激時,其機體內的 GST活性顯著升高(Blanchetteet al,2002),并通過催化過氧化氫的氧化還原反應,清除體內由代謝反應產生的氧自由基和過氧化物,保護細胞免受外界脅迫環境損傷(張永國等,2006),基于此敏感反應,GST可作為環境污染指示分子(Lenartovaet al,2000),并有研究證實GST活性與污染物水平顯著相關,可直觀反映環境的污染狀況(Martinez-Laraet al,1996)。在環境污染的評估和監測方面,貝類的pi型GST的作用尤為突出(Rheeet al,2007),Blanchette等(2003)對北方圓蛤pi型GST的研究是最早報道的海洋雙殼貝類 pi型 GST,結果指出pi型GST具有很強的解毒作用,從而使北方圓蛤能生活在污染嚴重的水體中,菲律賓蛤仔(Ruditapes philippinarum)的pi型GST亦是鎘等污染物的良好指示分子(Wanget al,2011);Feng等(2009)對硬殼蛤pi型GST研究發現,該基因沒有內含子,且這種無內含子的GST要比有內含子的GST解毒效果更明顯,而高速游動的烏賊、斑馬魚等水生生物GST均有內含子,說明活動性差的貝類 GST解毒效果更強,可成為很好的環境污染指示物。
厚殼貽貝作為典型的營附著生活的雙殼貝類,對環境污染具有很強的耐受性,是最常見的指示水體持久性污染的生物指示劑(Hoarauet al,2004)。本研究中,在銅和鎘持續脅迫下,厚殼貽貝GST基因的表達呈明顯上升狀態,說明 GST參與該生物的解毒過程,可以指示水環境中重金屬的污染狀況。Boutet(2004)曾指出pi型GST是農藥污染監測的有效標記物,而重金屬是農藥中的組成成分,本項研究結果可為重金屬污染的防治和檢測提供有效手段。因此,通過對厚殼貽貝等貝類體內 GST表達水平進行實時監測,可加強對養殖水體中重金屬污染程度的檢測,保證水產養殖業的持續健康發展。
何建瑜,趙榮濤,劉慧慧,2012.舟山海域厚殼貽貝軟體部分營養成分分析與評價.南方水產科學,8(4):37—42
張永國,亢晉勇,2006.谷胱甘肽硫轉移酶的生理功能述評.忻州師范學院學報,22(3):125—128
張 妍,2012.近江牡蠣(Crassostrea hongkongensis)谷胱甘肽S轉移酶和金屬硫蛋白基因 cDNA克隆及原核表達研究.廣東:暨南大學碩士論文,1—6
林 群,梁旭方,胡永樂等,2009.近江牡蠣4種類型GST基因 cDNA全序列的克隆與分析.生態毒理學報,4(2):237—243
羅凱婭,劉欣欣,葛端陽等,2012.鰻弧菌(Vibrio anguillarum)侵染對青蛤(Cyclina sinensis)谷胱甘肽硫轉移酶及其基因表達的影響.海洋與湖沼,43(4):735—740
周向紅,易樂飛,李信書等,2011.條斑紫菜谷胱甘肽 S-轉移酶基因的克隆與表達分析.水產學報,35(6):1354—1361
梁旭方,2006.魚蝦貝肝臟 sGST去毒酶基因的研究.水利漁業,26(5):4—5
程煒軒,梁旭方,李觀貴等,2009.鱖魚兩種谷胱甘肽 S-轉移酶基因 cDNA的克隆與分析.生態毒理學報,4(4):537—543
雷安平,陳 歡,黎雙飛等,2009.谷胱甘肽S轉移酶的功能、應用及克隆表達.環境科學與技術,32(12):85—91
Adamis P D B,Mannarino S C,Eleutherio E C A,2009.Glutathione and gamma-glutamyl transferases are involved in the formation of cadmium-glutathione complex.FEBS Letters,583(9):1489—1492
Blanchette B N,Singh B R,2002.Induction of glutathioneS-transferase in the Northern QuahogMercenaria mercenariaafter exposure to the polychlorinated biphenyl(PCB)mixture aroclor1248.Journal of Protein Chemistry,21(8):489—494
Blanchette B,Singh B R,2003.An enzyme based dechlorination of a polychlorinated biphenyl(PCB)mixture,Aroclor 1248,using glutathioneS-transferases from the northern quahogMercenaria mercenaria.Journal of Protein Chemistry,22(4):377—386
Boutet I,Tanguy A,Moraga D,2004.Characterisation and expression of four mRNA sequences encoding glutathioneS-transferases pi,mu,omega and sigma classes in the Pacific oysterCrassostrea gigasexposed to hydrocarbons and pesticides.Marine Biology,146(1):53—64
Edwards R,Dixon D P,Walbot V,2000.Plant glutathioneS-transferases:enzymes with multiple functions in sickness and in health.Trends in Plant Science,5(5):193—198
Feng X,Singh B R,2009.Molecular identification of glutathione S-transferase gene and cDNAs of two isotypes from northern quahog(Mercenaria mercenaria).Comparative Biochemistry and Physiology Part B,154(1):25—36
Fu J,Xu W,Mai Ket al,2012.Effects of dietary menadione on the activity of antioxidant enzymes in abalone,Haliotis discus hannaiIno.Chinese Journal of Oceanology and Limnology,30(1):118—123
Hoarau P,Garello G,Gnassia-Barelli Met al,2004.Effect of three xenobiotic compounds on Glutathione S-transferase in the clamRuditapes decussatus.Aquatic Toxicology,68(1):87—94
Kim M,Ahn IY,Cheon Jet al,2009.Molecular cloning and thermal stress-induced expression of a pi-class glutathioneS-transferase(GST)in the Antarctic bivalveLaternula elliptica.Comparative Biochemistry and Physiology Part A,152(2):207—213
Kumar S,Tamura K,Nei M,2004.MEGA 3:Integrated software for molecular evolutionary genetics analysis and sequence alignment.Briefings in Bioinformatics,5(2):150—163
Lenartova V,Holovska K,Javorsky P,2000.The influence of environmental pollution on the SOD and GST-isoenzyme patterns.Water Science And Technology,42(1—2):209—214
Liao W Q,Liang X F,Wang Let al,2006.Molecular cloning and characterization of alpha-class glutathioneS-transferase gene from the liver of silver carp,bighead carp and other major Chinese freshwater fishes.Joumal of Biochemical and Molecular Toxicology,20(3):114—126
Martinez-Lara E,Toribio F,Lopez Jet al,1996.Glutathione S-transferase isoenzyme patterns in the gilthead sea bream(Sparusaurata)exposed to environmental contaminants.Comparative Biochemistry and Physiology Part C,113(2):215—220
Masella R,Benedetto R D,Vari Ret al,2005.Novel mechanisms of natural antioxidant compounds in biological systems:involvement of glutathione and glutathonerelated enzymes.The Journal of Nutritional Biochemistry,16(10):577—586
Ohnuma T,Anan E,Hoashi Ret al,2011.Dietary diacetylene falcarindiol induces phase 2 drug metabolizing enzymes and blocks carbon tetrachloride induced hepatotoxicity in mice through suppression of lipid peroxidation.Biological and Pharmaceutical Bulletin,34(3):371—378
Pfaffl M W,2001.A new mathematical model for relative quantification in real-time RT-PCR.Nucleic Acids Research,29(9):2002—2007
Rhee J S,Lee Y M,Hwang D Set al,2007.Molecular cloning,expression,biochemical characteristics,and biomarker potential of theta class glutathione S-transferase(GST-T)from the polychaete Neanthes succinea.Aquatic Toxicology,83(2):104—115
Salinas A E,Wong M G,1999.GlutathioneS-transferases-a review.Current Medicinal Chemistry,6(4):279—310
Saranya Revathy K,Umasuthan N,Lee Yet al,2012.First molluscan theta-class Glutathione S-transferase:Identification,cloning,characterization and transcriptional analysis post immune challenges.Comparative Biochemistry and Physiology Part B,162(1):10—23
Umasuthan N,Saranya Revathy K,Lee Yet al,2012.A novel molluscan sigma-like glutathioneS-transferase from Manila clam,Ruditapes philippinarum:Cloning,characterization and transcriptional profiling.Comparative Biochemistry and Physiology Part C,1:539—550
Wan Q,Whang I,Lee J,2008.Molecular characterization of mu class glutathione-S-transferase from disk abalone(Haliotis discus discus),a potential biomarker of endocrine-disrupting chemicals.Comparative Biochemistry and Physiology Part B,150(2):187—199
Wang L,Pan L,Liu Net al,2011.Biomarkers and bioaccumulation of clamRuditapes philippinarumin responseto combined cadmium and benzo[a]pyrene exposure.Food and Chemical Toxicology,49:3407–3417
Yang H L,Zeng Q Y,Li E Qet al,2004.Molecular cloning,expression and characterization of glutathioneS-transferase fromMytilus edulis.Comparative Biochemistry and Physiology Part B,139(2):175—182

