基于線粒體16S rDNA和COΙ基因探討中國近海黃姑魚類的分子系統進化關系*
辛 儉 張玉榮 徐冬冬 樓 寶 詹 煒 毛國民
(浙江省海洋水產研究所 浙江省海水增養殖重點實驗室 舟山 316100)
黃姑魚屬(Nibea)魚類是世界重要的海洋經濟魚類之一,主要分布于印度洋和太平洋海域。該屬隸屬于鱸形目(Perciformes)石首魚科(Sciaenidae),首先由Jordan和 Thompson于 1911年在石首魚屬(Sciaena)內建立了黃姑魚亞屬(Nibea)(Jordanet al,1911),后由Trewavas(1977)將其提升為屬(Trewavas,1977)。黃姑魚屬魚類也是我國重要的海洋漁業資源,據文獻報道,分布在我國的黃姑魚屬魚類包括黃姑魚(Nibea albifora)、淺色黃姑魚(N.coibor)、半花黃姑魚(N.semifascitata)、雙棘黃姑魚(N.diacanthus)、日本黃姑魚(N.japonica)、狀黃姑魚N.miichthioides和尖頭黃姑魚(N.acuta)共7種(朱元鼎等,1963a,b;成慶泰等,1987)。此后,不斷有學者對黃姑魚屬的分類進行修訂:將雙棘黃姑魚改為雙棘原黃姑魚(Protonibea diacanthus),屬原黃姑魚屬(Protonibea);日本黃姑魚改為日本白姑魚(Argyrosomus japonicus),狀黃姑魚更改為廈門白姑魚(A.amoyensis),這兩種魚均屬白姑魚屬(Argyrosomus);尖頭黃姑魚改為尖頭黃鰭牙(Chrysochir aureus),屬金鰭屬(Chrysochir)(沈世杰等,1993;孟慶聞等,1995;莊平等,2006;伍漢霖等,2012)。盡管黃姑魚屬魚類的形態分類已經得到學者的認同,但黃姑魚類的同種多名、異名現象仍然較多,而且國內外對黃姑魚類的分子系統關系的研究報道較少。
隨著現代分子標記技術的發展,通過獲得物種特定遺傳標記的大量數據,構建分子系統樹,可以對傳統分類提供重要佐證以及補充或修正(Liuet al,2004;李琪等,2010)。線粒體DNA的分子結構簡單,而且具有進化速率快、幾乎不發生重組、嚴格母系遺傳等優點,成為開展分子系統學研究的重要工具。目前,常用于魚類分子系統分析的線粒體基因有:COI基因、16S rDNA、Cytb基因等(郭新紅等,2004;程國寶等,2012 ;Zhanget al,2012)。利用線粒體DNA探討石首魚科魚類的系統分化國內外已有一些報道(蒙子寧等,2004;童馨等,2007;Lakraet al,2009;柳淑芳等 2010;張永等,2011;馬春艷等,2012),主要是探討了石首魚科的分子系統進化,但對黃姑魚屬的系統分類問題卻鮮有涉及。
本研究測定了黃姑魚屬4個種30個個體的線粒體 16S rDNA和 COI基因的部分序列,并結合Genebank下載的其他石首魚科魚類相應的基因序列進行比較分析,構建分子系統樹,以此探討黃姑魚屬魚類的分子鑒定及其系統進化關系。
1 材料與方法
1.1 試驗材料
本研究所用的黃姑魚(HG)、淺色黃姑魚(QH)、日本黃姑魚(RH)、狀黃姑魚(MH)等4種黃姑魚類共計30尾,取樣地點和樣本數見表1。樣品根據朱元鼎等(1963a,b)和成慶泰等(1987)的報道進行形態學鑒定,剪取鰭條保存于75%的酒精中帶回實驗室提取總 DNA,DNA提取方法采用試劑盒提取(北京天根)。

表1 四種黃姑魚類的分類信息及采集地點Tab.1 Information of taxonomy and sampled localities for four Nibea fish species
1.2 16S rDNA和COΙ基因片段的擴增
實驗用的16S rDNA和COI片段擴增的引物由上海生工公司合成。16S rDNA的序列為16S-F1-5′CGCCTGTTTATCAAAAACAT 3′和16S-F2 5′CCGGTCTGAACTCAGATCACGT 3′(徐冬冬等,2010);COI基因片段引物序列為:COI-F1 5′-TCAACCAACCACAAAGACATTGGCAC-3′和 COI-F2 5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′(Wardet al,2005;Lakraet al,2009)。PCR擴增采用50μL反應體系:1×PCR緩沖液,0.2mmol/L dNTP,2mmol/L Mg2+,1μmol/L正反向引物,2 UTaqDNA聚合酶(Takara),DNA模板約400ng。反應程序為:預變性94°C,5min;35個循環,每個循環包括94°C變性30s;退火(16S rDNA 56°C;COI 52°C)30s;72°C延伸lmin;最后72°C再延伸10min,4°C保存。PCR產物經電泳、割膠回收、產物純化后獲得目的片段,純化后的目的片段采用ABI3730進行測序。
1.3 序列下載
從GenBank下載13種石首魚科魚類16S rDNA或COI基因序列,與本研究所檢測的4種黃姑魚類的基因同源序列一起分析。所分析物種的16S rDNA和COI基因序列及其相關信息如表2。
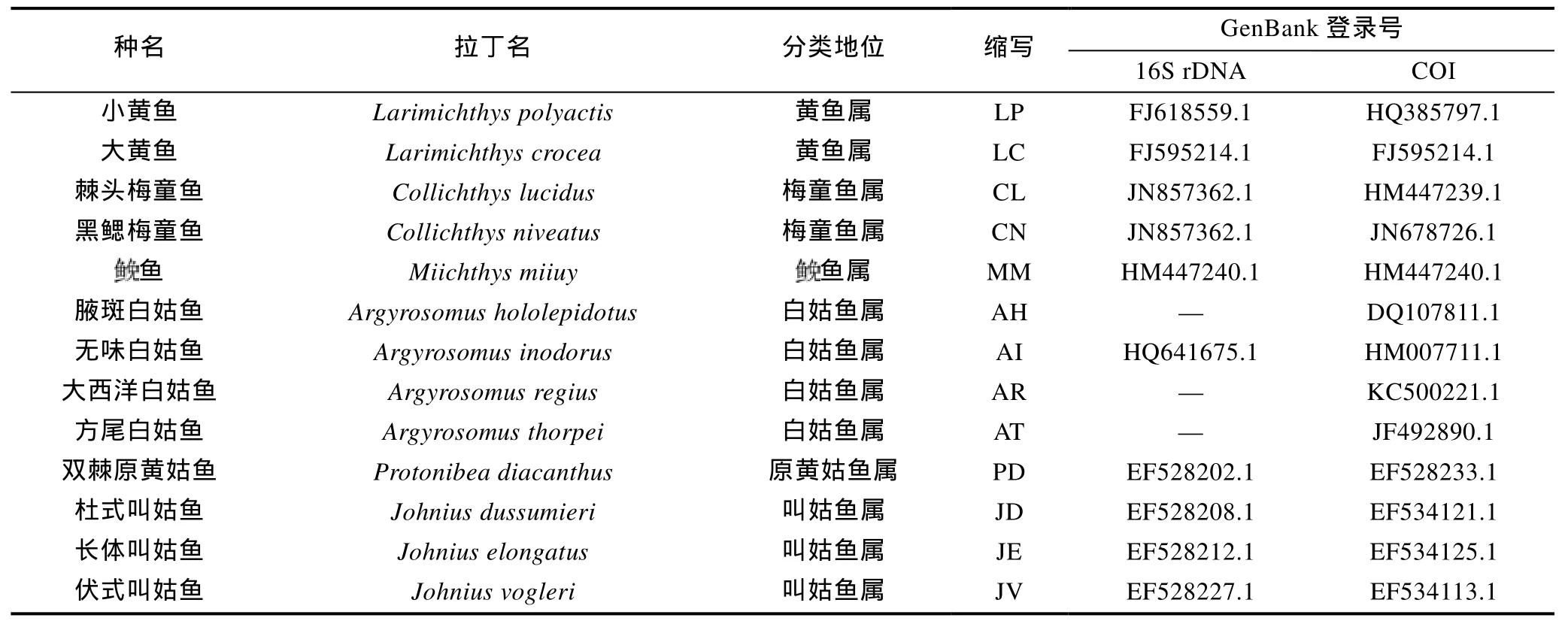
表2 實驗用的石首魚科魚類的16S rDNA和COΙ基因信息Tab.2 Information of 16S rDNA and COI gene in Sciaenidae species
1.4 序列分析
采用NCBI的Blastn工具(http://www.ncbi.nlm.nih.gov/blast)對所獲得的測序結果進行序列比對和分析,驗證所得序列是否為目的片段。應用Clustal X 1.83軟件對獲得的16S rDNA和COI基因序列進行聚類和比對,并輔以手工校正(Thompsonet al,1997)。采用DNASP 5.0軟件計算序列的堿基組成、轉換(Transition)和顛換(Transversion)比例等參數。利用Mega 4.0 軟件中的Kitumra-2-Parameter-Distance雙參數模型計算遺傳距離,并利用 Neibour–joining(NJ)方法構建分子系統進化樹(Tamuraet al,2007)。
2 結果與分析
2.1 四種黃姑魚類16S rDNA和COΙ基因序列特征
經PCR擴增、回收、測序等步驟得到4種黃姑魚類的16S rDNA部分序列,采用Clustal X 1.83對所得序列進行排序和聚類,保留同源序列,長度為553—569bp,編碼 184—189個氨基酸,其間存在插入/缺失位點,主要集中在137bp、192—205bp和550—551bp。四種黃姑魚類的16S rDNA基因序列的平均堿基組成為A=22.3%,T=29.2%,C=21.7%,G=26.8%,其中(C+G)的含量為 48.5%,與張永等(2011)計算的13種石首魚科魚類的GC平均含量略有差異(47.3%),而與蒙子寧等(2004)統計的 8種石首魚科魚類的 GC平均含量相似(48.3%)。四種黃姑魚類的 COI基因序列經排序和剪輯后得到637bp的同源序列,編碼212個氨基酸,其間沒有插入/缺失位點。四種黃姑魚類的平均堿基組成為 A=22.4%,T=27.0%,C=31.6%,G=19.0%。其(C+G)的含量為50.6%,比柳淑芳等(2010)統計的30種石首魚科魚類的GC平均含量略高48.3%。
4種黃姑魚類的16S rDNA序列中檢測到的多態位點數為72,約占總序列的12.91%,簡約信息位點數72個,其中,轉換位點24個,顛換位點9個,轉換顛換比(Ts/Tv)為2.6;COI基因檢測到131個多態信息位點,約占總序列的20.56%,簡約信息位點128個,包括轉換位點45個,顛換位點23個,單個突變位點3個,轉換顛換比2.0。由此可見,16S rDNA基因在所研究的4種黃姑魚類較COI基因保守,這在其他海洋動物的遺傳分析中也得到相似結果(劉君等,2009;Lakraet al,2009;Cuiet al,2010 ;Fenget al,2011)。這可能是由于線粒體基因組中不同區域的核苷酸突變速率不同或不同基因片段受到的選擇壓力不同,造成了不同基因片段的核苷酸變異存在差異(王巍等,2009;Teletchea,2009)。
2.2 四種黃姑魚類的種內和種間遺傳距離
根據4種黃姑魚類的16S rDNA和COI基因序列變異采用雙參數模型Kimura-2-parameter計算種內和種間遺傳距離(表3,表4)。基于16S rDNA計算的種內的平均遺傳距離為0.000—0.002,平均的種間遺傳距離為 0.064;淺色黃姑魚與狀黃姑魚的種間遺傳距離(0.098)最大,黃姑魚和淺色黃姑魚的種間遺傳距離(0.024)最小。基于COI基因計算的4種黃姑魚類平均的種內遺傳距離為 0.000—0.005,平均的種間遺傳距離為 0.121;淺色黃姑魚和日本黃姑魚的種間遺傳距離(0.192)最大;狀黃姑魚和日本黃姑魚的種間遺傳距離(0.002)最小。
線粒體DNA的COI基因已成為動物DNA條形碼研究的首選對象(Hebertet al,2003;Wardet al,2005;李琪等,2010 ;Malakaret al,2012)。應用COI基因進行石首魚科魚類的物種鑒定已有報道。如Lakra等(2009)利用COI基因分析印度洋7種石首魚科魚類的種間遺傳距離為 0.025—0.258;柳淑芳等(2010)分析了30種石首魚科魚類的種內遺傳距離平均值為 0.006,種間為 0.210,種間遺傳距離是種內的35倍;以上研究均表明COI基因可以作為上述石首魚科魚類物種鑒定的分子標記。但是,COI基因DNA條形碼技術在應用過程中也引起很大的爭議。Vences等(2005)通過對馬達加斯加蛙類的研究,發現兩棲類動物適合的條形編碼基因是 16S rRNA基因。Elias等(2007)利用COI序列只能準確鑒定出77%的蝴蝶種類。
本研究采用 COI基因序列計算 4種黃姑魚類的種內遺傳距離為0.000—0.005,種間遺傳距離為0.002—0.192,其中,淺色黃姑魚與日本黃姑魚、狀黃姑魚之間的遺傳距離為 0.115—0.192,處于種間遺傳分化的水平;而日本黃姑魚和狀黃姑魚的種間遺傳距離僅為 0.002,尚未達到 Hebert等(2003)所推薦的物種鑒定的最小種間遺傳距離(0.02)。柴學軍等(2009)比較日本黃姑魚和狀黃姑魚的 cytb基因序列也發現二者具有很高的同源性(99.82%)。利用16S rDNA序列計算的日本黃姑魚和狀黃姑魚的種間遺傳距離為0.026。因此,16S rDNA可以用于日本黃姑魚和狀黃姑魚的分子鑒定(Venceset al,2005);而COI基因能否作為這兩種魚分子鑒定的有效標記值得商榷。一般而言,采用單基因作為 DNA條形碼進行物種鑒定可能存在一定的局限性,同時使用多個分子標記進行分析會增加結果的可靠性(郭新紅等,2004 ;Eliaset al,2007 ;Zhanget al,2012)。
2.3 分子系統樹分析
基于 Kimura雙參數法分別根據 16S rDNA和COI基因序列計算了石首魚科魚類的遺傳距離(表3,表4),并應用鄰位相連法(Neighbor-joining,NJ)構建了分子系統進化樹(圖1,圖2),枝上的數值是 1000次重復抽樣檢驗的置信度值。由16S rDNA片段構建的NJ樹來看,黃姑魚與淺色黃姑魚聚為一枝,狀黃姑魚首先與無味白姑魚(A.inodorus)聚類,然后與日本黃姑魚聚為一枝,具有很高的支持率。由COI基因片段構建的 NJ樹來看,黃姑魚與淺色黃姑魚首先聚為一枝,日本黃姑魚和狀黃姑魚聚為一枝。由拓撲結構來看,日本黃姑魚和狀黃姑魚與黃姑魚屬代表種黃姑魚和淺色黃姑魚的親緣關系較遠。將日本黃姑魚與狀黃姑魚以及黃姑魚和淺色黃姑魚劃分為兩個類群,基于16S rDNA和COI基因序列計算出兩者間的遺傳距離為0.102和0.180,表明日本黃姑魚和狀黃姑魚與黃姑魚屬間存在較大的遺傳分化,它們之間的親緣關系已經超過了同屬內的遺傳分化水平(表3,表4)。因此,研究結果支持黃姑魚屬不包括日本黃姑魚和狀黃姑魚的分類學觀點。
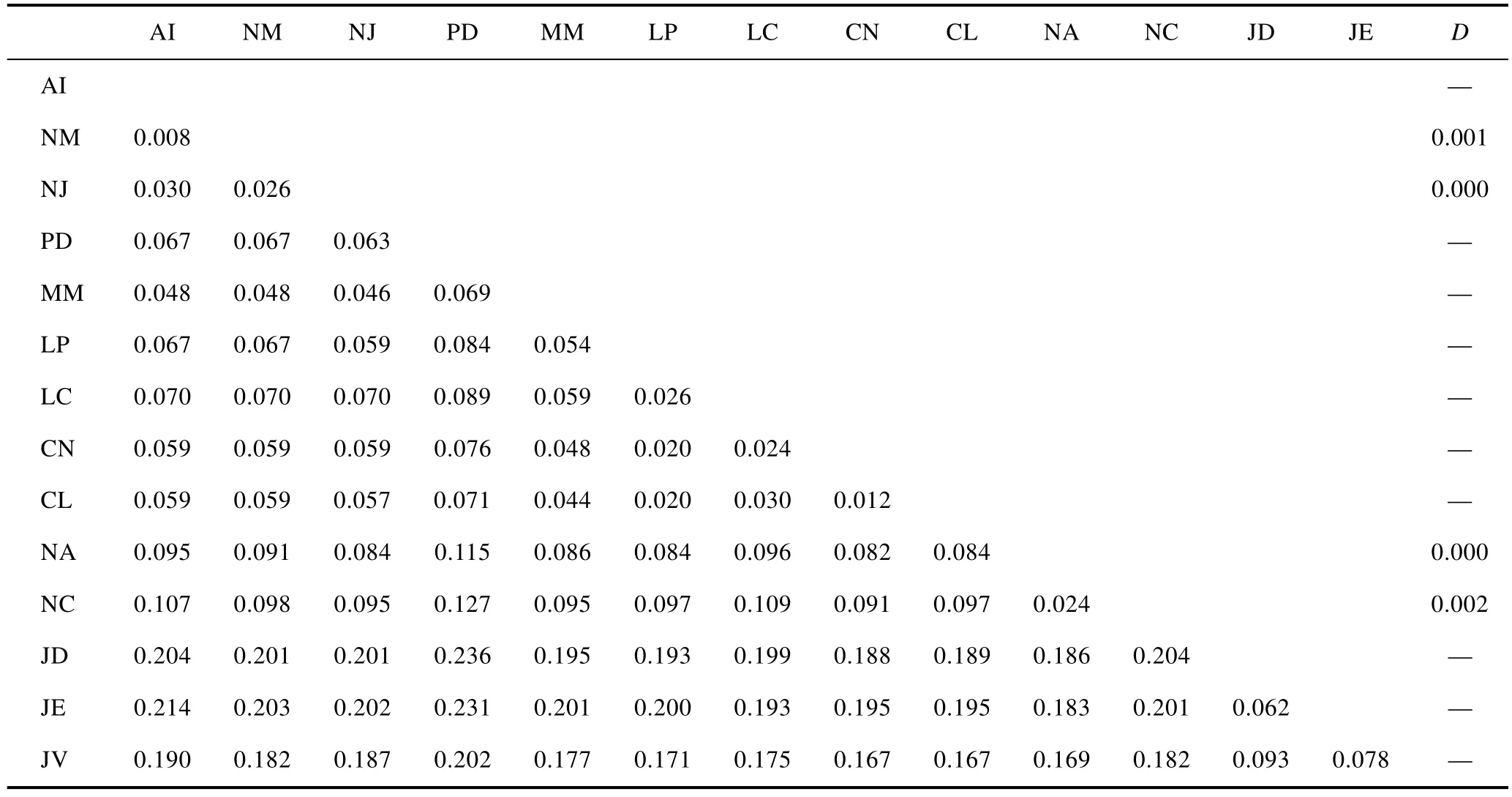
表3 基于16S rDNA的石首魚科魚類的種內和種間遺傳距離Tab.3 Intraspecific and interspecific genetic distance among Sciaenidae species based on 16S rDNA

表4 基于COΙ基因的石首魚科魚類的種內和種間遺傳距離Tab.4 Intraspefic and interspefic genetic distance among Sciaenidae species based on COI gene
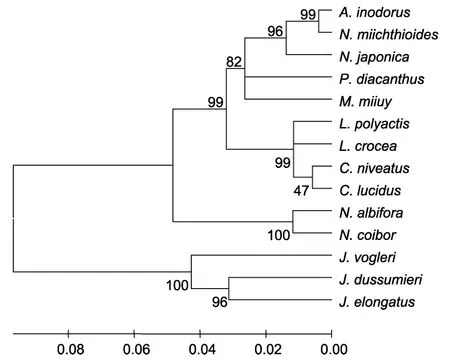
圖1 基于16S rDNA構建的石首魚科魚類分子系統樹(NJ法)Fig.1 Molecular phylogenetic tree of Sciaenidae fishes based on the fragment of the 16S rDNA(NJ method)
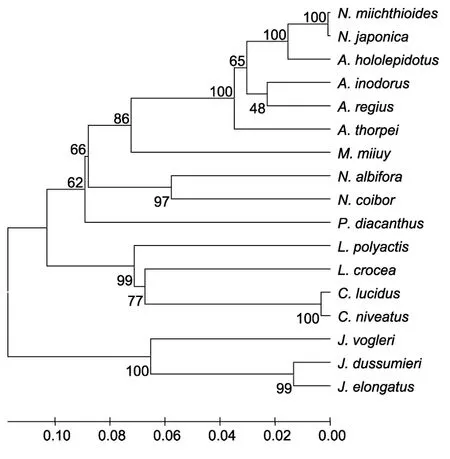
圖2 基于COI構建的石首魚科魚類分子系統樹(NJ法)Fig.2 Molecular phylogenetic tree of Sciaenidae fishes based on the fragment of the COI gene(NJ method)
綜上,本研究結果從分子系統學角度為黃姑魚屬的重新修訂提供科學依據,支持日本黃姑魚和狀黃姑魚屬白姑魚屬的分類學觀點;利用16S rDNA可以對4種黃姑魚類進行分子鑒定,但是,基于COI基因計算的日本黃姑魚和狀黃姑魚的種間遺傳距離尚未達到種間分化水平,應用該基因片段進行這兩種魚的分子鑒定值得商榷。然而,本研究所獲得黃姑魚屬的種類及數量有限,希望以后能采集到盡可能多的種類及樣品,利用多種分子標記從屬間、種間以及種內不同水平進行黃姑魚屬的系統發育研究,進一步闡明中國石首魚科魚類的物種多樣性及其進化關系,深化對石首魚科魚類的進化和中國近海海洋魚類區系的認識。
馬春艷,馬凌波,倪 勇等,2012.基于RAG基因的中國近海13種石首魚科魚類系統進化關系.水產學報,36(1):9—16
王 巍,朱 華,胡紅霞等,2009.五種鱘魚線粒體控制區異質性和系統發育分析.動物學研究,30(5):487—496
成慶泰,鄭葆珊,1987.中國魚類系統檢索.北京:科學出版社,317—324
朱元鼎,張春霖,成慶泰,1963a.石首魚科.東海魚類志.北京:科學出版社,268—293
朱元鼎,羅云林,伍漢霖,1963b.中國石首魚類分類系統的研究和新屬新種的敘述.上海 :上海科學技術出版社,47—56
伍漢霖,邵廣詔,賴春福等,2012.拉漢世界魚類系統名典.北京:北京出版社,105—535
莊 平,王幼槐,李圣法等,2006.長江口魚類.上海:上海科技出版社,348—349
劉 君,李 琪,孔令鋒等,2009.基于線粒體 COI的 DNA條形碼技術在貽貝科種類鑒定中的應用.水生生物學報,35(5):874—881
李 琪,鄒山梅,鄭小東等,2010.DNA條形碼及其在海洋生物中的應用.中國海洋大學學報(自然科學版),40(8):43—47
沈世杰,1993.臺灣魚類志.臺灣 :國立臺灣大學動物學系出版,383—392.
孟慶聞,陳力行,1995.石首魚科.魚類分類學.北京:中國農業出版社,713—728
柳淑芳,陳亮亮,戴芳群等,2010.基于線粒體 COI基因的DNA條形碼在石首魚科(Sciaenidae)魚類系統分類中的應用.海洋與湖沼,41(2):223—232
柴學軍,李鵬飛,吳祖杰,2009.日本黃姑魚和狀黃姑魚細胞色素 b基因序列的比較分析.現代漁業信息,24(4):18—20
徐冬冬,尤 鋒,樓 寶等,2010.8種鲆鰈魚種間遺傳距離與雜交親和性的相關性分析.水產學報,34(2):178—184
郭新紅,劉少軍,劉 巧等,2004.魚類線粒體DNA研究新進展.遺傳學報,31(9):983—1000
程國寶,李三磊,徐冬冬等,2012.梭魚和鯔魚線粒體 16S rRNA和 COI基因片段的比較分析.浙江海洋學院學報:自然科學版,31(2):103—106
童 馨,杜 博,喻達輝等,2007.淺色黃姑魚線粒體16S rRNA基因片段序列特征分析.海洋水產研究,28(3):85—91
蒙子寧,莊志猛,丁少雄等,2004.中國近海 8種石首魚類的線粒體16S rRNA基因序列變異及其分子系統進化.自然科學進展,14(5):514—521
Cui Z,Liu Y,Liu Jet al,2010.Molecular identification ofPampusfishes(Perciformes,Stromateidae).Ichthyological Research,57(1):32—39
Elias M,Hill R I,Willmott K Ret al,2007.Limited performance of DNA barcoding in a diverse community of tropical butterflies.Proceedings of the Royal Society B:Biological Sciences,274(1627):2881—2889
Feng Y W,Li Q,Kong L Fet al,2011.DNA barcoding and phylogenetic analysis of Pectinidae(Mollusca:Bivalvia)based on mitochondrial COI and 16S rRNA genes.Molecular Biology Reports,38:291—299
Hebert,Paul D N,Sujeevan Ratnasingham,et al,2003.Barcoding animal life:cytochrome c oxidase subunit I divergences among closely related species.Proceedings of the Royal Society of London.Series B:Biological Sciences,270(S1):S96—S99
Jordan D S,Thompson W F,et al,1911.A review of the sciaenoid fishes of Japan.Proceedings of the United States National Museum,39(1787):241—261
Lakra W S,Goswami M,Gopalakrishnan A,2009.Molecular identification and phylogenetic relationships of seven Indian Sciaenids(Pisces:Perciformes,Sciaenidae)based on 16S rRNA and cytochrome c oxidase subunit I mitochondrial genes.Molecular Biology Reports,36(5):831—839
Liu Z J,Cordes J F,2004.DNA marker technologies and their applications in aquaculture genetics.Aquaculture,238(1):1—37
Malakar A K,Lakra W S,Goswami Met al,2012.Molecular identification of threeOmpokspecies using mitochondrial COI gene.Mitochondrial DNA,23(1):20—24
Teletchea F,2009.Molecular identification methods of fish species:reassessment and possible applications.Reviews in Fish Biology and Fisheries,19(3):265—293
Trewavas E,1977.The sciaenid fishes(croakers or drums)of the Indo-West-Pacific.Transactions of the Zoological Society of London,33(2):253—541
Thompson J D,Gibson T J,Plewniak Fet al,1997.The CLUSTAL–X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools.Nucleic Acids Research,25(24):4876—4882
Tamura K,Dudley J,Nei Met al,2007.MEGA4:Molecular evolutionary genetics analysis(MEGA)software version 4.0.Molcular Biology Evolution,24(8):1596—1599
Vences M,Thomas M,Van der Meijden A,et al.2005.Comparative performance of the 16S rRNA gene in DNA barcoding of amphibians.Frontiers in Zoology,2(1):5
Ward,R,Zemlak T,Innes Bet al,2005.DNA barcoding Australia’s fish species.Philosophical Transactions of the Royal Society B:Biological Sciences,360(1462):1847—1857
Zhang J,Hanner R,2012.Molecular approach to the identification of fish in the South China Sea.PloS One,7(2):e30621

