基于BQPSO算法的癌癥特征基因選擇與分類
范方云, 孫俊
(江南大學物聯網工程學院,江蘇無錫214122)
基于BQPSO算法的癌癥特征基因選擇與分類
范方云, 孫俊*
(江南大學物聯網工程學院,江蘇無錫214122)
提出了基于二進制編碼的量子行為粒子群優化算法(BQPSO)的癌癥特征基因選擇方法,利用BQPSO對樣本數據進行特征選擇。使用選出的特征基因訓練支持向量機進行留一法交叉驗證。實驗結果表明,基于BQPSO算法的癌癥特征基因選擇方法是一種行之有效的方法。
微陣列數據;特征基因;二進制編碼的量子行為粒子群優化算法;支持向量機;留一法交叉驗證
現代社會,癌癥已經成為威脅人類生命的重要因素之一。漏診和誤診使很多患者錯過了最佳治療時機。因此,人們迫切需要更多可靠的輔助方法,結合醫療診斷,最大限度地提高癌癥診斷的正確率。隨著信息科學和分子生物科學的飛速發展,基因芯片技術因其微型化,高通量等特點為人們提供了大量的微陣列DNA表達數據,被廣泛應用于癌癥診斷、臨床檢驗等方面。然而,DNA微陣列數據維度高,樣本量很少,且分布不均勻。過多的分類特征不一定能夠得到較好的分類結果,而且增加了計算復雜度。為此,在利用DNA微陣列表達數據進行癌癥分類之前必須進行特征選擇,選擇出對分類有積極作用的特征基因。
目前常用的特征選擇方法可以從兩方面進行分類[1],即評價準則和搜索策略。在基于評價準則劃分的特征選擇方法中,又可根據特征選擇是否獨立于后續的學習算法分為過濾式(Filter)和封裝式(W rapper)兩種。Filter與后續學習算法無關,而W rapper利用后續學習算法的訓練準確率評估特征子集。在基于搜索策略劃分特征選擇方法時,按照特征子集的形成過程,可分為全局搜索,隨機搜索和啟發式搜索3種。一個具體的搜索算法會采用兩種或多種基本搜索策略[2-4]。張靖等[5]利用信噪比指標過濾無關基因,再采用迭代Lasso方法進行冗余基因的剔除,結合SVM分類器在數據集Leukemia,Prostate,colon上分別獲得了98.61%, 96.08%,90.32%的分類正確率。張煥萍等[6]提出了離散粒子群和支持向量機封裝模式的BPSO-SVM特征基因選擇方法,在數據集colon上用34個特征基因子集獲得了89.67%的平均正確率。目前結合多種特征選擇方法雖比單獨使用有一定改善,但存在的問題依然很明顯,如第二階段的纏繞過程如何在特征子集規模、所選特征的分類能力和其他約束條件等多個目標下求得最優解。
文中提出的基于BQPSO的癌癥特征基因選擇方法屬于全局搜索和啟發式搜索的結合,依靠改變BQPSO的初始搜索空間的大小和BQPSO的強搜索能力,本方法在分類正確率上獲得了較大的提高,同時也得到了規模更小的特征子集。
1 二進制編碼的量子行為粒子群優化算法
1.1 粒子群優化算法
粒子群優化算法(Particle Swarm Optimization, PSO)[7]是在1995年由Kennedy和Eberhart提出的基于群體智能理論的優化算法,它模擬了鳥群的覓食過程,通過個體間的合作與競爭產生的群體智能指導優化搜索。PSO算法首先初始化一組隨機解,通過迭代搜尋最優值。每個優化問題的解視為搜索空間的一只鳥,稱為“粒子”。所有的粒子對應一個優化問題的適應值,粒子的速度決定其飛行的方向和距離,粒子通過追尋群體中的最優粒子來完成在解空間的搜索。
1.2 量子行為粒子群優化算法
在PSO算法的基礎上,Efron B等[8]從量子力學的角度出發,提出了一種新的PSO算法模型,即量子行為粒子群優化算法(Quantum-Bahaved Particle Swarm Optimization,QPSO)。QPSO的量子模型,參考量子物理學,將粒子的運動狀態用波動函數表示。同時,在QPSO算法的模型中,利用波動函數φ給定的概率密度函數確定粒子在某個時刻某個位置出現的概率。
1.3 二進制編碼的量子行為粒子群優化算法
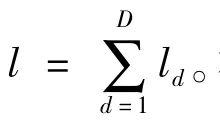

其中,dH(·)是計算Hamming距離的函數,它的值為兩個位串對應位的不同值的總個數。
與文獻[9]中式(2)不同,在BQPSO中,平均最優位置mbest的每個二進制位由群體中每個粒子最優個體pbesti對應的二進制位上0,1出現的情況決定。具體為:統計所有粒子pbesti二進制串每一位上0,1出現的次數。如果出現0的次數多,則mbest對應位為0;反之,則為1。若0,1出現的次數相同,mbest對應位隨機出現0或者1。將獲得mbest的函數記為
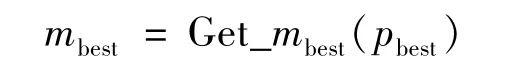
輸入為粒子最優個體pbest;輸出為平均最優位置mbest。
在BQPSO算法中,父代粒子最優個體pbesti和群體最優個體gbest隨機交叉產生粒子的局部吸引子,即Pi。Pi值的函數表示為
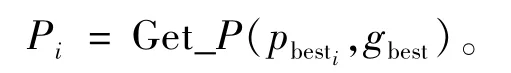
而粒子的新位置Xid由Pid變異而來。變異的概率為
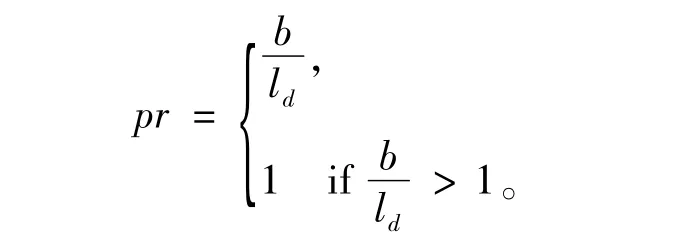
式中:ld為粒子第d維的長度;
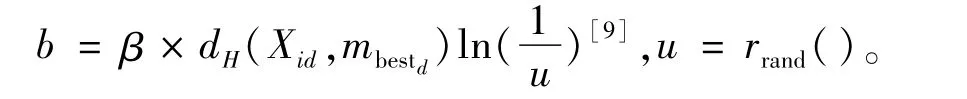
其中,β為BQPSO算法的系數。由于Hamming距離為整數,對b值[·]取整再使用[7]。計算Xid的函數表示為
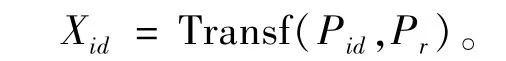
BQPSO算法的步驟[9]如下:
1)用二進制位串的形式初始化群體中的每個粒子Xi,并使得pbesti=Xi;
2)根據方程mbest=Get_mbest(pbest)計算群體平均最優位置mbest的值;
3)根據適應度函數值(例如最大化問題)計算群體中每一個粒子的適應值,并與前次的粒子最優值比較,如果f(Xi)?f(pbesti),則pbesti=Xi;反之,則不更新;
4)計算群體中全局最優粒子pbestg,并與前次的全局最優值gbest比較,f(pbestg)>f(gbest),則gbest= pbestg;反之,則不更新;
5)根據方程Pi=Get_P(pbesti,gbest),計算局部吸引子Pi的值;

計算pr的值;
7)根據方程Xid=Transf(Pid,pr)計算Xid的值,并連接生成X;
8)重復2)~7),直到滿足算法結束條件。
2 實驗設計
2.1 數據集
實驗采用BRB-ArrayTools[10]主頁上公開的3個DNA微陣列基因表達譜數據集,分別為急性白血病數據集Leukemia,前列腺癌數據集Prostate和結腸癌數據集Colon。這3個數據集均可以從如下的地址下載:http://linus.nci.nih.gov/~brb/DataArchive_ New.htm l
數據集中每個樣本一定屬于兩類中的一種,根據SVM分類器的要求,分別將兩類標識為0和1。每個數據集情況見表1。
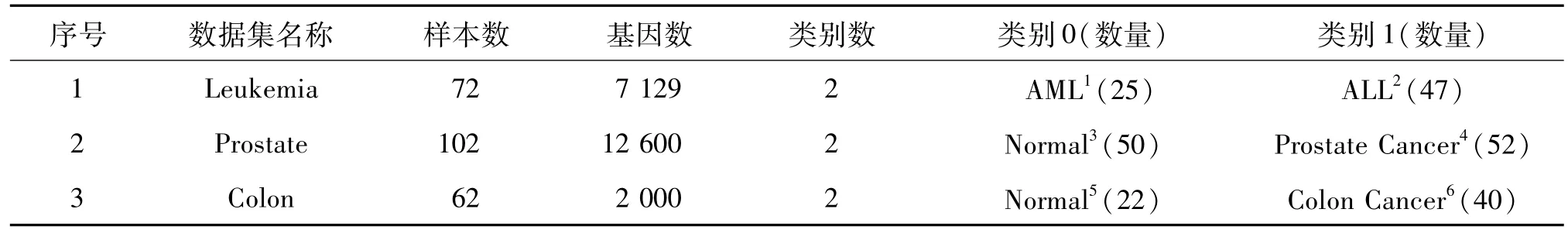
表1 實驗數據集描述Tab.1 Description of experim ental datasets
2.2 預處理
首先,對數據進行標準化處理,消除量綱對分類的影響,再對所有的特征進行T檢驗。取P值較小的前d個特征進行初步篩選,結果作為BQPSO算法的全局搜索空間。文中分別對d=20,30,40,50進行實驗比較,為不同的數據集找出合適的d值。因為在BQPSO算法中,初始特征的個數就是粒子的二進制編碼長度,若不用T檢驗進行初步篩選,粒子的二進制串長度會有幾千甚至上萬,這樣不僅增加了計算復雜度,而且影響BQPSO的搜索效果。因為初始的搜索空間越大,BQPSO最終選擇出的特征基因越多。所以,在保證較好的分類效果的同時,為每個數據集選擇出盡量小的d值,以選擇出最少的特征基因,同時具有較高的分類正確率。
2.3 BQPSO算法選擇特征基因
用BQPSO算法對這d個基因進行第二次篩選,提取出真正具有分類信息的基因。根據數據集樣本數量較少的特點,為了得到可靠穩定的分類模型,使用SVM分類器進行留一交叉驗證(Leave-One-Out Cross-Validation,LOOCV)。采用數據集的留一分類正確率作為BQPSO的適應值,即f(·)等于SVM采用留一法分類數據集得到的分類正確率。若一個數據集有n個樣本,留一交叉驗證是指只使用所有樣本中的一個作為預測集,剩下n-1個樣本作為訓練集,訓練SVM并預測。重復,直到所有的樣本都被當做一次預測集。留一分類正確率就是這n次分類正確率的均值。
實驗設計群體共有20個粒子,每個粒子只有1個決策變量,即粒子的維數為1。每個粒子的長度為d(d=20,30,40,50),即每個粒子用長度為d的0,1串表示,1代表選中該特征,0代表沒有選中。初始時,隨機產生20個長度為d的0,1串Xi(i=1,2,…, 20),且設初始粒子最優位置

用函數 mbest=Get_mbest(pbest)
計算群體平均最優位置mbest。根據每個粒子適應值是否增大維護粒子最優位置pbesti,即如果本次迭代中f(Xi)>f(pbesti),則更新pbesti=Xi,否則不更新。根據所有的pbesti更新全局最優位置gbest。
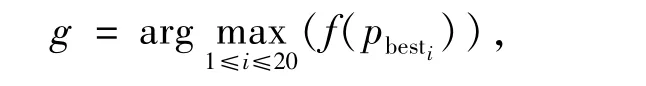
如果f(pbestg)>f(gbest),更新gbest=pbestg。更新完pbesti和gbest之后,采用函數

計算局部吸引子。根據

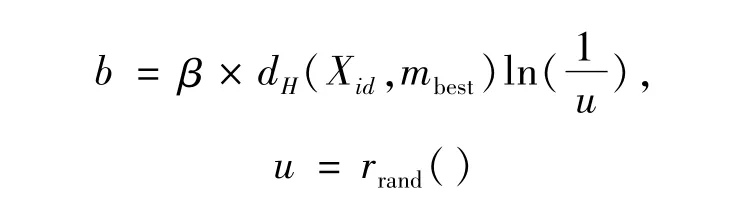
計算的概率,在局部吸引子上進行變異,得到新的粒子種群。其中l為粒子二進制表示的長度,即l= d。將以上過程重復進行200代,或者當f(gbest)>99.99%時退出迭代。
3 結果分析
對每個數據集,取d為20,30,40,50分別進行實驗,每個實驗重復50次,記錄這50個LOOCV分類正確率的最高值和平均值。得到如圖1所示的不同d值時的最高和平均分類正確率。
由圖1可以看出,在分類不同的數據集時,得到最優結果時的d值不盡相同。數據集Leukemia中,當d為40時,數據集Prostate中,當d為50時,數據集Colon中,當d為20時,平均正確率和最好正確率都可達到最優。
表2給出了3個數據集在最優d值下經過BQPSO進一步篩選后提取的基因數和正確率。這里的基因個數是50次實驗結果的平均值。最高、平均正確率同樣來自這50次實驗。
將實驗結果與其他方法相比較,具體結果見表3。
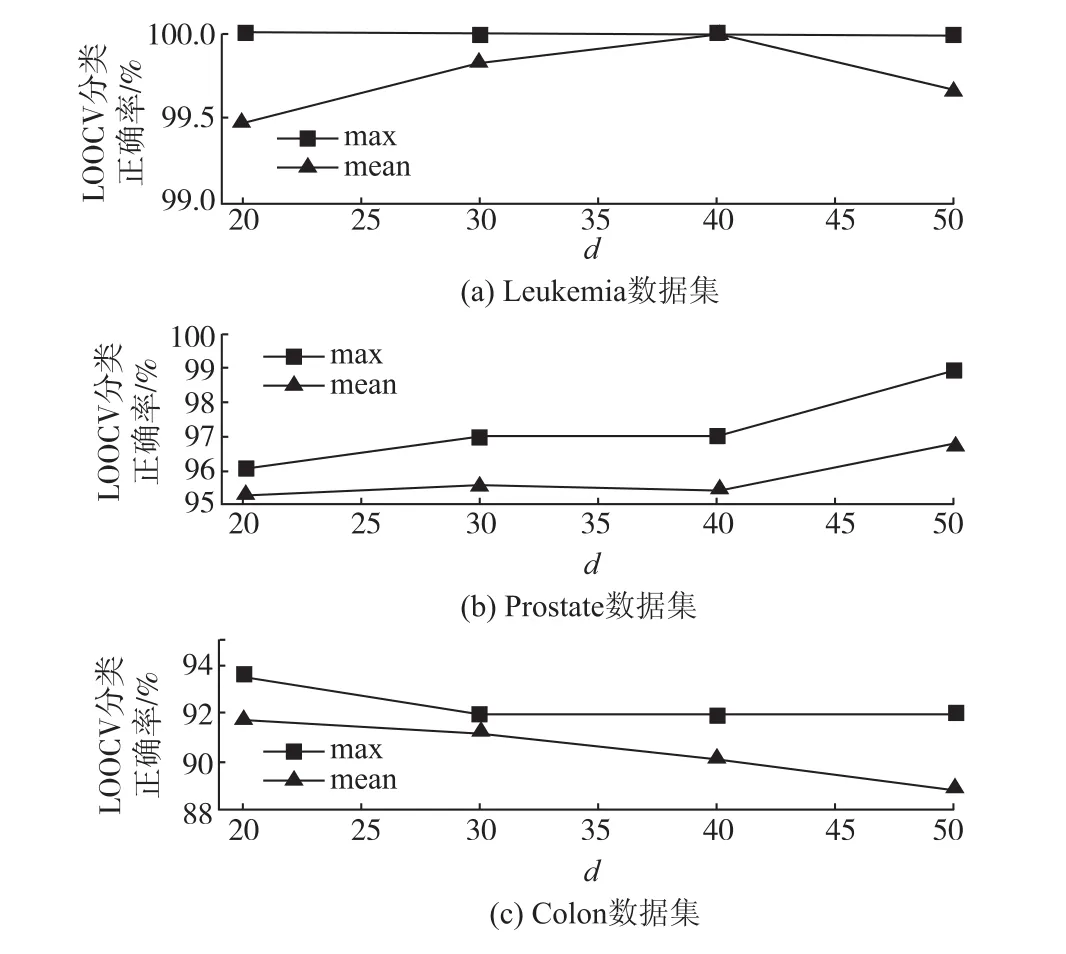
圖1 不同d值時的最高和平均分類正確率Fig.1 Best and the average classification accuracy w ith different d
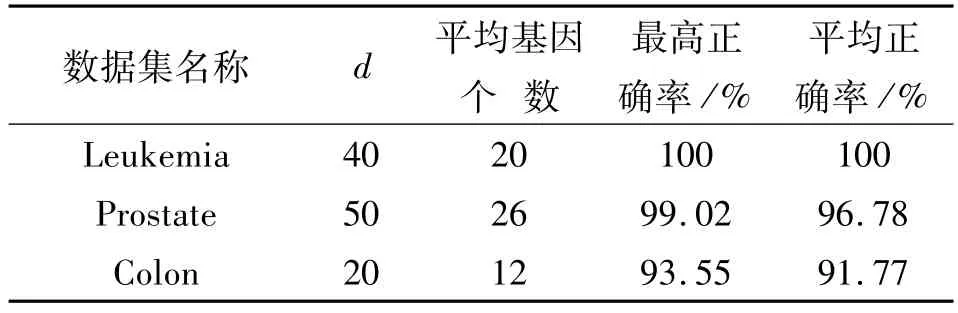
表2 BQPSO特征選擇實驗結果Tab.2 Experimental results of gene selection based on BQPSO
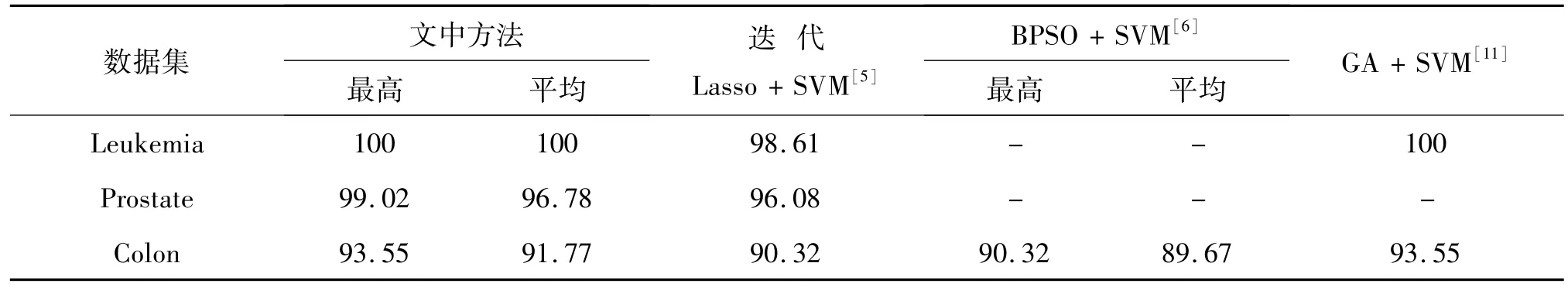
表3 不同方法實驗結果分類正確率的比較Tab.3 Com parison of experiment results from differentmethods 單位:%
由表3可知,對于數據集Leukemia,文中提出的BQPSO+SVM方法得到的LOOCV分類正確率,與GA+SVM方法一樣都能達到100%,且優于迭代Lasso+SVM方法的98.61%;對于Prostate數據集,文中得到的最高和平均分類正確率均高于迭代Lasso+SVM方法得到的96.08%的正確率;對于colon數據集,文中得到的LOOCV正確率與GA+ SVM方法相同,高于迭代Lasso+SVM得到的90.32%,也高于BPSO+SVM得到的最高正確率。
由上述分析可知,文中提出的基于BQPSO的癌癥特征基因選擇方法在特征選擇效果上具有明顯的優勢。
4 結 語
提出了基于BQPSO的用于高維微陣列數據的特征基因選擇與分類方法。并且將實驗結果與迭代Lasso+SVM、BPSO+SVM和GA+SVM相比較。T檢驗結合BQPSO+SVM的方法從微陣列數據成千上萬的基因中選擇出了10~20個最具分類信息的基因,并且得到了較高的分類正確率。由此可知,基于BQPSO算法的微陣列數據特征基因選擇與分類方法是一種行之有效的方法。
[1]姚旭,王曉丹,張玉璽,等.特征選擇方法綜述[J].控制與決策,2012,27(2):161-166.
YAO Xu,WANG Xiaodan,ZHANG Yuxi,et al.Summary of feature selection algorithms[J].Control and Decision,2012,27(2): 161-166.(in Chinese)
[2]劉金勇,鄭恩輝,陸慧娟.基于聚類和微粒群優化的基因選擇方法[J].數據采集與處理,2014,29(1):83-89.
LIU Jinyong,ZHENG Enhui,LU Huijuan.Gene selection based on clusteringmethod and particle swarm optimization[J].Journal of Data Acquisition and Processing,2014,29(1):83-89.(in Chinese)
[3]于彬,張巖.基于GA-SVM方法的結腸癌基因表達譜數據分析[J].青島科技大學學報:自然科學版,2013,33(6): 587-592.
YU Bin,ZHANG Yan.Analysis of colon cancer gene expression profiles based on GA-SVM method[J].Journal of Qingdao University of Science and Technology:Natutral Science Edition,2013,33(6):587-592.(in Chinese)
[4]徐久成,徐天賀,孫林,等.基于鄰域粗糙集和粒子群優化的腫瘤分類特征基因選取[J].小型微型計算機系統,2014,35 (11):2528-2532.
XU Jiucheng,XU Tianhe,SUN Lin,et al.Feature celection for cancer classification based on neighborhood rough set and particle swarm optimization[J].Journal of Chinese Computer Systems,2014,35(11):2528-2532.
[5]張靖,胡學鋼,李培培,等.基于迭代Lasso的腫瘤分類信息基因選擇方法研究[J].模式識別與人工智能,2014,27(1): 49-59.
ZHANG Jing,HU Xuegang,LIPeipei,etal.Informative gene selection for tumor classification based on iterative lasso[J].Pattern Recognition and Artificial Intelligence,2014,27(1):49-59.(in Chinese)
[6]張煥萍,宋曉峰,王惠南.基于離散粒子群和支持向量機的特征基因選擇算法[J].計算機與應用化學,2007,24(9): 1159-1162.
ZHANG Huanping,SONG Xiaofeng,WANG Huinan.Feature gene selection based on binary particle swarm optimization and support vectormachine[J].Computers and Applied Chemistry,2007,24(9):1159-1162.(in Chinese)
[7]孫俊,方偉,吳小俊,等.量子行為粒子群優化:原理及其應用[M].北京:清華大學出版社,2011.
[8]SUN Jun,FENG Bin,XUWenbo.Particle swarm optimization with particles having quantum behavior[C]//The 2004 Congress on Evolutionary Computation.Oregon:IEEE,2004:325-331.
[9]奚茂龍,孫俊,吳勇.一種二進制編碼的量子粒子群優化算法[J].控制與決策,2010,25(1):99-104.
XIMaolong,SUN Jun,WU Yong.Quantum-behaved particle swarm optimization with binary encoding[J].Control and Decision, 2010,25(1):99-104.(in Chinese)
[10]Efron B,Hastie T,Johnstone I,et al.Least angle regression[J].The Annals of Statistics,2004,32(2):407-499.
[11]LIS,WU X,HU X.Gene selection using genetic algorithm and support vectorsmachines[J].Soft Computing,2008,12(7):693-698.
(責任編輯:邢寶妹)
Cancer Feature Gene Selection and Classification Based on BQPSO A lgorithm
FAN Fangyun, SUN Jun*
(School of Internet of Things Engineering,Jiangnan University,Wuxi214122,China)
In this paper,the cancer feature gene selectionmethod based on BQPSO(Quantum-Behaved Particle Swarm Optimization with Binary Encoding)is proposed where BQPSO algorithm is applied to select feature genes from example data and feature genes selected are used to train SVM classifiers and to make LOOCV(leave-one-out cross-validation).The experiment results show that the cancer feature selectionmethod based on the BQPSO algorithm is effective.
microarray data,feature gene,BQPSO,SVM,LOOCV
*通信作者:孫 俊(1971—),男,江蘇無錫人,副教授,碩士生導師。主要從事智能計算、圖像處理與模式識別等研究。Email:sunjun_wx@hotmail.com
TP 181
A
1671-7147(2015)01-0011-05
book=15,ebook=18
2014-08-15;
2014-10-16。
國家自然科學基金項目(61170119)。
范方云(1989—),女,江蘇揚州人,計算機科學與技術專業碩士研究生。

