大黃魚(Larimichthys crocea)新品種“東海1號”體長相關的DArT標記篩選*
徐圣釗 林 勉 閆松松 史雨紅 苗 亮 李明云 陳 炯
(寧波大學海洋學院 生物化學與分子生物學實驗室 寧波 315211)
大黃魚(Larimichthys crocea)屬硬骨魚綱、鱸形目、石首魚科、黃魚屬,是我國“四大海水經濟魚類”之一(姚康等,2008)。20世紀90年代人工繁育成功后,大黃魚成為我國人工育苗量和養殖規模最大的海洋水產養殖品種,其經濟效益顯著。由于不重視種質保護和人工選育,目前人工養殖大黃魚在體形、生長、肉質、性成熟、抗逆性、抗病性等多鐘性狀上出現了衰退,遺傳多樣性降低,在一定程度上限制了其養殖業的可持續發展(Gaoet al,2010)。因此,有必要對大黃魚進行品質改良,培育出具有生長快、抗病強、體型肉質好、耐低溫等性狀的優良品種。
DNA分子標記是根據個體間基因組DNA的多態性發展起來的一類遺傳標記技術,相比形態學標記、細胞學標記、生物化學標記具有一定的優越性(魏東旺等,2001),已廣泛應用于水產動物遺傳育種。大黃魚遺傳育種也普遍采用DNA分子標記技術。王志勇等(2002)采用 AFLP技術(Amplified fragment length polymorphism)分析顯示,大黃魚野生群體和 2個養殖群體,2個福建養殖大黃魚群體的遺傳多樣性低于野生群體。李明云等(2003)采用RAPD技術(Random amplified polymorphic DNA)分析表明,象山港網箱養殖大黃魚群體遺傳多樣性水平較低。李鵬飛等(2008)采用魚線粒體 DNA的細胞色素b(Cytochrome b,Cytb)基因片段多態性可以區分大黃魚、鯢魚和美國紅魚。王曉清等(2008)采用 AFLP和 SSR技術(Simple sequence repeats)對親本與雜交子代分析結果表明,雜交子代與母本大黃魚之間的遺傳同質性極高,屬于異源精子誘導大黃魚雌核發育個體。寧岳(2007)分離獲得的AFLP和SSR標記應用于大黃魚的雌性和雄性連鎖圖譜的構建,同時確定了大黃魚性別決定機制。此外,也陸續篩選獲得許多有價值的SSR標記(Guoet al,2005; Changet al,2009)。早期DNA分子標記技術主要應用于大黃魚遺傳多樣性檢測、系譜確認、遺傳連鎖圖譜的構建中(Yeet al,2014)。近年來,研究者主要致力于采用DNA分子標記技術篩選性狀相關標記。劉賢德等采用微衛星標記技術對不同大黃魚家系和群體進行分析,篩選到與大黃魚生長性狀緊密相關的微衛星標記(劉賢德等,2012,2013; 葉華等,2014)。薛良義等(2013)研究表明大黃魚肌肉生長抑制素基因 3’端非編碼區微衛星序列多態性與大黃魚體長、體質量之間的相關系數沒有達到顯著水平。但生長性狀相關標記發掘較少,此外是否可用于生產實踐還需進一步驗證,因此還需繼續篩選生長性狀相關標記。
多樣性芯片技術(Diversity arrays technology,DArT)是一種基于基因芯片技術的 DNA指紋圖譜分析方法,可廣泛用于檢測和分析動物、植物和微生物的DNA差異以及構建遺傳圖譜、QTL定位和品種指紋圖譜鑒定(Sánchez-Sevillaet al,2015)。與常規技術相比,DArT不需要明確物種的基因組DNA序列信息,具有高通量和低成本的顯著特點,克服了以往跑電泳凝膠為主的標記技術產量低、成本高、耗時長、自動化程度低等缺點,只用少量成本就可進行全基因組的高通量圖譜分析。目前該技術已成功用于水稻、大麥、小麥、油菜、桉樹、蘋果、木薯、擬南芥、木豆、大麥病原菌、沙門氏菌等生物的遺傳連鎖圖譜以及基因定位研究中(Hacklet al,2010; Schoutenet al,2012)。在水產動物的研究中僅見該技術應用于三疣梭子蟹地理種群多樣性分析(榮曄婧等,2014)。
本研究旨在采用DArT技術鑒定與大黃魚體長相關的DArT標記。首先按分離群體標記關聯分析法篩選“東海1號”大黃魚體長相關的DArT標記,后續進一步驗證其相關性,以期為大黃魚選育和種質資源利用提供有用參考資料。
1 材料與方法
1.1 樣品采集
2012年11月從寧波象山港灣水產苗種有限公司網箱養殖的“東海 1號”大黃魚(1齡)中挑選健康無損傷的大黃魚199尾,測量每條魚的體長,并在魚鰓蓋內側較軟部位植入電子標記。然后分別將其放入水泥池中暫養。使用SPSS 17.0軟件統計分析,分別建立體長的正態分布圖,取體長位于 10%的高值個體記為“極端大群體”(20尾),10%低值個體記為“極端小群體” (20 尾)。
1.2 基因組代表性DNA片段文庫構建
文庫構建方法參照 Jaccoud等(2001)描述。采用酚-氯仿法提取基因組 DNA,并將“極端大群體”和“極端小群體”的大黃魚基因組DNA等量混合。500ng混合基因組DNA用切割頻率低的限制性內切酶PstI分別與切割頻率高的AluI、BanⅡ、Bsp1286I、BstNI、HaeIII、RsaI和TaqI組合進行酶切。在T4 DNA連接酶作用下,純化的 DNA連接上PstI特異性接頭(Wenzlet al,2004)。連接產物作為模板用于后續PCR擴增,所用引物為DArT-PstI引物(Wenzlet al,2004),反應程序如下: 94°C變性5min后,以下程序重復35個循環,94°C變性30s,53°C復性30s,72°C延伸1min,循環完成后 72°C延伸反應 10min。擴增產物克隆至載體pMD19-T,轉化大腸桿菌TOP10F并涂布于含氨芐青霉素和X-gal的LB培養基上。
1.3 基因組DNA復雜性降低方法的優化
1.3.1探針制備及芯片點制從代表性基因組DNA文庫中隨機挑選單菌落,利用質粒載體上的通用引物M13F-47和M13R-48對插入的DNA片段進行 PCR擴增,產物用 1倍體積異丙醇沉淀。每個 7種降低基因組復雜性方法基因組代表性DNA片段數為 840個。在芯片中布置質控探針,陰性對照探針,陽性對照探針,空白對照探針,工作探針。每個探針包含三個重復。每張芯片包含4個點陣,每個點陣25行、27列,每個點的位置用“行標-列標”表示。其中1-1—1-3為質控探針,1-19—1-21為陽性對照探針,1-7—1-18、1-22—2-3為陰性對照探針,2-4—25-12為工作探針,1-4—1-6、25-13—25-27為空白對照探針。從PstI/AluI、PstI/BanII、PstI/Bsp1286I、PstI/BstNI、PstI/HaeIII、PstI/RsaI、PstI/TaqI文庫中獲得的 840個基因組代表性DNA片段依次排布在4個點陣中。采用晶芯SmartArrayerTM48點樣儀進行點制。
1.3.2熒光標記基因組代表性 DNA片段的制備用M13F-47和M13R-48引物對未插入外源DNA片段的載體進行PCR擴增,該PCR產物作為reference DNA。經乙醇沉淀后,加入 20μL滅菌水溶解,放–30°C冰箱保存備用。
采用酚-氯仿法提取大黃魚“極端大群體”與“極端小群體”基因組DNA,兩組 DNA先分別使用7組限制性內切酶(PstI/AluI、PstI/BanII、PstI/Bsp1286I、PstI/BstNI、PstI/HaeIII、PstI/RsaI、PstI/TaqI)降低基因組復雜性,再加PstI特異性接頭,接著用PstI引物對加接頭產物進行 PCR擴增,最后擴增產物使用DNAmate沉淀濃縮10倍體積。
取 150ng基因組代表性 DNA片段變性后用DecaLabel DNA Labeling Kit進行Cy3標記,加入含十堿基隨機引物的 5×緩沖液、MixC、Cy5-dCTP、exo-Klenow fragment共 2.1μL,37°C 孵育 10min 后,加入 dNTPs 0.4μL,37°C 孵育 30min,加入 0.1μL EDTA (pH 8.0)終止反應。取150ng reference DNA變性后用DecaLabel DNA Labeling Kit進行Cy5標記。
1.3.3芯片雜交Cy3和Cy5標記反應產物混合,再加入1μL鮭精DNA(10g/L)和50μL ExpressHyb?雜交液混合后,96°C變性 3min,冰浴驟冷 1min。將上述反應產物加入預處理的 DArT芯片中,在晶芯?雜交儀中進行雜交(65°C孵育過夜。雜交后先用0.3×SSC,0.1% SDS清洗一次,再用0.06×SSC清洗兩次,離心甩干。
1.3.4芯片的掃描與數據處理雜交后采用晶芯? LuxScanTM10K-A雙通道激光共聚焦掃描儀進行掃描,并用LuxScan3.0軟件進行數據的提取。若對應的reference DNA雜交熒光強度較弱,則屬于壞點,棄之。質量符合條件的探針,其對應的熒光強度按照lg[Cy3 Target/Cy5 Reference]進行計算均一化。利用模糊 C-均值聚類分析法(模糊度為 1.5)將歸一化后的芯片信號值分為 2組聚類簇(cluster),如果計算出的聚類簇之間方差至少大于總方差的 80%,則認為此探針具有多態性,模糊 C-均值聚類分析法可將其在不同芯片樣本內分成0/1兩組類別(Wenzlet al,2004)。采用ANOVA單側檢驗(one-way ANOVA)分析組差異標記。計算P值、q值和FDR,并制作相應的散點圖。
1.4 大黃魚生長相關分子標記的篩選
從PstI/RsaI代表性基因組DNA文庫中隨機挑選3360個克隆,PCR擴增插入片段,重新點制芯片。在芯片中布置質控探針,陰性對照探針,陽性對照探針,空白對照探針,工作探針。每個探針包含三個重復。每張芯片包含16個點陣,每個點陣的設置同1.3.1。各取500ng基因組DNA,分別用PstI與RsaI組合進行雙酶切。酶切后加PstI特異性接頭,用 DArT-PstI引物引物對酶切產物進行 PCR擴增。熒光標記基因組DNA代表性片段的制備方法同1.3.2。芯片預處理、雜交、掃描、數據提取及數據初步處理方法同 1.3.3和 1.3.4。
1.5 大黃魚生長相關DArT標記的驗證
在后期驗證實驗中,重新取177尾網箱養殖的健康、無損傷1齡“東海1號”大黃魚。使用SPSS 17.0軟件統計分析建立體長的正態分布圖。這個群體每個個體基因組DNA代表性片段制備、芯片預處理、雜交、掃描、數據提取及數據初步處理方法同 1.3.2—1.3.4。篩選獲得的候選DArT標記進行測序,同時用BLAST2GO (http://www.blast2go.org)軟件對鑒定的DArT標記進行基因功能注釋。
2 結果
2.1 大黃魚體長數據統計
隨機選擇199個大黃魚樣本測量其體長數據。實驗群體中的體長最大值為16.30cm、最小值為11.50cm,均值為 13.45cm,標準偏差 1.0337。經過 Shapiro-Willie過程進行正態分布檢驗,峰度為0.192,偏度為–0.210,計算得到P=0.257,樣品符合正態分布(P>0.05)。因而根據采用分離群體分組分析法(Bulked Segregate Analysis,BSA)建立關于體長的正態分布圖(圖 1),在各群體中選取 10%的高值個體即體長大于14.80 cm的記為極端大群體,選取10%的低值個體即體長小于 12.00 cm的記為極端小群體,兩組之間體長差異極顯著(P<0.01)。可用于與生長相關 DArT標記的初步篩選。
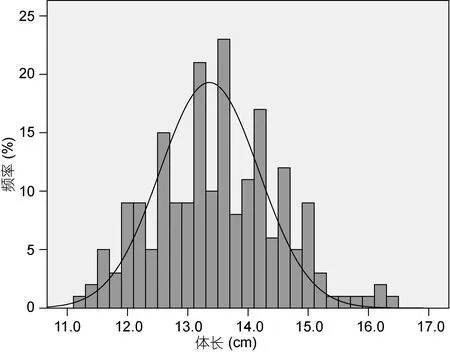
圖1 大黃魚體長正態分布頻率直方圖Fig.1 Normal distribution frequency histogram of body length of large yellow croaker
2.2 大黃魚基因組代表性DNA片段文庫的構建
本研究中大黃魚基因組 DNA完整,純度高,無RNA 污染(圖 2A)。將不同體長的大黃魚混合后,分別用PstI/AluI、PstI/BanII、PstI/Bsp1286I、PstI/BstNI、PstI/HaeIII、PstI/RsaI、PstI/TaqI酶切后純化(圖 2B)。在 T4DNA連接酶作用下,純化的 DNA酶切片段與PstI特異性接頭連接。連接產物作為模板進行后續 PCR擴增(圖 2C)。PCR產物克隆至pMD19-T載體后轉化大腸桿菌Top10F’,所得7個基因組代表性DNA文庫滴度≥105(表1),陽性克隆平均插入片段長度>500bp(圖 2D—J),符合 DArT芯片點制要求。

圖2 大黃魚基因組代表性DNA片段文庫構建Fig.2 Construction of genomic representations library of large yellow croaker
2.3 降低基因組復雜性方法
大黃魚“極端大群體” 與“極端小群體”樣品提取基因組 DNA,分別采用 7種酶切組合(PstI/AluI、PstI/BanII、PstI/Bsp1286I、PstI/BstNI、PstI/HaeIII、PstI/RsaI、PstI/TaqI)降低基因組復雜性。經標記后的DNA片段分別與各自DArT芯片雜交,雜交結果清晰可靠。7種酶切組合中,PstI/RsaI不僅可降低基因組復雜性,且多態性率最高(17.62%)(表1)。因此,選擇PstI/RsaI基因組代表性DNA片段文庫用于大黃魚體長相關DArT標記的篩選。
2.4 大黃魚生長相關DArT標記的篩選
重新點制的PstI/RsaI DArT芯片,其克隆數增加至3360個(圖3A)。并按照上述方法制備“極端大群體”與“極端小群體”PstI/RsaI大黃魚基因組代表性 DNA片段,并加上Cy3熒光標記,reference DNA為Cy5熒光標記。雜交后進行芯片掃描(圖3A)和數據提取。每張芯片經過歸一化處理后,通過用模糊 C-均值聚類分析法獲得0/1矩陣。根據計算所得的p值獲得散點圖(圖2B)。散點圖中黑色為無差異位點,紅色為差異顯著位點(P≤0.05),各位點集中在對角線附近,偏離對角線越大越容易呈現紅色。上述差異位點中只有18個DArT候選標記在“極端大群體”與“極端小群體”中聚類結果穩定,且組間P<0.01(表2),其中17個為“極端大群體”DArT候選標記,1個“極端小群體”DArT候選標記(表2)。
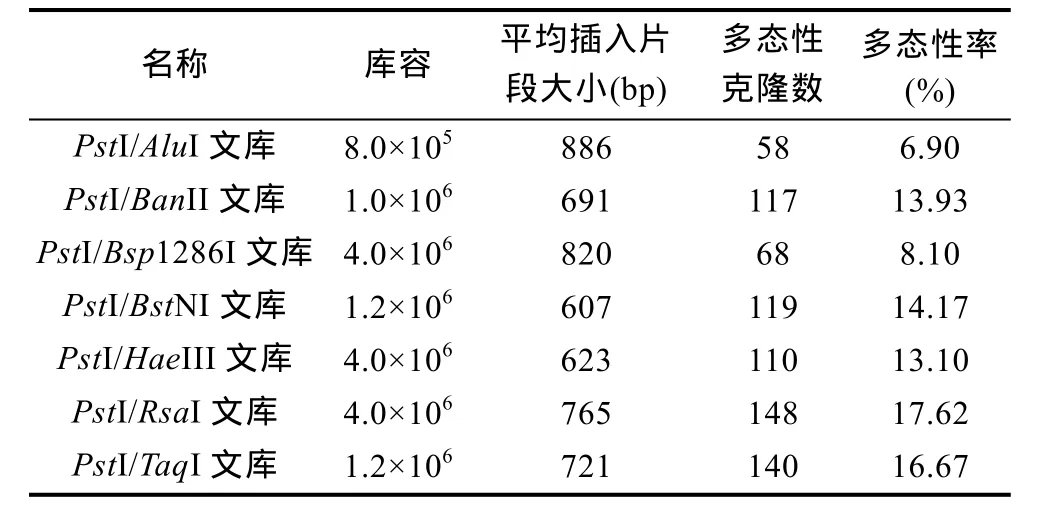
表1 7個基因組DNA代表性文庫由于所用限制性內切酶組合不同而造成的差異克隆數和多態性率Tab.1 The number of unique clone and polymorphism level in genomic representative library differing in enzymes used for co-digestion
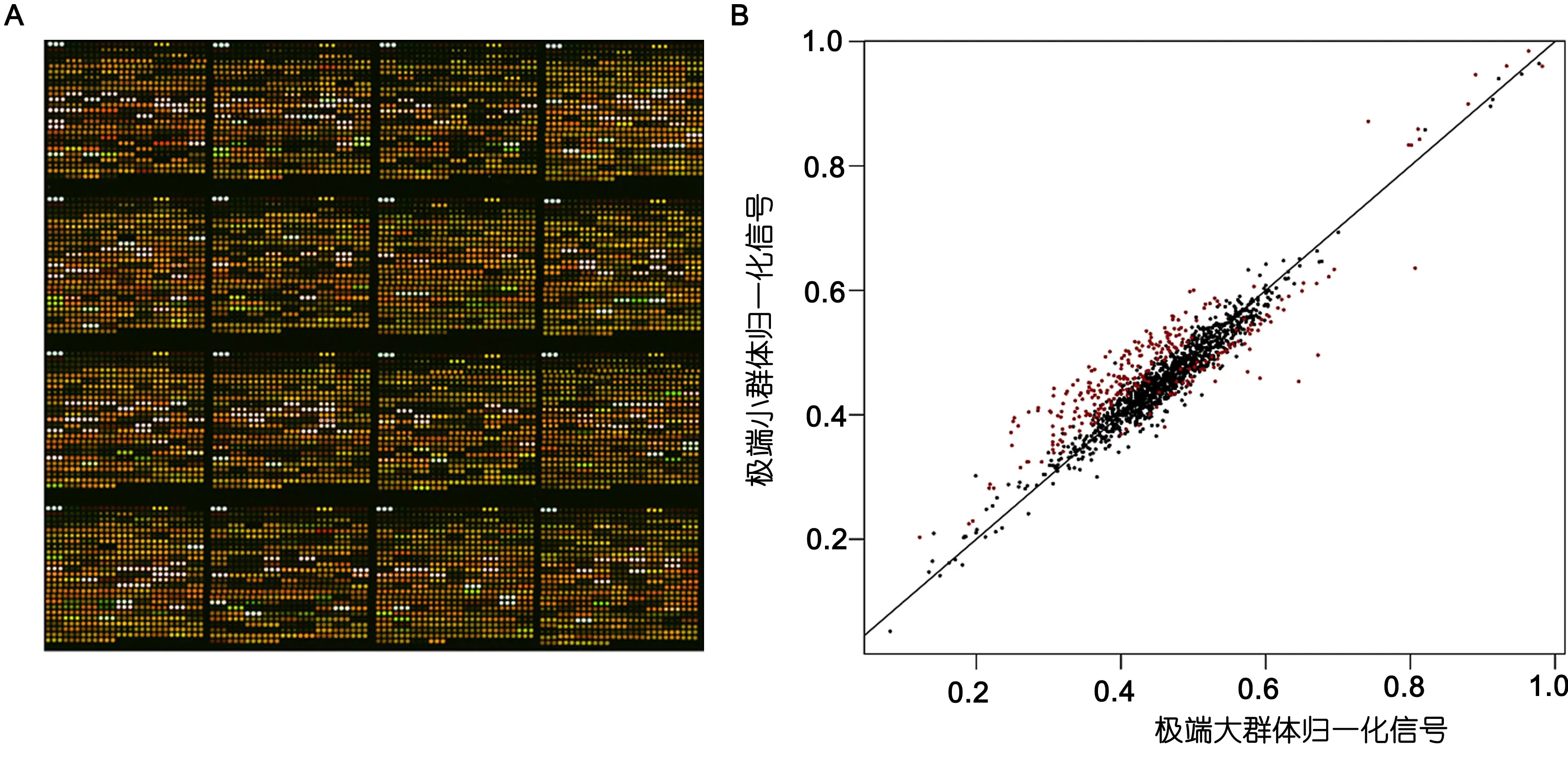
圖3 x827樣品雜交結果(A)及雜交歸一化信號P值散點圖(B)Fig.3 Result of DArT microarray assay of x827 (A) and the P value scatter plot of normalization signals from maximal length group and minimal length group (B)

表2 “極端大群體”和“極端小群體”0/1矩陣及統計分析Tab.2 The 0/1 matrix of maximal length group and minimal length group,and the statistics
2.5 大黃魚生長相關DArT標記的驗證
為驗證所篩選 DArT候選標記的有效性,又重新挑選了浙江象山港灣網箱養殖的“東海1號”F6代大黃魚(1齡) 177尾大黃魚,這個群體體長經過Shapiro-Willie過程進行正態分布檢驗,也符合正態分布(P>0.05)。從檢驗結果可以看到,RsaI1-23等8個DArT標記仍與體長性狀緊密相關(P<0.01) (表3)。為了進一步確定篩選獲得的大黃魚 DArT候選標記,將上述候選標記進行測序,測序結果用BLAST2GO軟件進行Blastn分析。結果顯示測定的序列中6個為已知序列,2個為未知序列(表3)。
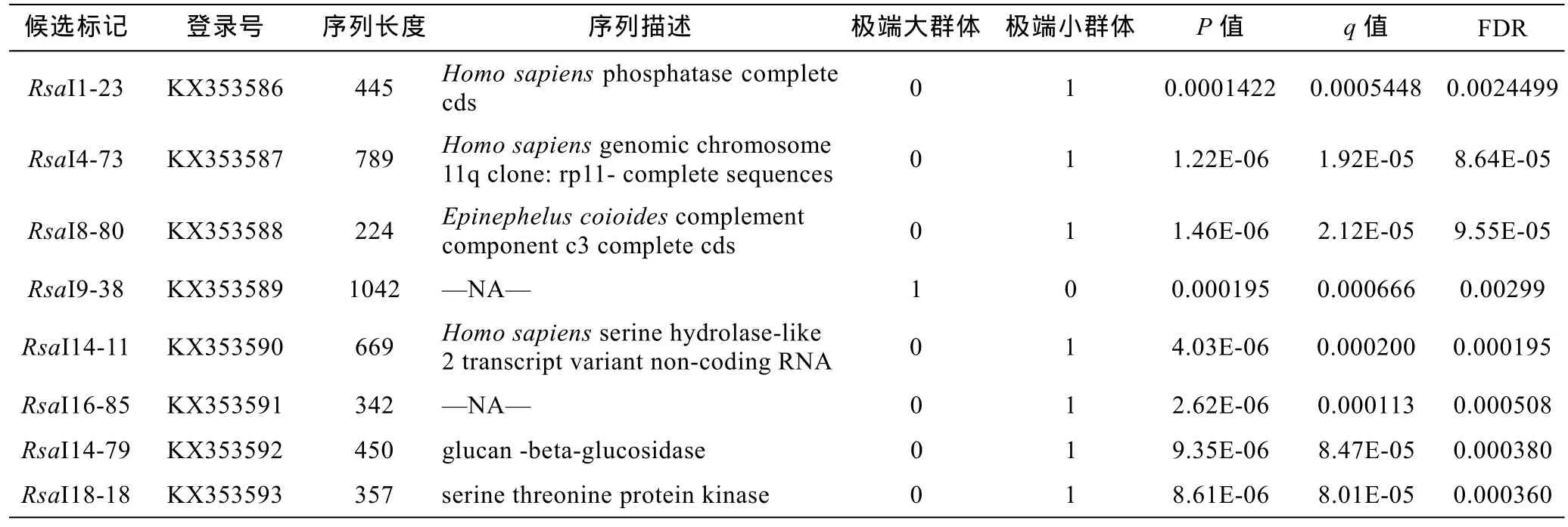
表3 大黃魚體長相關DArT標記再次驗證結果Tab.3 Re-verification for body-length-related DArT markers in large yellow croaker
3 討論
傳統選育需要經歷多個生命周期才能分離出穩定遺傳的經濟性狀。隨著分子生物學技術的迅猛發展,運用 DNA分子標記技術進行選育,從分子水平研究與水產動物優良經濟性狀相連鎖的分子標記極大地縮短了選育時間(劉賢德等,2013)。目前,SSR技術常用于尋找與生長、抗逆等性狀緊密連鎖或相關標記。如樊佳佳等(2009)關聯分析得到 7 個微衛星位點與體重、體長和體高顯著相關(P<0.05)或極顯著相關(P<0.01)。Yi等(2015)從100個SSR標記中找到8個基因座上 9個基因型與鱖魚生長性狀(體重、體長和體高)相關。研究者也試圖通過生長、抗逆等性狀緊密連鎖或相關標記指導大黃魚遺傳育種實踐。高國強等(2010)進行了大黃魚耐低溫標記的篩選,找到一個標記(LYC0002)可能與耐低溫有關。劉賢德等采用微衛星標記技術鑒定了LYC0088和LYC0143與大黃魚不同家系和群體生長性狀緊密相關(劉賢德等,2012,2013; 葉華等,2014)。薛良義等(2008)研究表明大黃魚肌肉生長抑制素基因 3’端非編碼區微衛星序列多態性與大黃魚體長、體質量無相關性。Ni等(2012)在浙江養殖大黃魚生長基因內含子 1的 196位SNP(Single nucleotide polymorphysim)與體長和體高相關,在兩個群體大黃魚生長基因內含子2的692位SNP與體重全長顯著相關。本研究中首次采用DArT技術鑒定了8個大黃魚體長相關DArT標記,其中7個為“極端大群體”DArT候選標記,1個“極端小群體”DArT候選標記。
一般有兩種方法篩選和鑒定水產動物目標性狀相關聯的分子標記,一種是分離群體標記關聯分析法,該方法可快速獲得與目的性狀連鎖的分子標記,缺點是靈敏度和精確度都較低; 另一種為隨機選擇群體標記關聯分析法,該方法可全面分析所選用的標記,準確度和精確度較好,缺點是需要檢測的樣本量大和分析費用較高(樊佳佳等,2009)。本研究采用分離群體標記關聯析法進行初步篩選,根據“東海 1號”大黃魚體長數據挑出“極端大群體”和“極端小群體”,從兩個群體中獲得18個與體長相關DArT候選標記。隨后新群體中驗證確認RsaI1-23等8個DArT標記仍與體長性狀緊密相關。由于仍不能保證本研究中所篩選到的標記在其它群體適用,因而在后續研究中尚需擴大群體的規模和類型,進行多方比較進行進一步驗證,為下一步基因輔助育種、進一步提高性狀選擇的準確性提供依據。
4 結論
本研究從PstI/RsaI基因組代表DNA片段文庫中擴增獲得3360個基因組代表性DNA片段,制備大黃魚體長相關多樣性芯片。雜交信號轉換為0/1矩陣并進行統計分析篩選,初步獲得 18個大黃魚體長相關DArT候選標記。之后經過驗證表明仍有8個DArT標記與新群體的體長相關。本研究成功應用DArT技術篩選到與體長相關標記,該方法也可以應用于其它生長相關性狀標記篩選。同時本研究成果可直接指導大黃魚人工選育工作; 也可作為進一步研究大黃魚生長相關的遺傳連鎖圖譜提供基礎,并對其它水產經濟動物的遺傳育種的建立提供理論依據與實踐參考。
王志勇,王藝磊,林利民等,2002. 福建官井洋大黃魚 AFLP指紋多態性的研究. 中國水產科學,9(3): 198—202
王曉清,王志勇,謝中國等,2008. 大黃魚(♀)與鮸(♂)雜交的遺傳分析. 水產學報,32(1): 51—57
葉華,劉洋,劉賢德等,2014. 大黃魚微衛星標記與生長性狀的相關分析. 西南大學學報: 自然科學版,36(3):27—33
寧岳,2007. 大黃魚遺傳連鎖圖譜的構建. 集美: 集美大學碩士學位論文,23—52
劉賢德,韋信鍵,蔡明夷等,2012. 大黃魚22個微衛星標記在F1家系中的分離方式及與生長性狀的相關分析. 水產學報,36(9): 1322—1330
劉賢德,隋班良,王志勇等,2013. 大黃魚快長相關微衛星標記的篩選與驗證. 水生生物學報,37(6): 1036—1043
李明云,張海琪,薛良義等,2003. 網箱養殖大黃魚遺傳多樣性的同工酶和RAPD分析. 中國水產科學,10(6): 523—525
李鵬飛,周永東,徐漢祥,2008.大黃魚、鮸魚及美國紅魚線粒體DNA的Cytb基因序列比較. 南方水產,4(3): 43—47
榮曄婧,陳強,史雨紅等,2014. 基于DArT標記的三疣梭子蟹地理種群遺傳多樣性分析. 生物學雜志,31(2): 18—21
姚康,王文策,耿梅梅等,2008. 大黃魚緊密連鎖α-和β-珠蛋白基因間序列功能分析. 水生生物學報,32(3): 413—416
高國強,常玉梅,韓啟霞等,2010. 大黃魚耐低溫性狀相關微衛星標記的篩選. 遺傳,32(3): 248—253
樊佳佳,白俊杰,李小慧等,2009. 大口黑鱸生長性狀的微衛星DNA標記篩選. 遺傳,31(5): 515—522
薛良義,孫升,肖章奎等,2008. 大黃魚肌肉生長抑制素基因微衛星序列多態性分析. 中國生物化學與分子生物學報,24(10): 980—985
魏東旺,樓允東,孫效文等,2001. 鯉魚微衛星分子標記的篩選. 動物學研究,22(3): 238—241
Chang Y M,Ding L,Wang W Wet al,2009. Isolation and characterization of 11 microsatellite markers for the large yellow croaker,Pseudosciaena crocea. Conservation Genetics,10(5): 1405—1408
Gao G Q,Chang Y M,Han Q Xet al,2010. Screening of microsatellite markers associated with cold tolerance of large yellow croaker (Pseudosciaena croceaR.). Hereditas,32(3): 248—253
Guo W,Wang Z Y,Wang Y Let al,2005. Isolation and characterization of six microsatellite markers in the large yellow croaker (Pseudosciaena croceaRichardson).Molecular Ecology Notes,5(2): 369—371
Hackl E,Konrad-K?szler M,Kilian Aet al,2010. Phage-type specific markers identified by Diversity Arrays Technology(DArT) analysis ofSalmonella entericassp.entericaserovars Enteritidis and Typhimurium. J Microbiol Meth,80(1): 100—105
Jaccoud D,Peng K M,Feinstein Det al,2001. Diversity arrays: a solid state technology for sequence information independent genotyping. Nucleic Acids Res,29(4): e25
Ni J,You F,Xu J Het al,2012. Single nucleotide polymorphisms in intron 1 and intron 2 ofLarimichthys croceagrowth hormone gene are correlated with growth traits. Chin J Oceanol Limnol,30(2): 279—285
Sánchez-Sevilla J F,Horvath A,Botella M Aet al,2015.Diversity Arrays Technology (DArT) marker platforms for diversity analysis and linkage mapping in a complex crop,the Octoploid cultivated strawberry (Fragaria × ananassa).PLoS One,10(12): e0144960
Schouten H J,van de Weg W E,Carling Jet al,2012. Diversity arrays technology (DArT) markers in apple for genetic linkage maps. Mol Breed,29(3): 645—660
Wenzl P,Carling J,Kudrna Det al,2004. Diversity Arrays Technology (DArT) for whole-genome profiling of barley.Proc Natl Acad Sci U S A,101(26): 9915—9920
Ye H,Liu Y,Liu X Det al,2014. Genetic mapping and QTL analysis of growth traits in the large yellow croakerLarimichthys crocea. Mar Biotechnol,16(6): 729—738
Yi T L,Fang L,Liang X F,et al,2015. Characterization of microsatellite markers and their correlations with growth traits in Mandarin fish (Siniperca chuatsi). Genet Mol Res,14(3): 8926—8934

